拟环纹豹蛛(Pardosa pseudoannulata)属于节肢动物门(Arthropoda)蛛形纲(Arachnida)蜘蛛目(Araneae)狼蛛科(Lycosidae),分布于我国贵州、河南、陕西、河北、北京、上海、甘肃、吉林、辽宁、西藏、广西、江西、云南等地区。拟环纹豹蛛是农田蜘蛛中的优势种,是稻田害虫重要的生物防治因子,可有效地控制稻飞虱、稻叶蝉[1]等害虫。拟环纹豹蛛的生长,发育,繁殖,存活率及其捕食行为受到周围环境温度的显著影响[2-5]。在稻田生态系统中,冬季温度往往低于0 ℃,夏季的温度往往高于40 ℃。当温度低于10 ℃,蜘蛛停止取食,并停止生长和发育;当温度高于40 ℃,蜘蛛活动迟钝,常常静伏于隐蔽物下,或钻入土内,尚能正常生活[6]。拟环纹豹蛛是一种对温度适应范围广,且能抗高温的蜘蛛,在30 ℃的条件下,拟环纹豹蛛的雌、雄成蛛对褐飞虱的捕食量可以达到100头和166.67头[7]。拟环纹豹蛛在39 ℃恒温条件下能正常蜕皮完成个体发育,在35 ℃恒温条件下还能产卵繁殖,使它能够在高温的夏天,保持田间较大种群数量,发挥其对害虫的控制作用[2]。但目前对于拟环纹豹蛛响应高低温胁迫的生理生化机制尚不清楚。
单核苷酸多态性(Single nucleotide polymorphism,SNP)是指由于单个核苷酸的变异所引起的DNA序列的多态性,即碱基的替换突变。在单个碱基对中,嘌呤突变为嘌呤或者嘌呤突变为嘧啶的改变方式称之为转换(比如AT→GC和GC→AT均为转换),根据已有的研究表明,SNP的碱基突变大多发生在T和C之间[8]。SNP占目前所有已知多态性的90%以上,是一种最常见的可遗传变异,也是生物进化的重要途径[9]。1996年,美国学者Lander[10]提出了第三代DNA遗传标记-SNP标记。由于SNP具有共显性、易于实现高通量和具有很高的遗传稳定性等特点,相比于前几代的标记方法,它能使DNA遗传标记更加快速有效[11-12]。目前SNP分子标记已被广泛运用于人类疾病[13-14]、药物[15]、 农业中动植物的遗传育种[16-18]等各个领域。
尽管温度因子对拟环纹豹蛛影响这一方面的研究逐步完善,但就目前的研究中,拟环纹豹蛛响应温度胁迫SNP位点的研究仍是一片空白。Xiao等[19]对拟环纹豹蛛在低温(10 ℃)和高温(40 ℃)胁迫后进行了转录组测序,获得了67 725条Unigene及拟环纹豹蛛在温度胁迫条件下Unigene差异表达及注释情况。但是转录组测序没有对拟环纹豹蛛SNP位点进行筛选和分析,因而本试验基于拟环纹豹蛛不同温度胁迫转录组测序数据基础上,对其进行SNP位点发掘,并进行相关分析及SNP位点所在Unigene进行基因注释和功能分析,为进一步开发拟环纹豹蛛适应温度变化、相关性状的分子标记打下基础,以期为拟环纹豹蛛响应温度胁迫的分子机制的进一步研究提供理论依据和基础数据。
1 材料和方法
1.1 试验材料
拟环纹豹蛛的采集和饲养:拟环纹豹蛛的亚成蛛采集于广州市华南农业大学实验田(113.37°E,23.17°N)。亚成蛛采回后单头放入玻璃试管(长10 cm,直径2.5 cm)中饲养,试管底部用一块蘸水的海绵保湿,管口用脱籽棉塞住,防止蜘蛛逃逸,置于人工气候箱内(PRX-350C,(25±0.5) ℃,相对湿度RH60%±10%,光周期为连续光照14 h明期,10 h暗期饲养。每周饲喂2次,每次提供4~6头家蝇(Muscadomestica)成虫。每隔1 d观察记录拟环纹豹蛛蜕皮及成熟情况。
转录组测序样品:当蜘蛛成熟14 d,成熟个体被转移到另外的人工气候箱保持在(10±0.5) ℃(低温处理组,TL)12 h,或(40±0.5) ℃(高温处理组,TH)12 h。其余蜘蛛一直保持在(25±0.5) ℃饲养的作为空白组(TC)。每个处理3个生物重复,每个重复包含3头雄蛛和3头雌蛛。处理好的整头蜘蛛立即用液氮冷冻并储存在-80 ℃冰箱以备总RNA提取。提取样品总RNA后,经质量检测合格的样品送百迈客生物科技有限公司利用Illumina HiseqTM2500进行转录组测序,测序结果用Trinity软件进行从头组装,共获得67 725条Unigene,包括3个转录组文库:空白组(TC,25 ℃),低温处理组(TL,10 ℃),高温处理组(TH,40 ℃)。转录组测序数据已上传至美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)的SRA数据库,数据序列号为转录组TC(SRR2404407),转录组TL(SRR2405992),转录组TH(SRR2406000)。
1.2 试验方法
使用软件SOAPsnp(http://soap.genomics.org.cn/SOAPsnp.html)对测序得到的67 725条Unigene序列进行SNP检测,存在SNP位点的Unigene称为SNP-Unigene。通过Blast(Blast的参数E-value不大于10-5)软件将所有SNP-Unigenes序列分别与Nr(Non-redundant protein database)、Swiss-Prot、GO(Gene Ontology)、COG(Clusters of Orthologous Groups of proteins)、KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库进行比对,获得各SNP-Unigene序列的注释信息,进而分析这些SNP位点是否影响了基因的表达水平或蛋白产物的种类。
2 结果与分析
2.1 SNP位点分析
使用软件SOAPsnp对转录组数据分析发现,3个转录组文库中总共存在402 910个SNP位点,其中低温处理组(TL)各基因型共存在220 235个SNP位点,高温处理组(TH)各基因型存在182 675个SNP位点(表1)。获得的67 725条Unigene中共存在7 421条SNP-Unigenes,其中低温处理组各基因型共存在6 360条SNP-Unigenes,高温处理组各基因型共存在5 469条SNP-Unigenes(表2)。SNP-Unigenes总长度为12 124 783 bp,编码区长度为7 100 355 bp,非编码区长度占5 024 428 bp,SNP发生频率为1/301 bp。两组间各基因型发生转换和颠换的情况见图1,空白组和低温处理组或高温处理组都是杂合基因型时发生转换和颠换的SNP-Unigene最多(图1)。
表1 两两样品间SNP位点数量统计
Tab.1 The number of SNP sites between two samples

样品Sample空白组:纯合基因型处理组:纯合基因型TC:homoTL/TH:homo空白组:杂合基因型处理组:纯合基因型TC:heteTL/TH:homo空白组:纯合基因型处理组:杂合基因型TC:homoTL/TH:hete空白组:杂合基因型处理组:杂合基因型TC:heteTL/TH:hete所有类型的统计信息TotalTL28141 80346 883131 268220 235TH39841 20734 451106 619182 675
注:homo.纯合基因型;hete.杂合基因型。表2同。
Note:homo.Homozygous genotype; hete.Heterozygous genotype. The same as Tab.2.
表2 两两样品间 SNP-Unigene数量统计表
Tab.2 The number of SNP-Unigene sites between two samples
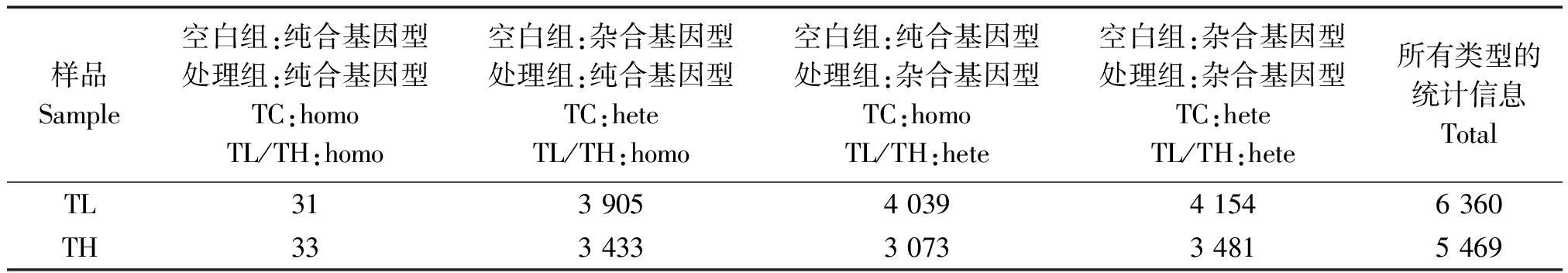
样品Sample空白组:纯合基因型处理组:纯合基因型TC:homoTL/TH:homo空白组:杂合基因型处理组:纯合基因型TC:heteTL/TH:homo空白组:纯合基因型处理组:杂合基因型TC:homoTL/TH:hete空白组:杂合基因型处理组:杂合基因型TC:heteTL/TH:hete所有类型的统计信息TotalTL313 9054 0394 1546 360TH333 4333 0733 4815 469
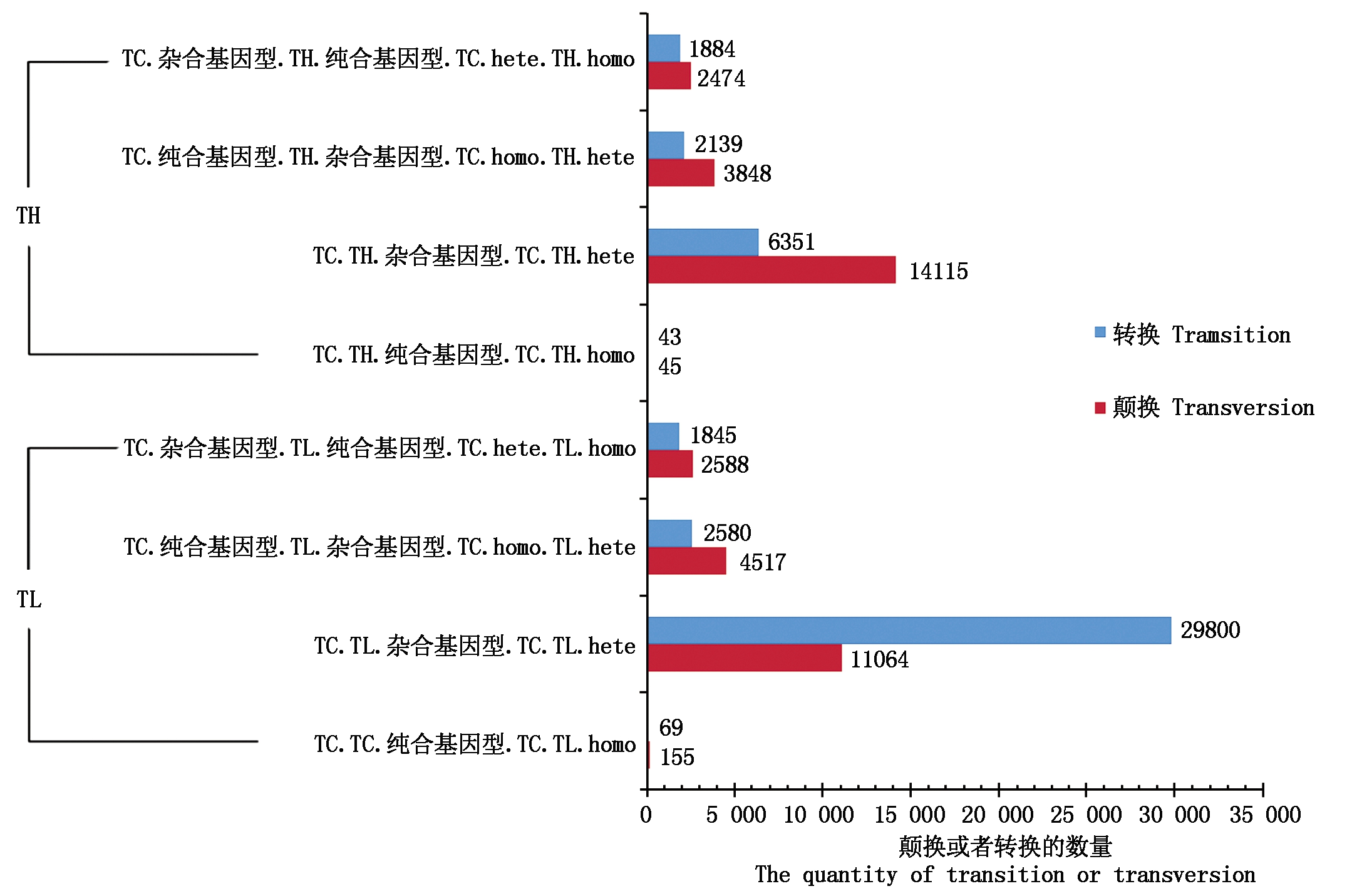
图1 拟环纹豹蛛转录组SNP 转换、颠换情况统计
Fig.1 The statistic of SNP transition or transversion in Pardosa pseudoannulata
2.2 SNP-Unigene的GO功能分类
对7 421条SNP-Unigenes进行GO功能分类,结果表明:在低温处理组中有2 097条SNP-Unigenes被注释到39个GO二级功能分类中(图2),其中841条SNP-Unigenes被注释到细胞组分(Cellular component)的11个子类中,635条SNP-Unigenes注释到分子功能(Molecular function)的13个子类中,621条SNP-Unigenes注释到生物进程(Biological process)的15个子类中。注释数目最多的功能分别是参与膜(Membrane,322条),占15.38%;其次是结合(Binding,311条)、催化活性(Catalytic activity,295条)及代谢过程(Metabolic process,267条),分别占14.84%,14.06%和12.75%。
在高温处理组中,1 923条SNP-Unigenes被注释到36个GO二级功能分类中(图2),其中有773条SNP-Unigenes注释到细胞组分的13个子类中,有553条SNP-Unigenes注释到分子功能的10个子类中,而有597条SNP-Unigenes则被注释到生物进程的13个子类中。数目最多的类别仍然是参与膜(Membrane,304条),占15.83%,其次是催化活性(Catalyticactivity,274条)、结合(Binding,273条)及代谢过程(Metabolic process,241条),分别占14.26%,14.17%和12.59%。
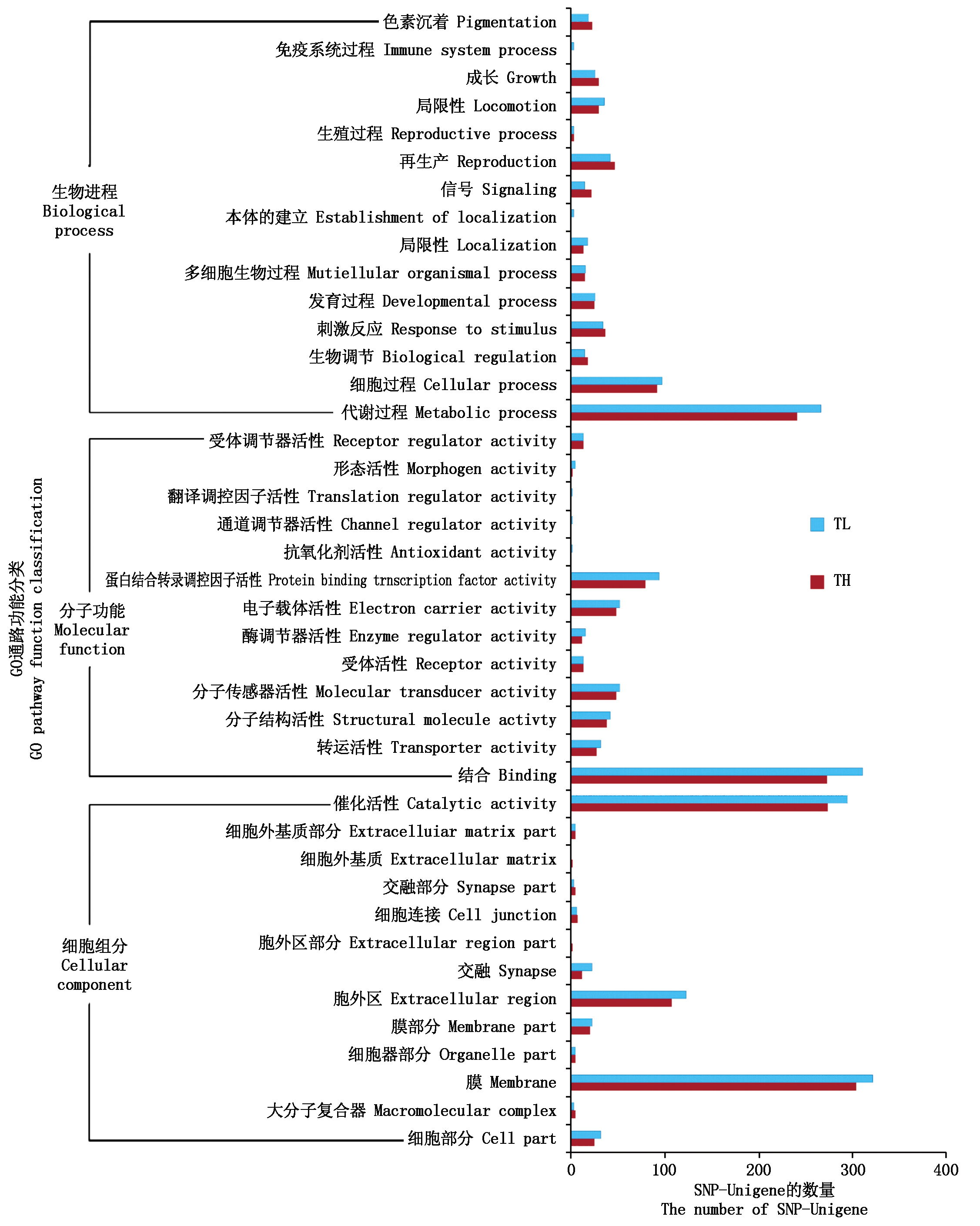
图2 SNP-Unigene的GO功能分类
Fig. 2 GO functional classification of SNP-Unigene
2.3 SNP-Unigene的COG功能分类
将SNP-Unigene序列比对到COG基因库中进行相关基因的注释和分类,结果表明:TL低温处理组有2 552条SNP-Unigenes在COG数据库中比对到了相应的注释信息,根据功能分为23类,其中大部分SNP-Unigenes都参与了两类或者两类以上的功能。从分析结果来看,这2 552条SNP-Unigenes参与了大多数的生命活动过程或功能。其中参与较多的功能是翻译后修饰,蛋白质翻转,分子伴侣(O,Posttranslational modification, protein turnover, and chaperones)、翻译,核糖体结构和生物转化 (J,Translation, ribosomal structure, and biogenesis)、和氨基酸转运和代谢(E,Amino acid transport and metabolism),分别占13.68%(349条),13.17%(336条)和12.03%(307条)(图3)。
在高温处理组中试验组中,有2 265条SNP-Unigeness在COG数据库中找到了相对应的注释信息,同低温处理组试验组类似,这2 265条SNP-Unigenes也参与了大多数的生命活动过程或功能。绝大多数参与了两类或者两类以上的功能,其中参与较多的功能也是翻译后修饰,蛋白质翻转,分子伴侣蛋白(O,Posttranslational modification, protein turnover, and chaperones)、翻译,核糖体结构和生物转化(J,Translation, ribosomal structure, and biogenesis)、和氨基酸转运和代谢(E,Amino acid transport and metabolism),分别占14.22%(322条),12.27%(278条)和11.83%(268条)(图3)。
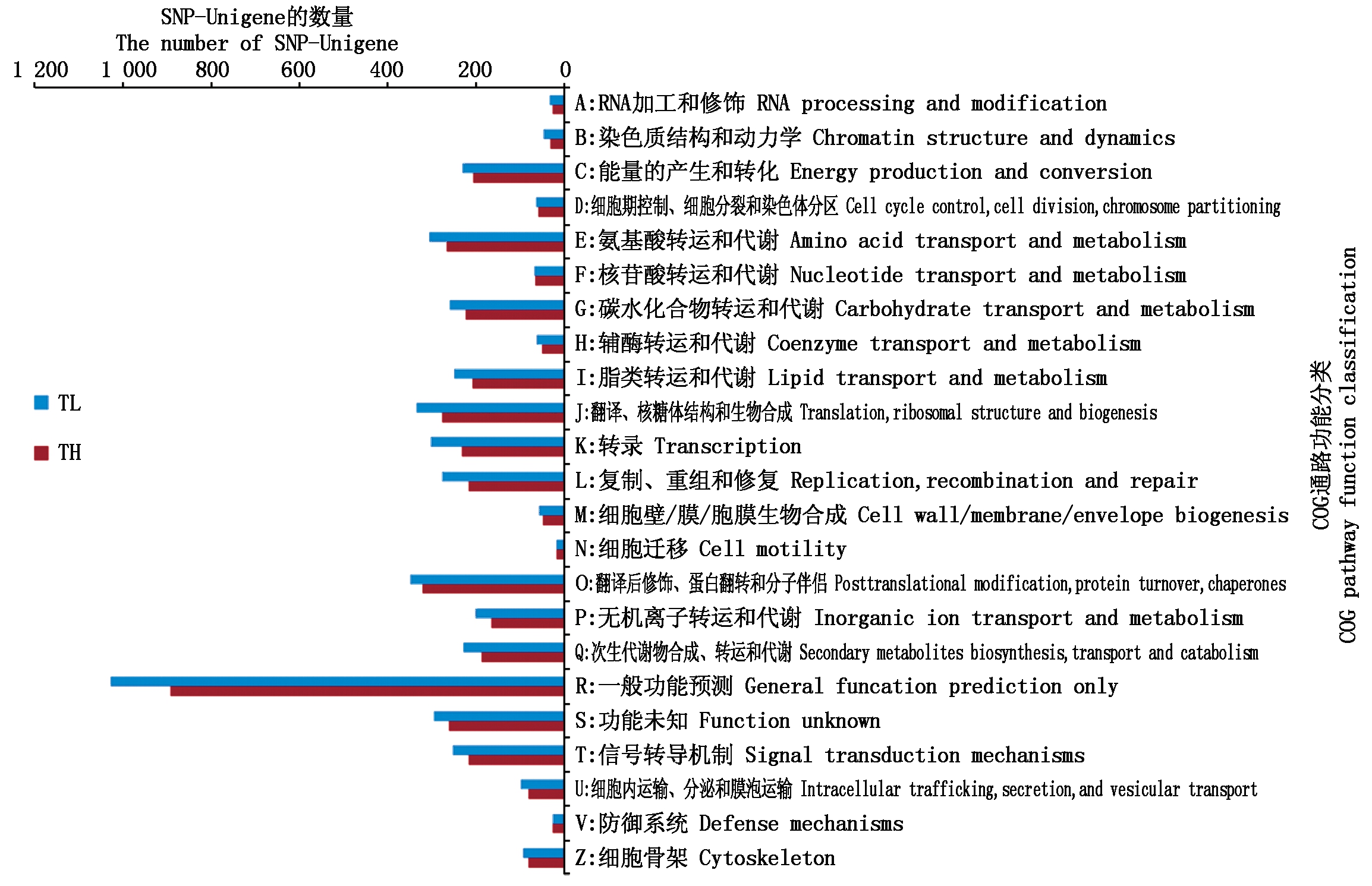
图3 SNP-Unigene的COG功能分类
Fig.3 COG functional classification of SNP-Unigene
2.4 SNP-Unigene的KEGG代谢通路分析
将7 421条SNP-Unigenes序列进行KEGG代谢通路分析,结果表明:在低温处理实验组,有2 530条SNP-Unigenes序列获得了注释,分别参与到物质代谢、遗传信息过程、细胞学过程等生物代谢途径。注释结果显示,占比例最大的分别是氧化磷酸化代谢通路(Oxidative phosphorylation),占6.40%(162条)。其次是核糖体代谢通路(Ribosome)和RNA转运代谢通路(RNA transport),分别占5.73%(145条)和5.45%(138条)(图4)。
高温处理试验组中,有2 266条SNP-Unigenes序列获得了注释,同低温处理试验组类似,它们涉及了物质代谢、遗传信息过程、细胞学过程等生物代谢途径。其中数目最多的是核糖体代谢通路(Ribosome),占6.44%(146条)。其次分别是氧化磷酸化代谢通路(Oxidative phosphorylation)和RNA转运代谢通路(RNA transport),占6.09%(138条)和5.16%(117条)(图4)。
3 讨论
SNP作为一种分子遗传标记,在动植物的遗传选育中发挥着重要作用[20-21]。拟环纹豹蛛SNP相关研究目前是未知的,因而本研究基于高通量测序从拟环纹豹蛛响应低温和高温胁迫转录组数据中筛选到了402 910个可能的SNP位点分布于7 421条SNP-Unigenes序列上,可为研究稻田蜘蛛响应温度胁迫分子机制提供理论依据。SNP-Unigenes序列总长度为12 124 783 bp,编码区长度为7 100 355 bp,非编码区长度占5 024 428 bp,并不是所有SNP位点都位于编码区,某些位于非编码区的SNP位点也可能对拟环纹豹蛛适应温度变化起到重要作用。已有研究发现陆地棉(Gossypium hirsutum)某些耐盐相关的SNP位于非编码区起重要作用[22],位于人类基因MIR196A启动子区域的SNP可以影响胃癌的发生和发展[23],及位于海湾扇贝(Argopecten irradians irradians 和Argopecten irradians concentricus)Hsp90基因启动子区的6 个SNP 都与其耐高温性相关[24]。所以,将来关注拟环纹豹蛛非编码区的SNP也很重要。

图4 SNP-Unigene的KEGG通路分析
Fig.4 KEGG metabolic pathway analysis of SNP-Unigene
拟环纹豹蛛转录组SNP发生频率为1/301 bp,也就是每301个碱基中,就可能存在一个突变的碱基,这个突变频率远远大于人类基因组中碱基的突变频率(1/1 000 bp)、辣椒基因转录组碱基突变频率(1/6 200 bp)[25]、玫瑰花基因转录组碱基的突变频率(1/1 173 bp)[26]和小麦(1/540)[27]等大多数动植物,低于茶树(1/200 bp)[28]、大麦(1/58 bp)[29]、葡萄转录组(1/64 bp)[30]等的突变频率,造成碱基突变频率差异主要取决于研究物种的差异和遗传背景差异。
本研究中,我们发现低温和高温胁迫后,SNP数目和位置不完全相同,且有些SNP只在低温胁迫或高温胁迫处理组存在。同样的,低温和高温胁迫后,SNP位点所在的SNP-Unigene也不完全相同,这说明拟环纹豹蛛遭受低温和高温胁迫后SNP发生位点变异是不同的,响应低温胁迫和高温胁迫的分子机制也是不同的,进而我们分析了拟环纹豹蛛响应低温和高温胁迫SNP-Unigene的基因功能。
经GO功能分类注释和COG功能注释,拟环纹豹蛛在响应低温胁迫和高温胁迫时,虽然具体的SNP-Unigene不相同,但主要SNP-Unigene参与的功能是相同的,说明当蜘蛛受到温度胁迫时,这些SNP-Unigene在帮助它们适应温度胁迫时发挥了重要作用。拟环纹豹蛛作为一种变温动物,能适应不同的温度范围被认为与其复杂的系统状态下各种酶和代谢模式息息相关[31]。变温动物遭受温度胁迫时,能量的产生和转运是必不可少的。蜘蛛遭受低温和高温胁迫时,氧化磷酸化代谢通路(Oxidative phosphorylation)、核糖体代谢通路(Ribosome)及RNA转运代谢通路(RNA transport)是识别的3个主要的代谢通路,且都与能量的产生有关。
高的线粒体基因浓度已经在其他冷适应的变温动物中有研究,比如说鱼类[32-33],Windisch等[33]证明了南极鱼Pachycara brachycephalum(Pappenheim)体内线粒体核糖体蛋白质在低温条件下具有高速的转录速率,对南极鱼适应低温环境产生积极的影响。蛋白质合成结构是细胞内最大的酶复合物之一,核糖体的结构及其功能与大多数通用的蛋白质息息相关[34]。当拟环纹豹蛛在高温胁迫下和低温胁迫下,核糖体代谢通路相关的SNP-Unigene数量相差不大。虽然本研究没有测量拟环纹豹蛛在温度胁迫下蛋白质生物合成的速率及代谢速率,但是这些SNP-Unigene有助于帮助蜘蛛提高蛋白质生物合成,从而补偿热应力下蛋白质的损伤,加快他们对温度变化的适应。从参与氧化磷酸化代谢通路的SNP-Unigene数量来看,蜘蛛在低温环境下的新陈代谢高于高温环境下。在水生变温动物中如贻贝在极端高温环境下将导致有氧范围的减少或诱导降低代谢速率[35-36]。温度胁迫下能量需求的增加通常是为了维持机体正常的功能,从而将更少的能量分配到生长,存储和繁殖[37-38]。当蜘蛛在10 ℃能正常活动的临界温度时,他们会产生更多的能量为了维持生存。相反的,当蜘蛛在40 ℃能正常活动和繁殖的温度时,会消耗更多的能量维持生长、发育和繁殖。因此,为了权衡能量的使用,蜘蛛相对于25 ℃温度条件下,会在10 ℃保持一个活跃的新陈代谢,而在40 ℃产生较少的能量。
本研究中,基于拟环纹豹蛛高低温胁迫后的转录组测序数据筛选SNP位点,并对含有SNP的Unigene (SNP-Unigene)进行功能注释。基于这些功能注释,筛选到响应温度胁迫相关的SNP-Unigene或者通路上的SNP可能与拟环纹豹蛛适应温度胁迫直接相关,为将来深入研究拟环纹豹蛛响应温度胁迫的分子机制奠定了理论基础。
[1] 林源, 周夏芝, 毕守东, 邹运鼎, 马飞, 程遐年, 柯磊, 杨林, 郭骅. 中稻田三种飞虱的捕食性天敌优势种及农药对天敌的影响[J].生态学报, 2013, 33(7): 2189-2199. doi: 10.5846/stxb201112171926.
Lin Y, Zhou X Z, Bi S D, Zou Y D, Ma F, Cheng X N, Ke L, Yang L, Guo H.The dominant species of predatory natural enemies of three kinds of planthoppers and impact of pesticides on natural enemies in paddy field[J]. Acta Ecologica Sinica, 2013, 33(7): 2189-2199.
[2] 赵敬钊, 袁爱荣, 余克庆. 温度对拟环纹豹蛛发育和繁殖力的影响[J]. 湖北大学学报(自然科学版), 1989(1): 1-9.
Zhao J Z, Yuan A R, Yu K Q. Effects of temperature on the development and fertility of Pardosa pseudoannulata [J]. Journal of Hubei University (Natural Science), 1989(1): 1-9.
[3] 赵敬钊. 温度对蜘蛛个体发育的影响[J]. 蛛形学报, 2001,10(1): 58-64.doi:10.3969/j.issn.1005-9628.2001.01.017.
Zhao J Z. Effects of temperature on spider individual ontogeny[J]. Acta Arachnologica Sinica, 2001,10(1):58-64.
[4] 赵敬钊, 刘凤想, 常瑾, 彭宇. 温度对蜘蛛繁殖力和实验种群的影响[J]. 生态学报, 2002,22(9):1470-1477. doi:10.3321/j.issn:1000-0933.2002.09.015.
Zhao J Z, Liu F X, Chang J, Peng Y. Effects of temperature on fecundity and the experimental population of spiders[J]. Acta Ecologica Sinica, 2002,22(9): 1470-1477.
[5] 孙继英, 彭光旭, 胡波, 付秀芹, 颜亨梅. 拟环纹豹蛛种群遗传多样性与其生境的关系[J]. 应用生态学报, 2007, 18(5): 1081-1085. doi: 10.13287/j.1001-9332.2007.0181.
Sun J Y, Peng G X, Hu B, Fu X Q, Yan H M. Genetic diversity of pardosa pseudoannulata population and its relationships with habitats[J]. Chinese Journal of Applied Ecology, 2007,18(5):1081-1085.
[6] 王洪全, 周家友, 刘贵匀. 拟环纹狼蛛的生物学研究[J]. 动物学报, 1982(1): 69-79.
Wang H Q, Zhou J Y, Liu G Y. Study on the ecology of Pardosa pseudoannulata [J]. Acta Zoologica Sinica, 1982(1): 69-79.
[7] 唐美蓉, 赵丽, 魏宝阳, 王智. 温度对拟环纹豹蛛的捕食功能反应的影响[J]. 湖南农业科学, 2016(9): 55-58.doi:10.16498/j.cnki.hnnykx.2016.09.017.
Tang M R, Zhao L, Wei B Y, Wang Z. Effect of temperature on the predation of Pardosa pseudoannulata[J]. Hunan Agricultural Sciences, 2016(9): 55-58.
[8] 唐梅森. 基于DNA单核苷酸多态性和甲基化修饰探讨早发冠心病血瘀证的相关易感基因的研究[D]. 长沙: 湖南中医药大学, 2012.
Tang M S. Research in susceptibility genes associated with premature coronaryheart disease blood stasis syndrome based on single nucleotide polymorphisms and DNA methylation[D]. Changsha: Hunan University of Chinese Medicine, 2012.
[9] 赵辉, 李启寨, 李俊, 曾长青, 胡松年, 于军. 相邻碱基组分与产生SNP的转换或颠换在植物基因组中的研究[J]. 中国科学C辑(生命科学), 2006,36(1): 1-8. doi: 10.3321/j.issn:1006-9259.2006.01.001.
Zhao H, Li Q Z, Li J, Zeng C Q, Hu S N, Yu J. Studies on conversion or transversion of neighboring base components and SNP generation in plant genomes[J], Science Inchina Ser C(Life Sciences), 2006,36(1): 1-8.
[10] Lander E S. The new genomics: global views of biology[J]. Science, 1996,274(5287):536-539. doi:10.1126/science.274.5287.536.
[11] 陈丝, 郭燕, 杨春, 李燕. 单核苷酸多态性在茶树中应用的研究进展[J]. 食品安全质量检测学报, 2018, 9(2): 243-247. doi: 10.3969/j.issn.2095-0381.2018.02.005.
Chen S, Guo Y, Yang C, Li Y. Review on single nucleotide polymorphisms application in tea plant (Camellia sinensis)[J]. Journal of Food Safety and Quality, 2018, 9(2): 243-247.
[12] Li R Q, Li Y R, Fang X D, Yang H M, Wang J,Kristiansen K, Wang J. SNP detection for massively parallel whole-genome resequencing[J]. Genome Research, 2009, 19(6): 1124-1132. doi:10.1101/gr.088013.108.
[13] Patterson N, Hattangadi N, Lane B, Lohmueller K E, Hafler D A, Oksenberg J R, Hauser S L , Smith M W, O′Brien S J, Altshuler D, Daly M J, Reich D. Methods forhigh-density admixture mapping of disease genes[J].The American Journal of Human Genetics, 2004, 74(5): 979-1000. doi:10.1086/420871.
[14] Liu R Z, Geng P L, Ma M H, Yu S Y, Wang X L, Zhang W, Di H. Association between endothelial nitric oxide synthase gene polymorphisms (T-786C) and ischemic stroke susceptibility: A meta-analysis[J]. International Jurnal of Neuroscience, 2014, 124(9):642-651. doi: 10.3109/00207454.2013.873978.
[15] Lai E. Application of SNP technologies in medicine: lessons learned and future challenges[J]. Genome Research, 2001, 11(6): 927-929. doi:10.1101/gr.192301.
[16] Septiningsih E M, Pamplona A M, Sanchez D L, Neeraja C N, Vergara G V, Heuer S, Ismail A M, Mackill D J. Development of submergence-tolerant rice cultivars: the Sub1 locus and beyond[J]. Annals of Botany, 2009, 103(2): 151-160. doi: 10.1093/aob/mcn206
[17] Naidoo R, Watson G M F, Derera J, Tongoona P, Laing M D. Marker-assisted selection for low phytic acid (lpa1-1) with single nucleotide polymorphism marker and amplified fragment length polymorphisms for background selection in a maize backcross breeding programme[J]. Molecular Breeding, 2012, 30(2): 1207-1217. doi: 10.1007/s11032-012-9709-8.
[18] Rahman M, Asif M, Shaheen T, Tabbasam N, Zafar Y, Paterson A H. Marker-assisted breeding in Higher Plants[D]. Faisalabad:National Institute for Biotechnology and Genetic Engineering, 2011: 39-76. doi:10.1007/978-94-007-0186-1_3.
[19] Xiao R, Wang L, Cao Y S, Zhang G R. Transcriptome response to temperature stress in the wolf spider Pardosa pseudoannulata (Araneae: Lycosidae)[J]. Ecology and Evolution, 2016, 6(11): 3540-3554. doi: 10.1002/ece3.2142.
[20] 陈柏湘, 王伟峰, 王卫民, 王焕岭. 团头鲂低氧耐受相关SNPs标记的开发[J]. 华中农业大学学报, 2019, 38(2): 23-29. doi: 10.13300/j.cnki.hnlkxb.2019.02.004.
Chen B X, Wang W F, Wang W M, Wang H L. Isolation of SNP markers associated with hypoxia tolerance in Megalobrama amblycephala [J]. Journal of Huazhong Agricultural University,2019, 38(2): 23-29.
[21] Hu W M. Development of 31 EST-SNP markers in Glycyrrhiza uralensis Fisch (Leguminosae) based on transcriptomics[J]. Conservation Genetics Resources, 2019: 1-5. doi:10.1007/s12686-019-01101-2.
[22] 王晓歌, 阴祖军, 王俊娟, 王德龙, 樊伟丽, 王帅, 叶武威. 陆地棉转录组耐盐相关SNP挖掘及分析[J].分子植物育种, 2016, 14(6): 1524-1532. doi: 10.13271/j.mpb.014.001524.
Wang X G, Yin Z J, Wang J J, Wang D L, Fan W L, Wang S, Ye W W. Mining and analyzing of SNP related to salinity stress in transcriptome of upload cotton (Gossypium hirsutum L.)[J]. Molecular Plant Breeding, 2016, 14(6):1524-1532.
[23] Xu M, Qiang F L, Gao Y, Kang M Y, Wang M L, Tao G Q, Gong W D, Zhu H X, Wu D G, Zhang Z D, Zhao Q H. Evaluation of a novel functional single-nucleotide polymorphism (rs35010275 G>C) in MIR196A2 promoter region as a risk factor of gastric cancer in a Chinese population[J]. Medicine, 2014, 93(26):e173. doi: 10.1097/MD.0000000000000173.
[24] Yang C Y, Wang L L, Liu C H, Zhou Z,Zhao X,Song L S. The polymorphisms in the promoter of HSP90 gene and their association with heat tolerance of bay scallop[J]. Cell Stress and Chaperones, 2015, 20(2): 297-308.doi: 10.1007/s12192-014-0546-z.
[25] Lu F H, Yoon M Y, Cho Y I, Chung J W, Kim K T, Cho M C, Cheong S R, ParkY J. Transcriptome analysis and SNP/SSR marker information of red pepper variety YCM334 and Taean[J]. Scientia Horticulturae, 2011, 129(1): 38-45.doi:10.1016/j.scienta.2011.03.003.
[26] 梁芳,张继,吕平,龙凌云,黄惠芳,檀小辉,韦丽君. 基于EST序列的玫瑰EST-SNP位点发掘与分析[J]. 南方农业学报,2016,47(3): 325-331.doi:10.3969/j:issn.2095-1191.2016.03.325.
Liang F, Zhang J, Lü P, Long L Y, Huang H F, Tan X H, Wei L J. Discovery and analysis of Rosa rugosa EST-SNP site based on EST sequences[J]. Journal of Southern Agriculture, 2016, 47(3): 325-331.
[27] Somers D J, Kirkpatrick R, Moniwa M, Walsh A. Mining single-nucleotide polymorphisms from hexaploid wheat ESTs[J]. Genome, 2003, 46(3): 431-437. doi: 10.1139/g03-027.
[28] 王丽鸳, 张成才, 成浩, 韦康. 茶树EST-SNP分布特征及标记开发[J]. 茶叶科学, 2012, 32(4): 369-376. doi:10.13305/j.cnki.jts.2012.04.014.
Wang L Y, Zhang C C, Cheng H, Wei K. Characterization of EST-derived SNPs and development of SNP-markers in tea (Camellia sinensis)[J]. Journal of Tea Science, 2012, 32(4): 369-376.
[29] Varshney R K, Beier U, Khlestkina E K, Kota R, Korzun V, Graner A, Börner A. Single nucleotide polymorphisms in rye (Secale cereale L.): discovery, frequency, and applications for genome mapping and diversity studies[J]. Theoretical and Applied Genetics, 2007, 114(6): 1105-1116. doi:10.1007/s00122-007-0504-6.
[30] Lijavetzky D, Cabezas J A, Ib
 ez A, Rodríguez V, Martínez-Zapater J M. High throughput SNP discovery and genotyping in grapevine (Vitis vinifera L.) by combining a re-sequencing approach and SNPlex technology[J]. BMC Genomics, 2007, 8(1): 424. doi:10. 1186/1471-2164-8-424.
ez A, Rodríguez V, Martínez-Zapater J M. High throughput SNP discovery and genotyping in grapevine (Vitis vinifera L.) by combining a re-sequencing approach and SNPlex technology[J]. BMC Genomics, 2007, 8(1): 424. doi:10. 1186/1471-2164-8-424.
[31] Wieser W. Temperature relations of ectotherms: a speculative review[M]//Effects of temperature on ectothermic organisms.Springer, 1973:1-23. doi:10.1007/978-3-642-65703-0_1.
[32] Clarke A, Johnston N M. Scaling of metabolic rate with body mass and temperature in teleost fish[J]. Journal of Animal Ecology, 1999, 68(5): 893-905. doi:10.1046/j.1365-2656.1999.00337.X.
[33] Windisch H S, Frickenhaus S, John U, Knust R, Pörtner H O, Lucassen M. Stress response or beneficial temperature acclimation: transcriptomic signatures in Antarctic fish(Pachycara brachycephalum)[J]. Molecular Ecology, 2014, 23(14): 3469-3482. doi:10.1111/mec.12822.
[34] Korobeinikova A V, Garber M B, Gongadze G M. Ribosomal proteins: structure, function, and evolution[J]. Biochemistry (Moscow), 2012, 77(6): 562-574. doi:10.1134/S0006297912060028.
[35] Pörtner H. Climate change and temperature-dependent biogeography: oxygen limitation of thermal tolerance in animals[J]. Naturwissenschaften, 2001, 88(4): 137-146. doi:10.1007/s001140100216.
[36] Pörtner H O. Oxygen-and capacity-limitation of thermal tolerance: a matrix for integrating climate-related stressor effects in marine ecosystems[J]. The Journal of Experimental Biology, 2010, 213: 881-893. doi:10.1242/jeb.037523.
[37] Múgica M, Sokolova I M, Izagirre U, Marigómez I . Season-dependent effects of elevated temperature on stress biomarkers, energy metabolism and gamete development in mussels[J]. Marine Environmental Research, 2015, 103: 1-10. doi:10.1016/j.marenvres.2014.10.005.
[38] Kooijman B, Kooijman S. Dynamic energy budget theory for metabolic organization[D].Cambridge: Cambridge University Press, 2010.