三七(Panax notoginseng (Burk) F.H. Chen )别名田七等,广泛分布在云南省文山州地区,是我国传统名贵中药材。《本草纲目》中记载三七具有止血散血定痛之功效[1],研究表明三七还具有抗血小板聚集、溶栓、降低血压、抗炎等多种功效[2]。三七性喜温暖阴湿,其独特的生长环境和较长的生长周期易诱发病害发生,尤其是根腐病等真菌病害[3]。三七根腐病的病原菌具有多样性,不但真菌病原可侵染,细菌和线虫亦可侵染致病,但以真菌为主。目前报道的三七根腐病的病原主要包括尖孢镰刀菌(Fusarium oxysporum)[4]、茄腐镰刀菌(F. solani)[5]、双孢柱孢(Cylindrocarpon didynum)[6]、毁灭柱孢菌(C. destructans)[7]、恶疫霉(Phytophthora cactorum)、草茎点霉(Phoma herbarum)、假单孢杆菌(Pseudomonas sp.)和小杆线虫(Rhabditis elegarts)[8]。
三七病原菌的传统检测方法有形态学鉴定、血清学和分子生物学检测等方法。龙月娟等[9]从三七根腐病植株根部中分离获得纯培养菌株,通过显微镜观察其形态特征并进行了致病性测定,根据形态特征结合致病性分析结果将分离到的菌株鉴定为毁灭柱孢菌,并通过rDNA-ITS序列分析验证了这一结论。Miao等[10]通过分离培养从三七腐烂和软化的三七根部组织中分离到一株真菌,通过rDNA-ITS序列扩增和同源性分析表明该真菌的序列与柔毛镰孢菌(F. flocciferum)具有100%的相似性。汪静等[11]从三七根腐病组织中获得分离物,纯化培养后运用科赫法则进行病原菌回接感染,得到3株致病明显的菌株,然后对菌株进行18s rDNA全序列分析鉴定,结果表明,它们分别是尖孢镰刀菌、茄腐镰刀菌和链格孢菌(Alternaria alternata)。Zheng等[12]从文山采集的三七腐烂根样品中分离得到了多株真菌,通过形态学及rDNA-ITS序列扩增分析的方法进行鉴定,结果表明分离得到了人参链格孢(A. panax)、尖孢镰刀菌、茄腐镰刀菌、草茎点霉和槭菌刺孢(Mycocentrospora acerina)。宋丽敏[13]制备了28种具有免疫原性的植物病毒蛋白抗血清,运用DAS-ELISA对皱缩症状的三七叶片进行血清学检测,在样品PN-1中检测到了ToMV病毒的存在。但在随后的样品中没有再次检测到阳性,认为ToMV病毒不是主要病原。
病原菌的形态学鉴定和核糖体序列分析耗时长,且PCR产物易产生假阳性结果。血清学检测的灵敏度较低,并且容易发生漏检。实时荧光定量PCR技术(Real-time PCR)是一种利用荧光信号积累实时监测整个PCR进程并通过标准曲线对未知模板进行定量分析的方法[14]。由于它具有很高的灵敏度、特异性、重现性和准确性,它已成为检测和定量植物病原菌的一种非常有效的方法[15]。Lin等[16]设计了FOCSC-1/FOCSC-2引物组,利用实时荧光定量PCR建立了尖孢镰刀菌的特异性定量检测方法,绘制了标准曲线并进行了实时荧光PCR扩增的熔融曲线分析,结果显示,利用FOCSC-1/FOCSC-2引物组可以准确检测出有症状的香蕉假茎和叶片中尖孢镰刀菌FOCg DNA片段的数量。此外,诊断结果还显示严重症状香蕉假茎和叶片中的DNA的含量分别比轻度症状香蕉中的高6 946倍和26.69倍。
尽管报道的三七根腐病的病原种类多,但是近年来的研究表明包括尖孢镰刀菌在内的镰刀属真菌是三七根腐病的重要致病菌。本研究采用实时荧光定量PCR对尖孢镰刀菌进行快速检测分析,旨在建立一种准确、快速的三七根腐病菌的检测方法,为田间三七根腐病、带菌土壤以及种子种苗的快速分子检测提供依据和技术支持。
1 材料和方法
1.1 试验材料
尖孢镰刀菌、茄腐镰刀菌、轮枝镰刀菌(F. verticillioides)、禾谷镰刀菌(F. graminearum)分离自三年生三七根腐病病株,人参链格孢分离自三年生三七黑斑病病叶。烟草赤星病菌(A. alternata)、核盘菌(Sclerotinia sclerotiorum)、葡萄座腔菌(Botryosphaeria dothidea)等病原真菌保存于云南省三七资源可持续利用重点实验室。所有三七试验样品采自云南省文山州三七种植基地。本试验所有试剂均购自上海生工生物有限公司。
1.2 尖孢镰刀菌特异性基因位点筛选及荧光定量特异性验证
下载GenBank数据库中已公开的尖孢镰刀菌的核酸序列,从中随机挑选出17个基因,以它们为模板设计PCR引物,以上述7种真菌的纯培养菌株的基因组DNA为模板进行PCR扩增,筛选特异性基因位点。PCR反应体系 (20 μL),包含2×Rapid Taq Master Mix 10 μL、无菌双蒸水7 μL、上游引物和下游引物各1 μL、DNA模板 1 μL。PCR扩增条件为:95 ℃ 3 min,然后94 ℃ 30 s,57 ℃ 30 s,72 ℃ 40 s,28个循环,72 ℃ 5 min。PCR产物通过1.2%琼脂糖凝胶电泳检测,对于只在尖孢镰刀菌中产生特异扩增的条带,将条带进行胶回收并进行TA克隆,然后测序并分析序列的同源性。
根据上述PCR筛选得到的尖孢镰刀菌特异的基因序列,设计特异性荧光定量引物对Fo-QF和Fo-QR。以7种真菌基因组DNA为模板,进行实时荧光定量PCR检测引物对Fo-QF和Fo-QR的特异性。荧光定量PCR反应体系(20 μL),包含无核酸酶水7 μL、2×SYBR Green Mix 10 μL、Fo-QF 2 μmol/L、Fo-QR 2 μmol/L和DNA模板各1μL。反应程序为95 ℃ 2 min,然后进行45个循环反应,每个循环反应为95 ℃ 1 min,62 ℃ 30 s,72 ℃ 1 min,于72 ℃时测定荧光值。每个真菌DNA模板设置3次重复,并设置空白对照(无模板)。Real-time PCR使用的设备为美国Bio-Rad iCyclerTM CFX96 Touch 荧光定量PCR检测系统。
1.3 实时荧光定量PCR标准曲线的建立
从筛选得到的尖孢镰刀菌特异扩增产物的阳性克隆子中提取重组质粒,测定质粒浓度,并计算质粒质量。将质粒DNA进行10倍梯度稀释制作10-1~10-5 系列标准样品,稀释介质为无核酸酶水。以标准品为模板进行实时荧光定量PCR反应,每个梯度设置3次重复,并根据Ct值与浓度梯度构建标准曲线。此外,将质粒DNA 再进行更低浓度的稀释(10-6~10-9)后进行实时荧光定量PCR反应,每个梯度设置3次重复,以确定该方法所能检测的最低核酸量。荧光定量PCR反应体系和程序与1.2相同。
1.4 三七病株以及种植土壤的实时荧光定量PCR检测
在文山州三七主产区采集具有根腐病、黑斑病典型症状的三七病株及三七种植土壤,运用本研究设计的引物及其检测方法对多个样品进行分析。用CTAB法提取各样品(0.1 g)的总DNA,按1.2反应体系及程序进行荧光定量PCR反应,每个样品的检测设置3次重复。将得到的Ct值均值代入标准曲线中,计算样品中尖孢镰刀菌的含量。
2 结果与分析
2.1 尖孢镰刀菌特异性基因位点分析
通过多个基因的筛选,获得了一个能特异性扩增尖孢镰刀菌的基因,尖孢镰刀菌脂肪酸ω-羟化酶基因CYP505 (GenBank No.AB030037.1),设计的普通PCR引物为Fo-F (5′-CATCCAACGCCGCCCA CTTTATC-3′)和Fo-R (5′-CAGCAACCACCAACGCT TCTTCG-3′)。PCR产物的凝胶电泳结果如图1所示。这对引物只在以尖孢镰刀菌DNA为模板的PCR扩增中产生378 bp的扩增产物,而在其他镰刀属真菌中不引起有效扩增。对扩增产物进行TA克隆和测序,序列的同源性分析显示扩增产物与尖孢镰刀菌CYP505脂肪酸ω-羟化酶基因有97%的相似性,证明PCR扩增产物为阳性产物。
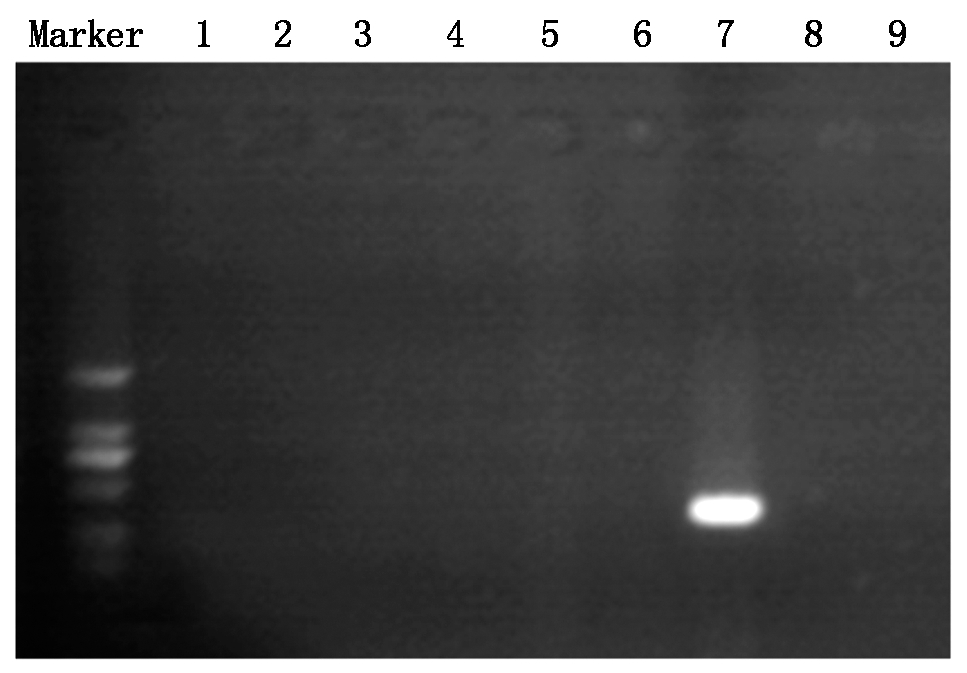
Marker. DL2000; 1.核盘菌; 2.烟草赤星病菌; 3. 人参链格孢; 4.轮枝镰刀菌; 5.禾谷镰刀菌; 6.茄腐镰刀菌; 7.尖孢镰刀菌; 8.葡萄座腔菌;9.阴性对照(无模板)。
Marker. DL2000; 1.S. sclerotiorum; 2.A. alternate; 3.A. panax; 4.F. verticillioides; 5.F. graminearum; 6.F. solani; 7.F. oxysporum; 8.B. dothidea; 9.Negative control (without template).
图1 验证尖孢镰刀菌脂肪酸ω-羟化酶基因CYP505特异性的PCR产物电泳图
Fig.1 Agarose gel electrophoresis to verify the specificity of F.oxysporum CYP505 gene for fatty acid omega-hydroxylase
2.2 实时荧光定量PCR引物特异性分析
以尖孢镰刀菌脂肪酸ω-羟化酶基因CYP505序列设计荧光定量PCR的引物对Fo-QF和Fo-QR,上下游引物序列分别为5′-CTCAAACAGTGGTACAT GCGAGG-3′和:5′-CATCTAGGTCTTCCATCCACTT GA-3′。扩增片段长度为209 bp。该引物的实时荧光定量PCR扩增曲线如图2-A所示,引物Fo-QF和Fo-QR只在尖孢镰刀菌DNA中产生了有效的扩增,3次重复得到3条重合的扩增曲线,其Ct值分别为23.29,23.03,23.32。此外,熔解曲线图2-B峰型单一,峰值均在85 ℃左右,Tm值相差<0.1 ℃,可见本研究设计的Real-time PCR引物特异性强。
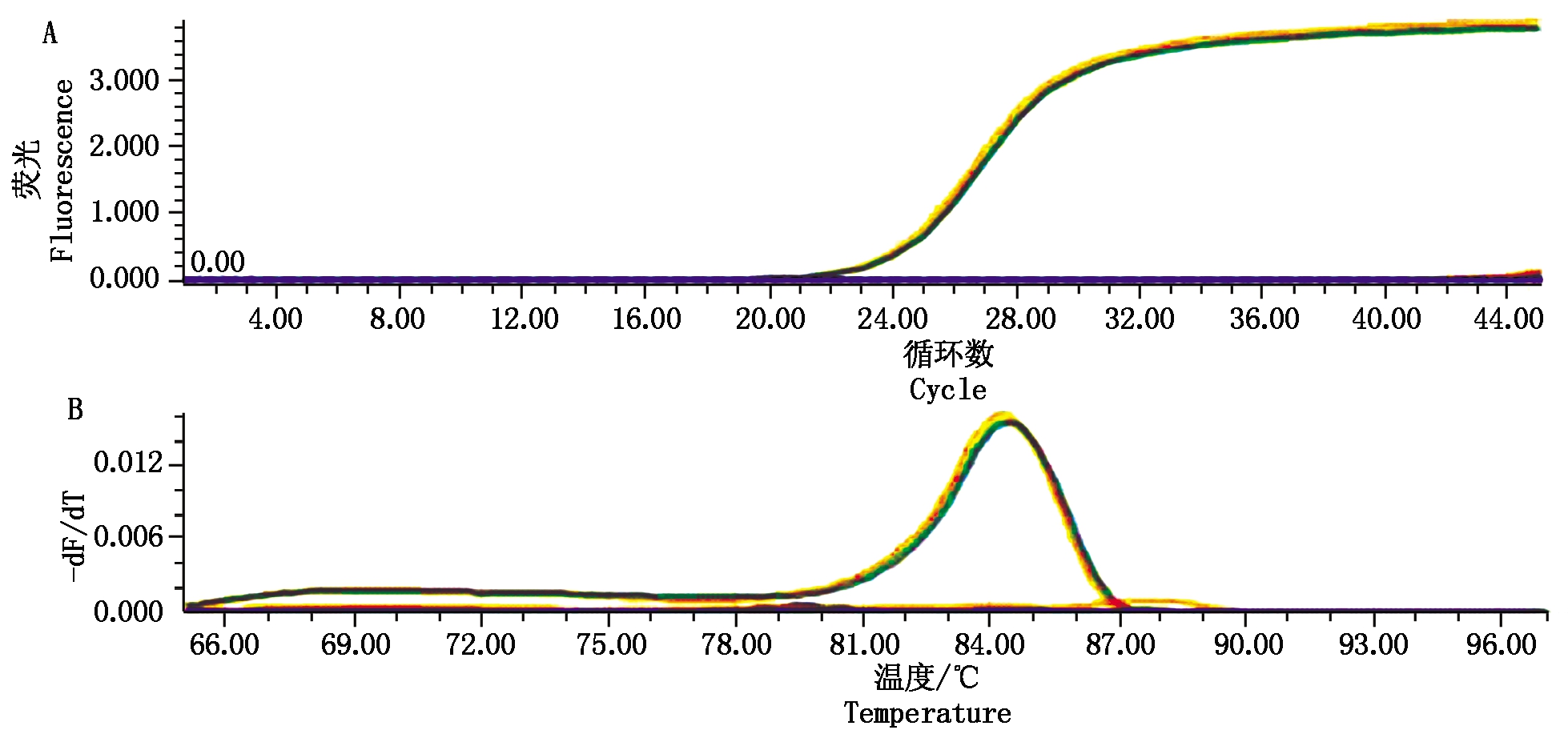
真菌DNA模板为人参链格孢、烟草赤星病菌、核盘菌、茄腐镰刀菌、尖孢镰刀菌、轮枝镰刀菌、禾谷镰刀菌、葡萄座腔菌。
Fungal DNA templates are A. panax,A. alternate,S. sclerotiorum,F. solani,F. oxysporum,F. verticillioides,F. graminearum,B. dothidea.
图2 引物对Fo-QF和Fo-QR对8种病原真菌DNA进行实时荧光定量PCR分析的扩增曲线图(A)以及熔解曲线图(B)
Fig.2 Amplification curve (A) and melting curve (B) of Real-time PCR of eight pathogenic
fungi DNA with primers Fo-QF and Fo-QR
2.3 实时荧光定量PCR标准曲线的建立
本试验以已知浓度的pGEM-T-CYP505质粒作为标准品进行10倍梯度稀释,所得5个梯度的质粒浓度见表1,对应的实时荧光定量PCR的扩增结果如图3所示,扩增曲线指数增长期的部分平行,反映了实时荧光PCR的扩增效率相近;而不同稀释度之间的Ct值相差均匀,反映了样本拷贝数与Ct值之间有良好的相关性。
5个标准品质粒质量对应的Ct均值见表1,并进一步构建标准曲线。以Ct均值为横坐标,即x值,以各梯度浓度对应的质粒质量对数值作为纵坐标,即y值,得到线性回归直线的方程y=-0.290 4x+4.344 5,其中相关系数R2=0.998 3 (图4)。不难发现荧光定量PCR中标准样品质粒DNA在10-1~10-5 的梯度稀释范围内与Ct均值具有很好的线性关系。
表1 尖孢镰刀菌质粒标准品的浓度梯度及其Ct值
Tab.1 Concentration of F. oxysporum plasmid standards and corresponding Ct values
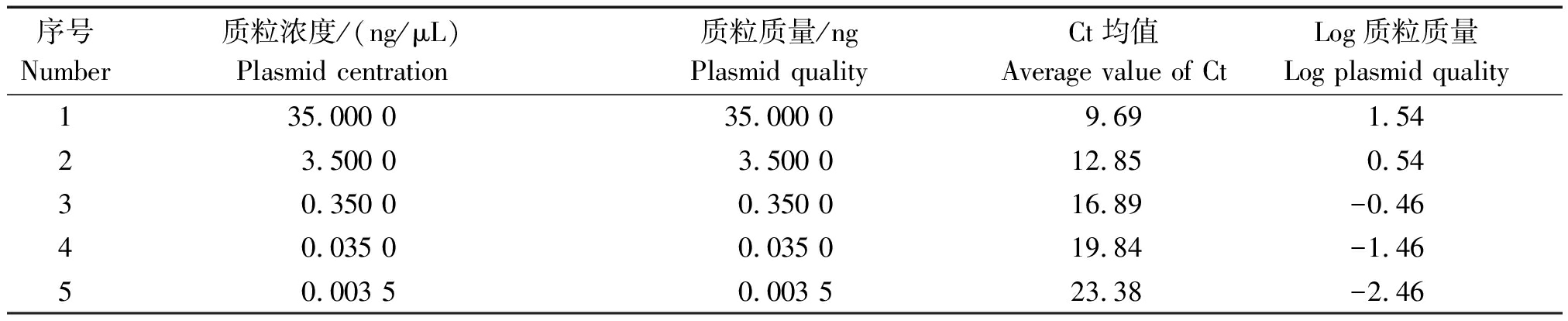
序号Number质粒浓度/(ng/μL)Plasmid centration质粒质量/ngPlasmid qualityCt均值Average value of CtLog质粒质量Log plasmid quality135.000 035.000 09.691.5423.500 03.500 012.850.5430.350 00.350 016.89-0.4640.035 00.035 019.84-1.4650.003 50.003 523.38-2.46
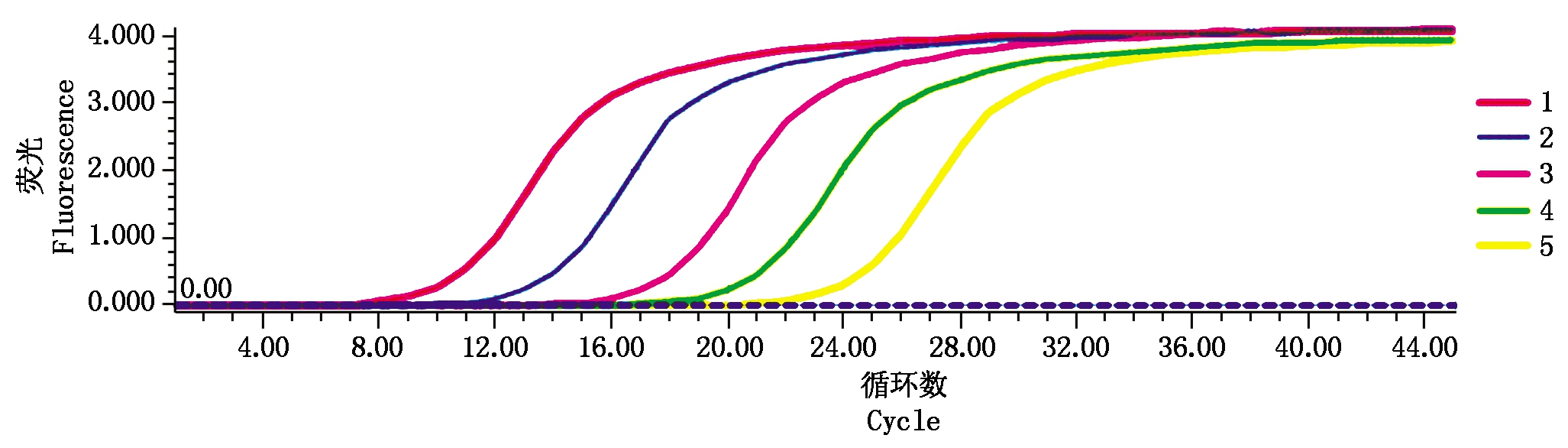
图3 pGEM-T-CYP5055质粒的实时荧光定量PCR的扩增曲线图
Fig.3 Amplification curve of Real-time PCR of pGEM-T-CYP5055 plasmids
此外,进一步试验结果显示当质粒浓度低于0.35 pg/μL的时候,质粒质量对数值与Ct值不再具有线性相关,因此表明本研究建立的尖孢镰刀菌实时荧光定量PCR检测方法的最低模板浓度为0.35 pg/μL,可见本研究的实时荧光定量PCR检测方法灵敏度高。
2.4 三七病株以及种植土壤的实时荧光定量PCR检测
从三七根腐病、黑斑病病株及种植土壤中提取总DNA,然后进行实时荧光定量PCR以检测尖孢镰刀菌的含量。各样品的扩增曲线见图5,将各样品3次重复的Ct均值带入标准曲线计算得到尖孢镰刀菌含量(表2)。几个三七根腐病样品(7~10号)有明显扩增,Ct值平均值分别为29.53,34.20,33.77,33.22,尖孢镰刀菌的含量均大于7 pg/g样品,含量高的多达177 pg/g样品,可见尖孢镰刀菌在三七根腐病病株的根部样品中大量存在。其余样品均无扩增,说明在健康的三七根部及其种植土壤、三七黑斑病病叶及健康叶片和根腐病土壤样品中均没有检测到尖孢镰刀菌。上述试验结果显示,本研究设计的引物以及建立的实时荧光定量PCR方法特异性强、灵敏、可靠,能准确诊断三七根腐病样品中是否含有尖孢镰刀菌以及测定尖孢镰刀菌的含量。
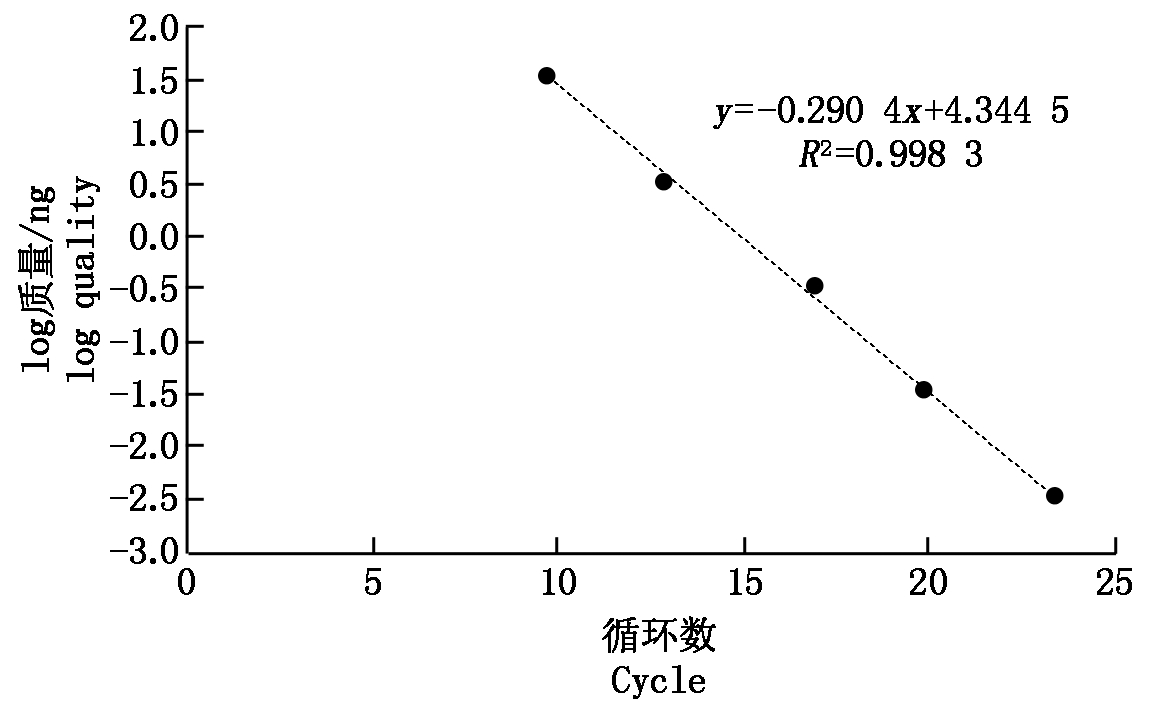
图4 标准曲线图
Fig.4 Standard curve
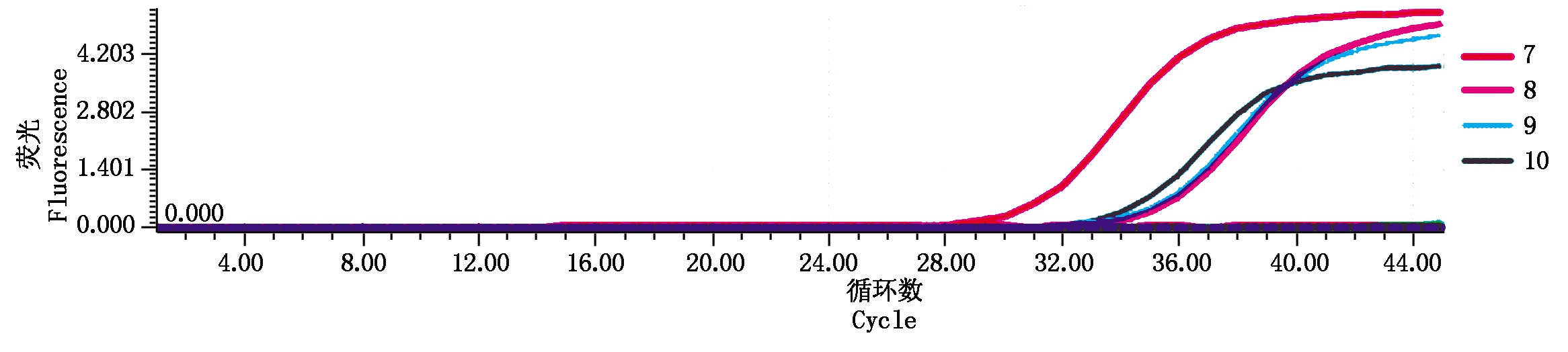
DNA模板为三七根腐病、黑斑病等典型症状的植株以及三七种植土壤等14个样本的DNA。
DNA templates are DNA from fourteen samples including P. notoginseng plants with typical symptoms
of root rot and black spot as well as soil of P. notoginseng planting.
图5 实时荧光定量PCR检测三七根部样品和土壤样品的扩增曲线图
Fig.5 Amplification curves of Real-time PCR for detection of P. notoginseng and soil samples
表2 14个三七及其种植土壤样品
的Real-time PCR检测结果
Tab.2 Real-time PCR results of 14 P. notoginseng
plant and soil samples
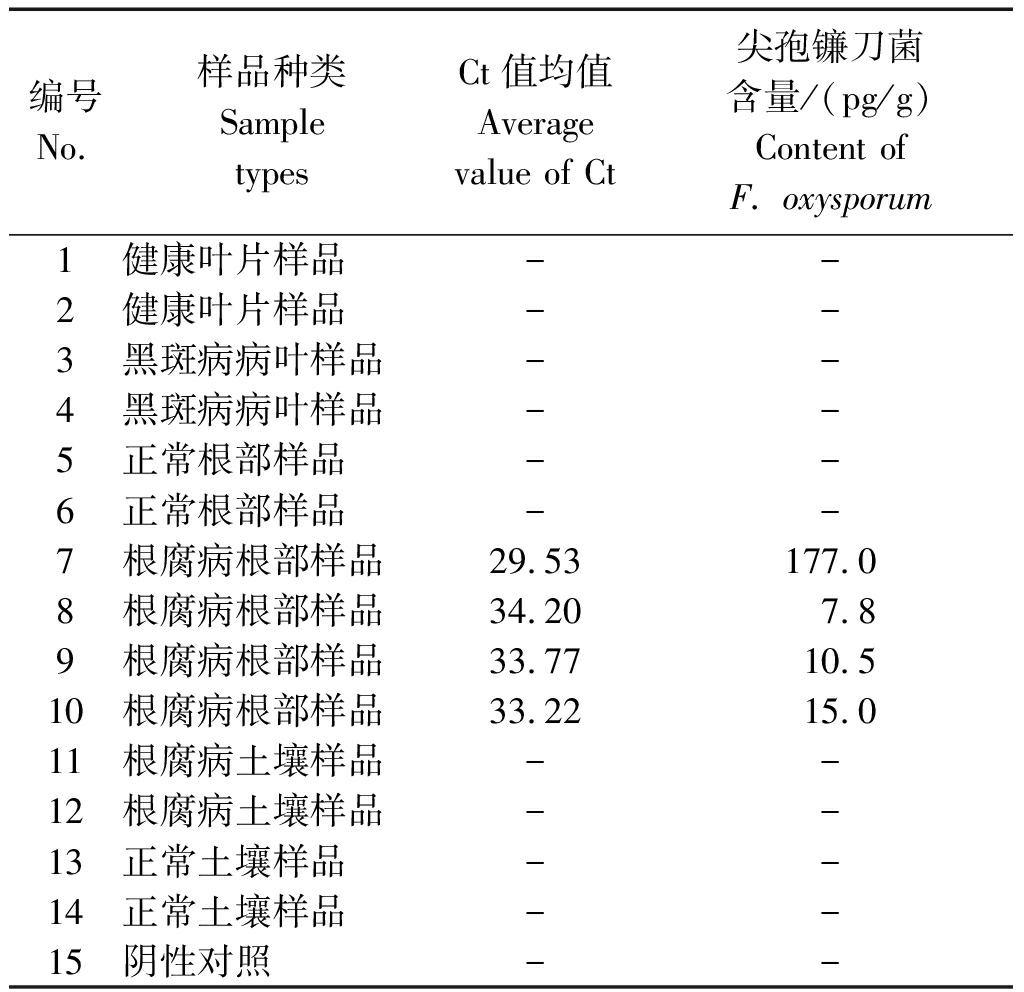
编号No.样品种类Sample typesCt值均值Average value of Ct尖孢镰刀菌含量/(pg/g)Content ofF. oxysporum1健康叶片样品--2健康叶片样品--3黑斑病病叶样品--4黑斑病病叶样品--5正常根部样品--6正常根部样品--7根腐病根部样品29.53177.08根腐病根部样品34.207.89根腐病根部样品33.7710.510根腐病根部样品33.2215.011根腐病土壤样品--12根腐病土壤样品--13正常土壤样品--14正常土壤样品--15 阴性对照--
3 讨论
本研究从三七根腐病致病真菌尖孢镰刀菌的多个基因中,筛选得到了特异扩增尖孢镰刀菌的脂肪酸ω-羟化酶基因CYP505,并针对此基因序列分别设计了普通PCR和实时荧光定量PCR的引物,采用SYBR Green Ⅰ实时荧光定量PCR技术[17]构建了尖孢镰刀菌定量检测的标准曲线,最终建立了三七根腐病菌尖孢镰刀菌的实时荧光定量PCR检测方法。该方法灵敏度高,检测模板的浓度可低至0.35 pg/μL。将本研究建立的尖孢镰刀菌检测方法用于检测一些三七病株和种植土壤,发现根腐病发病植株根部的尖孢镰刀菌含量显著高于健康植株及土壤,这说明尖孢镰刀菌在三七根腐病的发生过程中的确具有重要作用,这也验证了董鲜等[18]的研究结果。
尖孢镰刀菌是一种多寄主病原菌,普遍存在于农业生态系统中,能侵染多种作物并对其产生不同程度的伤害[19]。Kashiwa等[20]基于尖孢镰刀菌的一个内多聚半乳糖醛酸酶基因的序列设计了实时荧光定量PCR的特异引物和探针,运用Real-time PCR方法成功定量了卷心菜种植土壤中尖孢镰刀菌基因组DNA的数量,并发现当土壤中尖孢镰刀菌密度大于(2.0×103孢子/g土壤)时,卷心菜开始出现黄色枯萎病症状。汲珊珊等[21]针对尖孢镰刀菌β-tubulin基因中一段117 bp的序列,设计了特异引物和探针,对尖孢镰刀菌和其他9种临床常见的致角膜炎真菌的基因组DNA进行了检测。结果表明,该引物可以特异扩增尖孢镰刀菌,Ct值为17.178,而对其余真菌均无扩增。
关于三七根腐病致病菌尖孢镰刀菌快速分子检测方面目前还没有相关报道,本研究建立的实时荧光PCR检测体系能明确复杂三七种植土壤以及三七病株中尖孢镰刀菌的数量动态变化。本研究的方法对揭示三七根腐病的发病规律,以及建立该病害的防控技术提供了工作基础以及理论基础。同时,还可以为三七土壤处理、根腐病的早期诊断和动态监测以及带病三七种子、种苗的分子检测提供技术支持。后续需要进一步加强在种植基地实地检测的研究,并进一步认识尖孢镰刀菌的致病过程与机制,有针对性地研究高效、环保的防治方法。
[1] 张静静, 刘桂芹, 王庆鹏, 李兰杰. 植物三七中皂苷类化学成分的研究进展[J]. 聊城大学学报(自然科学版), 2018, 31(2): 43-59. doi: 10.19728/j.issn1672-6634.2018.02.006.
Zhang J J, Liu G Q, Wang Q P, Li L J. Advances in studies on chemical constituents of saponin from Panax notoginseng[J]. Journal of Liaocheng (Natural Science Edition), 2018, 31(2): 43-59.
[2] Wang J, Huang Z G, Cao H, Wang Y T, Hui P, Hoo C, Li S P. Screening of anti-platelet aggregation agents from Panax notoginseng using human platelet extraction and HPLC-DAD-ESI-MS/MS[J]. Journal of Separation Science, 2008, 31(6-7):1173-1180. doi: 10.1002/jssc.200700507.
[3] Wu Z X, Hao Z P, Sun Y Q, Guo L P, Huang L Q, Zeng Y, Wang Y, Yang L, Chen B D. Comparison on the structure and function of the rhizosphere microbial community between healthy and root-rot Panax notoginseng[J]. Applied Soil Ecology, 2016, 107:99-107. doi:10.1016/j.apsoil.2016.05.017.
[4] 毛忠顺, 龙月娟, 朱书生, 陈中坚, 魏富刚, 朱有勇, 何霞红. 三七根腐病研究进展[J]. 中药材, 2013, 36(12): 2051-2054.doi:10.13863/j.issn1001-4454.2013.12.004.
Mao Z S, Long Y J, Zhu S S, Chen Z J, Wei F G, Zhu Y Y, He X H. Research progress on root rot of Panax notoginseng[J]. Journal of Chinese Medicinal Materials, 2013, 36(12): 2051-2054.
[5] 缪作清, 李世东, 刘杏忠, 陈昱君, 李云华, 王勇, 郭荣君, 夏振远, 张克勤.三七根腐病病原研究[J]. 中国农业科学, 2006, 39(7):1371-1378. doi:10.3321/j.issn:0578-1752.2006.07.011.
Miao Z Q, Li S D, Liu X Z, Chen Y J, Li Y H, Wang Y, Guo R J, Xia Z Y, Zhang K Q. The causal microorganisms of Panax notoginseng root rot disease[J]. Scientia Agricultura Sinica,2006, 39(7):1371-1378.
[6] Wang W Y, Zhao C L, Chen Z J, Wen G S, Wei F G, Long T G, Li S W, Wang C D. Studies on the isolation, identification and in vitro growth rates of the three pathogenic fungi from Panax notoginseng cultivated in Wenshan Eparchy[J]. Agricultural Science and Technology, 2015, 16(6):1165-1171.doi:10.16175/j.cnki.1009-4229.2015.06.018.
[7] Mao Z S, Long Y J, Zhu Y Y, Zhu S S, He X H, Chen Z J. First report of Cylindrocarpon destructans var. destructans causing black root rot of Sanqi (Panax notoginseng) in China[J]. Plant Disease, 2014, 98(1):162. doi:10.1094/PDIS-11-12-1104-PDN.
[8] 蒋妮, 覃柳燕, 叶云峰. 三七病害研究进展[J]. 南方农业学报, 2011, 42 (9):1070-1074. doi:10.3969/j.issn.2095-1191.2011.09.010.
Jiang N,Qin L Y,Ye Y F. Research advances in diseases of Panax notoginseng[J]. Journal of Southern Sciences, 2011, 42 (9):1070-1074.
[9] 龙月娟, 毛忠顺, 朱书生, 陈中坚, 魏富刚, 尹兆波, 朱有勇, 何霞红. 三七锈斑病的病原菌[J]. 菌物学报, 2015, 34(2):177-185. doi: 10.13346/j.mycosystema.140013.
Long Y J, Mao Z S, Zhu S S, Chen Z J, Wei F G, Yin Z B, Zhu Y Y, He X H.The pathogens of Panax notoginseng root rust spot[J]. Mycosystema, 2015, 34(2):177-185.
[10] Miao C P, Qiao X G, Zheng Y K, Chen Y W, Xu L H, Guan H L, Zhao L X. First report of Fusarium flocciferum causing root rot of Sanqi (Panax notoginseng) in Yunnan, China[J]. Plant Disease, 2015, 99(11):1650.doi:10.1094/PDIS-11-14-1168-PDN.
[11] 汪静, 梁宗锁, 康冰, 罗美佳.文山三七根腐病病原真菌的鉴定与药剂防治[J]. 西北林学院学报, 2015, 30(1):158-163.doi:10.3969/j.issn.1001-7461.2015.01.26.
Wang J, Liang Z S, Kang B, Luo M J. Identification of root rot pathogen of Panax notoginseng from Wenshan[J]. Journal of Northwest Forestry University, 2015, 30(1):158-163.
[12] Zheng Y K, Miao C P, Chen H H, Huang F F, Xia Y M, Chen Y W, Zhao L X. Endophytic fungi harbored in Panax notoginseng: diversity and potential as biological control agents against host plant pathogens of root-rot disease[J]. Journal of Ginseng Research, 2017, 41(3):353-360. doi:10.1016/j.jgr.2016.07.005.
[13] 宋丽敏. 三七病毒病害病原的初步鉴定[D]. 北京:中国农业大学, 2005. doi: 10.7666/d.y774579.
Song L M. Detection and identification of the viruses infecting Panax notoginseng[D]. Beijing:China Agricultural University College of Agronomy and Biotechnology, 2005.
[14] 龚美蓉, 陈凤丽, 曹晨, 孙亦农, 王明艳. 实时荧光定量PCR技术及其在中医药研究中的应用[J]. 中国实验方剂学杂志, 2014, 20(22):238-241. doi: 10.13422/j.cnki.syfjx.2014220238.
Gong M R,Chen F L,Cao C,Sun Y N,Wang M Y. Application of Real-time fluorescence quantitative PCR in Chinese medicine research[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2014, 20(22):238-241.
[15] Zheng S, Shan L Y, Zhuang Y L, Shang Y. Identification of pyrG used as an endogenous reference gene in qualitative and Real-time quantitative PCR detection of Pleurotus ostreatus[J]. Journal of Food Science, 2018, 83(3): 750-755. doi: 10.1111/1750-3841.14072.
[16] Lin Y H, Su C C, Chao C P, Chen C Y, Chang C J, Huang J W, Chang P F. A molecular diagnosis method using Real-time PCR for quantification and detection of Fusarium oxysporum f. sp.cubense race 4[J]. European Journal of Plant Pathology, 2013, 135(2):395-405. doi: 10.1007/s10658-012-0096-0.
[17] Kędrak-Jab ońska A, Budniak S, Krupa M, Szczawińska A, Reksa M, Szulowski K, Iwaniak W. Detection of Listeria spp. and Listeria Monocytogenes in biological samples by SYBR Green I and TaqMan probe-based Real-time PCRs[J]. Journal of Veterinary Research, 2017, 61(4):427-432. doi: 10.1515/jvetres-2017-0069.
ońska A, Budniak S, Krupa M, Szczawińska A, Reksa M, Szulowski K, Iwaniak W. Detection of Listeria spp. and Listeria Monocytogenes in biological samples by SYBR Green I and TaqMan probe-based Real-time PCRs[J]. Journal of Veterinary Research, 2017, 61(4):427-432. doi: 10.1515/jvetres-2017-0069.
[18] 董鲜, 马晓惠, 陈传娇, 耿连用, 杨志慧, 鲁荣昌, 徐福荣. 三七根腐病尖孢镰刀菌分离鉴定及致病作用研究[J]. 中药材, 2018, 41(1):8-12. doi: 10.13863/j.issn1001-4454.2018.01.002.
Dong X,Ma X H,Chen C J,Geng L Y,Yang Z H,Lu R C,Xu F R. The isolation and identification of Fusarium oxysporum and the study about its pathogenesis effect on Panax notoginseng[J].Journal of Chinese Medicinal Materials, 2018, 41(1):8-12.
[19] 高晓敏, 王琚钢, 马立国, 云兴福. 尖孢镰刀菌致病机理和化感作用研究进展[J]. 微生物学通报, 2014, 41(10):2143-2148. doi:10.13344/j.microbiol.china.140406.
Gao X M, Wang J G, Ma L G, Yun X F. Research advances on the mechanism of pathogenesis and allelopathy of Fusarium oxysporium[J]. Microbiology China, 2014, 41(10):2143-2148.
[20] Kashiwa T, Inami K, Teraoka T, Komats K, Arie T. Detection of cabbage yellows fungus Fusarium oxysporum f. sp. conglutinans in soil by PCR and Real-time PCR[J]. Journal of General Plant Pathology, 2016, 82(5):240-247. doi: 10.1007/s10327-016-0668-5.
[21] 汲珊珊, 李君文, 安娜, 谌志强, 陈照立, 邱志刚, 刘先宁, 王新为, 郭长江, 金敏. 尖孢镰刀菌TaqMan探针荧光定量PCR快速检测方法的建立及应用[J]. 现代检验医学杂志, 2012, 27(6):30-33. doi: 10.3969/j.issn.1671-7414.2012.06.010.
Ji S S, Li J W, An N, Shen Z Q, Chen Z L, Qiu Z G, Liu X N, Wang X W, Guo C J, Jin M. Establishing and applying of TaqMan probe Real-tirne PCR detection of Fusarium oxysporum[J]. Journal of Modern Laboratory Medicine, 2012, 27(6):30-33.