miRNA是一类生物体内源性的长度21~24 nt的非编码小分子RNA。miRNA通过序列互补与特定靶基因的mRNA结合,引起mRNA降解或抑制mRNA翻译或造成DNA的甲基化,对基因的表达起到负调节作用[1-3]。miRNA最初在秀丽新小杆线虫中被发现[4-5],随后被证明广泛存在于动物、植物和病毒中。2002年,最初在植物中发现miRNA的存在[6],随后,通过克隆测序、生物信息学分析和高通量测序等方法在拟南芥、水稻、大豆、棉花等作物中鉴定到大量miRNA,并证明miRNA在植物的生长发育及适应逆境胁迫中有重要作用。miRNA在植物发育、开花、熟性等方面相关研究也有报道。拟南芥中,miR172通过其AP2家族靶基因调控拟南芥从营养生长到生殖生长的转换和开花的早晚[7-8]。miR156通过调控SPL家族的转录因子调节拟南芥开花,过表达miR156导致拟南芥延迟开花 [9-10]。
随着高通量测序技术的发展和棉花基因组测序的不断完善,棉花中miRNA的鉴定和相关功能研究越来越多。2007年,Zhang和Qiu等[11-12]分别利用生物信息学的方法最先在棉花中发现miRNA的存在。2008年,Abdurakhmonov等[13]以开花后0~10 d的棉花胚珠为材料分别构建了11个小RNA库,鉴定了新的miRNA,分析了相关miRNA在开花后不同时期的表达情况。随后,通过高通量测序技术,大量棉花纤维发育相关miRNA被鉴定[14-15]。另外,miRNA在棉花非生物胁迫和抗病菌等方面的作用也有报道。2015年,Xie等[16]利用高通量测序技术在棉花中鉴定到大量对盐胁迫和干旱胁迫响应的miRNA。Zhu等[17]研究发现,棉花中miR482家族的4个miRNA,通过自身表达的下调提高其靶基因NBS-LRR的表达,从而增强自身对病菌的抵抗。
棉花是我国重要的经济作物,随着棉粮争地矛盾的突出,越来越多的早熟棉品种审定并推广,棉花早熟性相关研究也越来越重要。目前,棉花中miRNA研究主要集中在棉花纤维发育、逆境胁迫等方面相关研究。本研究中,以棉花早熟品种和中熟品种为材料,通过高通量测序技术构建2个小RNA文库,鉴定在早熟棉花中表达的miRNA,比较miRNA在早熟棉和中熟棉中表达的差异,为miRNA在棉花早熟性调控作用研究中提供依据。
1 材料和方法
1.1 材料培养及高通量测序
本研究选用石早1和冀棉958 2个棉花品种作为研究材料。其中石早1是河北省审定的早熟棉品种,冀棉958是河北省审定的中熟棉品种。石早1和冀棉958棉花种子播种于蛭石和营养土3∶1混合基质的培养盆中,温度为28 ℃,相对湿度为70%。苗期取幼嫩叶片,利用mirVana-miRNA Isolation试剂盒(Ambion, Austin, TX, USA)提取小RNA。2个样品的小RNA委托上海美吉生物公司,利用Illumina Hiseq测序平台进行高通量测序,获得SZ1(石早1)和JM958(冀棉958)2个小RNA文库。
1.2 数据分析
利用SeqPrep软件,对高通量测序得到的原始数据去除接头序列、3′端低质量碱基序列,含N比率超10%的序列、超短序列(<18 bp)、超长序列(>32 nt)等污染序列,得到18~32 bp的高质量干净序列。
使用Rfam数据库(http://Rfam.sanger.ac.uk/)对测得的小RNA序列进行注释,去除其中非miRNA序列的干扰,如 rRNA、scRNA、snoRNA、snRNA、tRNA等序列。
rRNA、scRNA、snoRNA、snRNA、tRNA等非已知miRNA序列去除后,利用Bowtie软件将小RNA序列定位到参考基因组上,分析这些序列在基因组上的分布情况。
将去除rRNA、scRNA、snoRNA、snRNA、tRNA等序列后的小RNA序列与miRBase数据库(http://www.mirbase.org/)中棉花已知miRNA及前体序列进行比对,能完全比对上的序列为已知miRNA。
小RNA比对到参考基因组上,截取其周围序列作为小RNA的前体序列,利用Mfold软件(http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form)对前体序列的二级结构、能量值特征进行预测[18],鉴定新的miRNA。判断标准为:小RNA 前体序列形成一个发夹式的二级结构,该二级结构的 dG 值尽可能的低;小RNA序列在二级结构的 5′或3′臂部,而不能在环上;在前体发夹结构上,miRNA 最多只能有6 nt的错配,且不能有大的间断[19]。将得到的新的miRNA序列在miRbase数据库中与棉花和其他物种中的已知miRNA进行比对,分析新的miRNA所属的miRNA家族。
将2个样品归一化到同一个量级,公式:归一化的表达量=miRNA 表达量/样品总 miRNA 表达量×归一量级。使用标准化后的结果对石早1和冀棉958中鉴定到的miRNA表达差异进行分析。
2 结果与分析
2.1 高通量测序数据初步分析
利用Illumina Hiseq测序平台对石早1和冀棉958进行高通量测序,构建石早1和冀棉958 2个小RNA文库。结果,在石早1和冀棉958中小RNA总读取次数(reads)分别为7 529 830 和 7 541 032。将接头序列、低质量碱基序列,超短序列等污染序列去除后,2个文库中分别得到5 916 831,5 941 660个18~32 nt的干净reads。这些reads主要集中在长度21~24 nt的序列,其中长度为24 nt的序列最多,其次为21 nt序列,然后依次为22, 23 nt序列(图1)。这种模式的长度分布和以往的研究一致[20-21]。
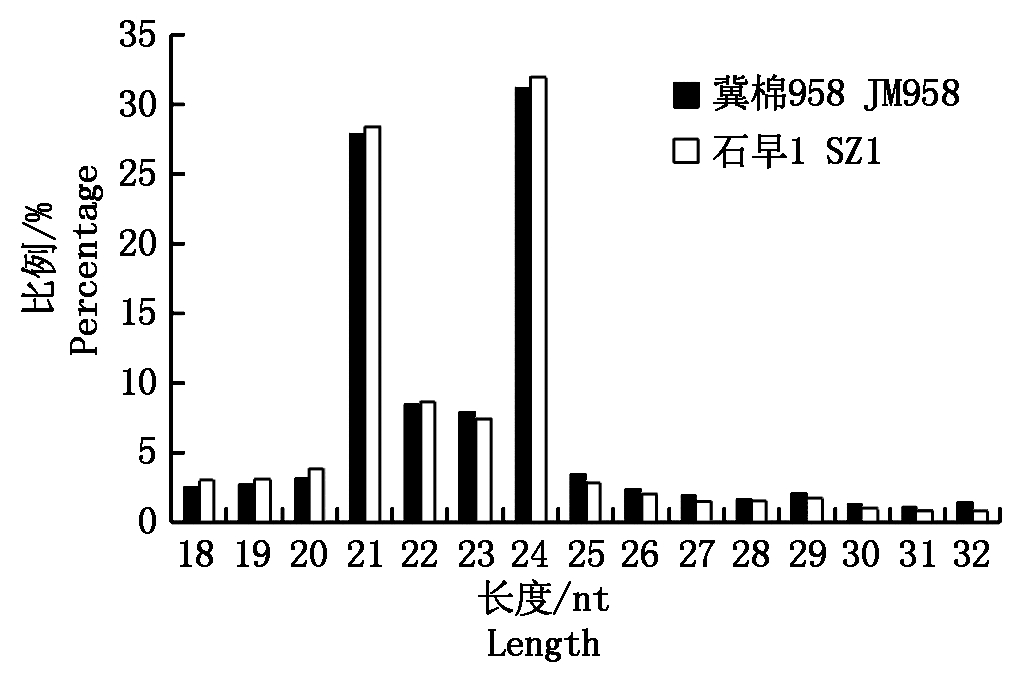
图1 石早1和冀棉958中小RNA序列长度分布
Fig.1 Size distribution of small RNA raw
reads from SZ1 and JM958
对得到的干净reads,合并完全相同的序列后,2个小RNA库中共得到3 585 751个Unique序列。统计发现,大部分Unique序列在2个库中共有,占77%;石早1中特有Unique序列占12%,冀棉958中特有Unique序列占11%。另外,将这些序列在棉花各个染色体上的分布进行了统计,发现这些小RNA序列在Chr11染色体上占比例最大,为15%;Chr3染色体上占比例最小,为4%(图2)。这说明,小RNA在石早1和冀棉958 2个品种中表达存在较大的差异,且在不同染色体上的分布不同。
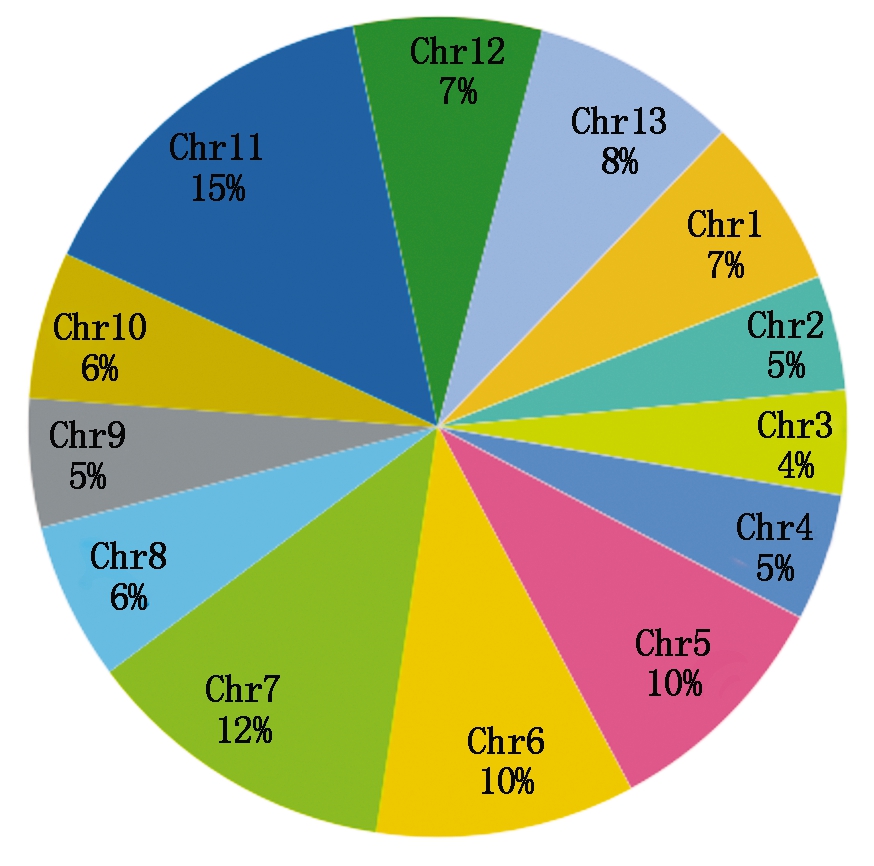
图2 小RNA序列染色体分布
Fig.2 The distribution of small RNA on chromosomes
2.2 miRNA鉴定
将测序得到的小RNA序列在Rfam数据库中进行比对,去除其中的rRNA、scRNA、snoRNA、snRNA、tRNA等非miRNA序列。将剩余的小RNA序列在miRBase数据库中与棉花的miRNA前体及成熟体序列进行比对,鉴定已知miRNA。结果,共得到属于41个不同家族的64个已知的miRNA。
表1 新miRNA鉴定
Tab.1 The identification of novel miRNAs
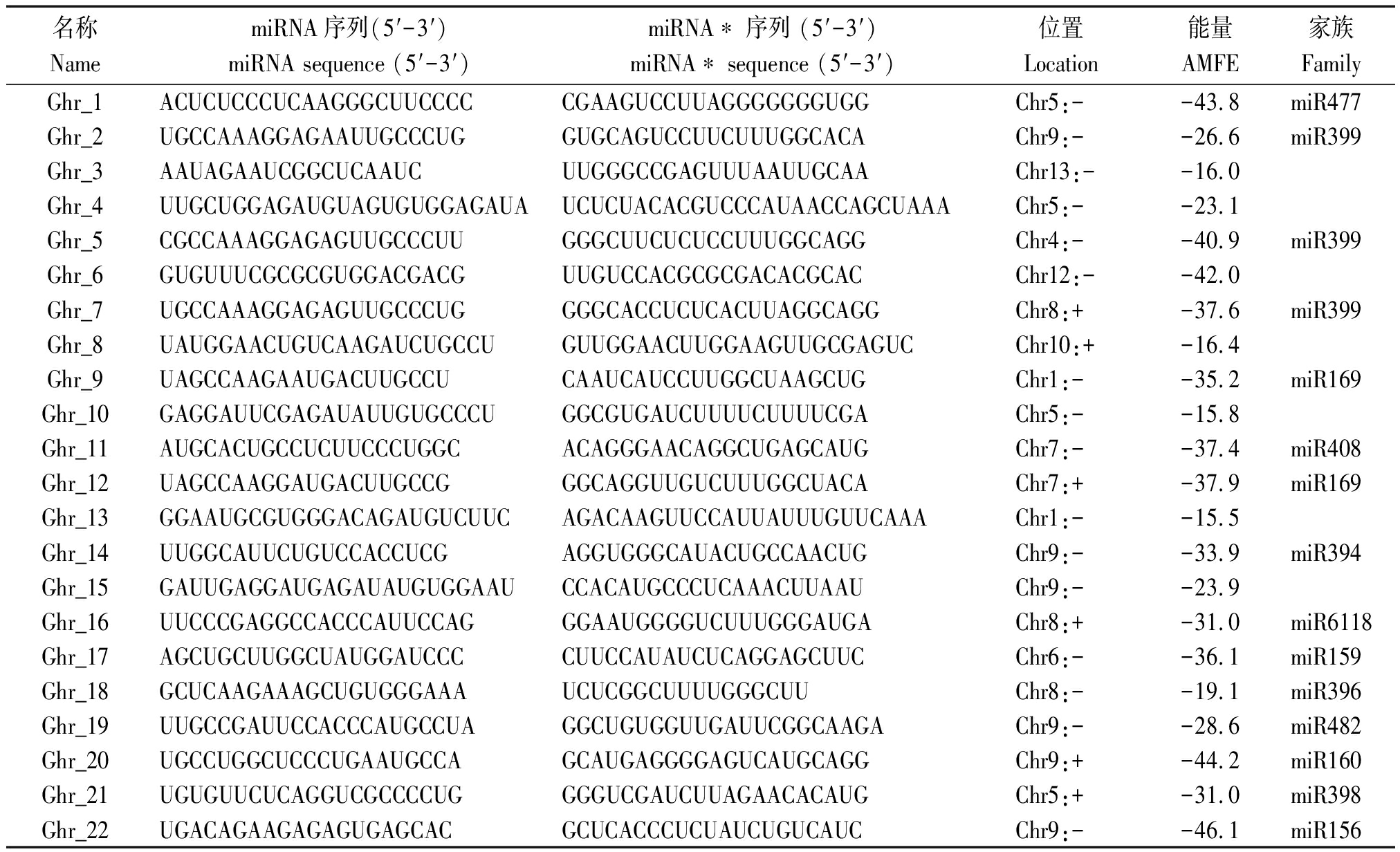
名称NamemiRNA序列(5′-3′)miRNA sequence (5′-3′)miRNA∗ 序列 (5′-3′)miRNA∗ sequence (5′-3′)位置Location能量AMFE家族FamilyGhr_1ACUCUCCCUCAAGGGCUUCCCCCGAAGUCCUUAGGGGGGGUGGChr5:--43.8miR477Ghr_2UGCCAAAGGAGAAUUGCCCUGGUGCAGUCCUUCUUUGGCACAChr9:--26.6miR399Ghr_3AAUAGAAUCGGCUCAAUCUUGGGCCGAGUUUAAUUGCAAChr13:--16.0Ghr_4UUGCUGGAGAUGUAGUGUGGAGAUAUCUCUACACGUCCCAUAACCAGCUAAAChr5:--23.1Ghr_5CGCCAAAGGAGAGUUGCCCUUGGGCUUCUCUCCUUUGGCAGGChr4:--40.9miR399Ghr_6GUGUUUCGCGCGUGGACGACGUUGUCCACGCGCGACACGCACChr12:--42.0Ghr_7UGCCAAAGGAGAGUUGCCCUGGGGCACCUCUCACUUAGGCAGGChr8:+-37.6miR399Ghr_8UAUGGAACUGUCAAGAUCUGCCUGUUGGAACUUGGAAGUUGCGAGUCChr10:+-16.4Ghr_9UAGCCAAGAAUGACUUGCCUCAAUCAUCCUUGGCUAAGCUGChr1:--35.2miR169Ghr_10GAGGAUUCGAGAUAUUGUGCCCUGGCGUGAUCUUUUCUUUUCGAChr5:--15.8Ghr_11AUGCACUGCCUCUUCCCUGGCACAGGGAACAGGCUGAGCAUGChr7:--37.4miR408Ghr_12UAGCCAAGGAUGACUUGCCGGGCAGGUUGUCUUUGGCUACAChr7:+-37.9miR169Ghr_13GGAAUGCGUGGGACAGAUGUCUUCAGACAAGUUCCAUUAUUUGUUCAAAChr1:--15.5Ghr_14UUGGCAUUCUGUCCACCUCGAGGUGGGCAUACUGCCAACUGChr9:--33.9miR394Ghr_15GAUUGAGGAUGAGAUAUGUGGAAUCCACAUGCCCUCAAACUUAAUChr9:--23.9Ghr_16UUCCCGAGGCCACCCAUUCCAGGGAAUGGGGUCUUUGGGAUGAChr8:+-31.0miR6118Ghr_17AGCUGCUUGGCUAUGGAUCCCCUUCCAUAUCUCAGGAGCUUCChr6:--36.1miR159Ghr_18GCUCAAGAAAGCUGUGGGAAAUCUCGGCUUUUGGGCUUChr8:--19.1miR396Ghr_19UUGCCGAUUCCACCCAUGCCUAGGCUGUGGUUGAUUCGGCAAGAChr9:--28.6miR482Ghr_20UGCCUGGCUCCCUGAAUGCCAGCAUGAGGGGAGUCAUGCAGGChr9:+-44.2miR160Ghr_21UGUGUUCUCAGGUCGCCCCUGGGGUCGAUCUUAGAACACAUGChr5:+-31.0miR398Ghr_22UGACAGAAGAGAGUGAGCACGCUCACCCUCUAUCUGUCAUCChr9:--46.1miR156
表1(续)

名称NamemiRNA序列(5′-3′)miRNA sequence (5′-3′)miRNA∗ 序列 (5′-3′)miRNA∗ sequence (5′-3′)位置Location能量AMFE家族FamilyGhr_23AUUCGGUAAACUCCGGUAAUGCCUGUGUUACAGUUUACCUAUCAAChr12:+-33.6Ghr_24CAGGGCUUCUCUGCAUUGGCAUGCCAAAGGAGAGUUGCCCUUChr8:--30.8miR399Ghr_25UGAAAUGGAUUCUACGGUAUCAACCGUAGAUGUCAAUUUGCUChr8:--15.8Ghr_26UUGACAGAAGAUAGAGAGCACGCUCUCUAUGCUUCUGUCAUCChr1:+-45.9miR157Ghr_27AAUUUCUUUCUUACUUGGGACACCAAGUGAGAAGGAAAUChr7:+-22.8Ghr_28CUGAAGUGUUUGGGGGAACUCGUUCCUCUGAUCACUUCAUUGChr13:--36.1miR395Ghr_29UGUCGCAGGAGCGAUGGCACUGGUGCCUUCGGCCUGCGACAAGChr7:+-40.6miR5786Ghr_30UUGGACAGAGUAAUCACGGUCGAUCGUGUUACUCCGUCCAAACChr5:--29.1miR3954Ghr_31UGCCUGGCUCCCUGUAUGCCAGCGUAUGAGGAGCCAAGCAUAChr12:+-49.4miR160Ghr_32UUCCAUCUCUUGCACACUGGAUGGUGUGCAGGGGGUGGAAUAChr7:--33.9miR2950Ghr_33UAAUCUGCAUCCUGAGGUUUAGUCCUUGGGGUGCAGAUUACCChr6:+-32.8miR2111Ghr_34UCAACCGGUUGGAACUCUUCCAAGAAUUUCAGCUGGUUGUGCCAChr6:--35.3miR5027Ghr_35UGUCGCAGGAGAGAUGGCACUGGUGCCGUCGCCUUGUGACAAGChr6:--33.2miR5786Ghr_36UUGGACUUGGAUACGUUGAAGAAGCUUCUUCAACGUAUCCAAGUCChr7:+-41.3Ghr_37UGAUUGAGCCGUGCCAAUAUCUGUUGGAAUGGCUCAAUCAAAChr8:+-31.6miR171Ghr_38UUGACAGAAGAGAGUGAGCACGCUCACUUCUCUUUCUGUCAGCChr1:+-44.5miR156Ghr_39AAUCUAGUUUCUCUCUUGCUCAAGGGAAAGACUAGAUUCGUAscaffold_64:+-34.7miR8766Ghr_40UGACAGAAGAGAGGGAGCACAUGCUUCCUCUCUUCUGCCACGChr10:+-48.8miR156Ghr_41UGACAGAAGAGAGUGAGCACUUGCUUACUUCUCUUCUGUCGGCChr13:--43.4miR156Ghr_42UUGGACCAGGCUUCAUUCCCCGGAAUGUUGCCUGGUUCAAGGChr4:+-33.3miR166Ghr_43UCUUACCAACCCCUCCCAUACCUGUGGGAGAGUUGGGCAAGAAUChr9:--31.4miR482Ghr_44CAGUCGCAGAACUCCGUACCUGGUACGGAGUUUUGCAACUGCChr5:--49.3Ghr_45UCGGACCAGGCUUCAUUCCCCGGAAUGCUGUCUGGUUCGAGAChr6:--45.0miR166Ghr_46UUCCACGGCUUUCUUGAACUUGUCAAGAAAGCCGUGGGAAAAChr5:+-40.8miR396Ghr_47UUUAGAAAUCGUUCCUUCCUUAAUGGAUGAUUUCUAAAGCUUChr10:+-28.8miR7505Ghr_48UCCAUAUUUCACUAUCUCUUAAGAGAUAGUGCAAUAUGGAGGChr2:+-45.1miR8746Ghr_49AUGAAUCUAGUUUCUCUCUUGCAAGGGAGACCUAGAUUCAUAAscaffold_64:+-35.5miR8677Ghr_50UUGAAUCUAGUUUCUCUCUUGCUAAGGGAGAUCUAGAUUCAUAAscaffold_64:+-37.7miR8677Ghr_51ACAGCUUUAGAAAUCACUCCUAUGAUUUCUAAAGCUUUAGAUChr10:+-26.0miR7505Ghr_52UUCCCAAAACCUCCAAUUCCAAGGAAUCGCGUUUUUGGGAAGAChr1:+-20.2miR482Ghr_53AAAUUUUUCAUCUUCACUCGUUAGUGAGGAUGGGAAAUUUGUChr2:--38.7Ghr_54UUAGAUUCACGCACAAACUCGAGUUUUGUGCGUGAAUCUAAUAChr8:--36.6miR403Ghr_55UCGAUAAACCUCUGCAUCCAGUGGAGGCAGCGGUUCAUCGAUCChr3:--33.6miR162Ghr_56UUCAGAAACCAUCCCUUCCUUGGAAGGAAUGGUUUCUGAAGCChr10:+-42.2miR7505Ghr_57UGAAGCUGCCAGCAUGAUCUUGAUCAUGUGGUAGCUUCACCChr1:+-36.2miR167Ghr_58UUUCCAAUUCCUCCCAUUCCACAAUGGAGGAGUUGGAAAGAUUChr2:--30.2miR482Ghr_59UGAAGCUGCCAGCAUGAUCUAGAUCAUAUGGCAGUUUCGUCChr7:--35.4miR167Ghr_60UUCCCGGCUUCUUUUCUUGCUCAAGAAAAGAAGUCGGGAGAGChr7:+-39.7miR7508Ghr_61UCCAAAGGGAUCGCAUUGAUCCAUCAUGCUAUCCCUUCGGAUUChr12:+-25.3miR393Ghr_62UGACAACGAGAGAGAGCACGUGUGCUCUCUAUCGGUGUCAUGChr1:+-44.0miR535Ghr_63UCGGACUGGAUUUGUUGACAAGUCAACAAAUUCAGUUUGAGCChr3:--32.6miR3476Ghr_64UCGGACCAGGCUUCAUUCCCCGGAAUGUUGUCUGGUUCGAUGChr4:--38.9miR166Ghr_65UAUACCGUGCCCAUGACUGUACGUUAUGGGCACGGUAUGGAChr2:--45.3miR2947Ghr_66UCGGACCAGGCUUCAUUCCGGAAUGUUGGCUGGCUCGAAGChr12:--36.8miR166Ghr_67UCUUCCCAACACCUCCCAUACCUAUGGGGGGUAUGGGCAGGUUChr5:+-31.7miR482
miRNA前体具有标志性发夹结构,可以用来预测新的miRNA。小RNA比对到参考基因组上,截取周围序列作为其前体序列,对其进行二级结构预测,根据预测结果结合Dicer酶切位点、能量值等特征信息鉴定新的miRNA。结果,在2个小RNA库中共得到67个新的miRNA(表1),丰富了棉花中miRNA数量。这些新的miRNA在5号和7号染色体上分布最多,均为8个,其次为1,8,9号染色体,均为7个(表1)。另外。将这些新的miRNA在miRbase数据库中与棉花及其他物种中的已知miRNA序列进行比对,发现有54个新的miRNA属于35个已知的miRNA家族,13个miRNA属于新的miRNA家族。其中,ghr_39、ghr_47、ghr_48、ghr_49、ghr_50、ghr_51、ghr_56、ghr_60、ghr_63和ghr_65 10个新的miRNA所属家族只在棉花中保守。这些家族的miRNA只在棉花中发现,在棉花生长发育过程中可能存在特殊的作用[22]。
对鉴定到miRNA的reads数进行统计,找到8个高表达的已知miRNA和7个高表达的新miRNA,reads数均超过10 000。其中,ghr_45 reads数最高,在2个小RNA库中均超过22万次,其次为miR166b,在2个小RNA库中均超过19万次,这2个miRNA均属于miR166家族。这15个高表达miRNA中,只有miR398家族的ghr_21,在2个小RNA文库中表达差异较大,在冀棉958中resds数是石早1的3.4倍;其余14个高表达miRNA在石早1和冀棉958 2个小RNA文库中表达差异不明显(图3)。这15个高表达miRNA分别属于miR166、miR7505、miR396、miR482、miR156、miR535、miR398 7个miRNA家族。
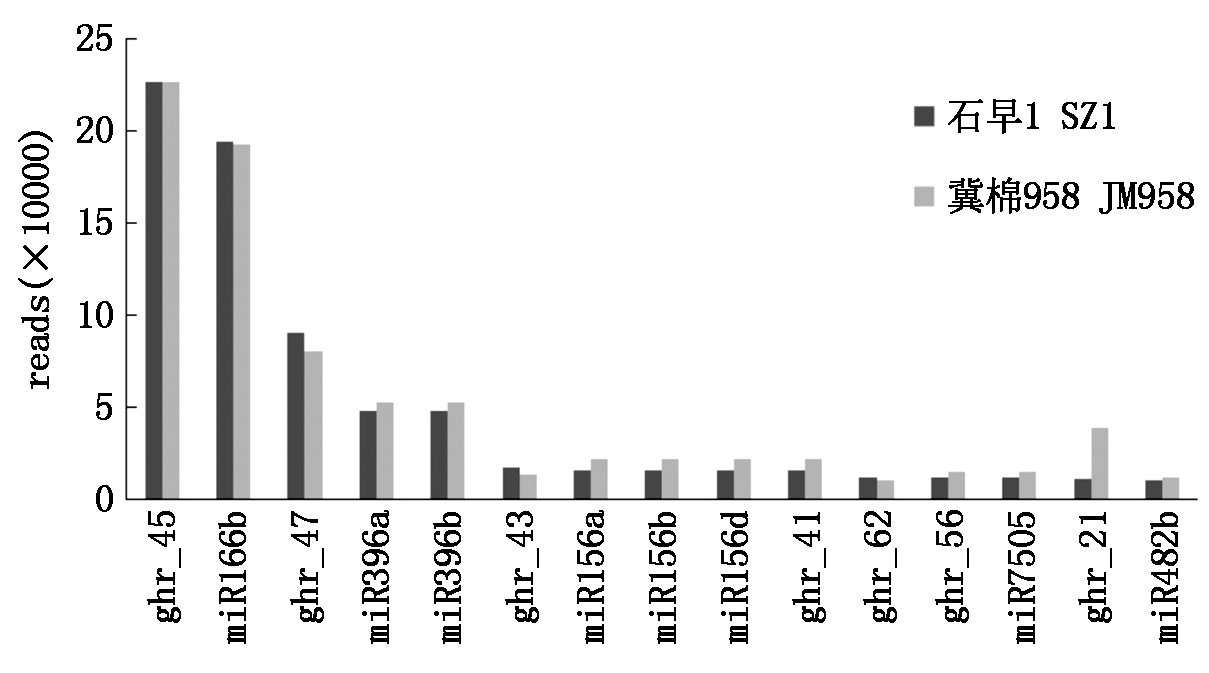
图3 高表达miRNA reads分布
Fig.3 The distribution of miRNAs with high expression
2.3 两品种中差异表达miRNA分析
通过比较石早1和冀棉958 2个文库中miRNA的reads数,分析了131个miRNA的差异表达情况。结果,共有39个miRNA在2个品种中的表达差异超过2倍,占总miRNAs数的29.8%;9个miRNA在2个品种中表达差异超过5倍,占总miRNA数的6.9%;另外,有2个miRNA在2个品种中表达差异超过10倍(图4)。在64个已知miRNA中,15个miRNA在石早1中比在冀棉958中表达超过2倍,其中miRNA394最高,达到6.7倍;4个miRNA在冀棉958中表达比石早1中超过2倍,其中,miR399d最高,达到8倍(图5)。在67个新miRNA中,14个miRNA在石早1中比在冀棉958中表达超过2倍,其中ghr_27最高,达到11.9倍;8个miRNA在冀棉958中比石早1中表达超过2倍,其中ghr_2最高,达到10.7倍(图6)。另外,miR2950只在石早1中检测到,miR172 和 miR7500 只在冀棉958中检测到,这3个特异表达的miRNA表达量都很低。
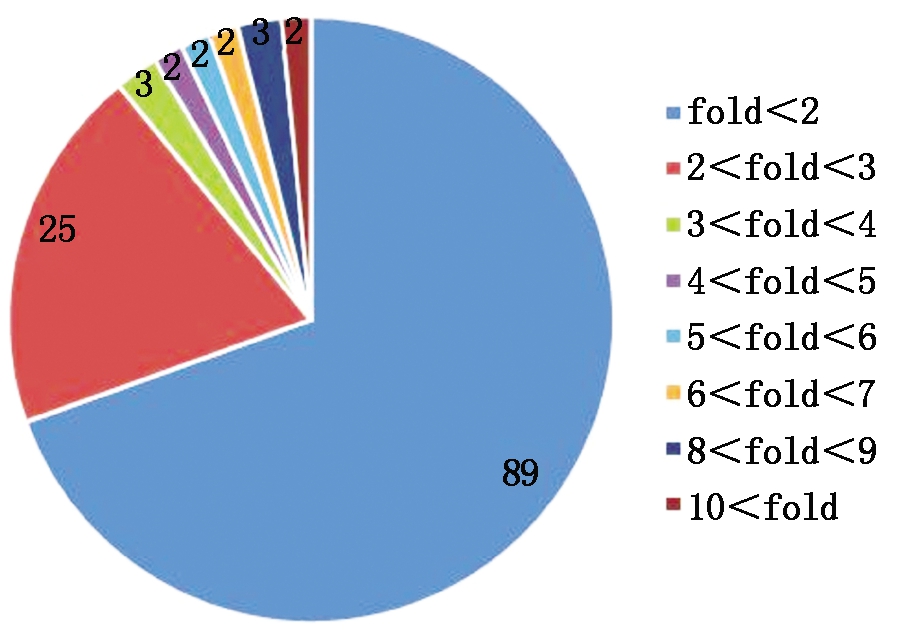
图4 miRNA表达差异分布
Fig.4 The distribution of miRNAs with fold change
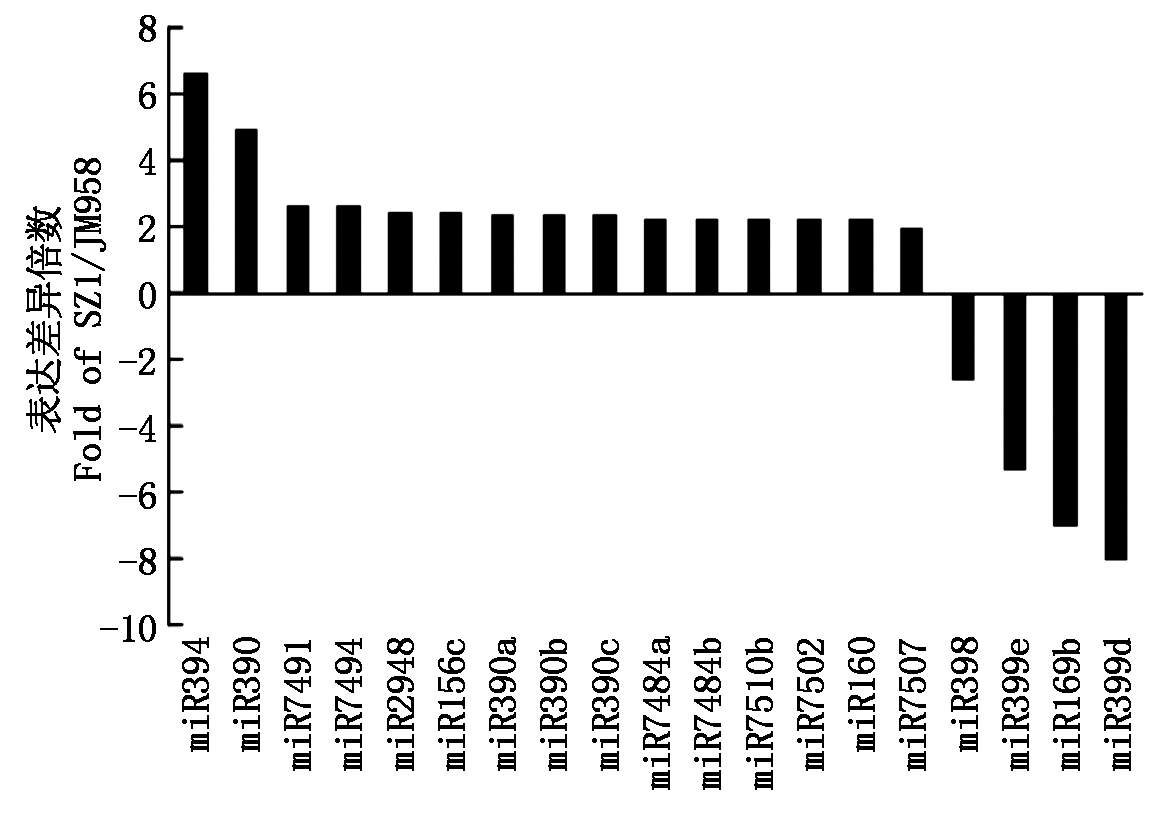
图5 差异表达超过2倍的已知miRNA
Fig.5 The known miRNAs with obviously different
expression (>2 fold) between SZ1 and JM958
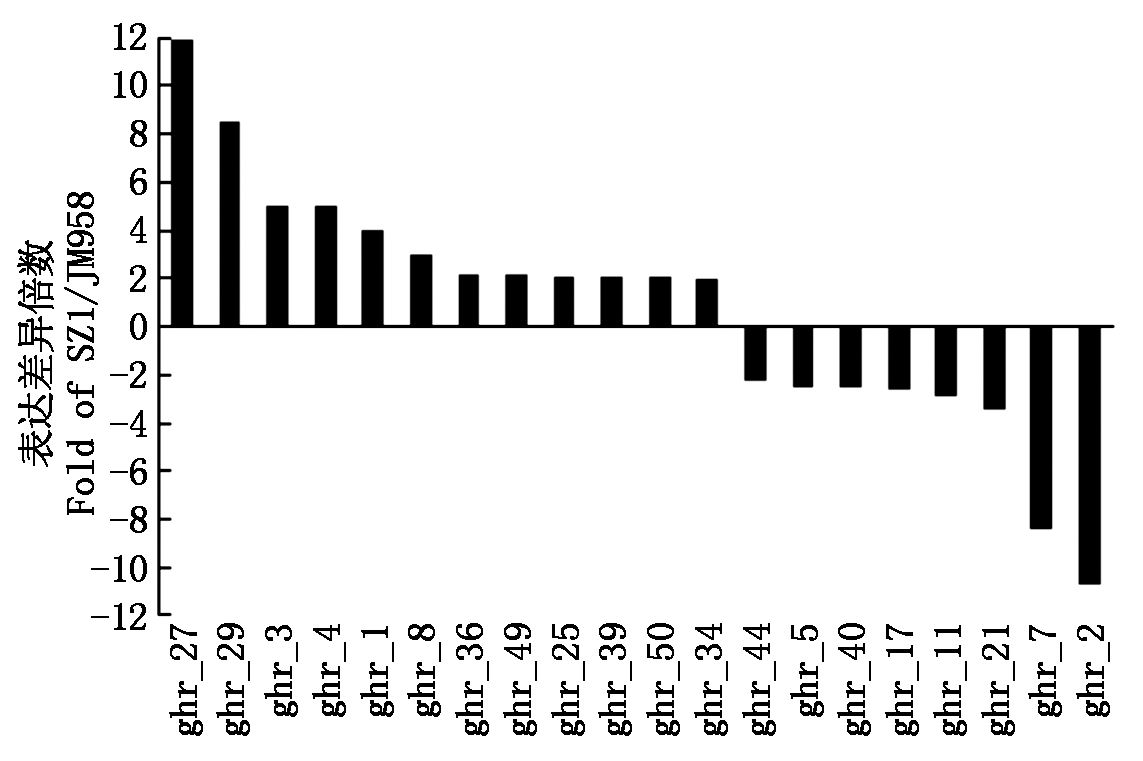
图6 差异表达超过2倍的新的miRNA
Fig.6 The novel miRNAs with obviously different
expression (>2 fold) between SZ1 and JM958
鉴定到的miRNA中:miR156家族有8个miRNA,其中,4个已知miRNA 和 4个新的miRNA;miR399家族有7个miRNA,其中,3个已知miRNA和4个新的miRNA;miR169家族有4个miRNA,其中, 2个已知miRNA和2个新的miRNA;miR394家族有4个miRNA,其中,3个已知miRNA和 1个新的miRNA。对相同家族的不同miRNA在2个品种中的表达差异进行比较。结果,miR156家族的8个miRNA中,只有miR156c和ghr_40在两品种中表达差异超过2倍,其余差异不明显;miR399家族的7个miRNA中,miR399c和ghr_24在两品种中表达差异不明显,其余5个差异明显,超过2倍;miR169家族的4个miRNA中,miR169b在两品种中表达差异达到7倍,其余差异不明显;miR394家族的4个miRNA中,miR394在两品种中表达差异达为6.7倍,其余差异不明显(表 2)。这说明,同一家族的不同miRNA,在同一品种中或相同环境下存在着不同的表达模式,同一家族的不同miRNA,可以发挥不同的生物学功能。
表2 同一家族不同miRNA表达差异分析
Tab.2 The analysis of miRNAs′
expression in the same miRNA family
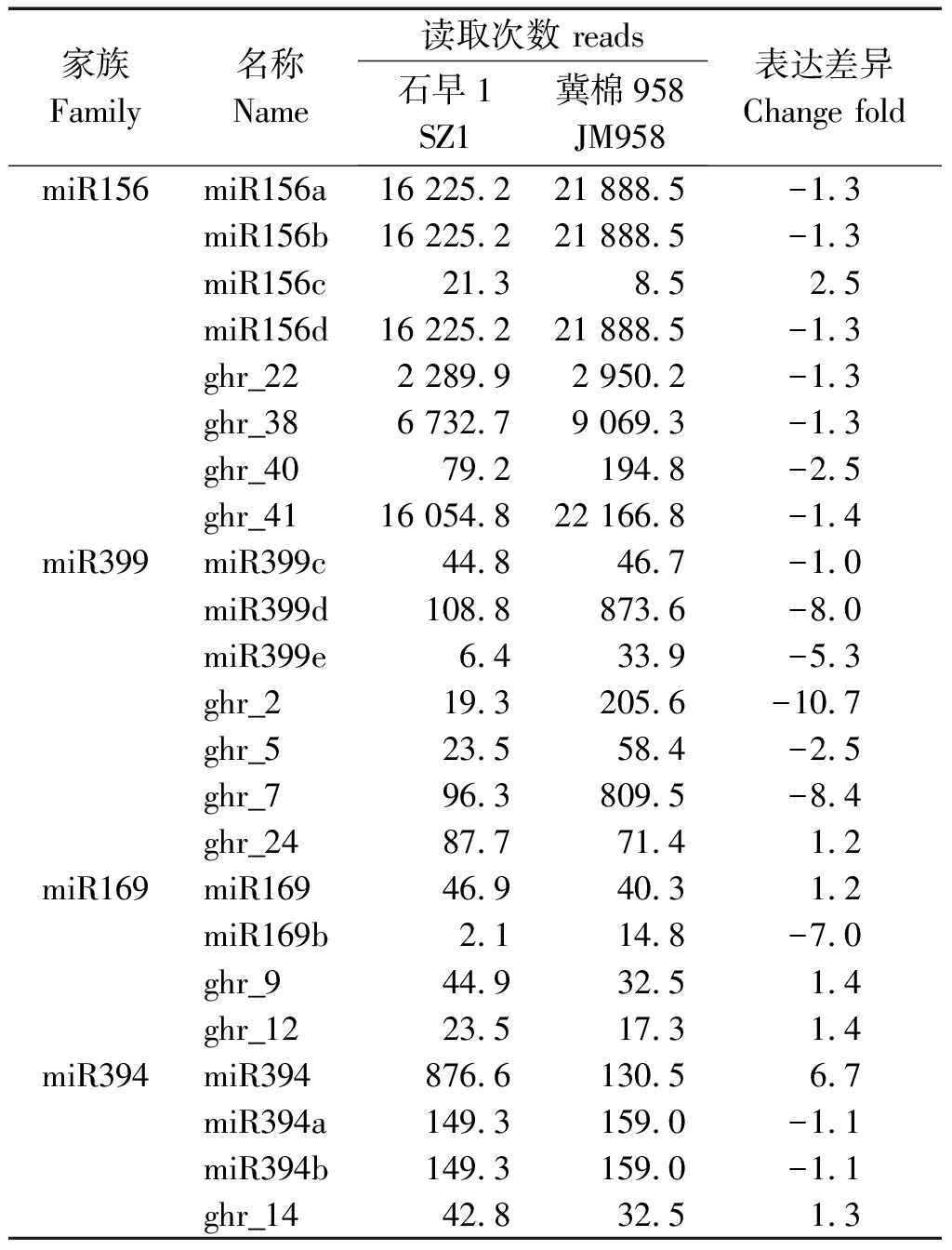
家族Family名称Name读取次数 reads石早1 SZ1冀棉958JM958表达差异Change foldmiR156miR156a16 225.2 21 888.5 -1.3 miR156b16 225.2 21 888.5 -1.3 miR156c21.3 8.5 2.5 miR156d16 225.2 21 888.5 -1.3 ghr_222 289.9 2 950.2 -1.3 ghr_386 732.7 9 069.3 -1.3 ghr_4079.2 194.8 -2.5 ghr_4116 054.8 22 166.8 -1.4 miR399miR399c44.8 46.7 -1.0 miR399d108.8 873.6 -8.0 miR399e6.4 33.9 -5.3 ghr_219.3 205.6 -10.7 ghr_523.5 58.4 -2.5 ghr_796.3 809.5 -8.4 ghr_2487.7 71.4 1.2 miR169miR16946.9 40.3 1.2 miR169b2.1 14.8 -7.0 ghr_944.9 32.5 1.4 ghr_1223.5 17.3 1.4 miR394miR394876.6 130.5 6.7 miR394a149.3 159.0 -1.1 miR394b149.3 159.0 -1.1 ghr_1442.8 32.5 1.3
3 结论与讨论
随着棉花基因组测序的不断完善和高通量测序技术的不断发展,棉花中越来越多的miRNA被发现。然而,目前棉花中鉴定到的miRNA数量仍远少于水稻、大豆等作物,甚至少于基因组较小的拟南芥。本研究以早熟棉为材料,鉴定到67个新的miRNA,丰富了棉花中miRNA的数量。将这67个miRNA在miRbase数据库中进行比对,发现10个miRNA只在棉属中保守,其他物种中未发现与其相同家族的miRNA,这10个miRNA可能在棉花生长发育过程中有特殊作用;另外,13个miRNA在棉花及其他物种中未发现与其同源的miRNA,这些miRNA是否属于新的miRNA家族尚有待于进一步验证。
相同家族的miRNA在不同物种中具有很高的保守性,更说明miRNA在植物生长发育过程中具有非常重要的作用。本研究中,利用高通量测序技术比较了早熟棉与中熟棉品种中miRNA表达的差异。发现,在2个品种中有29.8%的miRNA表达差异超过2倍。这说明,miRNA在棉花熟性发育过程中具有重要的调节作用,且较多miRNA参与了相关过程。miRNA在植物熟性及开花调控中的作用已有报道,miR172可以通过对靶基因SMZ(AP2家族)的调节,抑制拟南芥的开花[7-8];miRNA156可以通过SPL家族的靶基因调控拟南芥幼苗期到成熟期的转变过程,其高表达对拟南芥较早开花有抑制作用[9-10]。在本研究中,miR156c和属于miR156家族的一个新的miRNA(ghr_40)在石早1和冀棉958中表达差异均超过2倍,靶基因预测发现其靶基因均属于SPL家族;而miR172在本研究中只在冀棉958中检测到,且靶基因预测发现其靶基因属于AP2家族。推测,miR156和miR172分别通过对SPL和AP2家族靶基因的调控,在棉花熟性和开花调控中发挥重要作用,其具体过程有待于进一步研究。
对较高表达miRNA进行分析,发现分别属于miR166、miR7505、miR396、miR482、miR156、miR535、miR398等7个miRNA家族的15个miRNA表达较高(reads>10000)。与以往研究比较,发现miR166、miR156、miR482家族的miRNA在棉花及其他作物中也有较高的表达[23-25]。进一步分析发现,这15个高表达miRNA中,只有miR398家族的ghr_21,在2个小RNA文库中表达差异较大,超过2倍(3.4倍),明显少于中量和低量表达的miRNA。这说明高表达miRNA在两品种中表达差异较小,具有较稳定的表达。这些高表达家族的miRNA可能在棉花及其他物种中有更基础的生物学作用。
对相同miRNA家族的不同miRNA在2个品种中的表达差异进行了比较,结果发现相同家族的不同miRNA,存在不同的表达模式。推测相同家族的不同miRNA存在不同的生物学功能。相同家族的miRNA可以调控不同家族的靶基因,同1个miRNA也可以调控多个家族的靶基因[26-27]。推测在植物发育过程中,相同家族的不同miRNA可以通过调控不同家族的靶基因发挥不同的生物学功能。
[1] Brodersen P, Sakvarelidze-Achard L, Bruun-Rasmussen M, Dunoyer P, Yamamoto Y Y, Sieburth L, Voinnet O. Widespread translational inhibition by plant miRNAs and siRNAs[J]. Science, 2008, 320(5880): 1185-1190.doi:10.1126/science.1159151.
[2] Chen X M. A microRNA as a translational repressor of APETALA2 in Arabidopsis flower development[J]. Science, 2004, 303(5666): 2022-2025. doi:10.1126/science.1088060.
[3] Sunkar R, Zhu J K. Novel and stress-regulated microRNAs and other small RNAs from Arabidopsis [J]. The Plant Cell, 2004, 16(8): 2001-2019.doi:10.1105/tpc.104.022830.
[4] Lee R C, Feinbaum R L, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843-854.doi:10.1016/0092-8674(93)90529-y.
[5] Reinhart B J, Slack F J, Basson M, Pasquinelli A E, Bettinger J C, Rougvie A E, Horvitz H R, Ruvkun G. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772): 901-906.doi:10.1038/35002607.
[6] Park W, Li J J, Song R T, Messing J, Chen X M. CARPEL FACTORY, a Dicer homolog, and HEN1, a novel protein, act in microRNA metabolism in Arabidopsis thaliana[J]. Current Biology,2002, 12(17): 1484-1495. doi:10.1016/s0960-9822(02)01017-5.
[7] Schwab R, Palatnik J F, Riester M, Schommer C, Schmid M, Weigel D. Specific effects of microRNAs on the plant transcriptome[J]. Developmental Cell, 2005, 8(4): 517-527. doi:10.1016/j.devcel.2005.01.018.
[8] Mathieu J, Yant L J, Mürdter F, Küttner F, Schmid M. Repression of flowering by the miR172 target SMZ[J]. PLoS Biology, 2009, 7(7):e1000148. doi:10.1371/journal.pbio.1000148.
[9] Wu G, Park M Y, Conway S R, Wang J W, Weigel D, Poethig R S. The sequential action of miR156 and miR172 regulates developmental timing in Arabidopsis[J]. Cell, 2009, 138(4): 750-759. doi:10.1016/j.cell.2009.06.031.
[10] Wang J W, Czech B, Weigel D. miR156-regulated SPL transcription factors define an endogenous flowering pathway in Arabidopsis thaliana[J]. Cell, 2009, 138(4): 738-749. doi:10.1016/j.cell.2009.06.014.
[11] Zhang B H, Wang Q L, Wang K B, Pan X P, Liu F, Guo T L, Cobb G P, Anderson T A. Identification of cotton microRNAs and their targets[J]. Gene, 2007, 397(1-2): 26-37. doi:10.1016/j.gene.2007.03.020.
[12] Qiu C X, Xie F L, Zhu Y Y, Guo K, Huang S Q, Nie L, Yang Z M.Computational identification of microRNAs and their targets in Gossypium hirsutum expressed sequence tags[J]. Gene,2007, 395(1-2): 49-61.doi:10.1016/j.gene.2007.01.034.
[13] Abdurakhmonov I Y, Devor E J, Buriev Z T, Huang L Y, Makamov A, Shermatov S E, Bozorov T, Kushanov F N, Mavlonov G T, Abdukarimov A. Small RNA regulation of ovule development in the cotton plant, G. hirsutum L.[J]. BMC Plant Biology, 2008, 16(8): 93. doi:10.1186/1471-2229-8-93.
[14] Zhao T L, Xu X J, Wang M, Li C, Li C, Zhao R B, Zhu S J, He Q L, Chen J H.Identification and profiling of upland cotton microRNAs at fiber initiation stage under exogenous IAA application[J]. BMC Genomics,2019, 20(1): 421. doi:10.1186/s12864-019-5760-8.
[15] Naoumkina M, Thyssen G N, Fang D D, Hinchliffe D J, Florane C B, Jenkins J.Small RNA sequencing and degradome analysis of developing fibers of short fiber mutantsLigon-lintles-1(Li 1) and -2(Li 2) revealed a role for miRNAs and their targets in cotton fiber elongation[J]. BMC Genomics,2016, 17:360. doi:10.1186/s12864-016-2715-1.
[16] Xie F L, Wang Q L, Sun R R, Zhang B H. Deep sequencing reveals important roles of microRNAs in response to drought and salinity stress in cotton[J]. Journal of Experimental Botany, 2015, 66(3):789-804. doi:10.1093/jxb/eru437.
[17] Zhu Q H, Fan L J, Liu Y, Xu H, Danny L, Wilson I. miR482 Regulation of NBS-LRR Defense Genes during Fungal Pathogen Infection in Cotton[J]. PLoS One, 2013,8(12): e84390. doi:10.1371/journal.pone.0084390.
[18] Zuker M. Mfold web server for nucleic acid folding and hybridization prediction[J]. Nucleic Acids Research, 2003, 31(13): 3406-3415. doi:10.1093/nar/gkg595.
[19] Meyers B C, Axtell M J, Bartel B, Bartel D P, Baulcombe D, Bowman J L, Cao X F, Carrington J C, Chen X M, Green P J, Griffiths-Jones S, Jacobsen S E, Mallory A C, Martienssen R A, Poethig R S, Qi Y J, Vaucheret H, Voinnet O, Watanabe Y, Weigel D, Zhu J K. Criteria for annotation of plant MicroRNAs[J]. The Plant Cell, 2008, 20(12): 3186-3190.doi:10.1105/tpc.108.064311.
[20] Tian B, Wang S C, Todd T C, Johnson C D, Tang G, Trick H N.Genome-wide identification of soybean microRNA responsive to soybean cyst nematodes infection by deep sequencing [J]. BMC Genomics, 2017, 18(1): 572. doi:10.1186/s12864-017-3963-4.
[21] Guo N, Zhang Y J, Sun X, Fan H H, Gao J S, Chao Y P, Liu A F, Yu X T, Cai Y P, Lin Y.Genome-wide identification of differentially expressed miRNAs induced by ethephon treatment in abscission layer cells of cotton(Gossypium hirsutum)[J]. Gene,2018, 676(15): 263-268. doi:10.1016/j.gene.2018.08.057.
[22] Wang Q L, Zhang B H.MicroRNAs in cotton: an open world needs more exploration[J]. Planta,2015, 241(6): 1303-1312. doi:10.1007/s00425-015-2282-8.
[23] Zhang Y J, Wang W, Chen J, Liu J B, Xia M X, Shen F F.Identification of miRNAs and their targets in cotton inoculated with Verticillium dahliae by high-throughput sequencing and degradome analysis[J]. International Journal of Molecular Sciences,2015, 16(7): 14749-14768. doi:10.3390/ijms160714749.
[24] An W Y, Gong W F, He S P, Pan Z E, Sun J L, Du X M.MicroRNA and mRNA expression profiling analysis revealed the regulation of plant height in Gossypium hirsutum[J]. BMC Genomics,2015, 16:886. doi:10.1186/s12864-015-2071-6.
[25] Wang M, Wang Q L, Zhang B H. Response of miRNAs and their targets to salt and drought stresses in cotton(Gossypium hirsutum L.)[J].Gene, 2013, 530(1): 26-32. doi:10.1016/j.gene.2013.08.009.
[26] 刘潮,褚洪龙,韩利红,代冬琴,陈欢欢,唐利洲.植物miR399家族分子特征及靶基因功能分析[J].华北农学报,2019,34(2):1-7. doi:10.7668/hbnxb.201751113.
Liu C, Chu H L, Han L H, Dai D Q, Chen H H,Tang L Z. Molecular characterization and target gene predication of plant miR399 family[J]. Acta Agriculturae Boreali-Sinica, 2019, 34(2):1-7.
[27] Millar A A, Lohe A, Wong G G.Biology and function of miR159 in plants[J]. Plants Basel, 2019, 8(8): 255. doi:10.3390/plants8080255.