作为亚洲栽培稻的起源中心之一,中国拥有类型丰富的稻种资源[1]。我国丰富稻种资源的主要组成部分是地方品种。地方品种不仅蕴含大量高产、优质、抗病、耐逆等优异基因,同时具有丰富的遗传多样性[2-4],是我国的宝贵资源。伴随着核心种质在水稻育种中被长期利用,育成品种遗传基础单一,多样性差的问题也日趋突出[5-7]。今后,水稻(Oryza sativa L.)育种能否取得实质性的突破,关键在于地方品种遗传多样性的挖掘与利用。随着世界人口的高速增长以及可耕地面积的持续下降,稳产一直是育种家最为关注的性状之一。然而不合理的灌溉及过度施肥,使得地球环境持续恶化,盐碱土地面积逐年增加,且遍及全球30多个国家。水稻作为中度感盐作物,土壤盐浓度对其产量和品质影响巨大。盐碱土地生长的水稻大多会减产,乃至绝收。因此,从地方品种中挖掘新的水稻耐盐基因,通过遗传改良等方法,耐盐水稻新品种的培育,对于提高我国盐碱土地的利用、保障水稻稳产具有重要意义。
早在20世纪60年代已有研究报道,水稻耐盐性是多基因控制的数量性状,同时外界环境和发育时期对其也有影响[8-11],其中二叶一心期最为敏感[12]。已有很多的研究者们利用数量性状定位群体及分子标记连锁图谱,进行QTL定位。结果表明大多耐盐QTL分布于第1,2,6,7号染色体上。第10,11号染色体也检测到少量耐盐QTL。迄今仅几个耐盐基因被克隆。位于第1染色体上的耐盐基因SKC1第1个被克隆。当水稻处于高盐胁迫,茎秆积累大量Na+时,SKC1编码的细胞膜定位Na+转运蛋白能够将茎秆中过量Na+运回至根系,从而减轻Na+水稻植株的毒害,提高水稻植株对盐胁迫的耐受性[13]。DST是Huang等[14]利用图位克隆获得的新型锌指转录因子。其来源于耐盐突变体,可以通过负调控增强水稻的耐盐或耐旱性。突变后的DST降低了过氧化氢代谢基因表达,使得过氧化氢在保卫细胞中过积累,导致气孔关闭,从而减少水分蒸发,植株表现出耐盐性或耐旱性。HST1是最新报道的一个耐盐基因,编码一个B型反应调节蛋白OsRR22,与调节渗透和离子转运相关基因的表达有关。相比SKC1基因,HST1基因具有更强的耐盐性[15]。
虽然对水稻耐盐性基因的克隆已取得一些进展,但真正用于耐盐品种培育上基因资源还很少。挖掘和定位新的水稻耐盐性QTL,提高地方品种的可利用性,对改善现有品种的遗传多样性具有积极意义。本研究利用云南籼稻品种扎西玛和江苏优质水稻品种南粳46构建的重组自交系,分析水稻幼苗期耐盐性QTL,以期获得新的耐盐性QTL,为耐盐性水稻育种提供理论基础。
1 材料和方法
1.1 试验材料
以云南籼稻品种扎西玛和江苏优质水稻品种南粳46为亲本,单粒传法,构建扎西玛/南粳46重组自交系群体,共143个家系。
1.2 水稻幼苗期盐胁迫处理
利用0.5% NaCl盐浓度对143个家系及其亲本扎西玛和南粳46幼苗进行耐盐性鉴定。直径9 cm培养皿用于水稻种子催芽。每个培养皿加入12 mL dH2O,28 ℃暗培养3 d。将芽长基本一致的种子置于96孔板上,每行8粒,每个材料5行。96孔板放置于加水周转箱中,光照培养箱内生长。培养箱的设置为:光照14 h/d,白天28 ℃,夜间24 ℃。幼苗长至2叶1心时,在营养液中添加0.5% NaCl进行盐胁迫处理,营养液每3 d更换1次。盐胁迫处理持续时间为21 d,复水10 d。试验设置3次重复,每个重复设置3个重复材料,以3次重复的幼苗成活率平均值作为统计数据。
1.3 苗期耐盐性调查
调查各家系及亲本幼苗处理21 d、复水10 d后的幼苗存活率,平均值作为耐盐性评价数据。
存活率=存活株数/总株数×100%。
1.4 QTL分析
QTL分析采用Wang等[16]开发的QTL检测软件 IciMapping v3.2,其基于完备复合区间作图,LOD值阈值设定为2.0。分子标记检测结果与幼苗存活率比值相结合,对重组自交系群体各株系进行全基因组范围内苗期耐盐性QTL分析,QTL的命名遵循McCouch等[17]的原则。
2 结果与分析
2.1 亲本与143个家系的苗期耐盐性分析
亲本扎西玛的苗期存活率较低,为4.93%,南粳46的苗期存活率较高,为57%,差异极显著。重组自交系群体苗期存活率在0~90%,表现出连续偏正态分布(图1),偏度为0.456,峰度为-1.041。有一定数量超亲类型的存在,表明该性状为多基因控制的数量性状。
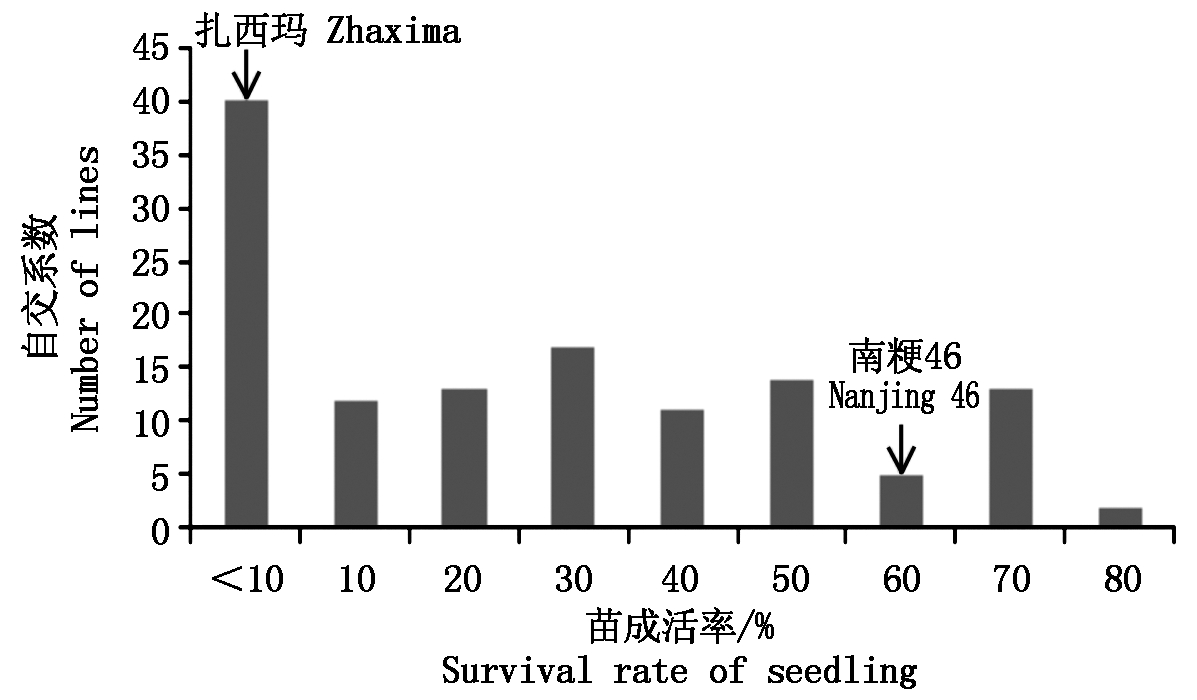
图1 亲本及重组自交系群体经NaCl
处理后幼苗成活率的表型分析
Fig.1 Phenotypes of seedling survival rate of
parents and the RIL population after NaCl stress
2.2 苗期耐盐性QTL分析
本实验室前期已利用均匀分布于水稻12条染色体上的202对SSR多态性分子标记,构建了该重组自交系群体的分子连锁图谱。图谱全长1 437.3 cM,平均图距8.1 cM,符合QTL定位要求。利用QTL IciMapping v3.2软件,结合表型数据,分析苗期耐盐性QTL的位置及遗传效应,共检测到4个苗期耐盐性相关QTL(qSST-1、qSST-3、qSST-5、qSST-11),分别位于第1,3,5,11号染色体上(图2、表1),共解释34.03%的表型变异。贡献率最高的苗期耐盐性是qSST-3,达到11%。本次试验检测出的QTL加性效应均为负值,表明苗期耐盐性的增效位点均来源于亲本南粳46。

图2 水稻苗期耐冷性QTL检测
Fig.2 QTL detection of salt tolerance at seedling stage of rice
表1 RIL群体中检测到的苗期耐冷性QTL
Tab.1 QTL affecting salt tolerance at seedling stage of rice detected in RIL population

QTL染色体遗传位置/cM标记区间LOD值可解释的表型变异/%加性效应ChromosomeGenetic locationMarker intervalLOD scorePVEAdditive effectqSST-1198RM11438~RM54972.127.86-7.14qSST-33145RM7000~RM35852.4411.00-8.35qSST-5544RM18451~RM184832.087.81-7.05qSST-111172RM1355~RM268302.017.36-6.92
3 讨论
大量优异基因资源蕴含于地方品种中,而生产上大面积推广的育成品种遗传基础日趋单一。如果能对地方品种中的优异基因资源充分挖掘,对提高现代育成品种遗传基础多样性将具有积极意义。水稻品种的产量、品质、抗病虫、耐逆境等众多重要农艺性状,大多是多基因控制的数量性状,遗传基础复杂。要想克隆数量性状基因,合适作图群体的构建,数量性状QTL的精准鉴定,QTL位置和效应的分析,是科技工作者首先需要考虑的;第2步通过不断回交将每个QTL分开,获得相应基因的近等基因系,把复杂数量性状分解成单个孟德尔因子。通过这种方法,克隆了很多水稻重要数量性状QTL[18-21]。
本研究利用扎西玛和南粳46为亲本构建的一套水稻重组自交系群体为研究对象,考察了苗期耐盐性,同时结合遗传图谱,利用IciMapping V 3.2软件进行苗期耐盐性QTL分析。分别在第1,3,5,11染色体上检测到一个QTL,共解释34.03%的表型变异。4个QTL均表现为加性效应,表明其增效位点均来自于高值亲本南粳46。其中位于第3染色体上的qSST-3效应值最大,达到11%。通过比较与前人研究报道,qSST-1与汪斌等[22]定位的QTL染色体区间重合,其位于第一染色体RM11438~RM5497区间内。其余3个耐盐QTLs均未找到相同区间报道,是本研究鉴定出的新QTLs。迄今,已经有大量水稻耐盐性QTL的定位报道,但研究结果大多不同[23]。作者分析盐胁迫处理浓度及时间会影响水稻芽期或苗期耐盐性表型鉴定结果,而遗传材料、群体类型以及遗传背景等会影响QTL分析结果;这些均会导致水稻苗期耐盐性QTL鉴定结果的不一致。
对水稻不同发育时期耐盐性的认识,不同研究者有不同看法,部分研究认为水稻芽期、苗期及成熟期的耐盐性具有一定的相关性[24-25],而另一些研究者认为同一品种在芽期和苗期的耐盐性表现不一致,两者相关性很低[26-27]。本研究仅初步提供了重组自交系群体苗期耐盐性遗传鉴定数据。其中,qSST-1被多个研究者利用不同的研究材料在不同环境中检测到,说明第一染色体RM11438~RM5497区域可能是影响水稻苗期耐盐性的重要区域,对分子标记辅助选育水稻苗期耐盐新品系具有一定的参考意义。
[1] 韩龙植,黄清港,盛锦山,庞汉华,裘宗恩,杨庆文,曹桂兰,赵江.中国稻种资源农艺性状鉴定、编目和繁种入库概况[J]. 植物遗传资源科学,2002,3(2):40-45.
Han L Z, Huang Q G, Sheng J S, Pang H H, Qiu Z E, Yang Q W,Cao G L, Zhao J. A survey on characterization,catalogue, multiplication and conservation of rice germplasm resources in China[J]. Journal of Plant Genetic Resources, 2002,3(2):40-45.
[2] Zhang H L, Sun J L, Wang M X, Liao D Q, Zeng Y W, Shen S Q, Yu P, Mu P, Wang X K, Li Z C.Genetic structure and phylogergraphy of rice landraces in Yunnan, China, revealed by SSR[J].Genome, 2007,50(1):72-83.doi:10.1139/g06-130.
[3] 杨志奇,杨春刚,汤翠凤,郭桂珍,余腾琼,张俊国,曹桂兰,阿新祥,徐福荣,张三元,戴陆园,韩龙植. 中国粳稻地方品种孕穗期耐冷性评价及聚类分析[J]. 植物遗传资源学报,2008,9(4):485-491.
Yang Z Q, Yang C G, Tang C F, Guo G Z, Yu T Q, Zhang J G, Cao G L, A X X, Xu F R, Zhang S Y, Dai L Y, Han L Z. Evaluation of cold tolerance at booting stage and cluster analysis for Japonica rice landraces in China[J]. Journal of Plant Genetic Resources, 2008,9(4):485-491.
[4] 孙建昌,曹桂兰,李亚非,马静,陈耀峰,韩龙植. 水稻地方品种群体内的遗传多样性分析[J]. 西北农林科技大学学报(自然科学版),2011,39(12):145-152.
Sun J C, Cao G L, Li Y F, Ma J, Chen Y F, Han L Z. Analysis of genetic diversity within populations of rice (Oryza sativa L.) landraces[J]. Journal of Northwest A&F University (Nat Sci Ed), 2011,39(12):145-152.
[5] 魏兴华,汤圣祥,江云珠,余汉勇,裘宗恩,颜启传. 中国栽培稻选育品种等位酶多样性及其与形态学性状的相关分析[J].中国水稻科学,2003,17(2):123-128.
Wei X H, Tang S X, Jiang Y Z, Yu H Y, Qiu Z E, Yan Q C. Genetic diversity of allozyme associated with morphological traits in Chinese improved rice varieties[J].Chinese J Rice Sci, 2003,17(2):123-128.
[6] 齐永文,张冬玲,张洪亮,王美兴,孙俊立,廖登群,魏兴华,裘宗恩,汤圣祥,曹永生,王象坤,李自超. 中国水稻选育品种遗传多样性及其近50年变化趋势[J]. 科学通报,2006,51(6):693-699. doi:10.3321/j.issn:0023-074X.2006.06.013.
Qi Y W, Zhang D L, Zhang H L, Wang M X, Sun J L, Liao D Q, Wei X H, Qiu Z E, Tang S X, Cao Y S, Wang X K, Li Z C. Genetic diversity of rice breeding varieties in China and its change trend in recent 50 years[J]. Chinese Science Bulletin, 2006,51(6):693-699.
[7] 邓宏中,王彩红,徐群,袁筱萍,冯跃,余汉勇,王一平,魏兴华. 中国水稻地方品种与选育品种的遗传多样性比较分析[J]. 植物遗传资源学报,2015,16(3):433-442. doi:10.13430/j.cnki.jpgr.2015.03.001.
Deng H Z, Wang C H, Xu Q, Yuan X P, Feng Y, Yu H Y, Wang Y P, Wei X H. Comparative analysis of genetic diversity in landrace and improved rice varieties in China[J]. Journal of Plant Genetic Resources, 2015,16(3):433-442.
[8] Pearson G, Ayers A, Eberhard D. Relative salt tolerance of rice during germination and early seeding development[J]. Soil Science, 1966, 102(3): 151-156. doi:10.1097/00010694-196609000-00003.
[9] 郭 岩,陈少麟,张耕耘,陈受宜. 应用细胞工程获得受主效基因控制的水稻耐盐突变系[J].遗传学报,1997,24(2):122-126.
Guo Y, Chen S L, Zhang G Y, Chen S Y.Salt-tolerance rice mutant lines controlled by a major effect gene were obtained by cell engineering technique[J]. Acta Genetica Sinica, 1997,24(2):122-126.
[10] 龚继明,何平,钱前,沈利爽,朱立煌,陈受宜. 水稻耐盐性QTL的定位[J]. 科学通报,1998,43(17):1847-1850.
Gong J M, He P, Qian Q, Shen L S, Zhu L H, Chen S Y. QTL location of salt tolerance in rice[J]. Chinese Science Bulletin, 1998,43(17):1847-1850.
[11] Lin H X, Zhu M Z, Yano M, Gao J P, Liang Z W, Su W A, Hu X H, Ren Z H,Chao D Y. QTLs for Na+ and K+ uptake of the shoots and roots controlling rice salt tolerance[J]. Theoretical and Applied Genetics, 2004, 108(2): 253-260. doi:10.1007/s00122-003-1421-y.
[12] 方先文,汤陵华,王艳平. 耐盐水稻种质资源的筛选[J]. 植物遗传资源学报,2004,5(3):295-298. doi:10.13430/J.cnki.jpgr.2004.03.018.
Fang X W, Tang L H, Wang Y P. Selection on rice germplasm tolerant to salt stress[J]. Journal of Plant Genetic Resources, 2004,5(3):295-298.
[13] Ren Z H, Gao J P, Li L G, Cai X L, Huang W, Chao D Y, Zhu M Z, Wang Z Y, Luan S, Lin H X. A rcie quantitative trait locus for salt tolerance encodes a sodium transporter[J]. Nature Genetics, 2005, 37(10):1141-1146. doi:10.1038/ng1643.
[14] Huang X Y, Chao D Y, Gao J P, Zhu M Z, Shi M, Lin H X. A previously unknow zinc finger protein, DST, regulates drought and salt tolerance in rice via stomatal aperture control[J]. Genes & Development, 2009,23(15):1805-1817. doi:10.1101/gad.1812409.
[15] Takagi H, Tamiru M, Abe A, Yoshida K, Uemura A, Yaegashi H, Obara T, Oikawa K, Utsushi H, Kanzaki E, Mitsuoka C, Natsume S, Kosugi S, Kanzaki H, Matsumura H, Urasaki N, Kamoun S, Terauchi R. MutMap accelerates breeding of a salt-tolerant rice cultivar[J]. Nature Biotechnology, 2015,33(5):445-449. doi:10.1038/nbt.3188.
[16] Wang J K, Wan X Y, Li H H, Pfeiffer W H, Crouch J, Wan J M. Application of identified QTL-marker associations in rice quality improvement through a design-breeding approach[J]. Theor Appl Genet,2007, 115(1): 87-100. doi:10.1007/s00122-007-0545-x.
[17] McCouch S R, Cho Y G, Yano M, Paule E, Blinstrue M, Morishima H M, Kinosita T. Report on QTL nomenclature[J]. Rice Genet Newsl, 1997, 14(11): 11-13.
[18] Weng J F, Gu S H, Wan X Y, Gao H, Guo T, Su N, Lei C L, Zhang X, Cheng Z J, Guo X P, Wang J L, Jiang L, Zhai H Q, Wan J M.Isolation and initial characterization of GW5, a major QTL associated with rice grain width and weight[J]. Cell Research, 2008, 18(12): 1199-1209. doi:10.1038/cr.2008.307.
[19] Zhang X J, Wang J F, Huang J, Lan H X, Wang C L, Yin C F, Wu Y Y, Tang H J, Qian Q, Li J Y, Zhang H S. Rare allele of OsPPKL1 associated with grain length causes extra-large grain and a significant yield increase in rice[J]. Proceedings of the National Academy of Sciences, 2012, 109(52): 21534-21539. doi:10.1073/pnas.1219776110.
[20] Ishimaru K, Hirotsu N, Madoka Y, Murakami N, Hara N, Onodera H, Kashiwagi T, Ujiie K, Shimizu B, Onishi A, Miyagawa H, Katoh E.Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield[J]. Nature Genetics, 2013, 45(6): 707-711. doi:10.1038/ng.2612.
[21] Li Y B, Fan C C, Xing Y Z, Yun P, Luo L J, Yan B, Peng B, Xie W B, Wang G W, Li X H, Xiao J H, Xu C G, He Y Q.Chalk5 encodes a vacuolar H+-translocating pyrophosphatase influencing grain chalkiness in rice[J]. Nature Genetics, 2014, 46(4): 398-404. doi:10.1038/ng.2923.
[22] 汪斌,兰涛,吴为人. 盐胁迫下水稻苗期Na+含量的QTL定位[J]. 中国水稻科学,2007,21(6):585-590.doi:10.16819/j.1001-7216.2007.06.005.
Wang B, Lan T, Wu W R, Mapping of QTLs for content in rice seedlings under salt stress[J]. Chinese J Rice Sci, 2007,21(6):585-590.
[23] 邢军,常汇琳,王敬国,刘化龙,孙健,郑洪亮,赵宏伟,邹德堂. 盐、碱胁迫条件下粳稻Na+、K+浓度的QTL分析[J]. 中国农业科学,2015,48(3):604-612. doi:10.3864/j.issn.0578-1752.2015.03.19.
Xing J, Chang H L, Wang J G, Liu H L, Sun J, Zheng H L, Zhao H W, Zou D T. QTL analysis of Na+ and K+ concentrations in Japonica rice under salt and alkaline stress[J]. Scientia Agricultura Sinica, 2015,48(3):604-612.
[24] Zang J P, Sun Y, Wang Y, Yang J, Li F, Zhou Y L, Zhu L H, Jessica R, Mohammadhosein F, Xu J L, Li Z K. Dissection of genetic overlap of salt tolerance QTLs at the seedling and tillering stages using backcross introgression lines in rice[J]. Science in China Series C:Life Sciences, 2008,51(7):583-591.doi:10.1007/s11427-008-0081-1.
[25] 陈亚萍. 粳稻种质资源不同生育时期的耐盐性评价[D]. 银川:宁夏大学,2016.doi:10.7666/d.Y3109504.
Chen Y P. Identification of salt tolerance of Japonica rice germplasm resources at different growth period[D]. Yinchuan: Ningxia University, 2016.
[26] 谢留杰,段敏,潘晓飚,唐兴国,朱长志,黄善军. 不同类型水稻品系苗期和全生育期耐盐性鉴定与分析[J]. 江西农业大学学报,2015,37(3):404-410. doi: 10.13836/j.jjau.2015062.
Xie L J, Duan M, Pan X B, Tang X G, Zhu C Z, Huang S J. Identification and analysis of salt tolerance in different type rice cultivars during seedling and whole plant growth stage[J]. Acta Agriculturae Universitatis Jiangxieensis, 2015,37(3):404-410.
[27] 郭望模,傅亚萍,孙宗修,郑镇一. 盐胁迫下不同水稻种质形态指标与耐盐性的相关分析[J]. 植物遗传资源学报,2003,4(3):245-251.doi:10.13430/j.cnki.jpgr.2003.03.014.
Guo W M, Fu Y P, Sun Z X, Zhen Z Y. The correlation analysis between the morphological indices and salt tolerance in different rice germplasm under the salt stress[J]. Journal of Plant Genetic Resources, 2003,4(3):245-251.