扁秆荆三棱(Bolboschoenus planiculmis)是河漫滩、湖岸、稻田等湿地类环境中的常见杂草。该物种在自然状态下能通过地下根茎和球茎等结构不断进行营养繁殖而产生大量无性系分株,这种过程被称为克隆生长[1-2]。克隆生长赋予该物种生理整合、内部分工等高效利用资源和适应环境的能力,使其经常在自然群落中占据绝对优势地位[2-3]。已有的研究通常将其视为水稻田中的恶性杂草而研究其防除技术[4-6]。钱希[7]建立了预测扁秆荆三棱再生苗和球茎生长量的线性回归方程,指出机械和人工移除虽然暂时抑制生长,却促进了再生苗的蔓延。康学耕和唐恩全等[8]认为其无性繁殖平均10 d一代,并且当年生的种子当年不能萌发,至次年6月中旬萌发出土形成实生苗,形成“即时型”和“延后型”相结合的爆发,对稻田生产极具威胁。该植物虽然在稻田中通常被视为恶性杂草,但其在不同类型的系统中却可能具有多种应用潜力。在自然湿地中,其球茎由于淀粉含量较高,成为国家一级保护动物白鹤(Grus leucogeranus)在我国东北地区湿地停歇时的主要植物性食物[9],并且有研究表明它可能是一种历史悠久的传统食物[10],具有食品加工方面的应用潜力。该物种还具有较好的去污染及净化水体能力,可以作为人工湿地的构建材料[11-12]。但是,目前的工作多属于经典生物学和实验生态学范畴,在分子水平的信息比较匮乏,不利于生物学机理的深入研究;因此,亟待补充相关数据,为农林相关工作提供科学依据。
近年来,基于高通量方法的转录组测序正成为植物生理生态研究领域的有力工具。转录组测序可以获得某一物种特定组织或器官在某一状态下的几乎所有转录本序列信息,进而得到的基因功能注释、蛋白质编码区序列、基因的表达量、代谢途径等大量信息,为进一步研究提供基础数据和重要参考[13-14]。该方法不仅能够用于有参考基因组序列的物种研究,如水稻[15-16]、葡萄[17]等,也能用于无参考基因组序列的物种[18-20],并且在探讨基因的差异性表达(Differential expression)和共表达(Co-expression)情况等方面具有较强功效。已有的研究显示,扁杆荆三棱的地下球茎网络系统在不同环境下具有很强的可塑性,根茎的长度、球茎的数量和营养成分都可能存在差异[3, 21-22],从而造成不同的种群扩张策略。对该物种进行转录组分析,有助于获取其生长调节相关的基因信息,并为进一步深入研究不同环境条件下的表达模式提供基础。
针对现有的研究中扁秆荆三棱转录组信息缺失的情况,本研究采用高通量测序技术对扁秆荆三棱的茎、叶、根茎和球茎的混合样品进行转录组测序,结合生物信息分析对获得的Unigene开展功能注释、代谢通路分析等方面的研究,并探讨其潜在的SSR位点情况,以期为扁秆荆三棱遗传多样性分析、功能基因挖掘和利用、分子辅助育种等方面的研究提供数据和理论支持。
1 材料和方法
1.1 试验材料
1.1.1 采集地点 样品采集于野外自然种群,具体位置为河北衡水湖湿地生态系统国家定位观测研究站湖岸生态监测区域内(115°38′39.84″,37°39′10.08″)。采样时间为2018年7月份,该时间内扁杆荆三棱多处于克隆生长的起始阶段,生长迅速[22],较为适合采集不同组织部位的样品。采样种群所在区域属暖温带大陆季风气候区,整个湖区的地形为浅碟状洼地,湖岸为自然平地,土壤以潮湿的黏土为主要类型。保护区内全年平均降水量为506.3 mm,主要集中于7-8月份,全年日照平均总时数为2 571.7 h[23]。
1.1.2 取样方法 选择长势均匀的扁秆荆三棱种群,在其范围内随机设置5个1 m×1 m的小样方,每个小样方内随机选择3个单株,分别采集其茎、叶、球茎和根茎部分,分开盛装并做好标记。总共采集15个扁秆荆三棱的单株,每个单株的取样量为5 g左右。各部分采样后立即使用去离子水冲洗去除杂质,然后封装放入足量的干冰环境中暂时保存,并在24 h内存于-80 ℃低温条件下,以避免RNA降解。
1.2 试验方法
1.2.1 RNA的提取、建库与测序 分别取茎、叶、根茎和球茎1 g的材料,采用RNeasy Plant Mini Kits(Qiagen, Inc., Valencia, CA, USA)试剂盒提取总RNA,对总RNA样品使用DNase(TaKaRa, Japan)纯化后,从每类组织的产物中取10 μg等量混合组成RNA池,然后使用Oligo(dT)磁珠分离和富集poly(A)mRNA,向富集后的mRNA中加入Fragmentation Buffer 并进行打断,再以片段化的mRNA为模板,用随机引物(Random hexamers)合成第一链cDNA,加入缓冲液、dNTPs、RNaseH 和DNA polymerase 合成第二链cDNA。经过试剂盒纯化回收、黏性末端修复后,在cDNA 的3′ 末端加上碱基“A”并连接接头,用琼脂糖凝胶电泳进行片段大小选择,然后进行PCR 扩增得到测序文库。构建好的文库用Agilent 2100 Bioanalyzer质检合格后,使用Illumina Solexa HiSeq 2000进行双端测序(Paired-end)。测序所获原始数据开放存储于figshare(doi:10.6084/m9.figshare.8150792)
1.2.2 测序数据的处理与拼装 测序得到的原始reads并不都是有效reads,在后续组装和分析前,首先对Raw reads进行预处理,移除含poly-N、接头和低质量的片段,获得Clean reads。然后使用Trinity,采用Paired-end的拼接方法,将具有一定长度overlap的reads 连成更长的片段,得到不含未知碱基的组装片段,之后利用TGICL软件聚类去冗余延伸得到一套Unigene,并最终选取每个Loci下最长的转录本作为确定的Unigene。
1.2.3 Unigene序列的同源比对及功能分类 把拼接获得的所有Unigene通过Blast比对(Evalue<1e-5)到主要蛋白数据库中,以获得其通路和功能等方面的注释。采用的数据库包括NR(非冗余蛋白数据库)、Swiss-Prot(蛋白质序列数据库)、GO(基因本体数据库)、KEGG(京都基因与基因组百科全书)和KOG(蛋白质真核直系同源数据库)等,得到跟给定Unigene具有最高序列相似性的蛋白(如有并列,取第1条),从而得到试验中所获得的Unigene序列的功能注释信息。
1.2.4 转录组中的SSR分析 使用MISA软件(http://pgrc.ipk-gatersleben.de/misa/)搜索所获得的Unigene上的潜在SSR位点。搜索条件为2个碱基的motif(基序),需要至少6个重复,3个碱基的motif需要至少5个重复,4~6个碱基的motif至少需要4次重复。如果2个SSR序列的距离短于100 bp,就会被认为是同一个潜在SSR标记。此外,为排除polyA结构的干扰,剔除了单核苷酸重复的情况。
2 结果与分析
2.1 测序与Denovo组装结果
基于Paired-end测序获得了大量的样本数据。为排除测序错误率的影响,使用滑动窗口法去除低质量片段,质量阈值20(错误率=1%),窗口大小5 bp,长度阈值35 bp,并切除reads中含N的部分序列。最终得到了有效RNA-seq数据共0.32亿条,GC平均含量为44.76%,共32 417 032条有效reads,平均长度95.77 bp。对比质控前数据,有效率为87.11%。测序数据质量控制前后对比情况见表1。
表1 测序数据质控前后对比
Tab.1 Comparison of sequence data after quality control

物种名称Species原始数据 Raw data质控后数据 Validated datareads数量reads amount碱基数量Base amountreads数量reads amount碱基数量Base amount平均长度/bpAverage length有效率/%Validation rate扁秆荆三棱37 212 6483 721 264 80032 417 0323 104 489 27795.7787.11Bolboschoenus planiculmis
注:表中reads数量为双端测序的结果。
Note: reads amount in the table is from pair-end sequencing.
将样本的有效reads合并进行de novo拼接,对拼接序列去重复,最终得到了79 098个长度大于200 bp的转录本,大小73 Mb。取每个Loci下最长的转录本作为Unigene,得到了59 788个Unigene,大小50 Mb,其最大长度、平均长度和N50分别为10 923,842,1 402 bp,长度大于500 bp和1 000 bp的Unigene数目分别为30 171和18 542。图1展示了所有Unigene的长度分布情况。
2.2 Unigene的注释
将所获得的Unigene序列比对到公共数据库(NR、 Swiss-Prot、 KEGG、Go、KOG),进行序列相似性分析(Evalue
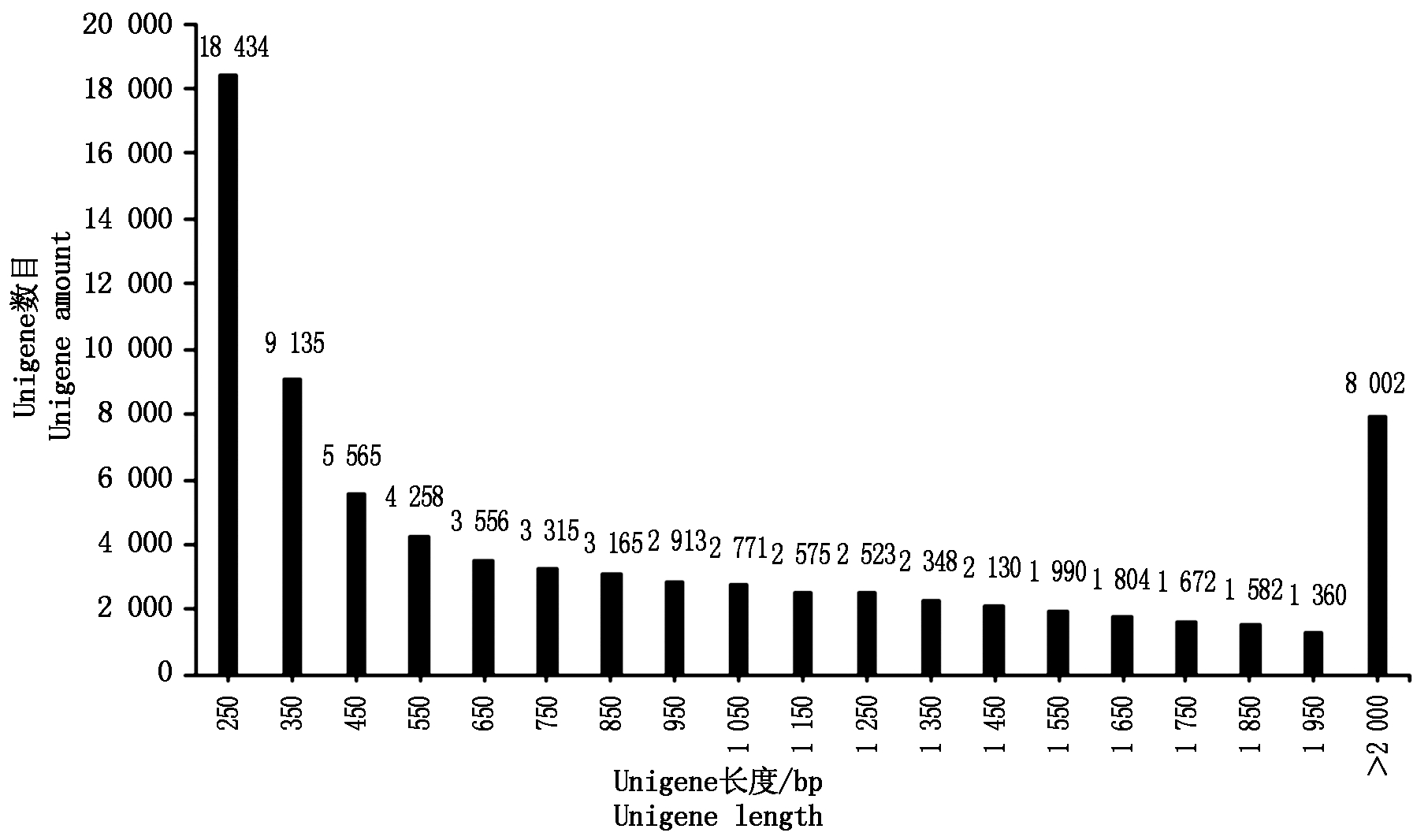
图1 所获得的Unigenes长度分布情况
Fig.1 The length distribution of acquired Unigenes
2.3 扁秆荆三棱Unigene的KOG分类
将获得的Unigene与KOG功能分类数据库进行比对,结果表明,有13 334个Unigene 被注释在25种功能分类中(图2)。其中,归类到信号传导机制(Signal transduction mechanisms)的Unigene数量最多(2 836个,4.74%),其次是蛋白质翻译后修饰与转运、分子伴侣(Posttranslational modification, protein turnover, chaperones)(2 465个,4.12%)及一般功能(General function prediction only)(2 142个,3.58%)。最小的类别是胞外结构(Extracellular structures)(33,<1‰)和细胞运动(Cell motility)(6,<1‰)。
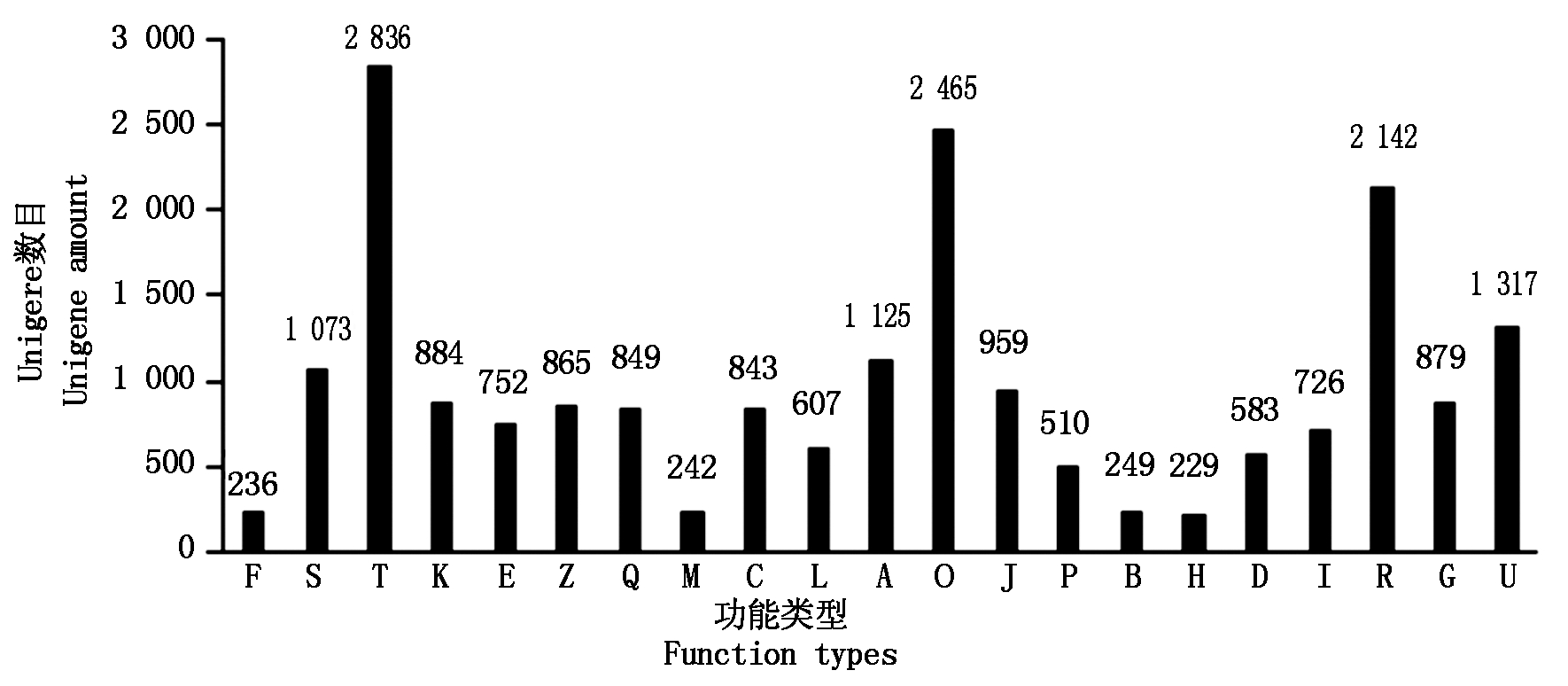
A.RNA 加工与修饰;B.染色质结构与变化;C.能量产生与转化;D.细胞周期调控与分裂,染色体重排;E.氨基酸运输与代谢;F.核苷酸运输与代谢;G.碳水化合物运输与代谢;H.辅酶运输与代谢;I.脂类运输与代谢;J.翻译,核糖体结构与生物合成;K.转录;L.复制、重组与修复;M.胞壁/膜生物发生;O.蛋白质翻译后修饰与转运、分子伴侣;P.无机离子运输与代谢;Q.次生产物合成,运输及代谢;R.一般功能;S.功能未知;T.信号传导机制;U.胞内分泌与膜泡运输;Z.细胞构架。
A.Nucleotide transport and metabolism; B.Chromatin structure and dynamics; C.Energy production and conversion; D.Cell cycle control, cell division, chromosome partitioning; E.Amino acid transport and metabolism; F.Nucleotide transport and metabolism; G. Carbohydrate transport and metabolism; H.Coenzyme transport and metabolism; I. Lipid transport and metabolism; J.Translation, ribosomal structure and biogenesis; K.Transcription; L. Replication, recombination and repair; M. Cell wall/membrane biogenesis; O.Posttranslational modification, protein turnover, chaperones; P.Inorganic ion transport and metabolism; Q.Secondary metabolites biosynthesis, transport and catabolism; R.General function prediction only; S.Function unknown; T.Signal transduction mechanism; U.Intracellular trafficking, secretion, and vesicular transport; Z. Cytoskeleton.
图2 Uingenes的KOG分类情况
Fig.2 KOG classification of Unigenes
2.4 扁秆荆三棱Unigene的GO分类
对Unigene进行GO功能分类预测,并有24 670个Unigene被注释上GO分类,并按照基因的分子功能,参与的生物过程和所处的细胞位置进行分类。如图3所示,在生物过程(Biological process)分类中,主要聚集于代谢过程(Metabiolic process)(28.66%)和细胞过程(Cellular process)(23.97%);在细胞组分(Cellular component)分类中,前三位依次是细胞(Cell)(16.00%)、细胞部分(Cell part)(16.00%)和细胞器(Organelle)(10.32%);在分子功能(Molecular function)分类中,主要聚集于结合(Binding)(25.85%)和催化活性(Catalytic activity)(23.12%)。

图3 Uingenes的GO分类
Fig.3 GO classification of Unigenes
2.5 扁秆荆三棱Unigene的KEGG通路分析
扁秆荆三棱共有13 141个Unigene映射到KEGG数据库中并得到注释,获得的参考代谢通路(Pathway)数目为291。其中包含Unigene最多的是代谢通路是核糖体(ko03010),共有405条Unigene,其次是剪切体(ko03040),共有360条Unigene。Pathway富集性分析的前10个Pathway数据列于表2。参与淀粉与糖代谢通路(ko00500)的Unigene有326条,共统计筛选出60条参与淀粉合成的Unigene,编码9个关键酶(表3),其中5个Unigene编码α-糖苷酶;11个Unigene编码β-呋喃果糖苷酶;13个Unigene编码己糖激酶;6个Unigene编码果糖激酶;4个Unigene编码葡萄糖-6-磷酸异构酶;5个Unigene编码葡萄糖磷酸变位酶;6个Unigene编码葡萄糖-1-磷酸腺苷酰基转移酶;6个Unigene编码淀粉合成酶及4个Unigene编码1,4-α-葡聚糖分支酶,另外参与植物激素信号转导的Unigene为279条,参与根茎伸长生长的赤霉素受体GID1的Unigene有4条,与赤霉素(GA)生物合成过程中的关键酶赤霉素3-β-双氧化酶(GA3ox)同源的Unigene有5条,编码细胞分裂素合酶的Unigene有5条,共计14条Unigenes。
表2 扁秆荆三棱中包含Unigenes数目最多的10个代谢通路
Tab.2 Top ten metabolic pathways involving B.planiculmis Unigenes
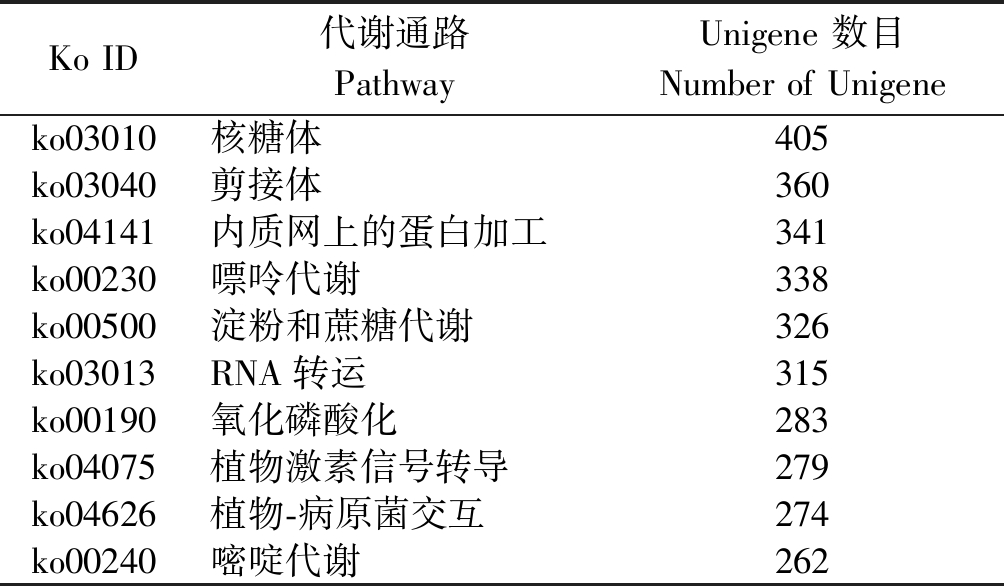
Ko ID代谢通路PathwayUnigene 数目Number of Unigeneko03010核糖体405ko03040剪接体360ko04141内质网上的蛋白加工341ko00230嘌呤代谢338ko00500淀粉和蔗糖代谢326ko03013RNA转运315ko00190氧化磷酸化283ko04075植物激素信号转导279ko04626植物-病原菌交互274ko00240嘧啶代谢262
表3 扁秆荆三棱中地下茎伸长及球茎淀粉合成相关基因
Tab.3 The genes and transcription factors related to sprouting and starch synthesis in B.planiculmis
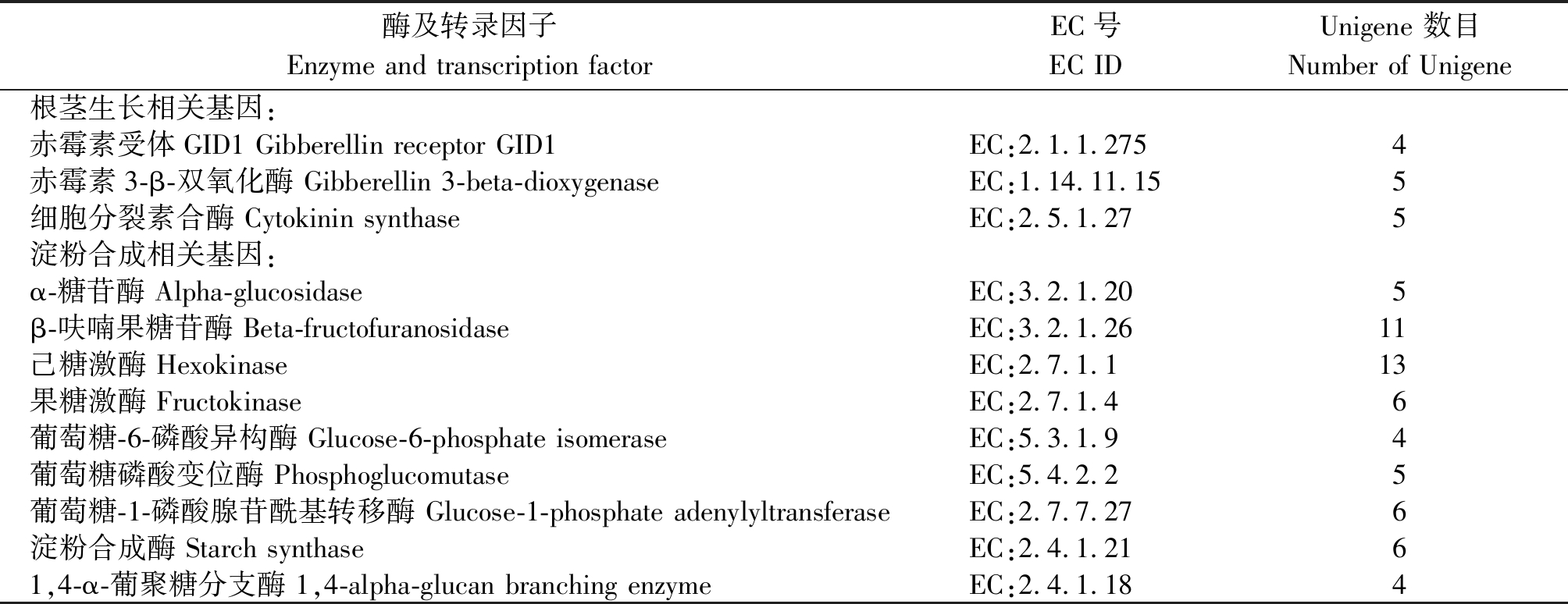
酶及转录因子Enzyme and transcription factorEC号EC IDUnigene 数目Number of Unigene根茎生长相关基因:赤霉素受体GID1 Gibberellin receptor GID1EC:2.1.1.2754赤霉素3-β-双氧化酶 Gibberellin 3-beta-dioxygenaseEC:1.14.11.155细胞分裂素合酶 Cytokinin synthaseEC:2.5.1.275淀粉合成相关基因:α-糖苷酶 Alpha-glucosidaseEC:3.2.1.205β-呋喃果糖苷酶 Beta-fructofuranosidaseEC:3.2.1.2611己糖激酶 HexokinaseEC:2.7.1.113果糖激酶 FructokinaseEC:2.7.1.46葡萄糖-6-磷酸异构酶 Glucose-6-phosphate isomeraseEC:5.3.1.94葡萄糖磷酸变位酶 PhosphoglucomutaseEC:5.4.2.25葡萄糖-1-磷酸腺苷酰基转移酶 Glucose-1-phosphate adenylyltransferaseEC:2.7.7.276淀粉合成酶 Starch synthaseEC:2.4.1.2161,4-α-葡聚糖分支酶 1,4-alpha-glucan branching enzymeEC:2.4.1.184
2.6 扁秆荆三棱转录组中潜在SSR位点的分布
利用MISA软件对扁秆荆三棱的59 788条Unigene 序列进行扫描,扫描的碱基容量为50 341 770。结果表明,12 823个Unigene 中含有潜在SSR位点,包含多于1个SSR位点的Unigene有2 531个,发生频率(含有SSR的Unigenes 数量与总Unigenes 数量之比)为21.45%。在剔除单核苷酸重复类型后,总共有9 698个SSR位点,平均0.193个SSR/kbp,SSR的分布频率(SSR的个数与总Unigenes 的数量比)为16.22%,其中二核苷酸motif(6 971个)和三核苷酸motif(2 332个)是主要的串联序列类型,分别占SSR总数的71.88%和24.05%,两者合计占所有SSR 位点的95.93%。所有潜在SSR位点的重复单元详细分布情况见表4。
表4 扁秆荆三棱转录组中潜在SSR位点重复单元的分布
Tab.4 Occurrence frequency of different microsatellites motifs in B.planiculmis transcriptome
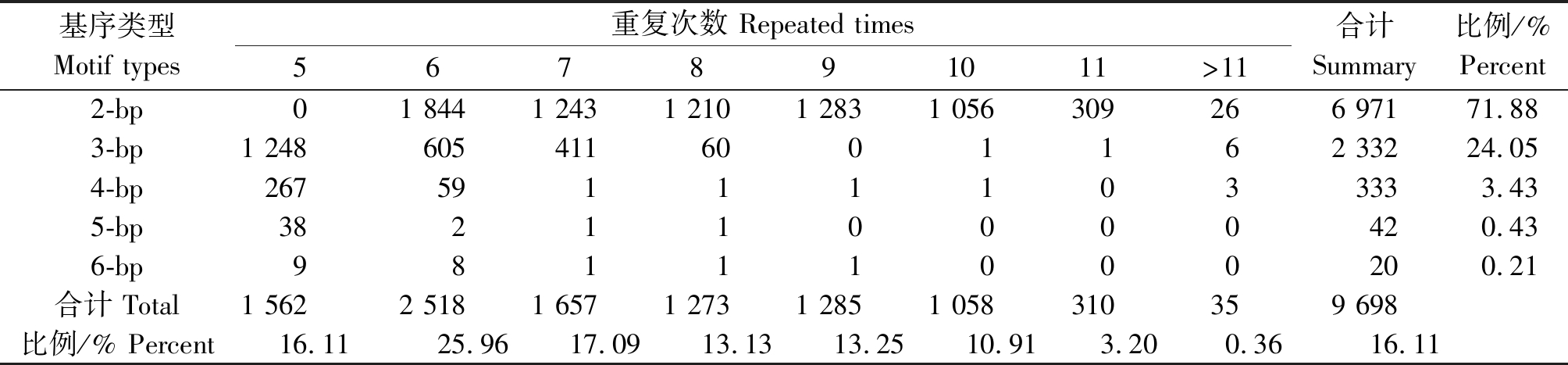
基序类型Motif types重复次数 Repeated times567891011>11合计Summary比例/%Percent2-bp01 8441 2431 2101 2831 056309266 97171.883-bp1 2486054116001162 33224.054-bp267591111033333.435-bp382110000420.436-bp98111000200.21合计 Total1 5622 5181 6571 2731 2851 058310359 698比例/% Percent16.1125.9617.0913.1313.2510.913.200.3616.11
3 结论与讨论
扁秆荆三棱为稻田和湿地环境中的常见杂草,隶属于莎草科(Cyperaceae)三棱草属(Bolboshoenus),属于无参考基因组序列的物种,目前尚未发现扁杆荆三棱或其同属物种的转录组研究先例。具体到本研究,在研究策略上,采用茎叶、球茎、根茎混合样品进行转录组测序,有助于在节约试验成本的基础上发掘到更多的转录本[24-25]。在数据质量上,通过高通量测序的技术手段,首次对扁秆荆三棱的转录组信息进行获取和分析。获得了59 788个Unigene,其最大长度为10 923 bp、平均长度为842 bp,N50为1 402 bp(即50%的数据分布在长度不小于1 402 bp的Unigene上),注释比率达到58.91%。以KEGG的代谢途径数据库为依据,可鉴别出291个代谢通路,并筛选到与地下茎伸长生长相关的Unigene 9条,与地下球茎中淀粉合成相关的Unigene 60条。此外,还获得了潜在的SSR位点9 698个,分布在12 823个Unigene中,发生频率为21.45%,其中二碱基和三碱基的motif为主要类型。类比已有的克隆植物转录组研究,如毛竹( Phyllostachys edulis) (均长612 bp,注释比率69.75%)[26]、巨龙竹(Dendrocalamus sinicus)(均长723 bp,注释比率69.22%)[27],本研究的Unigene长度较为进步,有助于获得较完整的基因表达信息。对于未获得注释的Unigene,可能是由于长度较短无法匹配,也有可能是非编码序列或者是新的基因[28],需要进一步的试验予以验证。在分析思路上,GO和KOG的功能分类对初步了解基因的功能起着重要作用,而KEGG数据库中的参考pathway不仅可以推测基因的功能,还可以研究基因在不同代谢通路中所在位置及作用,三者相辅相成,成为新物种中发掘功能基因的重要手段[29-30]。本研究通过KEGG 数据库中的pathway分析筛选与淀粉合成相关的基因,同时结合所筛选到的Unigene在相关数据库中予以功能注释,进一步确保了所获得基因的可靠性。
本研究结果有助于理解扁杆荆三棱的地下根(球)茎系统的发展过程。对克隆植物矢竹(Pseudosasa japonica)的研究表明,地下根茎的生长发育受到激素的调节,其中赤霉素在启动地下茎节间快速伸长上具有重要的作用,而细胞壁生长及细胞骨架组织等相关功能基因则在维持节间快速伸长生长上具有重要作用[31],扁秆荆三棱中也检测到了调控地下茎伸长生长的赤霉素代谢途径相关基因,但尚未发现细胞生长的相关基因。这可能是由于该物种在7月份正处于克隆生长的起始阶段,地下茎系统尚在形成过程中有关。该发现为防治该植物种群的过度扩张提供了新思路。在通路分析中,核糖体、剪接体和内质网上的蛋白加工居于前三位,表明可能在进行较大量的蛋白质合成工作,这与其地下根茎系统的快速扩张需求是相符合的[9, 22]。嘌呤代谢通路也位于前列,作为核苷酸合成、能量物质及一些初级产物如蔗糖、多糖及磷脂类物质的前体物质,嘌呤及其代谢途径在许多生物学过程中具有重要的作用[32]。这也从另一方面印证了该植物地下茎系统的能量与物质需求。此外,本研究的数据也检测到较多淀粉合成相关的基因及通路,与木薯(Manihot esculenta)[33]、马铃薯(Solanum tuberosum)[34]等主要以地下块茎储存淀粉的植物相一致。已有的研究认为扁秆荆三棱球茎中的淀粉是一种“储备”行为,可支持其度过环境胁迫时期,并为球茎萌苗的快速生长提供能量[3, 22],结果进一步表明这种储备从地下系统扩张的早期就已经开始,为进一步挖掘该植物球茎的饲养价值或野生动物保护价值提供了支持。在研究展望方面,由于本研究采用的是不同组织材料等量混合后测序和组装,虽然有助于得到更多的转录组数据,成本控制也较优秀,但是由于可变剪切等特异性机制[35],本研究结果在解释组织差异化表达方面具有局限性。在未来的研究中,应注重对该植物不同生长发育阶段,或不同组织的转录组情况进行考察,具体探讨其对水淹、盐渍和种间竞争等生境中主要限制条件的响应机制。
[1] Yang Y Y, Kim J G. The optimal balance between sexual and asexual reproduction in variable environments: a systematic review[J]. Journal of Ecology and Environment, 2016, 40(1):12-18. doi:10.1186/s41610-016-0013-0.
[2] Dong M, Yu F H, Alpert P. Ecological consequences of plant clonality[J]. Annals of Botany, 2014, 114(2): 367-368. doi:10.1093/aob/mcu137.
[3] 王沫竹,董必成,李红丽,于飞海. 根茎型植物扁秆荆三棱对光照强度和养分水平的生长响应及资源分配策略[J]. 生态学报, 2016,36(24): 8091-8101. doi:10.5846/stxb201505060938.
Wang M Z, Dong B C, Li H L, Yu F H. Growth and biomass allocation responses to light intensity and nutrient availability in the rhizomatous herb Bolboschoenus planiculmis[J]. Acta Ecologica Sinica, 2016, 36(24): 8091-8101.
[4] Kraehmer H, Jabran K, Mennan H,Chauhan B S. Global distribution of rice weeds-A review[J]. Crop Protection, 2016, 80: 73-86. doi:10.1016/j.cropro.2015.10.027.
[5] 朱新云,施慎年,吴佳文. 水稻旱直播田47%氯吡丙异可湿性粉剂除草效果及安全性[J]. 杂草学报, 2016, 34(4): 39-42. doi:10.19588/j.issn.1003-935x.2016.04.008.
Zhu X Y, Shi S N, Wu J W.Efficacy and selectivity of halosulfuron-Methyl pretilachlor isoproturon 47% WP in dry seeded rice fields[J]. Journal of Weed Science, 2016, 34(4): 39-42.
[6] 陈宇博,赖朝晖,许燎原,金彬,赵丽隐,刘桂良. 直播早稻杂草发生规律研究[J]. 农学学报, 2015, 5(8): 27-30. doi:10.11923/j.issn.2095-4050.cjas14110012.
Chen Y B, Lai C H, Xu L Y, Jin B, Zhao L Y, Liu G L. Regularity of weeds in direct seeding early rice fields[J].Journal of Agriculture, 2015,5(8):27-30.
[7] 钱希. 扁秆藨草的生物学特性及防治研究[J]. 植物生态学报, 1988, 12(3): 205-215.
Qian X. A study on biology of flat stalk bulrush and its control[J]. Chinese Journal of Plant Ecology, 1988, 12(3): 205-215.
[8] 康学耕,唐恩全,张恕茗,王艳秋,高君.关于扁杆藨草种群疯长的研究[J]. 吉林农业大学学报, 1993, 15(3): 32-36.
Kang X G, Tang E Q, Zhang S M, Wang Y Q, Gao J. A study on intrinsic rate of natural increase of Scirpus planiculmis Fr. Schmidt[J]. Journal of Jilin Agricultural University, 1993, 15(3): 32-36.
[9] Liu B, Jiang M, Tong S Z, Zhang W G, Wu H T, Liu Y, Lu X G. Differential flooding impacts on Echinochloa caudata and Scirpus planiculmis: implications for weed control in wetlands[J]. Wetlands, 2016, 36(5): 979-984. doi:10.1007/s13157-016-0805-0.
[10] Wollstonecroft M M, Ellis P R, Hillman G C, Fuller D Q. Advances in plant food processing in the Near Eastern Epipalaeolithic and implications for improved edibility and nutrient bioaccessibility: an experimental assessment of Bolboschoenus maritimus(L.) Palla(sea club-rush)[J]. Vegetation History and Archaeobotany, 2008,17(S1):19-27. doi:10.1007/s00334-008-0162-x.
[11] Zhao L Y, Jiang J H, Chen C H, Zhan S E, Yang J Y, Shao Y. Efficiency and mechanism of the phytoremediation of decabromodiphenyl ether-contaminated sediments by aquatic macrophyte Scirpus validus[J]. Environmental Science and Pollution Research, 2017, 24(14): 12949-12962. doi:10.1007/s11356-017-8900-1.
[12] Chen T R, Liu X Y, Zhang X Y, Hou Y Y, Chen X, Tao K Y. Enhanced Scirpus triqueter phytoremediation of pyrene and lead co-contaminated soil with alkyl polyglucoside and nitrilotriacetic acid combined application[J]. Journal of Soils and Sediments, 2016, 16(8): 2090-2096. doi:10.1007/s11368-016-1394-5.
[13] Hrdlickova R, Toloue M, Tian B. RNA-Seq methods for transcriptome analysis[J]. Wiley Interdisciplinary Reviews: RNA, 2017, 8(1): 1364. doi:10.1002/wrna.1364.
[14] Todd E V, Black M A, Gemmell N J. The power and promise of RNA-seq in ecology and evolution[J]. Molecular Ecology, 2016, 25(6): 1224-1241. doi:10.1111/mec.13526.
[15] Chen R S, Cheng Y F, Han S Y, Handel B V, Dong L, Li X M, Xie X Q. Whole genome sequencing and comparative transcriptome analysis of a novel seawater adapted, salt-resistant rice cultivar-sea rice 86[J]. BMC Genomics, 2017, 18(1): 655-678. doi:10.1186/s12864-017-4037-3.
[16] 邵彩虹,李瑶,钱银飞,陈金,陈先茂,关贤交,刘光荣,彭春瑞,邱才飞. 氮素胁迫对水稻根系影响的转录组分析[J]. 华北农学报, 2018, 33(1): 168-175. doi:10.7668/hbnxb.2018.01.025.
Shao C H, Li Y, Qian Y F, Chen J, Chen X M, Guan X J, Liu G R, Peng C R, Qiu C F. Transcriptional analysis of rice root under nitrogen deficiency[J]. Acta Agriculturae Boreali-Sinica, 2018, 33(1):168-175.
[17] Ramos M J N, Coito J L, Fino J, Cunha J, Silva H, de Almeida P G, Costa M M R, Am ncio S, Paulo O S, Rocheta M. Deep analysis of wild vitis flower transcriptome reveals unexplored genome regions associated with sex specification[J]. Plant Molecular Biology,2017, 93(1-2): 151-170. doi:10.1007/s11103-016-0553-9.
ncio S, Paulo O S, Rocheta M. Deep analysis of wild vitis flower transcriptome reveals unexplored genome regions associated with sex specification[J]. Plant Molecular Biology,2017, 93(1-2): 151-170. doi:10.1007/s11103-016-0553-9.
[18] Li X, Sun X D, Yang S H, Qian M, Yang Y Q, Yang Y P. Molecular cloning and functional analysis of a novel phytoglobin gene from the alpine plant Stipa purpurea[J]. Plant Ecology & Diversity, 2017, 10(1): 17-27. doi:10.1080/17550874.2017.1302998.
[19] 姜福星,杨丽娟,高素萍,高顺,陈其兵. 碰碰香叶片的转录组测序分析[J]. 分子植物育种, 2015,13(10): 2310-2319. doi:10.13271/j.mpb.013.002310.
Jiang F X, Yang L J, Gao S P, Gao S, Chen Q B. Analysis on transcriptome sequenced of Plectranthus tomentosa leaf[J]. Molecular Plant Breeding, 2015,13(10): 2310-2319.
[20] 袁卫东,陆娜,陈青,宋吉玲,王伟科. 灰树花子实体转录组测序和分析[J]. 复旦学报(自然科学版), 2015, 54(5):673-678. doi:10.15943/j.cnki.fdxb-jns.2015.05.019.
Yuan W D, Lu N, Chen Q, Song J L, Wang W K. Analysis of transcriptome sequenced of Maitake[J]. Journal of Fudan University(Natural Science), 2015, 54(5):673-678.
[21] Karthikeyan P G, George S, Chithrima C R. Nutritive value and safety of greater club rush as livestock feed[J]. Indian Journal of Weed Science, 2017, 49(1): 75-78. doi:10.5958/0974-8164.2017.00018.1.
[22] Ning Y, Zhang Z X, Cui L J, Zou C L. Adaptive significance of and factors affecting plasticity of biomass allocation and rhizome morphology: a case study of the clonal plant Scirpus planiculmis (Cyperaceae)[J]. Polish Journal of Ecology, 2014, 62(1): 77-88. doi:10.3161/104.062.0108.
[23] 王乃姗,张曼胤,崔丽娟,马牧源,颜亮,穆泳林,秦鹏. 河北衡水湖湿地汞污染现状及生态风险评价[J]. 环境科学, 2016, 37(5): 1754-1762. doi:10.13227/j.hjkx.2016.05.020.
Wang N S, Zhang M Y, Cui L J, Ma M Y, Yan L, Mu Y L, Qin P.Contamination and ecological risk assessment of mercury in Hengshuihu wetland, Hebei Province[J]. Environmental Science, 2016, 37(5): 1754-1762.
[24] 刘玉林,李伟,张志翔. 基于高通量测序的辽东栎转录组学研究[J]. 生物技术通报, 2014(7): 119-124. doi:10.13560/j.cnki.biotech.bull.1985.2014.07.028.
Liu Y L, Li W, Zhang Z X. Transcriptome analysis for Quercus liaotungensis Koidz. based on high-throughput sequencing technology[J]. Biotechnology Bulletin, 2014(7): 119-124.
[25] Zhou Y J, Gao F, Liu R, Feng J C, Li H J. De novo sequencing and analysis of root transcriptome using 454 pyrosequencing to discover putative genes associated with drought tolerance in Ammopiptanthus mongolicus[J]. BMC Genomics, 2012, 13(1): 266-278. doi:10.1186/1471-2164-13-266.
[26] He C Y, Cui K, Zhang J G, Duan A G, Zeng Y F. Next-generation sequencing-based mRNA and microRNA expression profiling analysis revealed pathways involved in the rapid growth of developing culms in Moso bamboo[J]. BMC Plant Biology, 2013, 13(1): 119. doi:10.1186/1471-2229-13-119.
[27] Cui K, Wang H Y, Liao S X, Tang Q, Li L, Cui Y Z, He Y. Transcriptome sequencing and analysis for culm elongation of the world′s largest bamboo(Dendrocalamus sinicus)[J]. PLoS One, 2016, 11(6): e157362. doi:10.1371/journal.pone.0157362.
[28] 陈春旭,李琦,郭元新,杜传来,丁志刚. 基于高通量测序的发芽苦荞转录组学研究[J]. 生物技术通报, 2016, 32(7): 40-47. doi:10.13560/j.cnki.biotech.bull.1985.2016.07.006.
Chen C X, Li Q, Guo Y X, Du C L, Ding Z G. Transcriptome analysis of germinated tartary buckwheat based on high-throughput sequencing technology[J]. Biotechnology Bulletin, 2016, 32(7): 40-47.
[29] Unamba C I N, Nag A, Sharma R K. Next generation sequencing technologies: the doorway to the unexplored genomics of non-model plants[J]. Frontiers in Plant Science, 2015, 6(185): 1074-1085. doi:10.3389/fpls.2015.01074.
[30] Loke K K, Rahnamaie-Tajadod R, Yeoh C C, Goh H H, Mohamed-Hussein Z A, Noor N M, Zainal Z, Ismail I. RNA-seq analysis for secondary metabolite pathway gene discovery in Polygonum minus[J]. Genomics Data, 2016, 7: 12-13. doi:10.1016/j.gdata.2015.11.003.
[31] 魏强,丁雨龙. 矢竹地下茎转录组测序及节间生长相关基因表达分析[J]. 南京林业大学学报(自然科学版), 2017, 41(5): 42-48. doi:10.3969 /j.issn.1000-2006.201601006.
Wei Q, Ding Y L. Transcriptome sequencing, de novo assembly and expression analysis of several genes related to internode development in Pseudosasa japonica[J]. Journal of Nanjing Forestry University( Natural Sciences Edition), 2017, 41(5): 42-48.
[32] Jeilani Y A, Williams P N, Walton S, Nguyen M T. Unified reaction pathways for the prebiotic formation of RNA and DNA nucleobases[J]. Physical Chemistry Chemical Physics, 2016, 18(30): 20177-20188. doi:10.1039/C6CP02686A.
[33] Chen X, Xia J, Xia Z Q, Zhang H F, Zeng C Y, Lu C, Zhang W X, Wang W Q. Potential functions of microRNAs in starch metabolism and development revealed by miRNA transcriptome profiling of cassava cultivars and their wild progenitor[J]. BMC Plant Biology, 2015, 15(1): 33-45. doi:10.1186/s12870-014-0355-7.
[34] Ferreira S J, Senning M, Sonnewald S, Keβling P M, Goldstein R, Sonnewald U. Comparative transcriptome analysis coupled to X-ray CT reveals sucrose supply and growth velocity as major determinants of potato tuber starch biosynthesis[J]. BMC Genomics, 2010, 11(1): 93-105. doi:10.1186/1471-2164-11-93.
[35] Baralle F E, Giudice J. Alternative splicing as a regulator of development and tissue identity[J]. Nature Reviews Molecular Cell Biology, 2017, 18(7): 437-451. doi:10.1038/nrm.2017.27.
