胚胎发育晚期富集蛋白(Late embryogenesis abundant proteins, LEA)最早在棉花中发现,是在棉花种子成熟后期大量积累的一类亲水蛋白,在水稻、大麦、小麦、玉米和拟南芥等植物中均有LEA家族基因的报道。根据LEA家族基因氨基酸的序列特征,可以将LEA蛋白家族分为LEA1、LEA2、LEA3、LEA4、LEA5、LEA6、SMPS和dehydrins 8个亚组,每一个亚组都具有独特的氨基酸保守区域[1]。研究表明,LEA家族基因不仅存在于植物的种子中,在幼苗、芽和根等部位也能检测到该基因家族的存在[2-6]。同时,在细菌和无脊椎动物中也检测到了LEA蛋白的存在。LEA家族基因广泛参与植物生长发育、形态建成和衰老等生物学过程,在高盐、干旱等非生物胁迫下,LEA蛋白还能够对植株起到关键的保护作用[6-7]。Wang等[8]从丹参中分离得到LEA2家族的SmLEA2基因,该基因能显著提高转基因丹参的耐旱性和耐盐性。Lü等[9]从百慕大草中克隆得到了2个dehydrins亚家族基因,CdDHN4-L和CdDHN4-S,2个基因编码的氨基酸序列相差1个φ片段,均能够显著提高拟南芥对于高盐胁迫的抗性以及大肠杆菌对于高盐和极端温度的耐性,并且转CdDHN4-L比转CdDHN4-S的效果更加明显。LEA蛋白家族在植物应答生物胁迫中也发挥重要作用,玉米LEA3亚组成员在烟草中过表达后,提高了对假单胞致病菌的耐性[10],在大肠杆菌中过表达拟南芥LEA2和LEA4亚组成员能显著抑制细菌的生长[11]。
研究人员最早从棉花中发现LEA蛋白家族,并对LEA基因家族的序列特征和表达模式进行了初步的研究[4, 12-14]。Luo等[15-16]从棉花中分离得到LEA4亚组的D113基因的启动子片段,启动子序列中含有与LEA蛋白特异表达相关的顺式作用元件,并能够受ABA、高盐和干旱胁迫诱导表达。刘峰等[17]从棉花新陆早33中分离得到了D34基因启动子的同源序列,并验证了其表达模式。Magwanga等[6]利用公布的棉花基因组数据,分别从陆地棉、亚洲棉和雷蒙德氏棉中分离得到242,136和142个LEA家族基因,并将这些家族成员分为8个亚组,大多数LEA家族成员与棉花耐旱性有关,LEA基因家族成员在耐旱材料夏威夷棉中的表达量明显高于干旱敏感的陆地棉材料。Magwanga等[18]的研究表明,陆地棉LEA2家族成员,CotAD_20020、CotAD_21924和CotAD_59405能显著提高转基因拟南芥的根长和耐旱性。可见,棉花中LEA基因家族在棉花逆境胁迫应答中发挥着重要的作用。本研究从棉花均一化cDNA文库中筛选得到2个棉花LEA家族的基因,命名为GhLEA和GhLEAG1,对其进行了生物信息学分析和非生物胁迫条件下的表达模式分析,旨在为进一步研究棉花LEA家族基因的功能提供参考。
1 材料和方法
1.1 试验材料
供试陆地棉品种冀228为生物技术课题组保存。
1.2 试验方法
1.2.1 胁迫处理方法 参照Zhang等[19]、甄军波等[20-21]的方法种植冀228水培苗,并对幼苗进行NaCl处理(150 mmol/L)、ABA(100 μmol/L)处理和17% PEG6000处理,对照和胁迫处理的根、茎和叶分别于胁迫处理后0,1,2,3,6,24 h取样;纤维取样:棉花开花后8DPA、12DPA、16DPA、20DPA、24DPA、30DPA和35DPA样品分别取样。所有样品均液氮速冻,用于RNA提取。
1.2.2 棉花RNA的提取和反转录 采用天根试剂盒(DP441)提取棉花RNA;采用TaKaRa (RR0037A)试剂盒合成cDNA第一链。
1.2.3 LEA基因的克隆和序列分析 在对文库测序结果的分析过程中,从中筛选得到59号和378号2个克隆,用ORFfinder 对59号和378号克隆包含完整的开放阅读框进行预测,利用BlastX进行基因的同源性比对,用ClustalX和MEGA 4.0进行序列比对并构建系统进化树。利用DANMAN预测蛋白理化性质,NetPhos 3.1预测氨基酸序列磷酸化位点,SOMPA和Phyre2分别预测蛋白质的二级结构和三级结构,MEME预测氨基酸序列的保守motif,并用TBtools绘制motif组成图[21]。
1.2.4 棉花LEA基因表达分析 用Primer Premier 5软件设计LEA基因的荧光定量PCR引物(表1),选取Histone3为内参基因,qRT-PCR反应参照TB GreenTM Premix Ex Taq TM Ⅱ(Tli RNaseH Plus)试剂盒说明书:95 ℃ 30 s;95 ℃ 15 s,60 ℃ 15 s,72 ℃ 30 s,40个循环,最后进行溶解曲线分析,采用Bio-rad荧光定量PCR仪,试验进行3次重复。用Bio-Rad CFX Manager(2-ΔΔCT法)和Origin 6.0分析处理试验结果[21]。
表1 试验中所用的引物
Tab.1 Primers used in this study

引物名称Primer name引物序列(5′-3′)Sequence 引物用途PurposeqLEAG1FGCCACTAAAGCACAACAATACG荧光定量qLEAG1RGCTATGAGGATGAGAACGAGGA荧光定量qLEAFTCAGGCTTGATTCCAGATGC荧光定量qLEARTGATGCTGCCAGGCTTAATGT荧光定量H3FACCGTCCTGGAACTGTTGCTC内参引物H3RACCCACAAGGTATGCCTCTGC内参引物GhLEAG1FCCCAAAAGCAAATTAACGC全长扩增GhLEAG1RTATACAATCAAATCACCCA全长扩增GhLEAFTGCTTGCAGCTTTGCTATTA全长扩增GhLEARACCATCCCTGTTGATCTTTT全长扩增
2 结果与分析
2.1 棉花LEA基因的克隆和序列分析
通过对本研究室前期构建的cDNA文库进行分析,59号克隆和378号克隆均包含一个完整的开放阅读框。PCR扩增2个基因的全长,与cDNA文库测序结果完全一致。在NCBI网站,利用BlastX程序进行分析比对,59号克隆和378号克隆分别编码一个棉花胚胎发育晚期富集蛋白家族基因成员,根据比对结果,将其命名为GhLEA和GhLEAG1,登录号分别为KF906314和KF906316。GhLEA全长948 bp,编码315个氨基酸,等电点4.4,分子质量为35 ku,GhLEAG1全长756 bp,编码251个氨基酸,等电点10.76,分子质量27 ku,2个基因DNA序列相似度为36.93%,氨基酸序列相似度为10.44%。
2.2 棉花GhLEA和GhLEAG1基因进化树及保守基序分析
选取GhLEA、GhLEAG1、SmLEA(AAU29064.3)、SmLEA2(HQ676610)和部分拟南芥LEA家族成员构建了氨基酸序列进化树(图1),结果表明,GhLEA、SmLEA和SmLEA2同属于LEA2家族,GhLEA和SmLEA2具有更近的亲缘关系,其与At2G44060和SmLEA2的motif组成模式一致,均包含了motif1/2/6/7,motif6在LEA2家族中高度保守。GhLEAG1与拟南芥LEA3家族的成员划分在一个亚组,但分支成员之间的motif组成不完全相同,GhLEAG1由motif3/9组成,AT3G53770和AT4G15910由motif3/10组成,GhLEA5-D、AT4G02380和AT1G02820则由motif3/8组成,可见motif3在LEA3家族成员中高度保守。
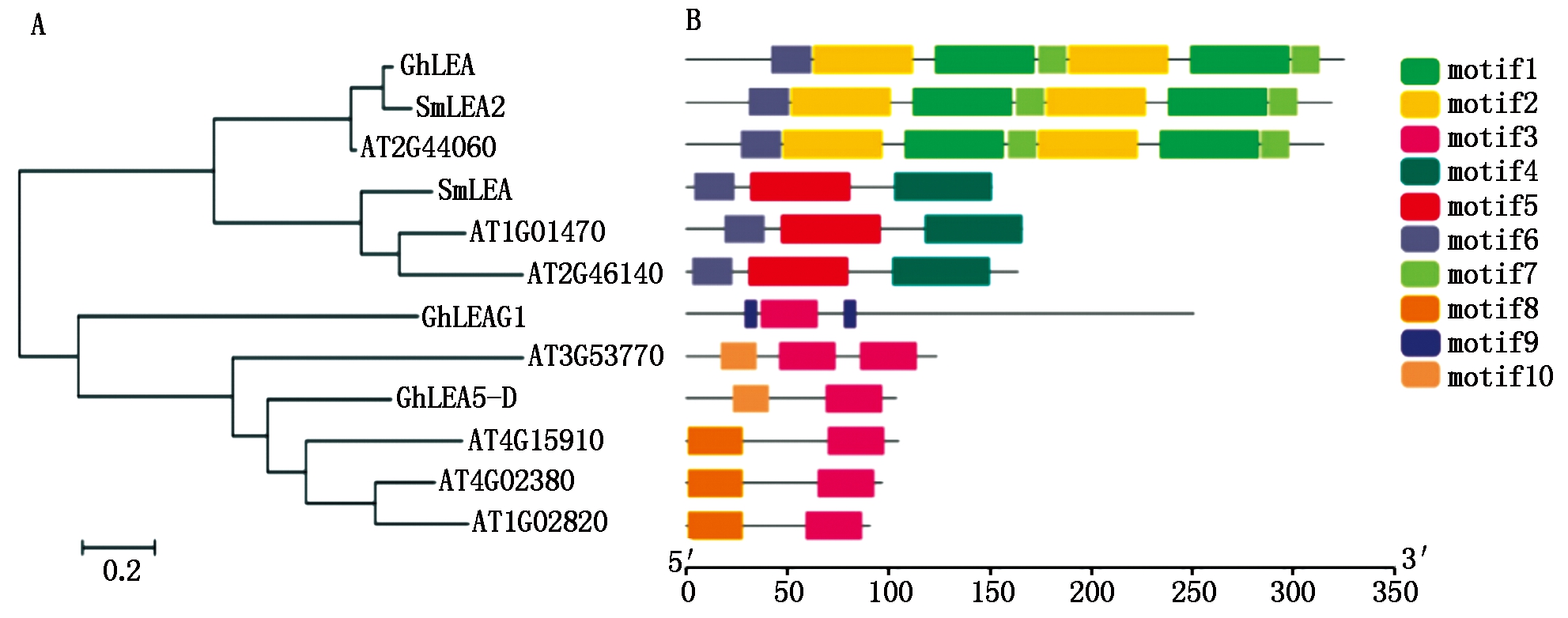
A.棉花GhLEA、GhLEAG1和部分拟南芥LEA氨基酸序列进化树分析; B.棉花GhLEA、GhLEAG1和部分拟南芥LEA蛋白保守基序组成。 A. Phylogenetic tree analysis of GhLEA, GhLEAG1 and closely related Arabidopsis LEA proteins; B. Motif analysis of GhLEA, GhLEAG1 and closely related Arabidopsis LEA proteins.
图1 GhLEA和GhLEAG1与部分拟南芥LEA进化及蛋白保守基序分析
Fig.1 Phylogenetic tree and motif analysis of GhLEA and GhLEAG1 with closely related LEA of Arabidopsis
2.3 棉花LEA基因蛋白质结构分析
参考甄军波等[21]的方法,利用NetPhos分析GhLEA和GhLEAG1的磷酸化位点(图2),GhLEA肽链具有较多的丝氨酸磷酸化位点,苏氨酸和酪氨酸磷酸化位点较少,GhLEAG1肽链中苏氨酸磷酸化位点明显增多,酪氨酸磷酸化位点较少。用SOPM法分析GhLEA蛋白的二级结构,GhLEA蛋白包括22.22%的α-螺旋,25.71%的延伸链,6.03%的β-转角和46.03%的无规则卷曲,GhLEAG1蛋白包括18.33%的α-螺旋,33.07%的延伸链,3.59%的β-转角和45.02%的无规则卷曲,通过预测2个蛋白的三级结构可以看出(图3),两者在三级结构上也存在明显的差别。
2.4 棉花LEA基因表达模式分析
分别研究了GhLEA和GhLEAG1 2个基因在纤维中的表达模式。结果表明,GhLEA在棉纤维发育20DPA时表达量达到峰值,同时,在根、茎和叶片中,GhLEA在根中的表达量最高,在叶片中的表达量最低(图4-A)。GhLEAG1在棉纤维中的表达量峰值同样出现在20DPA,在茎中的表达量最高(图4-B)。GhLEA可能只在棉纤维发育时期中的伸长期发挥作用,而GhLEAG1在伸长期及次生壁加厚期均可能发挥比较重要的作用。
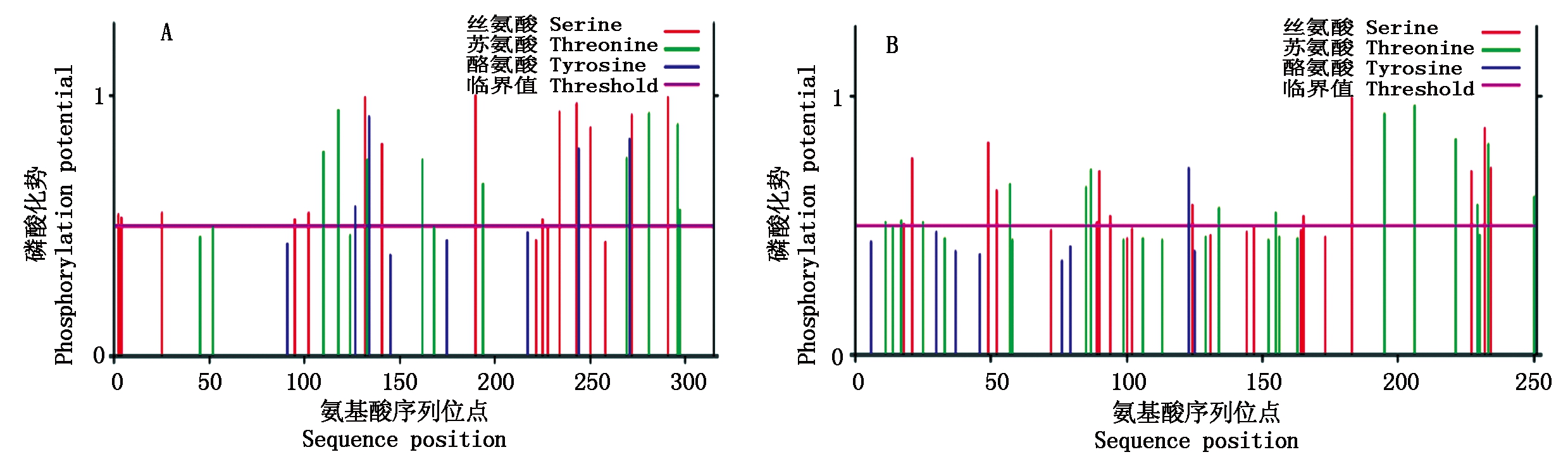
A.GhLEA; B.GhLEAG1。
图2 GhLEA和GhLEAG1蛋白磷酸化位点预测
Fig.2 The predicted phosphorylation sites in GhLEA and GhLEAG1
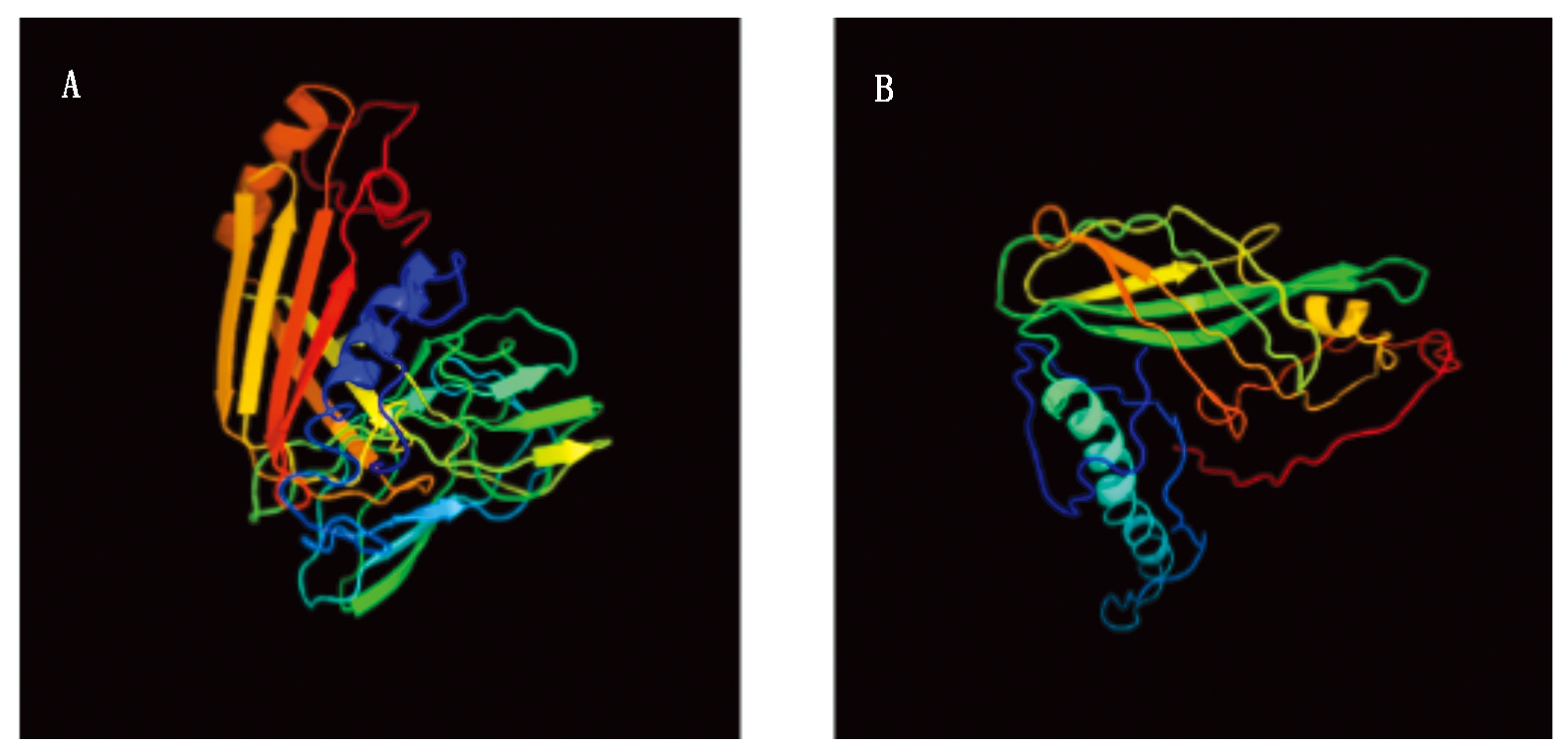
A.GhLEA三维结构; B.GhLEAG1三维结构。 A.Predicted 3D structure of GhLEA; B. Predicted 3D structure of GhLEAG1.
图3 GhLEA和GhLEAG1蛋白三维结构预测
Fig.3 The predicted tertiary structure of GhLEA and GhLEAG1
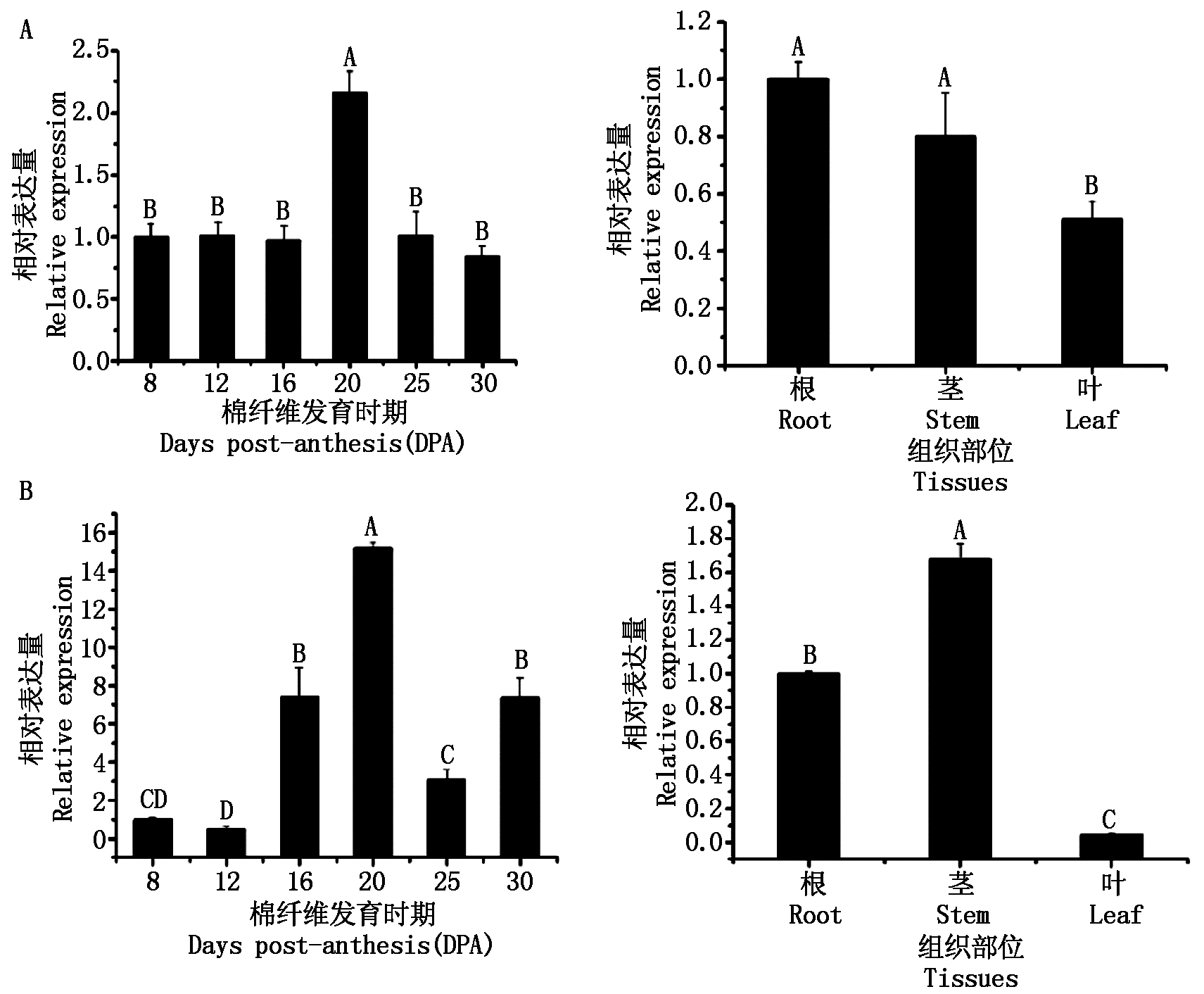
误差线上的不同大写字母表示0.01水平差异显著(P<0.01)。 A. GhLEA基因在不同组织中的表达模式; B.GhLEAG1基因在不同组织中的表达模式。 Bars superscripted by different capital letters mean significant difference(P<0.01). A.Tissue expression analysis of GhLEA; B.Tissue expression analysis of GhLEAG1.
图4 GhLEA和GhLEAG1在不同组织中的表达模式分析
Fig.4 Tissue expression analysis of GhLEA and GhLEAG1
分别验证了GhLEA和GhLEAG1在NaCl、PEG和ABA非生物胁迫条件下的表达模式。在NaCl处理条件下,GhLEA在根、茎和叶片中的表达量均有升高,在茎和叶片中的表达量峰值现在处理后3 h,在根中的表达量峰值则出现在处理后6 h。在PEG处理条件下,GhLEA在根的表达量逐渐下降,在茎中的表达量呈先降后升的趋势,在处理后6 h表达量达到最低,24 h后表达量恢复至对照水平。叶片中的表达量则呈上升趋势,峰值出现在处理后3 h。在棉花幼苗受到ABA处理条件下,GhLEA的表达量峰值出现在处理后3 h(图5-A)。通过图5-B可以看出,GhLEAG1的表达模式同GhLEA具有明显的区别。在高盐胁迫条件下,GhLEAG1在根中的表达量迅速下降,在茎中的表达量则是轻微的波动,而在叶片中的表达量则是显著提高,并且在处理后3 h达到峰值。在受到PEG处理时,GhLEAG1在根中的表达量同样是迅速下降,在茎中的表达量在6 h开始下降,在叶片中的表达量则出现在处理后6 h达到峰值。GhLEAG1同样受ABA诱导表达,表达量在处理后6 h达到峰值。

误差线上的不同大写字母表示0.01水平差异显著(P<0.01)。A.GhLEA基因表达模式分析; B.GhLEAG1基因表达模式分析。 Bars superscripted by different capital letters mean significant difference(P<0.01).A. Expression analysis of GhLEA; B.Expression analysis of GhLEAG1.
图5 GhLEA和GhLEAG1在非生物胁迫中的表达模式分析
Fig.5 Expression analysis of GhLEA and GhLEAG1 treated with different stress
3 讨论
棉花中前期已经报道了GhD-7、GhD11、GhD-19、GhD-29、GhLEA14-A、GhLEA5-D、GhLEAD-113、GhLEAD-34等8个LEA蛋白家族成员,根据其氨基酸序列的相似度和motif组成,这8个家族成员分别属于LEA1-5及Dehydrin家族成员[8]。陆地棉LEA2蛋白家族成员最多,有157个家族成员,根据其序列和motif结构组成可以将其分为6个亚组[18]。
本研究从棉花均一化cDNA文库中的2个LEA蛋白家族成员GhLEA和GhLEAG1,分别编码315,251个氨基酸,与棉花中已报道的8个LEA蛋白家族成员分析比对,GhLEA和GhLEAG1属于新的LEA家族成员。研究结果表明,GhLEA属于棉花LEA2中的一员,GhLEAG1则属于LEA3亚组,qRT-PCR结果表明,两者在棉花纤维、根、茎和叶片中均有表达,两者均受高盐、PEG和ABA等非生物胁迫诱导表达,但是GhLEA和GhLEAG1可能在棉纤维发育的伸长期和次生壁加厚期分别发挥作用,两者在蛋白质二级结构和三级结构上也具有比较明显的差异。Gao等[22]研究结果表明,即使在同一个亚组中,不同成员由于其motif组成及表达模式的差异,在功能上也会有所差异。从构建的系统进化树可以看出,GhLEA与丹参SmLEA和SmLEA2具有较近的亲缘关系,都属于LEA2家族成员,但是由于SmLEA由motif4/5/6组成,GhLEA、SmLEA2和SmLEA分别在不同的分支上。Hundertmark和Hincha[3]研究表明,At2G44060能够受胁迫诱导表达。Wang等[8]和Wu等[23]的研究结果也表明,SmLEA和SmLEA2均能够提高大肠杆菌和转基因丹参的耐盐性,但是其组织部位表达特异性存在差异,可能与其motif组成有关。GhLEA也能受到高盐等非生物胁迫的诱导表达,且GhLEA与At2G44060和SmLEA2的motif组成模式完全一致,推测其可能与SmLEA2具有相似的功能。植物LEA3家族成员也可能参与生物和非生物胁迫应答[5]。GhLEAG1则属于棉花LEA3家族,与棉花中已经克隆的GhLEA5-D同在一个亚组,但是motif组成模式具有一定的差别,可能属于LEA3亚家族中的一个小分支,与GhLEA5-D等基因发挥不同的作用。
[1] Battaglia M, Covarrubias A A. Late embryogenesis abundant(LEA) proteins in legumes[J]. Frontiers in Plant Science, 2013, 4: 190.doi:10.3389/fpls.2013.00190.
[2] Shao H B, Liang Z S, Shao M A. LEA proteins in higher plants: structure, function, gene expression and regulation[J]. Colloids and Surfaces B:Biointerfaces, 2005, 45(3-4): 131-135. doi:10.1016/j.colsurfb.2005.07.017.
[3] Hundertmark M,Hincha D K. LEA(late embryogenesis abundant) proteins and their encoding genes in Arabidopsis thaliana[J]. BMC Genomics, 2008, 9(1): 118. doi:10.1186/1471-2164-9-118.
[4] Galau G A, Hughes D W, Dure L Ⅲ. Abscisic acid induction of cloned cotton late embryogenesis-abundant(Lea) mRNAs[J]. Plant Molecular Biology, 1986, 7(3): 155-170. doi:10.1007/BF00021327.
[5] Liang Y, Xiong Z Y, Zheng J X, Xu D Y, Zhu Z Y, Xiang J, Gan J P, Raboanatahiry N, Yin Y T, Li M T.Genome-wide identification, structural analysis and new insights into late embryogenesis abundant(LEA) gene family formation pattern in Brassica napus[J]. Scientific Reports, 2016, 6: 24265. doi:10.1038/srep24265.
[6] Magwanga R O, Lu P, Kirungu J N, Lu H J, Wang X X, Cai X Y, Zhou Z L, Zhang Z M, Salih H, Wang K B,Liu F. Characterization of the late embryogenesis abundant(LEA) proteins family and their role in drought stress tolerance in upland cotton[J]. BMC Genetics, 2018, 19(1): 6. doi:10.1186/s12863-017-0596-1.
[7] Huang L P, Zhang M Y, Jia J, Zhao X X, Huang X X, Ji E, Ni L,Jiang M Y. An atypical late embryogenesis abundant protein OsLEA5 plays a positive role in ABA-induced antioxidant defense in Oryza sativa L.[J]. Plant and Cell Physiology, 2018, 59(5): 916-929. doi:10.1093/pcp/pcy035.
[8] Wang H Q, Wu Y C, Yang X B, Guo X R, Cao X Y. SmLEA2, a gene for late embryogenesis abundant protein isolated from Salvia miltiorrhiza, confers tolerance to drought and salt stress in Escherichia coli and S. miltiorrhiza[J]. Protoplasma, 2017, 254(2): 685-696. doi:10.1007/s00709-016-0981-z.
[9] Lü A M, Su L T, Liu X C, Xing Q, Huang B R, An Y, Zhou P. Characterization of Dehydrin protein, CdDHN4-L and CdDHN4-S, and their differential protective roles against abiotic stress in vitro[J]. BMC Plant Biology, 2018, 18(1): 299. doi:10.1186/s12870-018-1511-2.
[10] Liu Y, Wang L, Xing X, Sun L P, Pan J W, Kong X P, Zhang M Y, Li D Q. ZmLEA3, a multifunctional group 3 lEA protein from maize(Zea mays L.), is involved in biotic and abiotic stresses[J]. Plant and Cell Physiology, 2013, 54(6): 944-959. doi:10.1093/pcp/pct047.
[11] Campos F, Zamudio F,Covarrubias A A. Two different late embryogenesis abundant proteins from Arabidopsis thaliana contain specific domains that inhibit Escherichia coli growth[J]. Biochemical and Biophysical Research Communications, 2006, 342(2): 406-413. doi:10.1016/j.bbrc.2006.01.151.
[12] Galau G A, Dure L Ⅲ. Developmental biochemistry of cottonseed embryogenesis and germination: changing messenger ribonucleic acid populations as shown by in vitro and in vivo protein synthesis[J]. Biochemistry, 1981, 20(14): 4169-4178. doi:10.1021/bi00517a034.
[13] Galau G A,Wang H Y C,Hughes D W. Cotton Lea5 and Lea14 encode atypical late embryogenesis-abundant proteins[J]. Plant Physiology, 1993, 101(2): 695-696. doi:10.1104/pp.101.2.695.
[14] Baker J, Steele C V, Dure L Ⅲ. Sequence and characterization of 6 Lea proteins and their genes from cotton[J]. Plant Molecular Biology, 1988, 11(3): 277-291. doi:10.1007/BF00027385.
[15] Luo K M, Zhang G F, Deng W, Luo F T, Qiu K,Pei Y. Functional characterization of a cotton late embryogenesis-abundant D113 gene promoter in transgenic tobacco[J]. Plant Cell Reports, 2008, 27(4): 707-717. doi:10.1007/s00299-007-0482-9.
[16] 罗克明, 郭余龙, 肖月华, 侯磊, 裴炎. 棉花Lea 蛋白D-113基因启动子的克隆及序列分析[J]. 遗传学报,2002, 29(2): 161-165.
Luo K M, Guo Y L, Xiao Y H, Hou L, Pei Y. Cloning and characterization of D113 gene promoter from cotton[J]. Journal of Genetics Genomics, 2002, 29(2): 161-165.
[17] 刘峰, 汪小东, 赵彦鹏, 孙杰. 棉花种子特异表达的 LEA 启动子克隆及功能验证[J]. 棉花学报, 2014, 26(4): 310-317. doi:10.3969/j.issn.1002-7807.2014.04.005.
Liu F, Wang X D, Zhao Y P, Sun J. Isolation and functional characterization of the seed-specific promoter of LEA gene from cotton(Gossypium hirsutum L.) [J]. Cotton Science, 2014, 26(4): 310-317.
[18] Magwanga R O, Lu P, Kirungu J N, Dong Q, Hu Y G, Zhou Z L, Cai X Y, Wang X X, Hou Y Q, Wang K B, Liu F. Cotton late embryogenesis abundant(LEA2) genes promote root growth and confer drought stress tolerance in transgenic Arabidopsis thaliana[J].G3(Bethesda, Md.), 2018, 8(8):2781-2803. doi:10.1534/g3.118.200423.
[19] Zhang X, Zhen J B, Li Z H, Kang D M, Yang Y M, Kong J, Hua J P. Expression profile of early responsive genes under salt stress in upland cotton(Gossypium hirsutum L.) [J]. Plant Molecular Biology Reporter, 2011, 29(3): 626-637. doi:10.1007/s11105-010-0269-y.
[20] 甄军波, 蔡肖, 江振兴, 刘琳琳, 张建宏, 张香云, 迟吉娜. 棉花GhbHLH基因的克隆与表达分析[J]. 分子植物育种, 2017, 15(4): 1233-1239. doi:10.13271/j.mpb.015.001233.
Zhen J B, Cai X, Jiang Z X, Liu L L, Zhang J H, Zhang X Y, Chi J N. Molecular cloning and expression analysis of GhbHLH from upland cotton[J]. Molecular Plant Breeding, 2017, 15(4): 1233-1239.
[21] 甄军波,刘迪,宋世佳,蔡肖,刘琳琳,迟吉娜.棉花两个14-3-3基因的克隆和表达分析[J].分子植物育种,2019,17(5):1422-1429. doi:10.13271/j.mpb.017.001422.
Zhen J B, Liu D, Song S J, Cai X, Liu L L, Chi J N. Cloning and expression analysis of two 14-3-3 genes from upland cotton[J]. Molecular Plant Breeding, 2019,17(5):1422-1429.
[22] Gao J,Lan T. Functional characterization of the late embryogenesis abundant(LEA) protein gene family from Pinus tabuliformis(Pinaceae) in Escherichia coli[J]. Scientific Reports, 2016, 6: 19467. doi:10.1038/srep19467.
[23] Wu Y C, Liu C L, Kuang J, Ge Q, Zhang Y, Wang Z Z. Overexpression of SmLEA enhances salt and drought tolerance in Escherichia coli and Salvia miltiorrhiza[J]. Protoplasma, 2014, 251(5): 1191-1199. doi:10.1007/s00709-014-0626-z.
