腋生分生组织(Axillary meristem,AM)起源于茎顶端分生组织(Shoot apical meristem,SAM)旁的边界区(Boundary zone),为植物侧枝的生长和发育提供细胞来源,对植物器官发育和形态的建成极为重要[1-4]。SAM旁的边界区通常由一些小且低分裂速率的细胞组成,与SAM干细胞区不同,这些边界区细胞处于分化状态,但是在早期的边界区发育过程中,所有的边界区细胞都会低量表达标记细胞分化状态的SHOOT MERISTEMLESS(STM)基因,恢复未分化状态,起始形成腋生分生组织[5]。已有研究表明,许多转录因子通过与激素的共同作用调节着腋芽分生组织的发育和形成。如GRAS转录因子中的LATERAL SUPPRESSOR(LAS)/MOC/LS基因[6-8]、bHLH家族转录因子ROX/LAX1/BA[9-13]和Myb家族转录因子Blind/RAXs[14-17]等都被证实影响腋生分生组织的发育。
番茄中的Blind基因是最早用于研究腋生分生组织发育的空间标记之一,它是R2R3 Myb家族的一员,Blind基因在SAM旁的边界区域内的中央部分表达,调节腋生分生组织的形成和维持[15,17]。拟南芥中,Blind的同源基因RAX1、RAX2和RAX3的功能缺失突变体呈现出不同程度的侧芽缺失的表型,结合rax突变体中SHOOT MERISTEMLESS(STM)基因的转录表达检测结果表明,AtRAXs基因控制了腋生分生组织生长的很早的一个阶段,RAX1、RAX2和RAX3在侧枝发育的不同时期发挥作用,且具有部分功能冗余 [14,16]。前人研究表明,番茄和拟南芥中分别有9,6个Blind同源基因,这些基因大部分与分枝发育和叶型发育相关,Blind同源基因的前118个氨基酸具有高度的保守性,C端的同源性却较低,但是不同的物种中部分Blind同源基因的C端间却保持了特有的同源性[17]。
尽管高等植物中腋生分生组织维持相关的转录因子已经被大量发现和系统研究,但是烟草属中只有少数的侧生分生组织维持相关基因被报道[18-22]。由于普通烟草为异源四倍体,相对拟南芥等二倍体植物,其同源基因的数量通常加倍,同时存在功能冗余,只有发现更多相关基因才能解释同一通路或者多个通路共同影响下的腋生分生组织形成。本研究利用同源比对法在烟草的公开测序数据中克隆烟草Blind基因,对其进行生物信息学分析和非生物胁迫下的表达分析,为后续基因功能验证、创建烟草腋芽缺失植株打下基础。
1 材料和方法
1.1 材料与试剂
烟草种子K326(Nicotiana tabacum cv. K326)由四川农业大学农学院提供。TRNzol总RNA提取试剂(DP405)和反转录试剂盒 TIANScript Ⅱ RT Kit(KR107)购自天根生化科技有限公司;EasyTaq DNA polymerase购自全式金生物科技公司;PCR引物合成和测序均由北京擎科新业生物有限公司完成。
1.2 试验方法
1.2.1 烟草幼苗的胁迫处理 用5%次氯酸钠对烟草K326的种子进行表面消毒并播种于MS培养基上,播种后的培养基置于25 ℃的光照培养室内竖直培养14 d,然后转入MS液体培养基(无糖)中继续培养7 d,继而进行模拟干旱(20% PEG-6000)、1 μmol/L ABA、200 mmol/L NaCl的非生物胁迫处理,处理的时间间隔均为0,8,16,24 h,每个处理在4个取样点取100 mg烟苗全株用于RNA提取。
1.2.2 烟草NtBL1基因的克隆 提取K326苗期全株的总RNA,方法参照TRNzol(DP405)试剂的产品说明书;分别取2 μg总RNA合成cDNA,反转录引物采用oligo dT18,方法参照TIANScript Ⅱ RT Kit(KR107)的产品说明。以K326根部cDNA为模板,NtBL14F和NtBL1442R为引物,用EasyTaq DNA polymerase扩增NtBL1的CDS序列,采用普通三步法的PCR程序,琼脂糖凝胶检测PCR产物,并将PCR产物送出测序。
1.2.3 NtBL1的半定量和qRT-PCR表达分析 NtBL1基因的半定量分析。提取24 d苗期的根、全株、开花期的茎、叶、花等组织的RNA,反转录获得cDNA,方法同1.2.2中cDNA的合成方法。以cDNA为模板,NtBL1-qF和NtBL1-qR为引物扩增NtBL1的部分CDS序列,PCR产物大小约为150 bp,且引物设计跨内含子,采用烟草的ACTIN2为对照(引物为NtActinqF/R,序列见表1),琼脂糖凝胶电泳检测产物。
NtBL1基因的qRT-PCR表达分析。提取非生物胁迫处理后的K326幼苗RNA,反转录获得cDNA,以此cDNA为模板,NtBL1-qF/qR为引物进行相对定量分析,对照采用烟草ACTIN2为对照(引物为NtActinqF/R,序列见表1),表达差异的计算方法采用2-ΔΔCt法。
表1 引物序列
Tab.1 Primer sequences
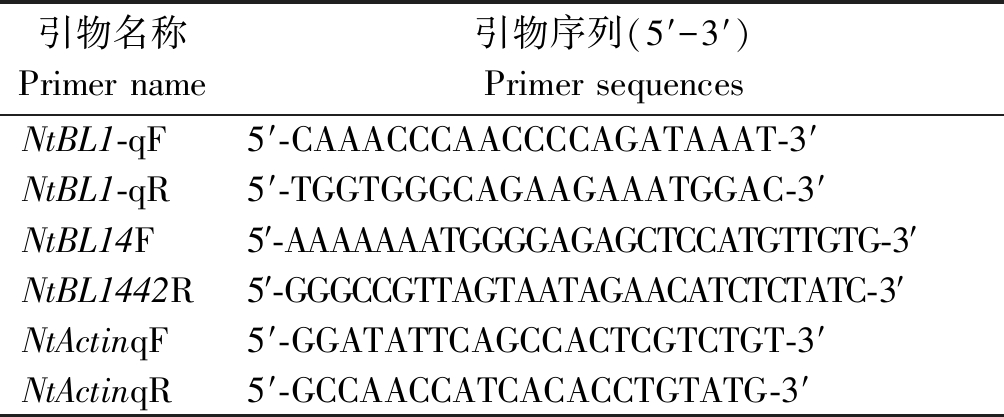
引物名称Primer name引物序列(5′-3′)Primer sequencesNtBL1-qF5′-CAAACCCAACCCCAGATAAAT-3′NtBL1-qR5′-TGGTGGGCAGAAGAAATGGAC-3′NtBL14F5′-AAAAAAATGGGGAGAGCTCCATGTTGTG-3′NtBL1442R5′-GGGCCGTTAGTAATAGAACATCTCTATC-3′NtActinqF5′-GGATATTCAGCCACTCGTCTGT-3′NtActinqR5′-GCCAACCATCACACCTGTATG-3′
1.2.4 NtBL1的生物信息学分析 从Sol Genomics Network、TAIR、NCBI网站下载烟草、拟南芥、番茄Blind同源基因的蛋白质序列,序列号分别为:绒毛状烟草mRNA_79528_cds(N.tomBL)、林烟草mRNA_10368_cds(N.sylBL)、本生烟Niben101Scf04560g07009.1(N.benBL1)、Niben101Scf01693g01004.1(N.benBl2)、渐狭叶烟草NIATv7_g13170.t1(N.attBL)、番茄Solyc11g069030.1(blind)、Solyc06g074910.2(blind-like2)、Solyc06g074920.1(blind-like6)、Solyc09g-008250.2(blind-like1)、Solyc04g077260.2(blind-like3)、Solyc02g091980.1(blind-like7)、Solyc07g-006750.2(blind-like8)、Solyc12g008670.1(blind-like4)、Solyc08g065910.1(blind-like5)、拟南芥AT5G57620.1(MYB36)、AT5G65790.1(MYB68)、AT3G49690.1(RAX3/MYB84)、AT2G36890.2(RAX2/MYB38)、AT2G36890.1(RAX2/MYB38)、AT5G23000.1(MYB37)、AT4G37780.1(MYB87),仅以番茄Blind、拟南芥RAX1、RAX2、RAX3 4个蛋白序列的前118个氨基酸为模板构建参考数据的隐马尔可科夫模型,然后利用Hmmer(Hmmsearch)软件在茄科基因组网站的烟草K326全基因组数据库中检索,获得包括NtBL1在内的14条氨基酸序列,序列号分别为:Nitab4.5_0001442g0010/NtBL1、Nitab4.5_0004993g0020/NtBL2、Nitab4.5_0001163g-0150、Nitab4.5_0001050g0010、Nitab4.5_0000578g-0120、Nitab4.5_0007679g0010、Nitab4.5_0003478g-0050、Nitab4.5_0006804g0010、Nitab4.5_0012173g-0010、Nitab4.5_0000063g0150、Nitab4.5_0000244g-0260、Nitab4.5_0001074g0040、Nitab4.5_0003781g-0060、Nitab4.5_0004621g0030。
利用ProtParam(ExPASy)在线分析软件分析NtBL1蛋白的理化性质;运用TMHMM和TMpred(ExpASy)进行基因的跨膜结构分析;利用PROSITE(ExPASy)对NtBL1进行蛋白质结构域分析;利用Sopma和Swissmodel分别预测蛋白二级结构和三级结构;用MEME软件对NtBL1及其同源蛋白分别进行了蛋白结构分析;利用ClustralW和DNAMan软件对氨基酸序列进行比对与分析,采用Mega 7.0 软件Neighbor-Joining构建系统进化树,Bootstrap设置1000次。
2 结果与分析
2.1 烟草NtBL1的克隆
以烟草K326的24 d苗期根部cDNA为模板,NtBL14F/NtBL1442R为引物进行PCR扩增,电泳结果显示在1 000 bp附近扩增得到目的大小的条带(图1),将PCR产物送出测序获得1 014 bp的测序序列,将测序结果与茄科基因组网站(http://www.Solgenomics.net//)上下载的NtBL1(序列号:Nitab4.5_0001442g0010)序列进行比对,结果显示序列一致。
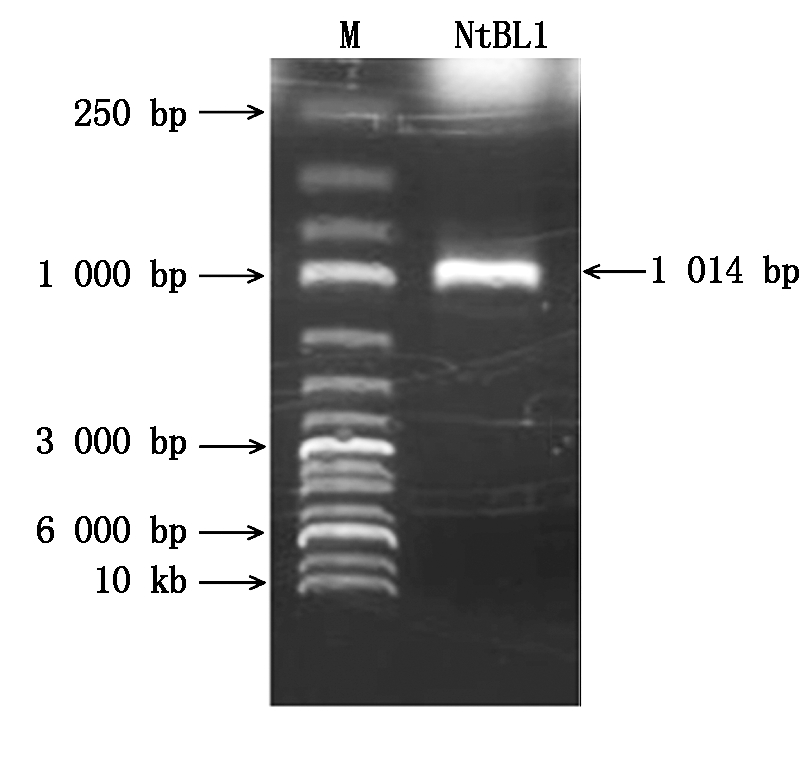
M.1 kb DNA Marker.
图1 NtBL1基因的克隆
Fig.1 Cloning of NtBL1 gene
2.2 烟草NtBL1基因及其同源基因的表达分析
对茄科基因组网站(Sol Genomics Network)上的烟草转录组数据进行分析,表明NtBL1基因在52 d的烟苗地上部分、根和茎顶端中均有表达,但在根中的表达量显著高于地上部分和茎顶端(图2);同时,对不同部位的烟株进行半定量PCR的结果同样显示,NtBL1基因在24 d苗期全株、苗期根、成熟期的花序、叶、茎中均有表达,但苗期全株和根中NtBL1的转录水平表达量明显高于花和叶(图3)。综合结果表明,NtBL1基因在普通烟草的多个时期和部位普遍表达,且在根中的表达量较高。
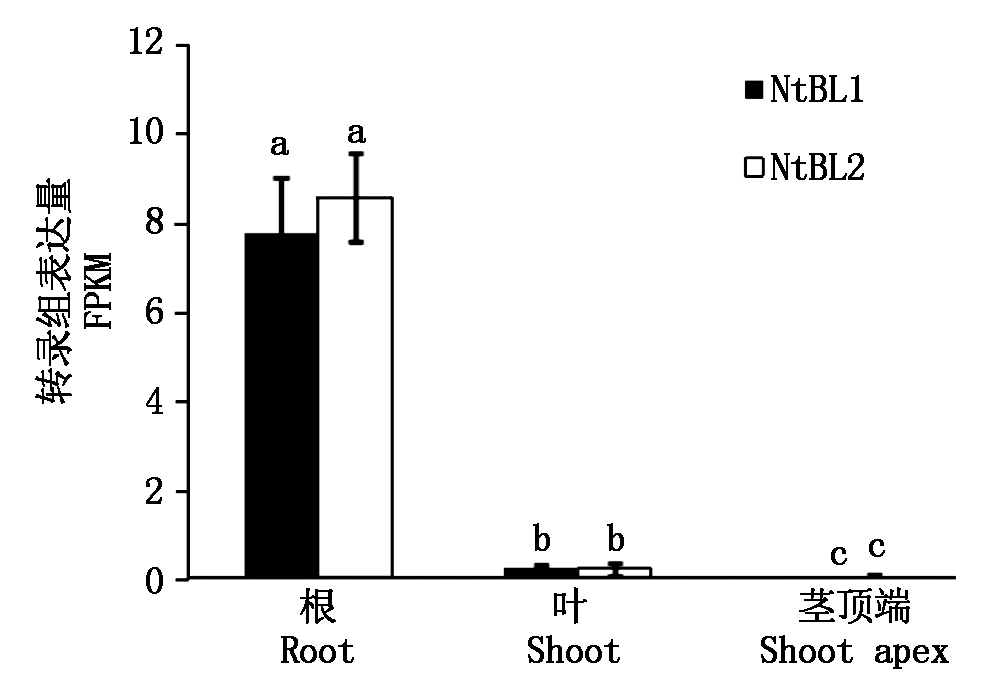
不同小写字母表示差异达到显著水平(P<0.05)。 Different lowercase letter show significant difference(P<0.05).
图2 NtBL1在不同组织中的转录组表达量分析
Fig.2 Expression analysis of NtBL1 in transcriptome of different tissues
2.3 烟草NtBL1蛋白质结构的生物信息学分析
2.3.1 NtBL1蛋白质的理化性质和跨膜结构分析 用ProtParam(ExPASy)在线分析了烟草NtBL1蛋白的理化性质,该蛋白含有337个氨基酸,其中天冬酰胺(Asn)含量最高,占比12.8%,其分子质量为3.8 ku, pI值为8.46,有 29个带负电荷的氨基酸残基和33个带正电荷的氨基酸残基,蛋白质的不稳定系数和平均总亲水性系数分别为52.96和-0.84,属于不稳定的亲水性蛋白。利用TMHMM和TMpred(ExpASy)进行基因的跨膜结构分析均表明NtBL1不存在跨膜结构。
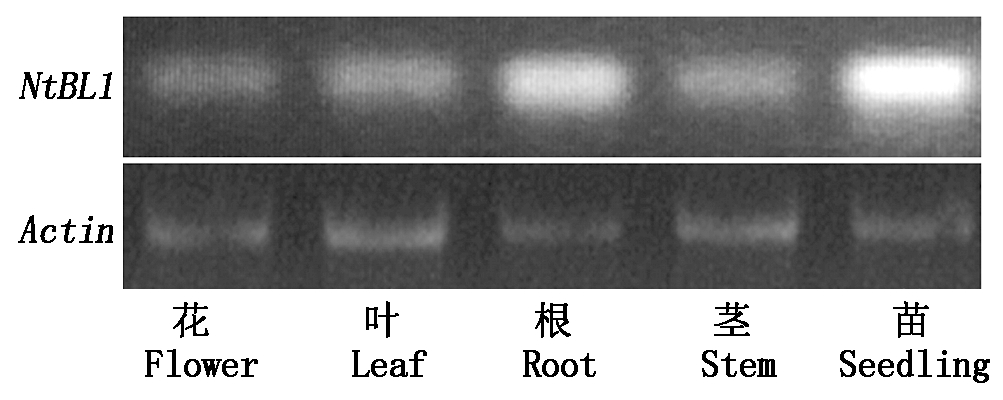
图3 NtBL1在不同组织中的表达分析
Fig.3 Analysis of relative expression of NtBL1 in different tissues
2.3.2 NtBL1蛋白二级、三级结构预测 利用PROSITE(ExPASy)和DNAMan进行结构域分析发现NtBL1的氨基酸序列包含2个Myb-type HTH DNA-binding domian,分别位于9-62,63-117的氨基酸区段,DNA结合区分别位于38-61和90-113的2个区段(图4)。利用Sopma进行蛋白质二级结构分析表明,62.61%的氨基酸组成了无规则卷曲(Random coil),21.07%的氨基酸组成了α-螺旋(Alpha helix),13.35%的氨基酸参与组成延伸链(Extended strand),2.97%的氨基酸参与组成β-转角(Beta turn)(图5-A)。利用Swissmodel预测得到NtBL1蛋白的三级结构(图5-B),与二级结构结果较为一致,表明NtBL1蛋白高级结构主要由无规则卷曲和α-螺旋组成。

图4 部分烟草属、番茄和拟南芥中Blind同源基因的多序列比对
Fig.4 Multiple sequences alignment of Blind homologues in several Nicotiana species,Tomato and Arabidopsis
2.3.3 NtBL1蛋白的同源性分析 从茄科基因组网站(Sol Genomics Network)和拟南芥网站(TAIR)上分别下载番茄、普通烟草K326和拟南芥的Blind/RAX同源蛋白序列,通过ClustalW进行多序列比对,然后用Mega 7.0软件构建系统进化树,同时利用MEME网站分析上述序列的模体结构(图6)。结果显示,NtBL1序列与番茄中的BL、Blind-like2、Blind-like6聚合在一起,且这些序列共同含有模体1,2,3,15,大部分含有模体5,8,9,11,12,相似的模体结构预示着番茄和烟草中BL同源基因可能拥有类似的功能,如NtBL1基因可能在烟草分支发育过程中起重要的调控作用。
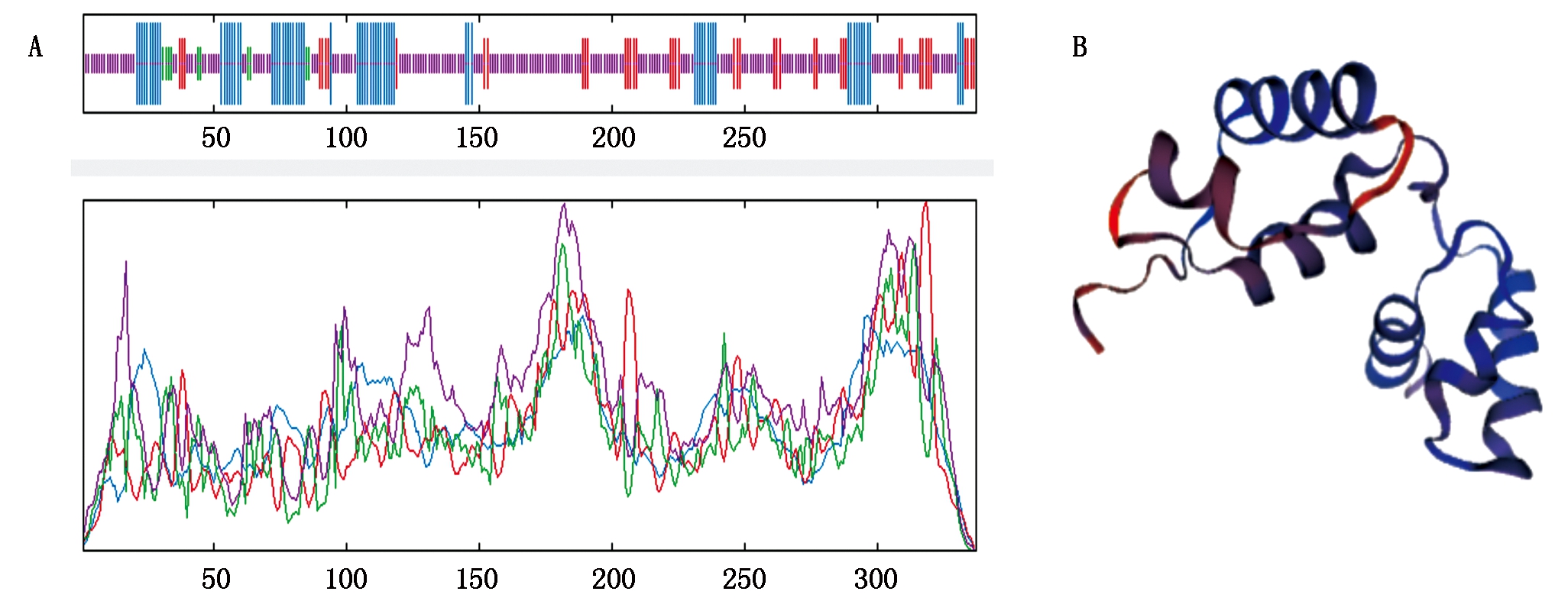
A.NtBL1二级结构预测图;B.NtBL1的三级结构预测图。 A.Secondary structure of NtBL1;B.Tertiary structure of NtBL1.
图5 NtBL1蛋白质的二级结构、三级结构预测
Fig.5 Secondary structure and tertiary structure prediction of NtBL1
利用DNAMan软件计算NtBL1与番茄、拟南芥和部分烟属Blind同源序列间的氨基酸相似度发现(图4),NtBL1基因与林烟草、绒毛烟草BL同源基因的蛋白序列同源性分别高达99.7%和97.3%,说明NtBL1来源于林烟草基因组;NtBL1基因与本生烟和渐狭叶烟草中3个Blind同源基因的全长氨基酸序列同源性为95.85%~96.13%,前118个氨基酸的同源性为98.28%~99.14%,证明Blind基因在烟属植物中普遍存在且序列保守程度高;NtBL1与番茄Blind的前118个氨基酸序列(Myb结构域)同源性为92.31%,与拟南芥RAXs的前118个氨基酸序列的同源性为82.05%~86.32%,进一步证实了NtBL1是番茄Blind基因在普通烟草中的同源基因之一。
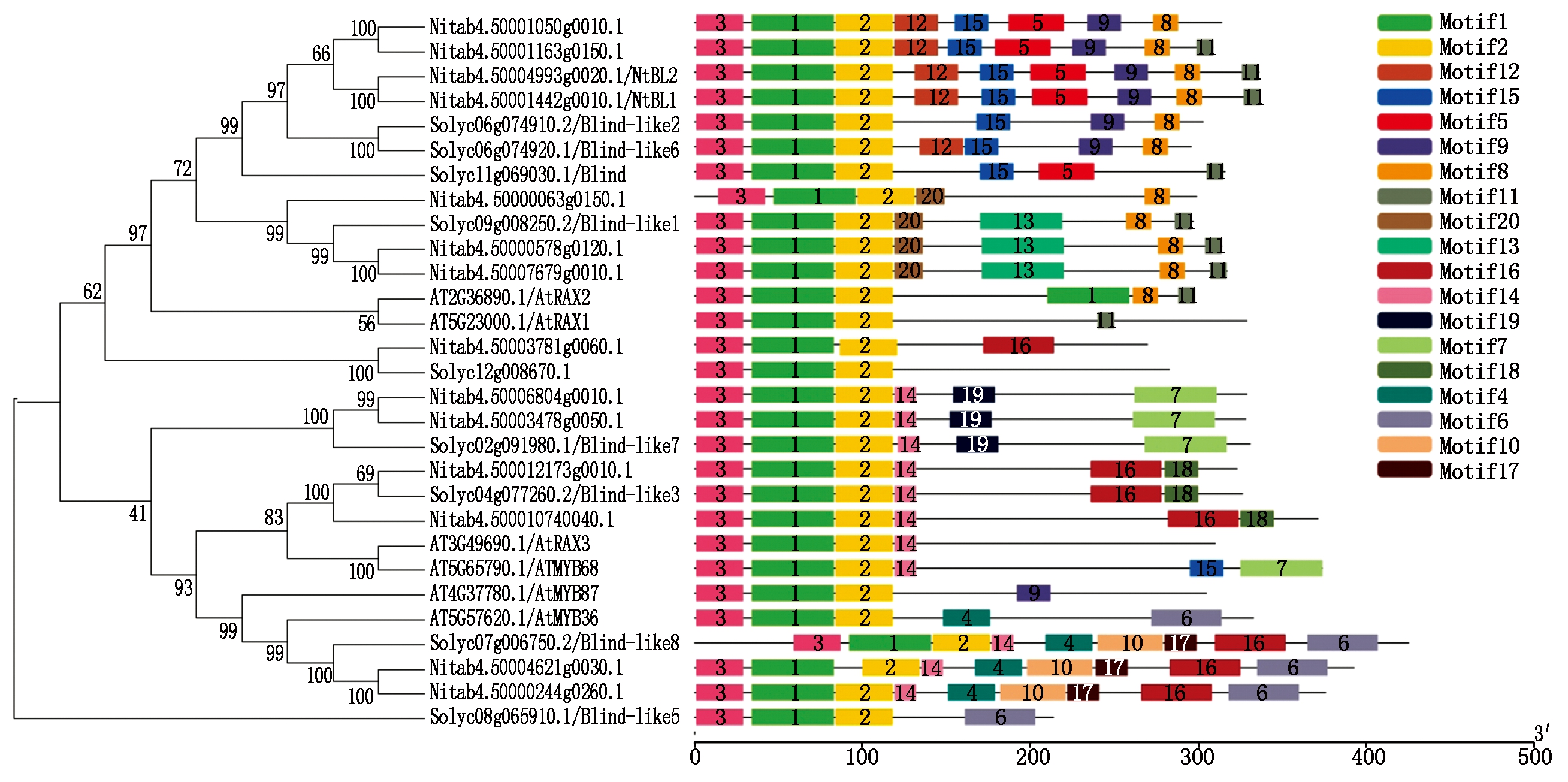
图6 NtBL1系统进化树和模体分析
Fig.6 The phylogenetic tree analysis and Motif prediction of NtBL1
2.4 非生物胁迫下NtBL1基因的表达分析
用qRT-PCR检测3种非生物胁迫处理下NtBL1基因的表达情况(图7),在ABA处理后,NtBL1基因的表达量在8 h时下调至对照的0.25倍,但是在随后的24 h内表达量随着时间延长而增加,最大值在24 h时出现,为对照的3.57倍。在NaCl处理后,NtBL1基因的表达量24 h内出现下调,最低值在24 h时出现,为对照的0.19倍。在PEG-6000处理后,NtBL1基因的表达量在24 h内与对照相比并未出现显著性差异。结果表明,NtBL1基因的表达受ABA处理的诱导,受NaCl处理的抑制,但是不受PEG-6000处理的影响。
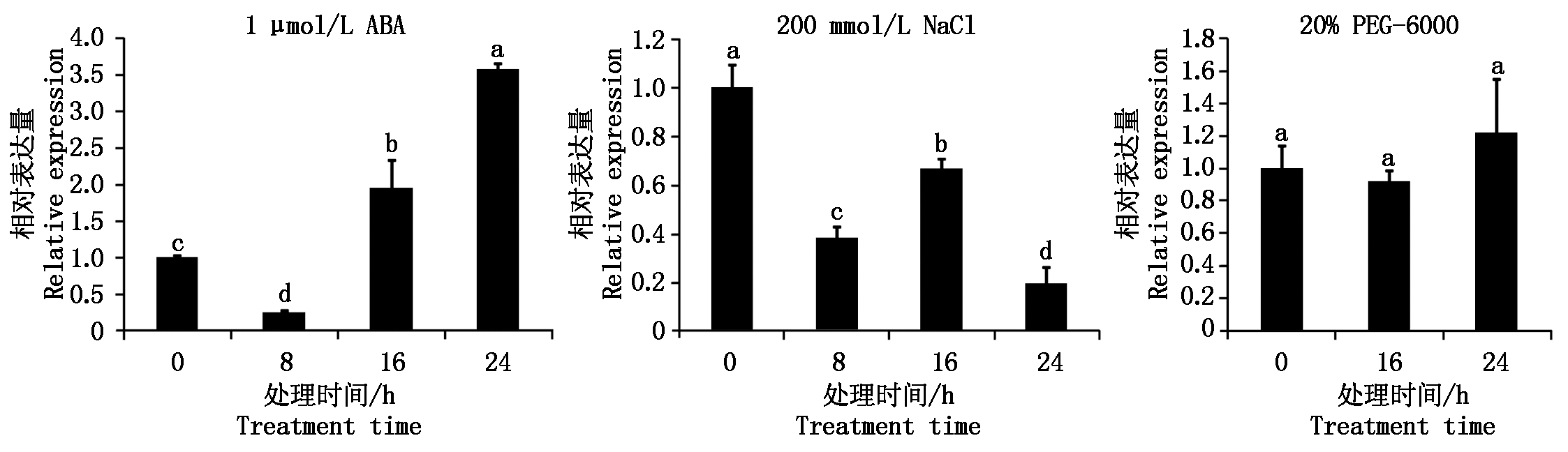
图7 NtBL1在3种非生物胁迫下的相对表达量分析
Fig.7 Analysis of relative expression of NtBL1 under three abiotic stresses
3 讨论
普通烟草是一种重要的叶用经济作物,为了收获高品质的烟叶制品,生产中通常采用人工打顶抹杈或者化学抑芽剂的方法去除腋芽,但是前者劳动强度大,后者对烟叶质量和环境均有不良影响,生物技术抑芽被公认为一条重要的探索途径,因此,对腋芽分化相关基因的克隆和功能研究极为重要。但是长期以来,普通烟草中相关遗传机制研究进展较慢。
本研究从普通烟草K326中克隆得到1个NtBL1基因的CDS序列,该基因全长1 014 bp,编码337个氨基酸。NtBL1蛋白与林烟草、绒毛状烟草、本生烟和渐狭叶烟草中的BL的同源性较高,说明其在烟草属中的进化相对保守。Blind基因在高等植物中普遍存在,且其N端具有高度保守的118个氨基酸残基,但是C端的序列却按结构和功能的不同而分别聚类[17],本研究中对番茄、拟南芥和烟草的Blind同源序列的蛋白结构分析验证了这种保守性以及不同进化分支在C端蛋白模体结构上的高度一致性。NtBL1蛋白前118个氨基酸序列与拟南芥和番茄中的Blind/RAX蛋白的同源性高达82.05%~92.31%,且与番茄Blind及其同源蛋白Blind-like2/C、Blind-like6有相似的蛋白模体结构和较近的进化距离,Blind和Blind-like2/C分别调控番茄的分枝发育和叶型结构,均参与到的分生组织的边界区域建立相关的通路中[17],预示着NtBL1基因可能也参与了分生组织边界区建立相关的进程。
本研究利用半定量PCR分析NtBL1在烟草苗期和成熟期不同组织中的表达水平,结果显示了其在多时期各部位均有表达,在根中的表达量最高,这与前人研究中拟南芥RAX2和RAX3的表达模式类似,但与番茄Blind的表达模式不完全相同,提示四倍体烟草基因组的复杂性,二倍体番茄和拟南芥中Blind/RAX基因在烟草中的同源基因可能存在多个,部分可能如番茄中Blind-like6基因一样,为无功能的假基因[17],系统进化树分析结果部分验证了这个猜想,与番茄Blind、Blind-like2、Blind-like6基因聚合在同一分支上的普通烟草Blind同源序列共计4个,其中包括NtBL1。在后续研究过程中,应该对烟草中剩余的13个Blind同源序列进行克隆,全面描述烟草Blind同源基因在腋芽分化过程中的功能。
部分与分支发育相关的基因同时参与植物非生物胁迫下的抗逆性,如AtRAX1/MYB37的超表达会影响一系列ABA应答相关基因的表达,同时超表达转化植株对外源ABA表现出超敏感表型,但是却能提高其耐旱性[23]。因此,本研究采用了3种非生物胁迫处理以研究NtBL1在胁迫处理下的应答,在不同的胁迫处理下,该基因的表达情况不同,推测NtBL1基因在不同的逆境条件下应答机制不同,同样仍不能排除NtBL1在对不同胁迫处理下是否存在翻译水平的不同响应,或者蛋白间的互作。因此,为了进一步明确NtBL1在腋芽分化和对抗非生物胁迫逆境的具体机制,预计构建NtBL1基因敲除和超表达载体,继而对林烟草和普通烟草进行遗传转化获得转基因植株,通过对转基因植株的遗传分析进一步明确NtBL1的功能。
[1] Chomicki G, Coiro M, Renner S S. Evolution and ecology of plant architecture: integrating insights from the fossil record, extant morphology, developmental genetics and phylogenies[J]. Annals of Botany, 2017, 120(6): 855-891.doi:10.1093/aob/mcx113.
[2] Wai A H, An G. Axillary meristem initiation and bud growth in rice[J]. Journal of Plant Biology, 2017, 60(5): 440-451. doi:10.1007/s12374-017-0088-x.
[3] Janssen B J, Drummond R S M, Snowden K C. Regulation of axillary shoot development[J]. Curr Opin Plant Biol, 2014, 17: 28-35. doi:10.1016/j.pbi.2013.11.004.
[4] Žádníková P, Simon R. How boundaries control plant development[J]. Current Opinion Plant Biology, 2014, 17: 116-125. doi:10.1016/j.pbi.2013.11.013.
[5] Long J, Barton M K. Initiation of axillary and floral meristems in Arabidopsis[J]. Development Biology, 2000, 218(2): 341-353. doi:10.1006/dbio.1999.9572.
[6] Schumacher K, Schmitt T, Rossberg M, Schmitz C, Theres K. The Lateral suppressor(Ls) gene of tomato encodes a new member of the VHIID protein family[J]. Proc Natl Acad Sci, 1999, 96(1): 290-295. doi:10.1073/pnas.96.1.290.
[7] Greb T, Clarenz O, Schāfer E, Müller D, Herrero R, Schmitz G, Theres K.Molecular analysis of the LATERAL SUPPRESSOR gene in Arabidopsis reveals a conserved control mechanism for axillary meristem formation[J]. Gene Dev, 2003, 17: 1175-1187. doi:10.1101/gad.260703.
[8] Zhang B, Liu X, Xu W N, Chang J Z, Li A, Mao X G, Zhang X Y, Jing R L. Novel function of a putative MOC1 ortholog associated with spikelet number per spike in common wheat[J]. Scientific Reports, 2015, 5: 12211. doi:10.1038/srep12211.
[9] Komatsu M, Maekawa M, Shimamoto K, Kyozuka J. The LAX1 and FRIZZY PANICLE2 genes determine the inflorescence architecture of rice by controlling rachis-branch and spikelet development[J]. Dev Biol, 2001, 231(2): 364-373. doi:10.1006/dbio.2000.9988.
[10] Ritter M K, Padilla C M, Schmidt R J. The maize mutant barren stalk1 is defective in axillary meristem development[J]. American Journal of Botany, 2002, 89(2): 203-210. doi:10.3732/ajb.89.2.203.
[11] Komatsu K, Maekawa M, Ujiie S, Satake Y, Furutani I, Okamoto H, Shimamoto K, Kyozuka J. LAX and SPA: major regulators of shoot branching in rice[J]. Proc Natl Acad Sci, 2003, 100(20): 11765-11770. doi:10.1073/pnas.1932414100.
[12] Gallavotti A, Zhao Q, Kyozuka J, Meeley R B, Ritter M K, Doebley J F, Pè E M, Schmit R J. The role of barren stalk1 in the architecture of maize[J]. Nature, 2004, 432(7017): 630-635. doi:10.1038/nature03148.
[13] Yang F, Wang Q, Schmitz G, Müller D, Theres K. The bHLH protein ROX acts in concert with RAX1 and LAS to modulate axillary meristem formation in Arabidopsis[J]. Plant J, 2012, 71(1): 61-70. doi:10.1111/j.1365-313X.2012.04970.x.
[14] Keller T, Abbott J,Moritz T, Doerner P. Arabidopsis REGULATOR OF AXILLARY MERISTEMS1 controls a leaf axilstem cell niche and modulates vegetative development[J]. The Plant Cell, 2006, 18: 598-611. doi:10.1105/tpc.105.038588.
[15] Schmitz G, Tillmann E, Carriero F, Fiore C, Cellini F, Theres K. The tomato blind gene encodes a MYB transcription factor that controls the formation of lateral meristems[J]. Proc Natl Acad Sci, 2002, 99(2): 1064-1069. doi:10.1073/pnas.022516199.
[16] Müller D, Schmitz G, Theres K. Blind homologous R2R3 Myb genes control the pattern of lateral meristem initiation in Arabidopsis[J]. The Plant Cell, 2006, 18(3): 586-597. doi:10.1105/tpc.105.038745.
[17] Busch B L, Schmitz G, Rossmann S, Piron F, Ding J, Bendahmane A, Theres K. Shoot branching and leaf dissection in tomato are regulated by homologous gene modules[J]. The Plant Cell, 2011, 23(10): 3595-3609. doi:10.1105/tpc.111.087981.
[18] 李文正, 高玉龙, 宋中邦, 李永平, 王丙武. 烟草NtMAX4-1基因的克隆及特征分析[J]. 基因组学与应用生物学, 2016, 35(11): 3147-3153. doi:10.13417/j.gab.035.003147.
Li W Z, Gao Y L, Song Z B, Li Y P, Wang B W. Cloning and characteristic analysis of NtMAX4-1 gene in tobacco[J]. Genomics and Applied Biology, 2016, 35(11): 3147-3153.
[19] 陈雅琼, 孙亭亭, 陈蕾, 丁安明, 陈涣, 孙玉合. 烟草BRANCHED1-Like基因的克隆及表达分析[J]. 植物遗传资源学报, 2015, 16(6): 1321-1329. doi:10.13430/j.cnki.jpgr.2015.06.026.
Chen Y Q, Sun T T, Chen L, Ding A M, Chen H, Sun Y H. Molecular cloning and expression analysis of BRANCHED1-Like gene in common tobacco[J]. Journal of Plant Genetic Resources, 2015, 16(6): 1321-1329.
[20] 陈雅琼. 烟草腋芽发育相关基因的克隆与功能分析[D]. 北京: 中国农业科学院, 2015.doi:10.7666/d.Y2787367.
Chen Y Q. Cloning and functional analysis of axillary bud development related genes in tobacco[D]. Beijing:Chinese Academy of Agricultral Sciences, 2015.
[21] 陈蕾. 烟草NtMAX3和NtMAX4基因克隆及RNA干扰研究[D]. 北京: 中国农业科学院,2016.
Chen L. Cloning and RNA interference of NtMAX3 and NtMAX4 gene in Nicotiana tabacum [D]. Beijing:Chinese Academy of Agricultral Sciences, 2016.
[22] 王姗姗, 杨军, 王中, 林福呈, 潘婷, 武明珠, 张剑锋, 曹培健, 谢小东. 利用CRISPR/Cas9技术的烟草NtLS基因敲除分析[J]. 烟草科技, 2018, 51(2):1-8. doi:10.16135/j.issn1002-0861.2017.0363.
Wang S S, Yang J, Wang Z, Lin F C, Pan T, Wu M Z, Zhang J F, Cao P J, Xie X D. Targeted mutagenesis of NtLS gene by using CRISPR/Cas9 system[J]. Tobacco Science & Technology, 2018, 51(2): 1-8.
[23] Yu Y T, Wu Z, Lu K, Bi C, Liang S, Wang X F, Zhang D P. Overexpression of the MYB37 transcription factor enhances abscisic acid sensitivity, and improves both drought tolerance and seed productivity in Arabidopsis thaliana[J]. Plant Mol Biol, 2016, 90(3): 267-279. doi:10.1007/s11103-015-0411-1.
