在自然界的各个小生境存在着多种真菌-细菌相互作用形成的真菌-细菌生物膜(Fungal-bacterial biofilm,FBB),其共同进化,发挥的作用超过任一单个成员,这些作用主要涉及动植物健康和发育、生物地球化学循环、生物技术和食品生产等[1],在农业生产领域则主要用于制备高效生物肥料接种剂、生防制剂等[2]。然而,制备FBB制剂耗时长,难以大规模生产。因此,非常有必要探讨FBB的形成机理。
真菌与细菌相互作用形成FBB包括2个步骤,即细菌通过趋化菌丝分泌物运动接近菌丝或到达丝际(Mycosphere),以及细菌和菌丝物理接触或附着[3-4]。生防菌荧光假单胞菌(Pseudomonas fluorescens)可在病原真菌尖刀镰孢菌(Fusarium oxysporum)菌丝表面定殖形成生物膜,并且该菌株对培养过尖刀镰孢菌的培养基、尖刀镰孢菌的次生代谢产物镰刀菌酸有趋化响应,细菌的趋化强度与镰刀菌酸浓度呈显著正相关。然而,该细菌的趋化缺失突变株在菌丝表面的定殖量显著降低,并且对培养过尖刀镰孢菌的培养基和镰刀菌酸无趋化响应。这些研究表明细菌在真菌菌丝表面的定殖与趋化有关,镰刀菌酸可能是细菌趋化菌丝的强趋化物[5]。草酸是大多数真菌的主要菌丝分泌物。真菌宿主分泌的草酸是食真菌的土壤细菌山冈单胞菌(Collimonas)及土壤中常见的FBB形成细菌土伯克霍尔德菌(Paraburkholderia terrae)趋化真菌菌丝的强趋化物,然而这些细菌对草酸的代谢利用能力较差[6-7]。土壤中真菌分泌的草酸对真菌具有多种生态功能,包括作为侵染致病毒力因子、降解木质素辅助因子及增加矿质元素的有效性和缓解重金属毒性等[8-9],因此,在许多真菌菌丝表面积聚有草酸钙结晶[7]。真菌菌丝分泌物中的其他物质是否是生物膜形成细菌的强趋化物尚未见报道。阐明细菌趋化真菌菌丝的强趋化物将有利于加快FBB制剂的生产。
双孢蘑菇(Agaricus bisporus)是世界上栽培范围最广、规模最大和生产量最高的一种人工栽培食用菌,由于色白味鲜,富含多种具有保健和药用功效的营养物质,其在国际市场中一直深受消费者喜爱[10]。同时,双孢蘑菇也是研究富含腐殖质的落叶层中真菌适应、定殖和生长的模式真菌[11]。双孢蘑菇有别于其他许多食药用菌的特征是,当培养料中的菌丝发满后,必须在培养料表面覆一层土才能出菇。在双孢蘑菇覆土中栖息着大量的细菌,优势菌群是恶臭假单胞菌(Pseudomonas putida)[12-13]。与土壤中其他细菌和真菌的相互作用相似,这些细菌也在双孢蘑菇菌丝表面形成菌膜,在双孢蘑菇菌丝表面同样积聚有大量的草酸钙结晶[14-15]。所以,双孢蘑菇-细菌生物膜是理想的FBB研究对象。然而,恶臭假单胞菌趋化双孢蘑菇菌丝的强趋化物还不清楚。
前期研究发现,双孢蘑菇的乙烯合成途径与高等植物相同,都经甲硫氨酸合成1-氨基环丙烷-1-羧酸(1-aminocyclopropane-1-carboxylate,ACC),ACC进而被氧化生成乙烯,合成途径中的关键酶是ACC合成酶和ACC氧化酶[16]。双孢蘑菇覆土出菇的机理是乙烯抑制双孢蘑菇原基的形成,覆土中的优势菌群假单胞菌能够产生ACC脱氨酶(ACC deaminase,AcdS),其降解乙烯合成中间代谢物ACC生成氨和α-酮丁酸,从而降低双孢蘑菇菌丝中乙烯的生物合成,进而解除乙烯对双孢蘑菇原基形成的抑制作用[17-20]。ACC是植物根际促生细菌(Plant growth promoting rhizobacteria,PGPR)趋化根际的强趋化物,PGPR在植物根际定殖,降解ACC,可有效降低植物乙烯的合成,解除逆境条件下植物合成的乙烯对植物的抑制作用[21]。因此,推测ACC可能也是恶臭假单胞菌趋化双孢蘑菇的强趋化物。为证实该假设,比较了恶臭假单胞菌UW4及其ACC脱氨酶缺失突变株UW4-AcdS-在双孢蘑菇菌丝表面的定殖情况及对双孢蘑菇菌丝主要分泌物的趋化特性,并对ACC引起的UW4趋化相关基因的表达量进行了转录组和定量RT-PCR分析,旨在进一步解析ACC在假单胞菌趋化双孢蘑菇菌丝中的作用机制,为开发新型高效生物膜肥料提供新的思路和方向。
1 材料和方法
1.1 试验材料
1.1.1 菌株 双孢蘑菇AS2796购自福建省农科院食用菌研究所;恶臭假单胞菌UW4及其ACC脱氨酶突变株UW4-AcdS-由加拿大滑铁卢大学Glick教授惠赠。
1.1.2 培养基和缓冲液 PDA培养基用于AS2796的活化和培养[17];TSB培养基用于细菌的活化和培养;胰蛋白胨水(TW)培养基(胰蛋白胨 10 g/L,NaCl 5 g/L)用于细菌的培养;甘油盐培养基(甘油 5 g (单独灭菌),K2HPO4 11.2 g,KH2PO4 4.8 g,(NH4)2SO4 2.0 g,MgSO4·7H2O 0.25 g,Fe2(SO4)3 0.5 g,超纯水 1 L)、细菌运动培养基(pH值7.0 磷酸缓冲液10-1 mol/L,EDTA 10-4 mol/L,琼脂糖 3 g,超纯水 1 L)和细菌趋化培养基(pH值7.0 磷酸缓冲液10-1 mol/L,EDTA 10-4 mol/L,琼脂糖 2 g,超纯水 1 L)用于细菌趋化试验[22]。趋化缓冲液(CMB)(pH值7.0 磷酸缓冲液10-1 mol/L,EDTA 10-4 mol/L)参考Adler[22]和de Weert[23]的方法配制,略有改动,主要用于细菌趋化试验。
1.1.3 主要试剂 ACC、柠檬酸和谷氨酰胺购自Sigma公司,氨基氧乙酸(AOA)购自阿拉丁公司。趋化试验所用其他试剂均购自国药集团化学试剂有限公司。
1.2 恶臭假单胞菌对双孢蘑菇菌丝生长速度的影响测定
将AS2796在PDA培养基中于25 ℃培养10 d,取菌丝边缘直径为1 cm的菌丝块,转接于PDA平板并添加不同浓度ACC合成酶抑制剂氨基氧乙酸(Aminooxyacetic acid,AOA)的PDA平板,培养6 d后,选取长势好、菌丝直径相同的平板用于UW4及其突变株接种试验。
将UW4和UW4-AcdS-分别接种于10 mL的TSB液体培养基中,28 ℃、220 r/min培养至OD600为1.9~2.0,取5 mL菌悬液,10 000 r/min离心1 min,再经5 mL灭菌超纯水洗涤3次后,用5 mL灭菌超纯水重悬菌体,用作UW4和UW4-AcdS-促生试验的菌悬液。
分别取制备好的UW4和UW4-AcdS-菌悬液10 μL均匀涂布在上述培养6 d的双孢蘑菇平板上,靠近菌丝边缘涂布,涂布圈直径大小约为1.5 cm,每个平板上涂布8个细菌圈,然后将平板置于25 ℃共培养,分别于共培养3,6,9 d时划线测量共培养菌丝直径。以涂布超纯水代替菌悬液的双孢蘑菇菌丝生长平板为对照(CK)。
1.3 恶臭假单胞菌在双孢蘑菇菌丝表面的定殖观察
待上述双孢蘑菇菌丝生长覆盖细菌涂布圈2/3时,挑取相互接合部位前端菌丝于显微镜下观察菌丝与细菌的结合情况。
待上述双孢蘑菇菌丝覆盖细菌涂布圈时,用5 mm打孔器取菌丝前端相互接合部位的菌丝-细菌块,经无菌水冲洗2次(3 min/次)。然后将菌丝-细菌块于3%的戊二醛溶液中4 ℃固定过夜后,用0.1 mol/L磷酸缓冲液漂洗3次(15 min/次),分别于30%,50%,70%,80%,90%,95%和100%的乙醇中梯度脱水,每梯度15 min,最后在乙酸异戊酯中置换15 min,经临界点干燥仪干燥并镀膜后,使用HITACHI S3400扫描电镜观察。
1.4 恶臭假单胞菌对趋化物质的趋化分析
将UW4及UW4-AcdS-接种于LB平板,于28 ℃培养2 d,挑单菌落接种于10 mL TW液体培养基中,28 ℃、150 r/min过夜培养。取1 mL菌液离心(4 ℃、8 000 r/min、5 min),弃上清,用1 mL无菌超纯水冲洗2次后悬浮于1 mL超纯水中,取200 μL接入250 mL甘油盐液体培养基中,于28 ℃、150 r/min培养至OD600约1.2,于4 ℃存放用于点滴趋化分析、毛细管趋化分析和游动平板分析。试验前用CMB缓冲液冲洗细菌并镜检,具有运动活性的细胞需大于90%。
点滴趋化分析:取上述250 mL菌悬液2瓶(500 mL),离心(4 ℃、8 000 r/min、5 min),去上清,用预冷CMB缓冲液20 mL冲洗2次后,悬浮于200 mL细菌趋化培养基中,混匀,分别取20 mL加入细菌趋化培养基平板进行趋化分析。试验采用点滴试验法[24],将10 μL浓度在500 mmol/L的趋化物质(谷氨酰胺、柠檬酸和ACC)加入平板中央,在2~6 h内观察趋化现象。
游动平板分析:取上述菌悬液15 mL,离心(4 ℃、8 000 r/min、5 min),去上清,用预冷CMB缓冲液10 mL冲洗3次后,取10 μL菌悬液加入含有3 mmol/L 各种趋化物质的细菌运动培养基(20 mL)平板中央,以不加趋化物质的运动培养基作对照。测定R值,即趋化物质的运动圈半径与对照的比值。
毛细管趋化分析:参照Adler[25]报道的方法进行。统计趋化组与对照组(CMB)中细菌数量的比值R,若R>1,表明趋化物质对细菌具有正向趋化效应,反之,则具有负趋化效应。
1.5 ACC处理的恶臭假单胞菌转录组测序
1.5.1 样品制备 将UW4单菌落接入10 mL TW液体培养基中,于28 ℃、150 r/min过夜培养,取1 mL菌液离心(4 ℃、8 000 r/min、5 min),用1 mL无菌超纯水冲洗2次后悬浮于1 mL超纯水中,取200 μL接种于250 mL甘油盐液体培养基中,于28 ℃、150 r/min培养至OD600约0.16时在细菌培养物中加入ACC(3 mmol/L),以UW4中加入等体积的无菌超纯水为对照,继续培养至OD600约1.2,离心收集菌体,用于转录组测序。
1.5.2 RNA的提取和检测 样品总RNA提取采用TRIzol法[26]。提取的总RNA经分光光度计和Agilent Bioanalyzer 2100电泳质检合格后,保存于-70 ℃备用。
1.5.3 cDNA文库构建和测序 首先从总RNA中纯化mRNA,按照RNeasy Micro kit操作流程纯化10 μg 总RNA,用带有Oligo(dT)的磁珠富集mRNA,所得产物用于测序文库构建。按照Encore Complete Prokaryotic RNA-Seq DR Multiplex Systems对纯化后的mRNA进行第一链cDNA合成、第二链cDNA合成,并采用Covaris S2系统超声打断cDNA,使目标DNA片段化的长度约300 bp,在End Repair Mix的作用下进行片段化DNA的末端修复,在3′末端添加poly(A)尾之后,连接用于测序的接头,富集cDNA,完成cDNA样本文库的构建。构建好的cDNA文库经Qubit®2.0 Fluorometer和FlashGel Dock系统质检合格后,使用Illumina测序平台进行测序(由上海伯豪生物技术公司完成)。
1.5.4 上机测序 按照cBot User Guide所示流程,使用IlluminaHiSeq 2000测序仪的cBot完成cluster生成和第一向测序引物杂交过程。按照HiSeq 2000 User Guide准备测序所需试剂,将带有cluster的flow cell上机测序。选用paired-end程序,进行paired end 2×50 nt multiplex测序。测序整个过程由Illumina提供的data collection software进行控制,进行实时数据监控分析。
1.5.5 数据分析 数据预处理:应用fastx(version:0.0.13)进行clean reads处理,包括去除质量大于20 bp所占比例小于50%的reads、去除长度小于20 bp的reads、去除仪器系统误差造成的准确度不高的reads、去除reads中含有的模糊的N碱基、去除3′端质量Q低于10 bp的碱基、去除reads中的接头序列、去除rRNA reads。应用bowtie 2 (version: 2-2.0.5)采用局部比对预处理后的reads进行Genome mapping,采用的基因组序列来自NCBI(Pseudomonas_putida_UW4_uid182733),下载地址为:ftp://ftp.ncbi.nlm.nih.gov/genomes/Bacteria/Pseudomonas_putida_UW4_uid182733。
基因表达分析:应用BEDTools (version:2.16.1)对bowtie 2的mapping结果进行基因覆盖reads定量,利用perl脚本进行基因定量。定量标准化公式如下:
其中,RPKM为Reads per kilobase of exon model per million mapped reads,transcription reads为覆盖整个gene exon的reads数目;transcription length为gene exon总长度;total mapped reads in run为该样本所有mapped genome的总reads数目。
差异表达基因分析挑选条件:①P-value<0.05;② log2(RPKM fold change)≥1。
1.6 恶臭假单胞菌趋化相关基因的荧光定量PCR(qRT-PCR)分析
用于qRT-PCR的UW4和UW4-AcdS-菌株的总RNA提取和cDNA样品制备参考转录组测序中相应的方法进行,cDNA合成采用cDNA mixture(TaKaRa,大连)。按照TB GreenTM Fast qPCR Mix(TaKaRa,大连)使用20 μL反应体系,参照qRT-PCR的方法进行PCR扩增[27]。qRT-PCR引物序列见表1。qRT-PCR反应条件:95 ℃ 10 min;95 ℃ 15 s,60 ℃ 15 s,72 ℃ 35 s,循环40次;95 ℃ 15 s,60 ℃ 30 s,60 ℃升温至95 ℃,20 min;95 ℃ 15 s;10 ℃ 1 min。以rpsl为内参基因,按照2-ΔΔCT方法对目标基因的表达水平进行相对定量。
表1 荧光定量PCR引物
Tab.1 The primer sets used for quantative Real time PCR
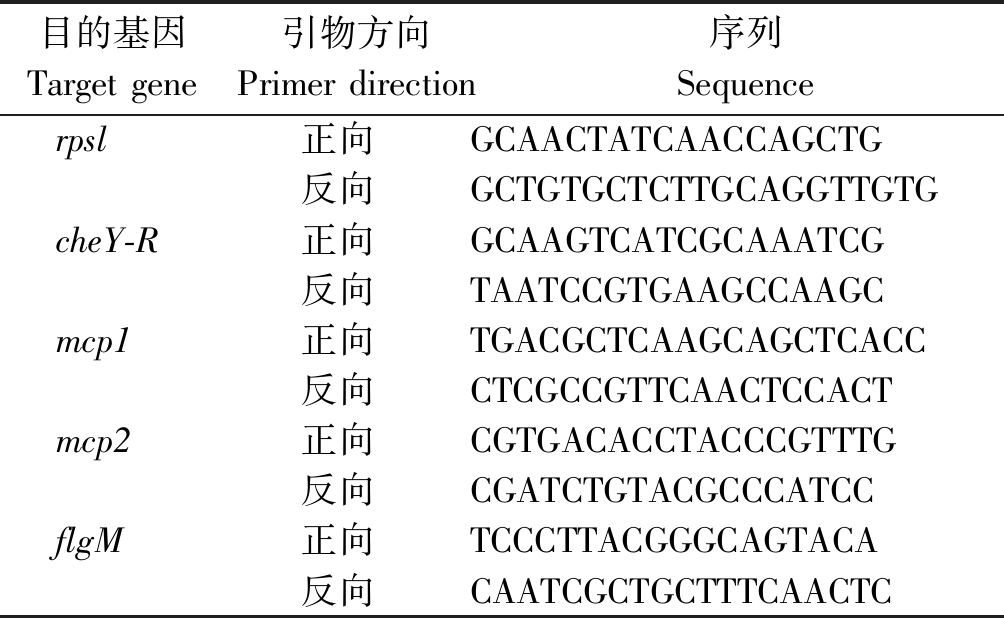
目的基因Target gene引物方向Primer direction序列Sequencerpsl正向GCAACTATCAACCAGCTG反向GCTGTGCTCTTGCAGGTTGTGcheY-R正向GCAAGTCATCGCAAATCG反向TAATCCGTGAAGCCAAGCmcp1正向TGACGCTCAAGCAGCTCACC反向CTCGCCGTTCAACTCCACTmcp2正向CGTGACACCTACCCGTTTG反向CGATCTGTACGCCCATCCflgM正向TCCCTTACGGGCAGTACA反向CAATCGCTGCTTTCAACTC
1.7 数据分析
所有数据来自至少3个独立重复试验,均使用GraphPad Prism 7计算其平均值和标准偏差(SD),数值=平均值±s,方差分析使用One-way ANOVA,P<0.05被认为具有统计学显著性。
2 结果与分析
2.1 恶臭假单胞菌对双孢蘑菇菌丝生长的影响
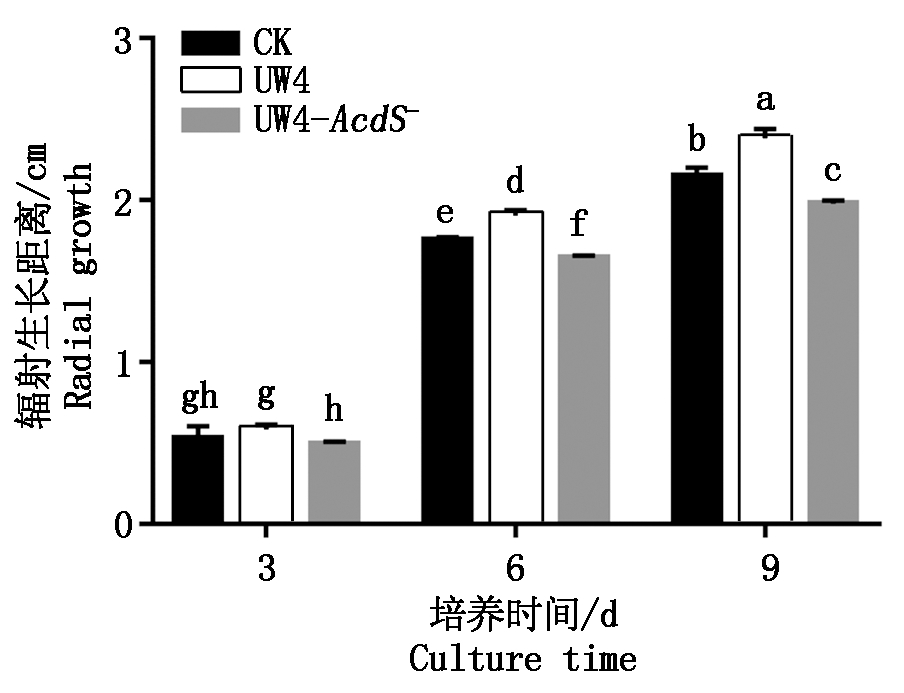
在PDA平板培养6 d的双孢蘑菇菌丝边缘涂布UW4及UW4-AcdS-的菌悬液进行共培养,比较细菌对菌丝生长的影响。在涂布菌悬液6 d和9 d时,涂布UW4菌悬液的双孢蘑菇菌丝生长距离显著大于涂布超纯水的双孢蘑菇菌丝对照组,其中9 d时,涂布UW4菌悬液的双孢蘑菇菌丝生长距离与对照组相比增加了10.85%。相反,涂布UW4-AcdS-菌悬液的双孢蘑菇菌丝生长距离则显著小于对照组,在9 d时的生长距离为对照组的92.09%(图1)。表明UW4对双孢蘑菇菌丝生长有促进作用,而UW4-AcdS-对双孢蘑菇菌丝生长则有显著的抑制作用。
2.2 恶臭假单胞菌在双孢蘑菇菌丝表面的定殖情况
为探索UW4对双孢蘑菇菌丝生长的促进机理,首先使用显微镜观察UW4是否在双孢蘑菇菌丝表面定殖。挑取PDA平板中与UW4共培养的双孢蘑菇菌丝进行显微观察,结果显示,UW4能够在双孢蘑菇菌丝表面形成一层具有一定厚度的菌膜(图2)。当在PDA中添加200 μg/mL或400 μg/mL的AOA抑制双孢蘑菇菌丝ACC合成酶活性时,UW4在双孢蘑菇菌丝表面定殖量减少,不能形成菌膜(图2)。与此结果相似,当使用UW4-AcdS-突变株时,双孢蘑菇菌丝表面也不能形成菌膜,且菌丝表面的细菌数量也较少(图2)。
不同小写字母表示差异达到显著水平(P<0.05)。图5同。 Different small letters indicate significant differences at P<0.05.The same as Fig.5.
图1 恶臭假单胞菌对PDA平板培养 双孢蘑菇菌丝生长的影响
Fig.1 The influences of Pseudomonas putida on mycelial growth of Agaricus bisporus grew on PDA plates

图2 恶臭假单胞菌在双孢蘑菇菌丝表面定殖的显微观察(10×100)
Fig.2 The microscopic examination of the Agaricus bisporus mycelia colonized by Pseudomonas putida
为进一步确认UW4在双孢蘑菇菌丝表面的定殖情况,使用电镜对UW4在双孢蘑菇菌丝表面的定殖情况进行了观察。UW4菌体在菌丝表面大量定殖形成清晰的菌膜。与此相反,UW4-AcdS-在双孢蘑菇菌丝表面几乎没有定殖(图3)。
2.3 恶臭假单胞菌UW4对几种双孢蘑菇菌丝分泌物的趋化反应
双孢蘑菇菌丝可分泌多种氨基酸,其中对恶臭假单胞菌有较强趋化作用的是谷氨酰胺和脯氨酸等[28],对恶臭假单胞菌有较强趋化作用的有机酸有柠檬酸、琥珀酸和苹果酸等[29]。因此,选择了谷氨酰胺、柠檬酸和ACC作为趋化物,测定了UW4和UW4-AcdS-对这3种物质的趋化反应。点滴趋化分析和游动平板分析结果均显示,UW4和UW4-AcdS-均能够趋化谷氨酰胺和柠檬酸,产生相似的趋化反应,然而,UW4可对ACC产生趋化,而UW4-AcdS-则不趋化ACC(图4、表2)。
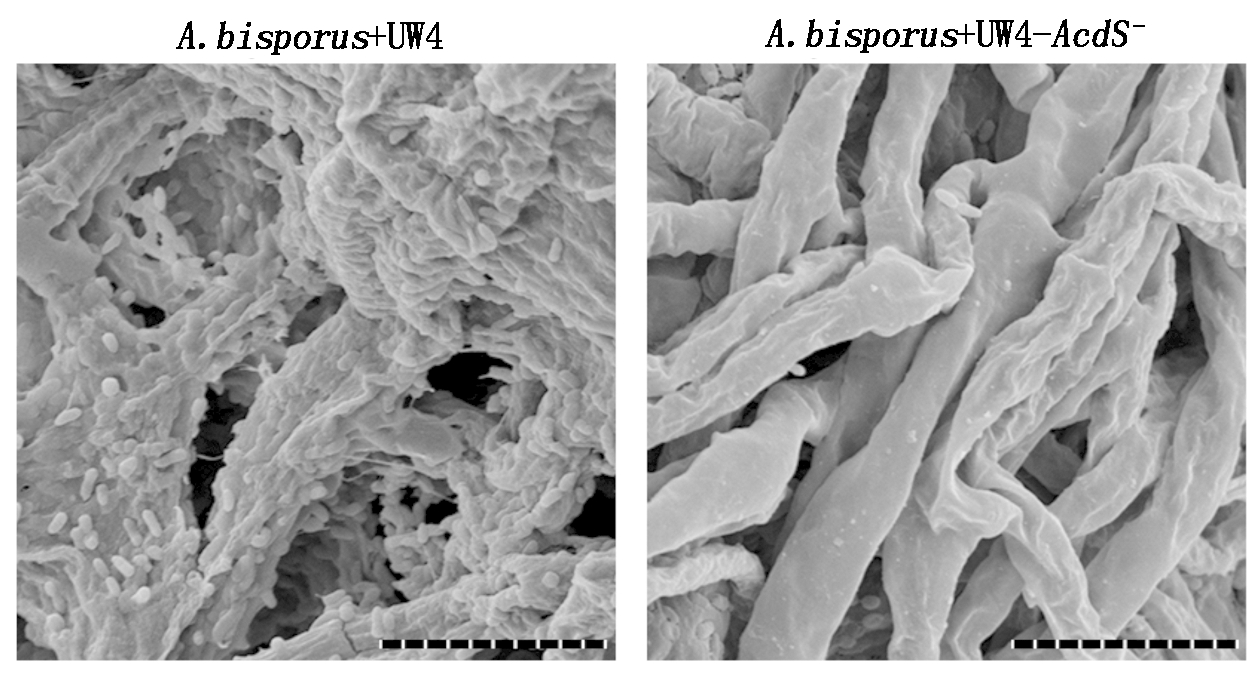
图3 恶臭假单胞菌在双孢蘑菇菌丝表面定殖 的扫描电镜观察(Bar=10 μm)
Fig.3 The scanning electron micrograph of the Agaricus bisporus mycelia colonized by Pseudomonas putida

图4 恶臭假单胞菌UW4和UW4-AcdS-对ACC等物质的趋化反应
Fig.4 The chemotactic response of Pseudomonas putida UW4 and UW4-AcdS- to ACC and other substances
表2 恶臭假单胞菌UW4和UW4-AcdS-对ACC等物质的趋化能力分析
Tab.2 Chemotactic ability of Pseudomonas putida UW4 and UW4-AcdS- to ACC and other substances

菌株Strain处理Treatment趋化圈半径/mmChemotaxis circle radiusR值R value是否正趋化Positive chemotaxis or notUW4CMB(CK)8.8±1.2b1.00柠檬酸15.8±1.9a1.80是1-氨基环丙烷-1-羧酸16.9±1.7a1.93是谷氨酰胺17.4±1.2a1.99是UW4-AcdS-CMB(CK)8.2±0.9b1.00柠檬酸15.2±2.3a1.85是1-氨基环丙烷-1-羧酸7.9±1.0b0.96否谷氨酰胺16.9±1.6a2.06是
注:R值为不同处理与对照的趋化圈半径之比。不同小写字母表示数据之间存在显著差异(P<0.05)。表3同。
Note: The R value is the ratio of chemotaxis radius between treatments and CK.The different letters represent the significance difference(P<0.05).The same as Tab.3.
在点滴趋化分析和游动平板分析的基础上,采用毛细管趋化分析法测定了ACC对UW4的趋化能力和最适趋化浓度。表3结果显示,ACC、柠檬酸和谷氨酰胺均可以诱导UW4产生正趋化效应。其中柠檬酸和谷氨酰胺的最适趋化浓度分别在10-5 mol/L和10-3~10-5 mol/L。与谷氨酰胺类似,ACC可以在各浓度下诱导UW4产生正趋化效应,在5×10-3 mol/L的浓度下,平板中的细菌数最多,达到28.4×103 cfu/mL(表3),表明ACC对UW4的最适趋化浓度在10-3 mol/L左右。ACC在5×10-3 mol/L时与对照相比的R值达到7.28,显著高于柠檬酸和谷氨酰胺在各浓度下的R值。这些结果表明,与柠檬酸和谷氨酰胺类似,ACC也是诱导恶臭假单胞菌趋化的一种强趋化物。
表3 恶臭假单胞菌UW4对ACC等物质的趋化浓度分析
Tab.3 The concentration analysis of Pseudomonas putida UW4 to ACC and other substances
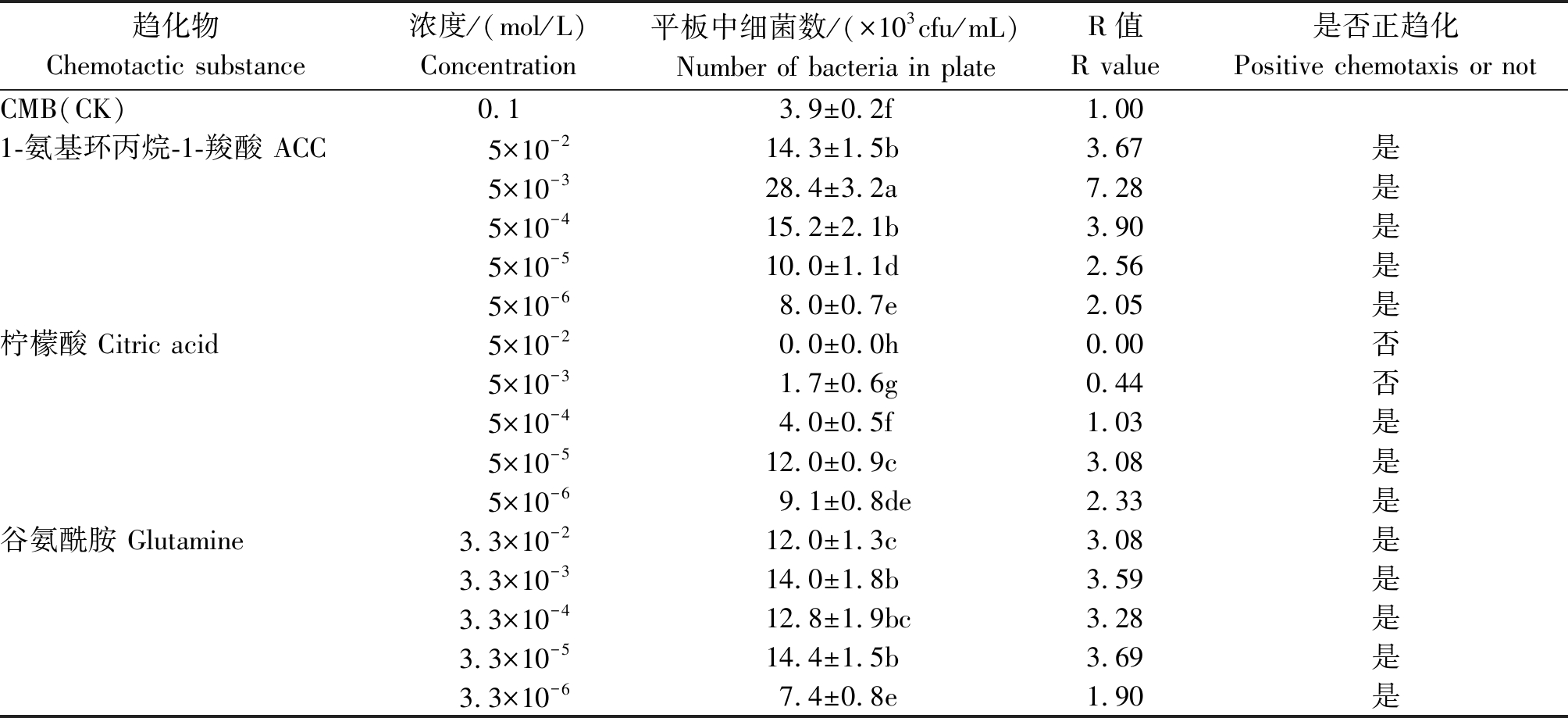
趋化物Chemotactic substance浓度/(mol/L)Concentration平板中细菌数/(×103cfu/mL)Number of bacteria in plate R值R value是否正趋化Positive chemotaxis or notCMB(CK)0.1 3.9±0.2f1.001-氨基环丙烷-1-羧酸 ACC5×10-214.3±1.5b3.67是5×10-328.4±3.2a7.28是5×10-415.2±2.1b3.90是5×10-510.0±1.1d2.56是5×10-68.0±0.7e2.05是柠檬酸 Citric acid5×10-20.0±0.0h0.00否5×10-31.7±0.6g0.44否5×10-44.0±0.5f1.03是5×10-512.0±0.9c3.08是5×10-69.1±0.8de 2.33是谷氨酰胺 Glutamine3.3×10-212.0±1.3c3.08是3.3×10-314.0±1.8b3.59是3.3×10-412.8±1.9bc3.28是3.3×10-514.4±1.5b3.69是3.3×10-67.4±0.8e1.90是
注:R值为趋化物处理中的细菌数与对照中细菌数之比。
Note: The R value is the ratio between the number of bacteria in chemoattractant treatments and in CK.
2.4 恶臭假单胞菌UW4对ACC产生趋化的差异表达基因分析
以UW4为对照组,以ACC处理的UW4为处理组,使用转录组测序分析方法筛选UW4对ACC产生趋化且与运动相关的差异表达基因。与对照相比,UW4经ACC处理后,筛选到5个上调趋化运动相关基因和23个下调趋化运动相关基因。其中,趋化信号传导基因cheY家族相关基因(14051552)呈现上调表达,而cheY家族相关基因(14051602)呈现下调表达。同时,与趋化运动相关的鞭毛组装基因(14055599和14054890)也呈现上调趋势。而下调基因多为趋化信号受体蛋白(甲基趋化性受体蛋白、甲基受体趋化感应传导蛋白和趋化感应传导蛋白)等相关基因,表明这些基因可能不是ACC趋化受体(表4)。
2.5 恶臭假单胞菌UW4与趋化运动相关基因表达的qRT-PCR分析
为进一步确认UW4中与趋化运动相关的关键基因的表达情况,根据转录组测序结果,使用qRT-PCR的方法对与趋化运动相关的关键基因的表达进行了验证。图5结果显示,与对照相比,ACC处理的UW4中与响应调节蛋白(cheY)相关的cheY-R基因(14051552)的表达量显著上调了6.90倍;鞭毛组装相关基因flgM(14055599)的表达量与对照相比也显著上调了6.38倍。然而,ACC处理的UW4中甲基趋化受体蛋白基因mcp1(14055832)和mcp2(14051596)的表达量分别是对照的0.29倍和0.25倍。同时,UW4-AcdS-中这些基因的表达量与对照相比都未发生明显变化(图5)。这些结果与转录组测序结果相符,表明ACC处理会诱导UW4中与趋化运动相关的基因表达量发生上升或下调变化,从而改变细菌的运动方向。
3 结论与讨论
自2003年首次通过人工培养得到植物根际促生细菌根瘤菌和土壤中常见丝状真菌曲霉形成的FBB制剂以来[30],由于FBB在固氮、溶磷、根际定殖、生防等方面表现出比其单一成员更优越的特性,在生物肥料研发领域受到极大地重视,发展迅猛[31]。能够形成FBB的丝状真菌除曲霉外,还有多种,包括青霉(Penicillium)、木霉(Trichoderma)、离褶伞(Lyophyllum)、外生菌根真菌双色蜡蘑(Laccaria bicolor)、疫霉(Phytophthora)、腐霉(Pythium)和蕈菌中的糙皮侧耳(Pleurotus ostreatus)等[2, 32]。在FBB中,真菌菌丝网络能够为细菌提供营养和运动载体[33-34],使得细菌能够沿着真菌菌丝高速公路进行远距离迁移[35],而细菌则能保护真菌免受抗真菌抗生素的作用[36-37]。另外,形成生物膜的细菌和真菌的代谢活动与游离的细胞相比也发生了较大的变化,二者相对抗的代谢活动减弱[38],碳氮代谢加强[39]。
表4 恶臭假单胞菌UW4响应ACC处理 的趋化运动相关的差异表达基因
Tab.4 The differentially expressed genes related with chemotaxis and movement responding to ACC treatment in Pseudomonas putida UW4
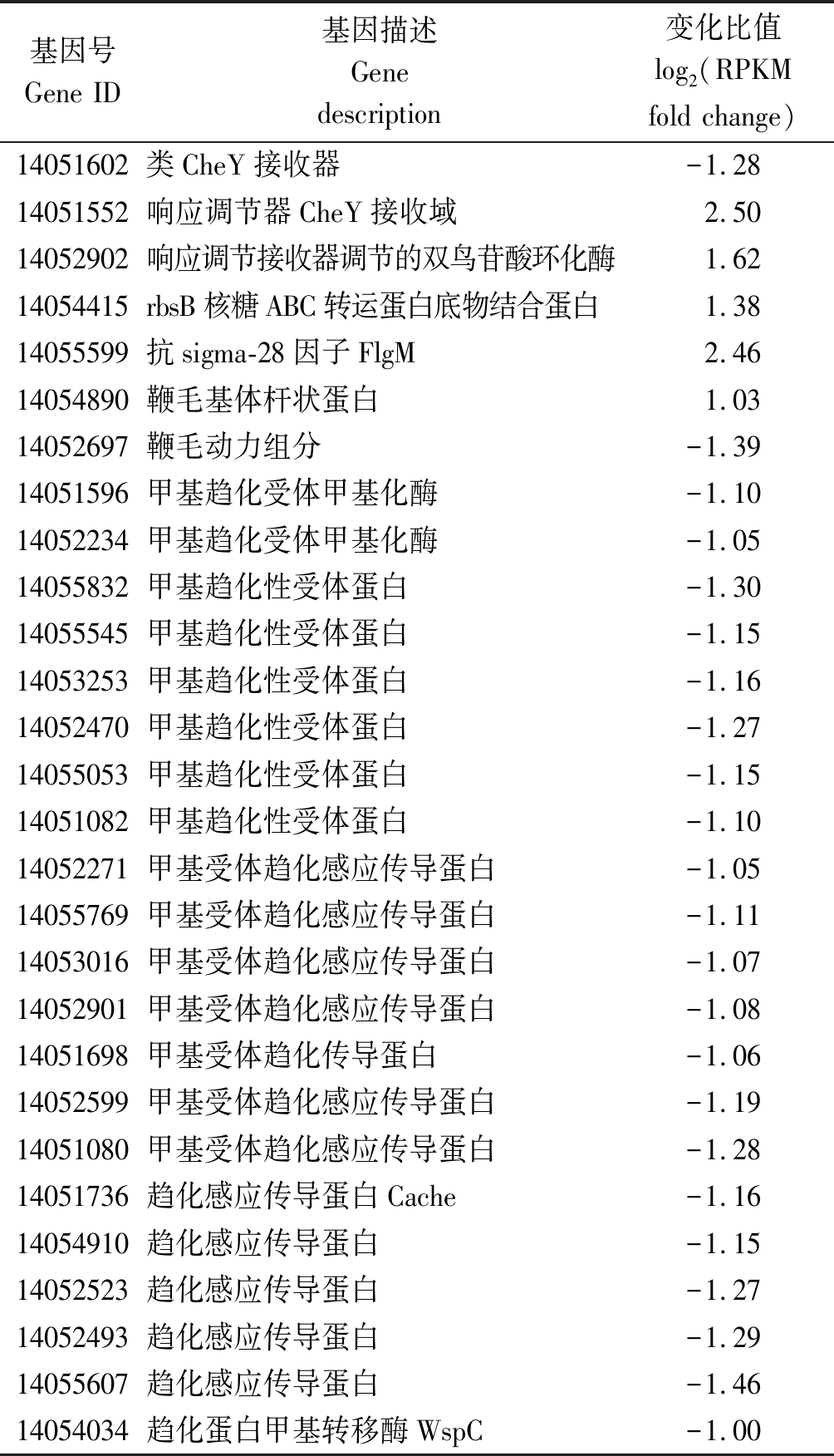
基因号Gene ID基因描述Gene description变化比值log2(RPKM fold change)14051602类CheY接收器-1.2814051552响应调节器CheY接收域2.5014052902响应调节接收器调节的双鸟苷酸环化酶1.6214054415rbsB核糖ABC转运蛋白底物结合蛋白1.3814055599抗sigma-28因子FlgM2.4614054890鞭毛基体杆状蛋白1.0314052697鞭毛动力组分-1.3914051596甲基趋化受体甲基化酶-1.1014052234甲基趋化受体甲基化酶-1.0514055832甲基趋化性受体蛋白-1.3014055545甲基趋化性受体蛋白-1.1514053253甲基趋化性受体蛋白-1.1614052470甲基趋化性受体蛋白-1.2714055053甲基趋化性受体蛋白-1.1514051082甲基趋化性受体蛋白-1.1014052271甲基受体趋化感应传导蛋白-1.0514055769甲基受体趋化感应传导蛋白-1.1114053016甲基受体趋化感应传导蛋白-1.0714052901甲基受体趋化感应传导蛋白-1.0814051698甲基受体趋化传导蛋白-1.0614052599甲基受体趋化感应传导蛋白-1.1914051080甲基受体趋化感应传导蛋白-1.2814051736趋化感应传导蛋白Cache-1.1614054910趋化感应传导蛋白-1.1514052523趋化感应传导蛋白-1.2714052493趋化感应传导蛋白-1.2914055607趋化感应传导蛋白-1.4614054034趋化蛋白甲基转移酶WspC-1.00
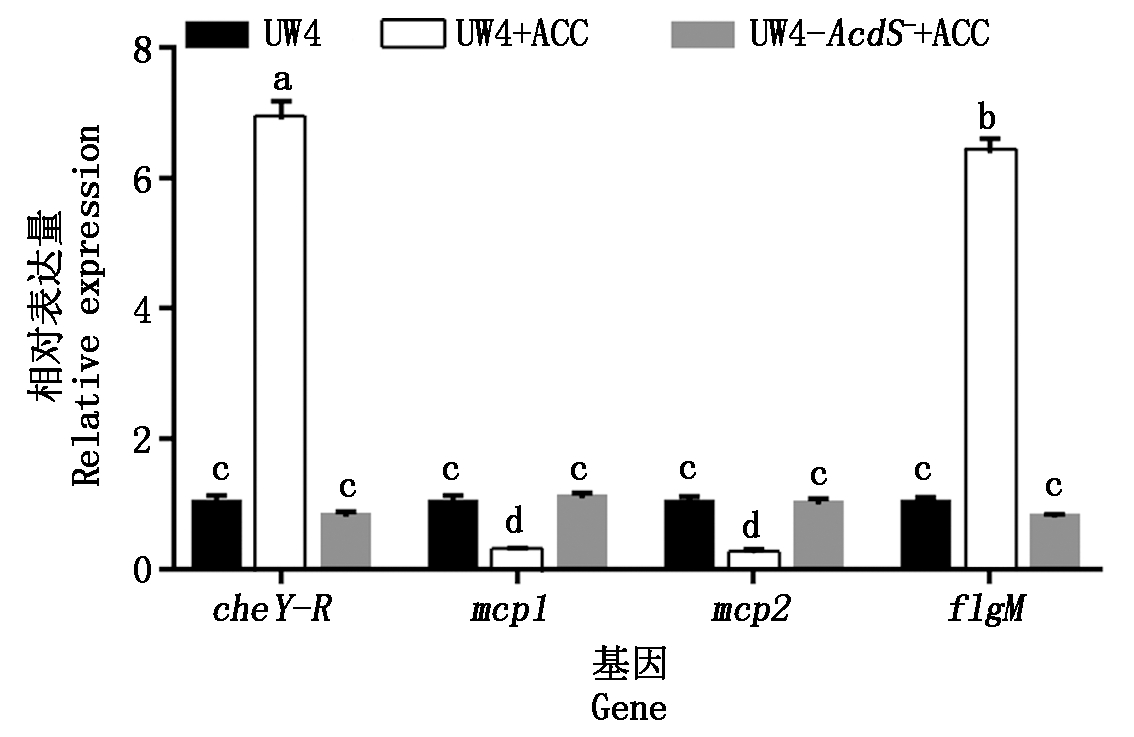
图5 恶臭假单胞菌UW4响应ACC诱导的 趋化运动相关基因表达的qRT-PCR分析
Fig.5 The analysis of chemotaxis- and movement-related genes responding to ACC treatment in Pseudomonas putida UW4 by qRT-PCR
PGPR识别真菌菌丝分泌物作为趋化物,通过趋化达到丝际,是形成FBB的关键第一步。厘清PGPR趋化丝际的关键趋化物,将为高效构建FBB提供理论依据。尽管荧光假单胞菌对尖刀镰孢菌分泌的真菌毒素镰刀菌酸表现出强的趋化性,然而,在镰刀菌酸合成量很少的尖刀镰孢菌突变株菌丝表面定殖的荧光假单胞菌的数量与其尖刀镰孢菌亲株没有差异,表明镰刀菌酸并不是荧光假单胞菌趋化菌丝的唯一菌丝分泌物[5]。草酸是植物根系和真菌菌丝常见的分泌物,许多PGPR能够趋化草酸,但趋化强度明显弱于苹果酸和柠檬酸等有机酸[21]。因此,PGPR趋化真菌菌丝分泌物形成FBB的强趋化物不是真菌毒素和草酸。
PGPR除具有能够在根际定殖的共同特征外,另一个重要的特征是绝大多数PGPR产AcdS,AcdS催化ACC裂解为氨和α-酮丁酸,使得PGPR能够利用ACC作为碳源和氮源。已发现的能够合成并分泌ACC的真菌有橘青霉(Penicillium citrinum)[40]和双孢蘑菇[16],但从许多真菌的基因组中发现有ACC合成酶基因,因此,合成并分泌ACC可能普遍存在于真菌中。本研究发现,恶臭假单胞菌UW4能够趋化ACC,趋化强度超过双孢蘑菇菌丝分泌物中的主要氨基酸和有机酸。当使用AOA抑制双孢蘑菇菌丝ACC合成酶活性,菌丝表面定殖的恶臭假单胞菌数量显著减少。恶臭假单胞菌ACC脱氨酶基因突变株丧失了对ACC的趋化,其在双孢蘑菇菌丝表面的定殖量同样显著减少。ACC可诱导恶臭假单胞菌趋化蛋白和少数趋化受体蛋白的高表达及大部分趋化受体蛋白的下调表达。因此,ACC可能是恶臭假单胞菌趋化双孢蘑菇菌丝的强趋化物或关键趋化物。
双孢蘑菇菌渣是良好的有机肥源[41],其覆土中形成的丰富的FBB也将成为FBB接种剂的一个简便高效的来源。ACC在假单胞菌趋化双孢蘑菇菌丝中作用机制的解析将为开发新型高效生物膜肥料提供新的思路和方向。
[1] Deveau A, Bonito G, Uehling J, Paoletti M, Becker M, Bindschedler S, Hacquard S, Hervé V, Labbé J, Lastovetsky O A, Mieszkin S, Millet L J, Vajna B, Junier P, Bonfante P, Krom B P, Olsson S, van Elsas J D, Wick L Y. Bacterial fungal interactions: ecology, mechanisms and challenges[J]. FEMS Microbiology Reviews, 2018, 42(3): 335-352. doi:10.1093/femsre/fuy008.
[2] Velmourougane K, Prasanna R, Saxena A K. Agriculturally important microbial biofilms: present status and future prospects[J]. Journal of Basic Microbiology, 2017, 57(7): 548-573. doi:10.1002/jobm.201700046.
[3] Haq I U, Zhang M Z, Yang P, van Elsas J D. Chapter five-the interactions of bacteria with fungi in soil: emerging concepts[J]. Advances in Applied Microbiology, 2014, 89: 185-215. doi:10.1016/B978-0-12-800259-9.00005-6.
[4] Haq I U, Calixto R O, Yang P, dos Santos G M P, Barreto-Bergter E, van Elsas J D. Chemotaxis and adherence to fungal surfaces are key components of the behavioral response of Burkholderia terrae BS001 to two selected soil fungi[J]. FEMS Microbiology Ecology, 2016, 92(11): 164. doi:10.1093/femsec/fiw164.
[5] de Weert S, Kuiper I, Lagendijk E L, Lamers G E, Lugtenberg B J. Role of chemotaxis toward fusaric acid in colonization of hyphae of Fusarium oxysporum f. sp. radicis-lycopersici by Pseudomonas fluorescens WCS365[J]. Molecular Plant-Microbe Interactions, 2004, 17(11): 1185-1191. doi:10.1094/MPMI.2004.17.11.1185.
[6] Haq I U, Zwahlen R D, Yang P, van Elsas J D. The response of Paraburkholderia terrae strains to two soil fungi and the potential role of oxalate[J]. Frontiers in Microbiology, 2018, 9: 989.doi: 10.3389/fmicb.2018.00989.
[7] Rudnick M B, van Veen J A, de Boer W. Oxalic acid: a signal molecule for fungus-feeding bacteria of the genus Collimonas? [J]. Environmental Microbiology Reports, 2015, 7(5): 709-714. doi:10.1111/1758-2229.12290.
[8] Dutton M V, Evans C S. Oxalate production by fungi: its role in pathogenicity and ecology in the soil environment[J]. Canadian Journal of Microbiology, 1996, 42(9): 881-895. doi:10.1139/m96-114.
[9] Heller A, Witt-Geiges T. Oxalic acid has an additional, detoxifying function in Sclerotinia sclerotiorum pathogenesis[J]. PLoS One, 2013, 8(8): e72292. doi:10.1371/journal.pone.0072292.
[10] 张亭, 韩建东, 李瑾, 任海霞, 任鹏飞, 宫志远. 金针菇菌渣提取液对双孢蘑菇蛋白质营养价值的影响[J]. 河南农业科学, 2016, 45(9):94-97.doi:10.15933/j.cnki.1004-3268.2016.09.019.
Zhang T, Han J D, Li J, Ren H X, Ren P F, Gong Z Y. Effects of Flammulina velutipes residue extracts on protein nutritional value of Agaricus bisporus[J]. Journal of Henan Agricultural Sciences, 2016, 45(9):94-97.
[11] Morin E, Kohler A, Baker A R, Foulongne-Oriol M, Lombard V, Nagy L G, Ohm R A, Patyshakuliyeva A, Brun A, Aerts A L, Bailey A M, Billette C, Coutinho P M, Deakin G, Doddapaneni H, Floudas D, Grimwood J, Hildén K, Kües U, Labutti K M, Lapidus A, Lindquist E A, Lucas S M, Murat C, Riley R W, Salamov A A, Schmutz J, Subramanian V, Wösten H A B, Xu J P, Eastwood D C, Foster G D, Sonnenberg A S, Cullen D, de Vries R P, Lundell T, Hibbett D S, Henrissat B, Burton K S, Kerrigan R W, Challen M P, Grigoriev I V, Martin F. Genome sequence of the button mushroom Agaricus bisporus reveals mechanisms governing adaptation to a humic-rich ecological niche[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(43): 17501-17506. doi:10.1073/pnas.1206847109.
[12] Nair N G, Hayes W A. Some effects of casing soil amendments on mushroom cropping[J]. Australian Journal of Agricultural Research, 1975, 26(1): 181-188.doi:10.1071/ar9750181.
[13] Rainey P B, Cole A L J, Fermor T R, Wood D A. A model system for examining involvement of bacteria in basidiome initiation of Agaricus bisporus[J]. Mycological Research, 1990, 94(2): 191-195.doi:10.1016/S0953-7562(09)80612-6.
[14] Masaphy S, Levanon D, Tchelet R, Henis Y. Scanning electron microscope studies of interactions between Agaricus bisporus (Lang) Sing hyphae and bacteria in casing soil[J]. Applied and Environmental Microbiology, 1987, 53(5): 1132-1137.
[15] Cochet N, Gillman A, Lebeault J M. Some biological characteristics of the casing soil and their effect during Agaricus bisporus fructification[J]. Acta Biotechnologica, 1992, 12(5): 411-419. doi:10.1002/abio.370120510.
[16] Zhang C H, Huang T, Shen C H, Wang X T, Qi Y C, Shen J W, Song A D, Qiu L Y, Ai Y C. Downregulation of ethylene production increases mycelial growth and primordia formation in the button culinary-medicinal mushroom, Agaricus bisporus (Agaricomycetes)[J]. International Journal of Medicinal Mushrooms, 2016, 18(12): 1131-1140. doi:10.1615/IntJMedMushrooms.v18.i12.80.
[17] Chen S C, Qiu C W, Huang T, Zhou W W, Qi Y C, Gao Y Q, Shen J W, Qiu L Y. Effect of 1-aminocyclopropane-1-carboxylic acid deaminase producing bacteria on the hyphal growth and primordium initiation of Agaricus bisporus[J]. Fungal Ecology, 2013, 6(1): 110-118. doi:10.1016/j.funeco.2012.08.003.
[18] 程雁,王景冒,张岩,杨潜龙,邱立友. 应用1-氨基环丙烷-1-羧酸脱氨酶产生菌提高双孢蘑菇产量[J]. 河南科学, 2015, 33(10): 1750-1755.
Cheng Y, Wang J M, Zhang Y, Yang Q L, Qiu L Y. Application of 1-aminocyclopropane-1-carboxylic acid deaminase producing bacterium for increasing the yield of the button mushroom[J]. Henan Science, 2015, 33(10): 1750-1755.
[19] 董晓雅,周巍巍,张继英,戚元成,高玉千,申进文,邱立友. 荧光假单胞菌对食用菌的促生作用及其机理[J]. 生态学报, 2010, 30(17): 4685-4690.
Dong X Y, Zhou W W, Zhang J Y, Qi Y C, Gao Y Q, Shen J W, Qiu L Y. The effect of growth promotion and its mechanism on edible fungi by fluorescent Pseudomonas[J]. Acta Ecologica Sinica, 2010, 30(17): 4685-4690.
[20] 张大飞,戚元成,高玉千,申进文,邱立友. 双孢蘑菇覆土出菇机理初步探讨[J]. 食用菌, 2010, 32(1): 9-11,16.doi:10.3969/j.issn.1000-8357.2010.01.004.
Zhang D F, Qi Y C, Gao Y Q, Shen J W, Qiu L Y. A primary analysis on the mechanism of casing soil triggering the sporophore formation of Agaricus bisporus[J]. Edible Fungi, 2010, 32(1): 9-11,16.
[21] Li T, Zhang J, Shen C H, Li H R, Qiu L Y. 1-aminocyclopropane-1-carboxylate: a novel and strong chemoattractant for the plant beneficial rhizobacterium Pseudomonas putida UW4[J]. Molecular Plant-Microbe Interactions, 2019, 32(6): 750-759. doi:10.1094/MPMI-11-18-0317-R.
[22] Adler J. A method for measuring chemotaxis and use of the method to determine optimum conditions for chemotaxis by Escherichia coli[J]. Journal of General Microbiology, 1973, 74(1): 77-91. doi:10.1099/00221287-74-1-77.
[23] de Weert S, Vermeiren H, Mulders I H, Kuiper I, Hendrickx N, Bloemberg G V, Vanderleyden J, De Mot R, Lugtenberg B J J. Flagella-driven chemotaxis towards exudate components is an important trait for tomato root colonization by Pseudomonas fluorescens[J]. Molecular Plant-Microbe Interactions, 2002, 15(11): 1173-1180. doi:10.1094/MPMI.2002.15.11.1173.
[24] Grimm A C, Harwood C S. Chemotaxis of Pseudomonas spp. to the polyaromatic hydrocarbon naphthalene[J]. Applied and Environmental Microbiology, 1997, 63(10): 4111-4115.
[25] Adler J. Chemotaxis in bacteria[J]. Science, 1966, 153(3737): 708-716. doi:10.1126/science.153.3737.708.
[26] Zhang G, Sun Z H, Ren A, Shi L, Shi D K, Li X B, Zhao M W. The mitogen-activated protein kinase GlSlt2 regulates fungal growth, fruiting body development, cell wall integrity, oxidative stress and ganoderic acid biosynthesis in Ganoderma lucidum[J]. Fungal Genetics and Biology, 2017, 104: 6-15. doi:10.1016/j.fgb.2017.04.004.
[27] Zhang G, Ren A, Shi L, Zhu J, Jiang A L, Shi D K, Zhao M W. Functional analysis of an APSES transcription factor (GlSwi6) involved in fungal growth, fruiting body development and ganoderic-acid biosynthesis in Ganoderma lucidum[J]. Microbiological Research, 2018, 207: 280-288. doi:10.1016/j.micres.2017.12.015.
[28] Grewal S I S, Rainey P B. Phenotypic variation of Pseudomonas putida and P. tolaasii affects the chemotactic response to Agaricus bisporus mycelial exudate[J]. Journal of General Microbiology, 1991, 137(12): 2761-2768. doi:10.1099/00221287-137-12-2761.
[29] Validov S Z, Kamilova F, Lugtenberg B J J. Pseudomonas putida strain PCL1760 controls tomato foot and root rot in stonewool under industrial conditions in a certified greenhouse[J]. Biological Control, 2009, 48(1): 6-11. doi:10.1016/j.biocontrol.2008.09.010.
[30] Seneviratne G, Jayasinghearachchi H S. Mycelial colonization by bradyrhizobia and azorhizobia[J]. Journal of Biosciences, 2003, 28(2): 243-247. doi:10.1007/BF02706224.
[31] Zakeel M C M, Safeena M I S. Biofilmed biofertilizer for sustainable agriculture[M]//Ansari R, Mahmood I. Plant health under biotic stress. Singapore:Springer,2019: 65-82.doi: 10.1007/978-981-13-6040-4_3.
[32] Seneviratne G, Weerasekara M L M A W, Seneviratne K A C N, Zavahir J S, Kecskés M L, Kennedy I R. Importance of biofilm formation in plant growth promoting rhizobacterial action[M]//Maheshwari D. Plant growth and health promoting bacteria. Berlin, Heidelberg: Springer, 2010: 81-95. doi:10.1007/978-3-642-13612-2_4.
[33] Ballhausen M B, Vandamme P, de Boer W. Trait differentiation within the fungus-feeding (mycophagous) bacterial genus Collimonas[J]. PLoS One, 2016, 11(6): e157552. doi:10.1371/journal.pone.0157552.
[34] Worrich A, Stryhanyuk H, Musat N, König S, Banitz T, Centler F, Frank K, Thullner M, Harms H, Richnow H H, Miltner A, Köstner M, Wick L Y. Mycelium-mediated transfer of water and nutrients stimulates bacterial activity in dry and oligotrophic environments[J]. Nature Communications, 2017, 8: 15472. doi:10.1038/ncomms15472.
[35] Simon A, Hervé V, Al-Dourobi A, Verrecchia E, Junier P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns[J]. FEMS Microbiology Ecology, 2017, 93 (1): w217. doi:10.1093/femsec/fiw217.
[36] Nazir R, Tazetdinova D I, van Elsas J D. Burkholderia terrae BS001 migrates proficiently with diverse fungal hosts through soil and provides protection from antifungal agents[J]. Frontiers in Microbiology, 2014, 5: 598. doi:10.3389/fmicb.2014.00598.
[37] Balbontin R, Vlamakis H, Kolter R. Mutualistic interaction between Salmonella enterica and Aspergillus niger and its effects on Zea mays colonization[J]. Microbial Biotechnology, 2014, 7(6): 589-600. doi:10.1111/1751-7915.12182.
[38] Benoit I, van den Esker M H, Patyshakuliyeva A, Mattern D J, Blei F, Zhou M M, Dijksterhuis J, Brakhage A A, Kuipers O P, de Vries R P,Kovács  . Bacillus subtilis attachment to Aspergillus niger hyphae results in mutually altered metabolism[J]. Environmental Microbiology, 2015, 17(6): 2099-2113. doi:10.1111/1462-2920.12564.
. Bacillus subtilis attachment to Aspergillus niger hyphae results in mutually altered metabolism[J]. Environmental Microbiology, 2015, 17(6): 2099-2113. doi:10.1111/1462-2920.12564.
[39] Lohberger A, Spangenberg J E, Ventura Y, Bindschedler S, Verrecchia E P, Bshary R, Junier P. Effect of organic carbon and nitrogen on the interactions of Morchella spp. and bacteria dispersing on their mycelium[J]. Frontiers in Microbiology, 2019, 10: 124. doi:10.3389/fmicb.2019.00124.
[40] Jia Y J, Kakuta Y, Sugawara M, Igarashi T, Oki N, Kisaki M, Shoji T, Kanetuna Y, Horita T, Matsui H, Honma M. Synthesis and degradation of 1-aminocyclopropane-1-carboxylic acid by Penicillium citrinum[J]. Bioscience, Biotechnology, and Biochemistry, 1999, 63(3): 542-549. doi:10.1271/bbb.63.542.
[41] Medina E, Paredes C, Pérez-Murcia M D, Bustamante M A, Moral R. Spent mushroom substrates as component of growing media for germination and growth of horticultural plants[J]. Bioresource Technology, 2009, 100(18): 4227-4232. doi:10.1016/j.biortech.2009.03.055.
