玉米大斑病(Northern corn leaf blight)是一种常见的玉米叶部病害,在我国的东北、华北北部以及南方冷凉山区常有发生[1]。引起该病害的病原菌为玉米大斑病菌(Setosphaeria turcica)。该病害直接影响玉米叶片的光合作用从而导致玉米减产,严重时减产可达50%[2-3],目前在生产上种植抗病玉米品种是最重要的防控措施,但近年来由于该病菌变异频繁,使得抗病玉米品种的抗性频频丧失[4]。因此,研发新型杀真菌制剂已成为目前研究的热点之一。
研究表明,真菌细胞壁是由糖蛋白、葡聚糖和几丁质所构成,其中几丁质在赋予细胞壁刚性、物理强度和特定形状中发挥重要作用[5-6]。几丁质是由膜蛋白几丁质合成酶通过催化N-乙酰葡糖胺单体聚合形成[7]。在酿酒酵母(Saccharomyces cerevisiae)中发现了3个编码几丁质合成酶的Scchs基因。Scchs1p在出芽过程中作为一种修复酶,修补酵母子细胞脱离母体后脆弱的细胞壁;Scchs2p参与初级隔膜的形成;Scchs3p参与芽痕和侧壁的几丁质合成[8-9]。在禾谷镰刀菌(Fusarium graminearum)中,FgChs2和其他的几丁质合成酶基因共同调控病菌的营养发育和毒力[10]。在稻瘟病菌(Magnaporthe oryzae)中,MoCHS1、MoCHS6和MoCHS7对于植物侵染是必需的,MoCHS5和MoCHS6在维持菌丝的极化生长过程中共同发挥作用[11]。另外,对酿酒酵母(S. cerevisiae)、禾谷镰刀菌(F. graminearum)、小巢状麴菌(Aspergillus nidulans)、灰葡萄孢菌(Botrytis cinerea)、粗糙脉孢菌(Neurospora crassa)和稻瘟病菌(M.oryzae)等真菌进行系统进化分析表明,这些真菌中的几丁质合成酶基因可为7类(Class Ⅰ~Class Ⅶ)[10]。河北省植物生理与分子病理学重点实验室植物病原信号转导课题组前期研究中在玉米大斑病菌中已鉴定得到8个几丁质合成酶基因(StCHS1~StCHS8),将这些几丁质合成酶基因同样分为7类(Ⅰ~Ⅶ),其中StCHS7属于Class Ⅰ, StCHS8属于Class Ⅱ, StCHS6属于Class Ⅲ, StCHS2属于Class Ⅳ, StCHS5属于Class Ⅴ, StCHS1属于Class Ⅵ, StCHS3、StCHS4属于Class Ⅶ。
本研究拟从玉米大斑病菌中克隆其关键的几丁质合成酶基因StCHS6(Class Ⅲ),并利用SMART、GSDS、Expasy、SOMPA和WoLF PSORT等软件对其基因结构及其所编码的蛋白进行分析,进一步利用RNA-Seq技术分析StCHS6在病菌不同发育时期的表达模式,旨在明确玉米大斑病菌几丁质合酶基因StCHS6的结构特征及表达模式,阐明该基因在病菌的生长发育及致病过程中的作用及研发以几丁质合成酶基因为靶标的新型抗真菌药物奠定基础。
1 材料和方法
1.1 供试菌株
玉米大斑病菌1号小种菌株01-23,由河北农业大学河北省植物生理与分子病理学重点实验室保存。
1.2 主要试剂
dNTP Mixture、10×PCR Buffer、Taq DNA聚合酶及PrimeScriptTM RT Reagent Kit with gDNA Eraser均购于宝生物工程(大连)有限公司;便捷型植物RNA快速提取试剂盒购于天津华力科析科技有限公司;胶回收试剂盒购于北京全式金生物技术有限公司;引物由上海生工生物工程有限公司合成。
1.3 试验方法
1.3.1 RNA的提取 玉米大斑病菌野生型菌株于25 ℃黑暗条件培养12 d后,收集病菌气生菌丝。RNA提取方法参照便捷型植物RNA快速提取试剂盒说明书。
1.3.2 第一链cDNA的合成 以1.3.1提取的病菌总RNA为模板,参照PrimeScriptTM RT Reagent Kit with gDNA Eraser试剂盒使用说明书合成第一链cDNA,并将反转录产物保存于-20 ℃备用。
1.3.3 玉米大斑病菌StCHS6基因的克隆 对玉米大斑病菌中几丁质合成酶的鉴定结果表明,StCHS6的蛋白ID为133408。利用该蛋白ID于玉米大斑病菌基因组数据库(http://genome.jgi.doe.gov/Settu1/Settu1.home.html)中下载StCHS6的CDS序列[12-13]。利用Primer 5.0软件设计引物CHS6-F/R用于StCHS6的CDS序列扩增(表1)。PCR反应体系为:模板cDNA(300 ng/μL) 1 μL,CHS6-F/R(10 μmol/L)各1 μL,TaKaRa Taq DNA聚合酶(5 U/μL) 0.25 μL,dNTP Mixture(2.5 mmol/L) 2 μL,10×PCR Buffer 2.5 μL,ddH2O 17.25 μL。PCR反应程序为:95 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸3 min,35个循环,72 ℃再延伸10 min。扩增产物于1%的琼脂糖凝胶上进行检测。
表1 基因克隆引物序列
Tab.1 Primer sequences used in gene cloning
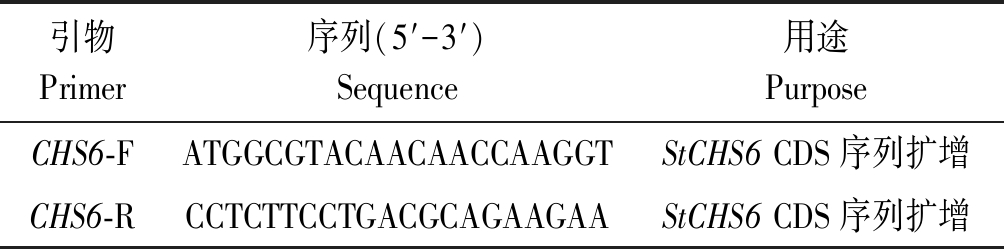
引物Primer序列(5′-3′)Sequence用途PurposeCHS6-FATGGCGTACAACAACCAAGGTStCHS6 CDS序列扩增CHS6-RCCTCTTCCTGACGCAGAAGAAStCHS6 CDS序列扩增
1.3.4 StCHS6基因及编码产物的生物信息学分析 StCHS6基因的结构特征及其所编码蛋白的保守结构域、理化性质、二级结构和亚细胞定位分析参考文献[14]。
1.3.5 玉米大斑病菌不同发育时期材料的收集 病菌不同发育时期材料的收集方法参考文献[14]。
1.3.6 StCHS6在玉米大斑病菌不同发育时期的表达模式分析 基于已经获得的玉米大斑病菌RNA-Seq数据库,分析StCHS6在病菌菌丝、分生孢子、芽管、附着胞和侵入钉5个典型的发育时期的表达情况。
2 结果与分析
2.1 玉米大斑病菌StCHS6基因的克隆及结构分析
利用StCHS6的蛋白ID从玉米大斑病菌基因组数据库进行搜索,结果表明其位于病菌基因组scaffold_1:2066293-2069876(-)的位置,基因全长为3 584 bp,CDS序列大小为2 703 bp。以玉米大斑病菌cDNA为模板,对StCHS6的CDS序列进行扩增,得到的PCR产物大小与预期相符(图1)。对目的条带进行回收测序,扩增产物序列与数据库所下载序列一致,表明在该病菌中克隆得到了StCHS6的CDS序列。
利用Gene Structure Display Server (GSDS)在线软件对StCHS6的基因结构进行分析,结果表明,StCHS6含有4个内含子,5个外显子(图2)。内含子相位通常用于分析基因结构的进化,0、1和2代表内含子在密码子中的位置。0代表内含子位于第1个碱基之前,1代表内含子位于第1个碱基之后,2代表内含子位于第2个碱基之后[15]。根据2个相邻内含子的组合是否引起移码突变定义了两类外显子:不对称外显子和对称外显子[16]。本研究发现,StCHS6基因不含有0相位,2个相邻内含子相位的组合分别为(1-2),(2-2)和(2-1),表明在StCHS6中不对称外显子和对称外显子均存在。
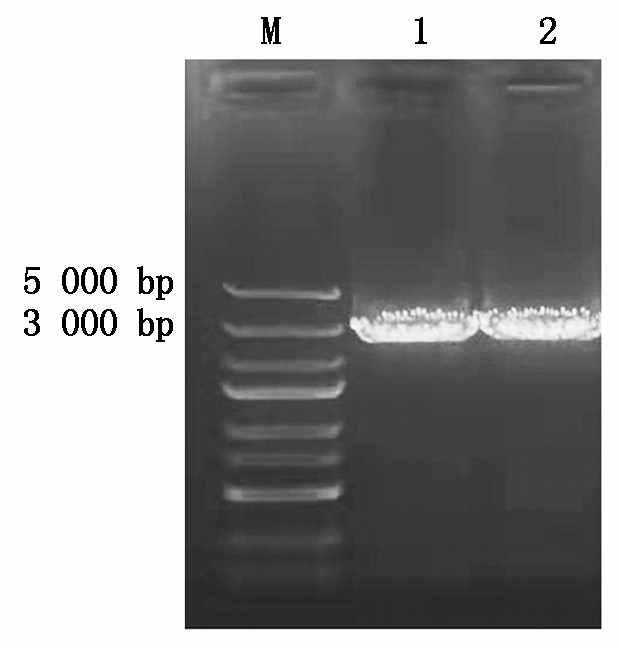
M.DNA Marker(100~5 000 bp); 1, 2.StCHS6基因扩增产物。 M.DNA Marker(100-5 000 bp); 1, 2.Amplification products of StCHS6.
图1 StCHS6基因CDS序列扩增
Fig.1 Amplification of CDS of StCHS6
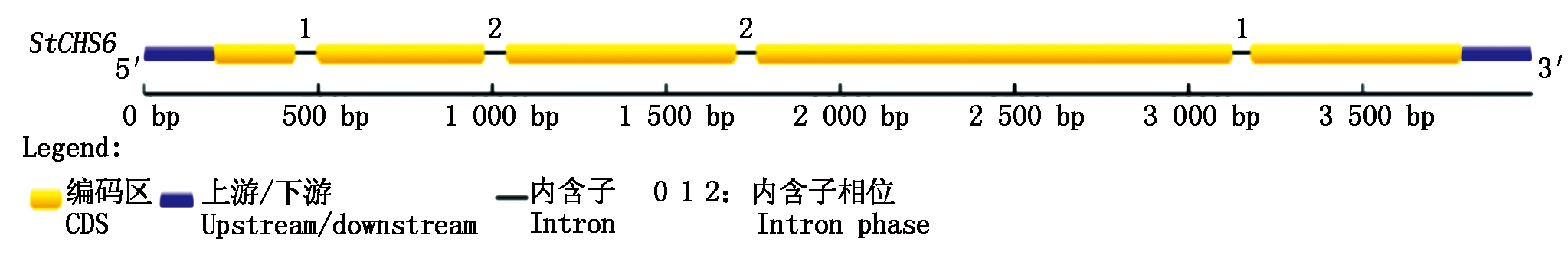
图2 StCHS6基因结构特征
Fig.2 The structure characteristics of StCHS6 gene
2.2 玉米大斑病菌StCHS6蛋白的理化性质分析及亚细胞定位
利用Expasy在线软件对StCHS6蛋白的理化性质进行分析,推测StCHS6蛋白的分子式为C4586H6985N1203O1309S30,编码900个氨基酸残基,分子质量为100.877 98 ku,等电点(PI)为8.36,表明StCHS6蛋白为碱性蛋白。该蛋白的脂肪指数为81.86,亲水性平均值为-0.178,不稳定指数为36.94,属于稳定型蛋白。利用SOMPA对StCHS6蛋白的二级结构进行预测,结果表明在该蛋白中,无规则卷曲(Random coil)占43.56%,α-螺旋(Alpha helix)占38.44%,延伸链(Extended strand)占13.44%,β-转角(Beta turn)占4.56%。利用SMART对StCHS6的保守结构域进行分析,发现StCHS6包含几丁质合酶催化结构域Chitin_synth_1(682-694aa)和多个跨膜结构域(图3)。利用WoLF PSORT对该蛋白进行亚细胞定位分析,推测其位于细胞膜上。
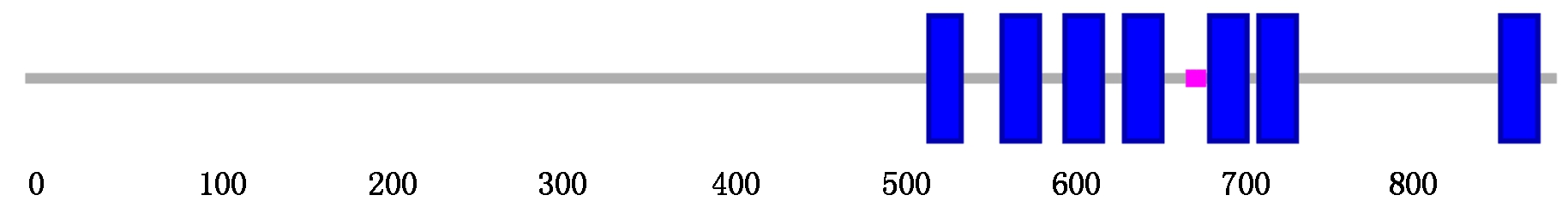
蓝色长方形代表跨膜结构域;粉色长方形代表几丁质合酶催化结构域Chitin_synth_1。 The blue rectangle represents transmembrane region; The pink rectangle represents the chitin synthase catalytic domain Chitin_synth_1.
图3 StCHS6保守结构域分析
Fig.3 The analysis of conserved domain for StCHS6
2.3 StCHS6在玉米大斑病菌不同发育时期的表达模式
基于RNA-Seq数据对StCHS6在菌丝、分生孢子、芽管、附着胞及侵入钉5个发育时期的表达模式进行分析。结果表明,StCHS6在以上5个发育时期均有表达,但其表达量有所差异(图4)。以芽管时期的表达量为1,菌丝、分生孢子、附着胞和侵入钉时期的表达量分别为菌丝时期的3.80,4.97,3.40,2.60倍,说明StCHS6在分生孢子时期表达量最高,在芽管时期表达量最低。以上结果表明,StCHS6在玉米大斑病菌生长发育及侵染过程中均发挥作用,但在各个发育时期的重要程度有所不同。
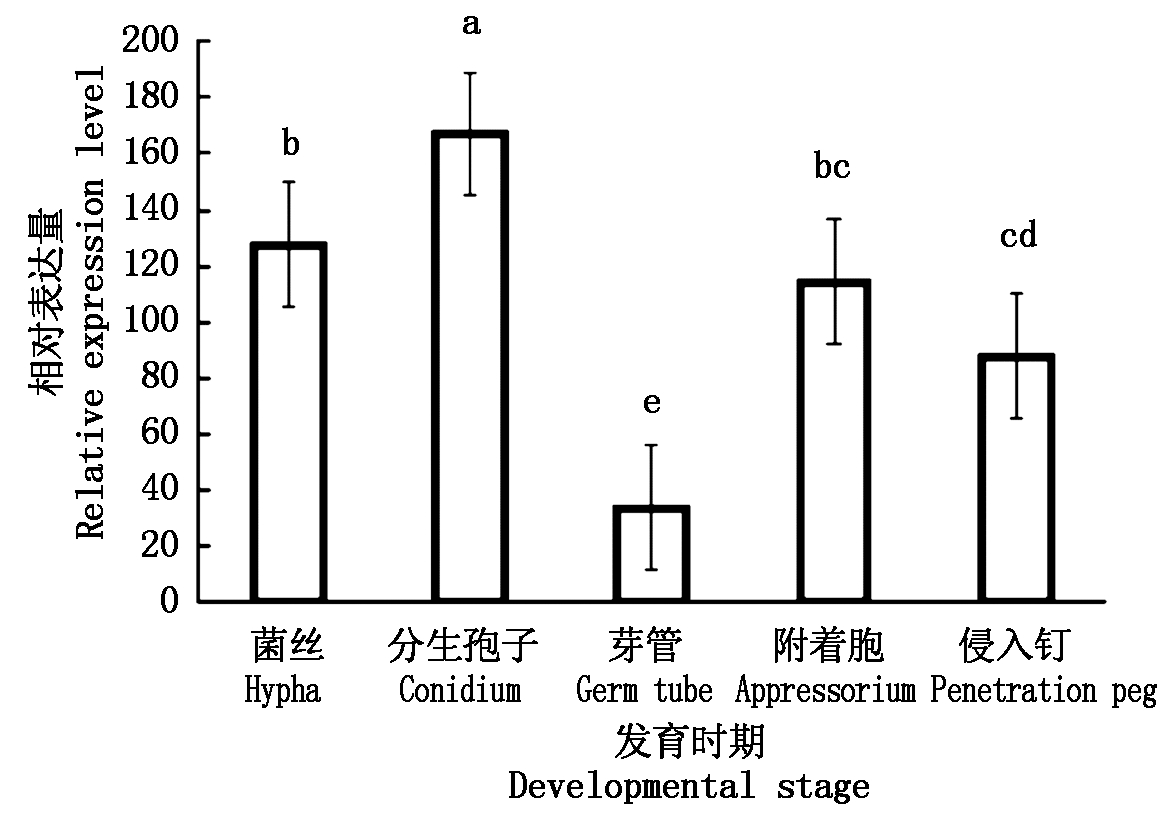
不同小写字母表示在0.05水平上差异显著。 Different small letters indicate significant difference at the 0.05 level.
图4 StCHS6基因不同发育时期表达量分析
Fig.4 Expression of StCHS6 in different developmental stages
3 讨论与结论
前人研究表明,真菌中的几丁质合成酶在维持细胞完整性、菌丝生长和寄主侵染等过程中均发挥重要作用[17-18]。在小巢状麴菌(A. nidulans)中,Class Ⅲ的几丁质合成酶对于菌丝生长是必需的[19];在禾谷镰刀菌(F. graminearum)中没有获得Fgchs3b(Class Ⅲ)的缺失突变体,表明该基因的缺失可能是致命的[20];在稻瘟病菌(M.oryzae)中,Class Ⅲ中的几丁质合成酶的缺失会导致超过90%的分生孢子形态异常[11];而在灰葡萄孢菌(B.cinerea)中,Class Ⅲ几丁质合成酶(Bcchs3a)的缺失导致病菌毒力显著降低[21];在构巢曲霉(Aspergillus nidulans)中,Class Ⅲ类几丁质合成酶基因AnchsB在其整个发育过程中都表达,当AnchsB敲除后,出现了菌株生长速率减慢、不产孢子、菌丝尖端膨大和侧壁异常等表型缺陷[22]。以上结果表明,真菌中ClassⅢ几丁质合成酶通过影响细胞壁的合成从而对病菌的生长发育及侵染和致病过程造成影响。本研究中,RNA-Seq数据表明StCHS6在病菌生长发育的各个时期都表达,但在菌丝和分生孢子时期表达量较高,由此推测,StCHS6可能参与玉米大斑病菌的菌丝生长及产孢过程,但需要进一步通过创制StCHS6基因敲除突变体进行验证。
前人研究表明,几丁质是植物病原真菌细胞壁的特有组分,该物质不存在于植物细胞壁中,因此,研发以抑制或干扰病原真菌几丁质合成酶合成或降低该酶活性的新型杀菌剂是目前植物病理学界研究的热点之一[23]。本研究只是分析了玉米大斑病菌几丁质合成酶家族中的1个关键基因StCHS6的表达模式,尚没有对整个基因家族的表达规律及功能进行系统分析。因此,在后续研究中,应利用基因敲除和过表达技术,明确该基因在病菌致病性方面的功能,进一步通过比较突变体与野生型菌株对细胞壁类杀真菌制剂的敏感性,明确杀真菌制剂的作用机制,并筛选创制新型杀真菌制剂。
本研究通过克隆得到了玉米大斑病菌几丁质合成酶基因StCHS6的CDS序列,发现该基因含有4个内含子,5个外显子。对其所编码的蛋白进行分析,发现该基因编码900个氨基酸,属于碱性稳定型蛋白。该蛋白含有几丁质合成酶催化结构域Ⅰ和多个跨膜结构域,定位于细胞膜上。对StCHS6在病菌菌丝、分生孢子、芽管、附着胞和侵入钉时期的表达量分析表明,该基因在分生孢子时期表达量最高,在芽管时期表达量最低,说明StCHS6可能在分生孢子时期发挥重要作用。
[1] 孙淑琴, 温雷蕾, 董金皋. 玉米大斑病菌的生理小种及交配型测定[J]. 玉米科学, 2005, 13(4): 112-113,123. doi:10.3969/j.issn.1005-0906.2005.04.032.
Sun S Q, Wen L L, Dong J G. Identification of physiological races and mating type of Exserohilum turcicum[J]. Joumal of Maize Sciences, 2005, 13(4): 112-113,123.
[2] Vieira R A, Mesquini R M, Silva C N, Hata F T, Tessmann D J, Scapim C A. A new diagrammatic scale for the assessment of northern corn leaf blight[J]. Crop Protection, 2014, 56: 55-57. doi:10.1016/j.cropro.2011.04.018.
[3] Technow F, Bürger A, Melchinger A E. Genomic prediction of northern corn leaf blight resistance in maize with combined or separated training sets for heterotic groups[J]. Genes Genomes Genetics, 2013, 3(2): 197-203. doi:10.1534/g3.112.004630.
[4] 王慧, 王翊, 姜丽静, 刘洪亮, 张志斌, 张铁强, 王仁杰. 黑龙江省主栽玉米品种对大斑病抗性评价[J]. 黑龙江农业科学, 2014(4): 8-10. doi:10.3969/j.issn.1002-2767.2014.04.003.
Wang H, Wang Y, Jiang L J, Liu H L, Zhang Z B, Zhang T Q, Wang R J. Resistance evaluation of main maize varieties for northern blight in Heilongjiang Province[J]. Heilongjiang Agricultural Sciences, 2014(4): 8-10.
[5] Morcx S, Kunz C, Choquer M, Assie S, Blondet E, Simond-C te E, Gajek K, Chapeland-Leclerc F, Expert D, Soulie M C. Disruption of Bcchs4, Bcchs6 or Bcchs7 chitin synthase genes in Botrytis cinerea and the essential role of class VI chitin synthase(Bcchs6)[J]. Fungal Genetics and Biology, 2013, 52: 1-8. doi:10.1016/j.fgb.2012.11.011.
te E, Gajek K, Chapeland-Leclerc F, Expert D, Soulie M C. Disruption of Bcchs4, Bcchs6 or Bcchs7 chitin synthase genes in Botrytis cinerea and the essential role of class VI chitin synthase(Bcchs6)[J]. Fungal Genetics and Biology, 2013, 52: 1-8. doi:10.1016/j.fgb.2012.11.011.
[6] Latgé J P, Beauvais A, Chamilos G. The cell wall of the human fungal pathogen Aspergillus fumigatus: biosynthesis, organization, immune response, and virulence[J]. Annual Review of Microbiology, 2017, 71: 99-116. doi:10.1146/annurev-micro-030117-020406.
[7] Cohen E. Chapter 2-Chitin biochemistry: synthesis, hydrolysis and inhibition[J]. Advances in Insect Physiology, 2010, 38: 5-74. doi:10.1016/S0065-2806(10)38005-2.
[8] Yang J K, Zhang K Q. Chitin synthesis and degradation in fungi: biology and enzymes[J]. Advances in Experimental Medicine and Biology, 2019, 1142: 153-167. doi:10.1007/978-981-13-7318-3_8.
[9] Shaw J A, Mol P C, Bowers B, Silverman S J, Valdivieso M H, Dur n A, Cabib E. The function of chitin synthases 2 and 3 in the Saccharomyces cerevisiae cell cycle[J]. Journal of Cell Biology, 1991, 114(1): 111-123. doi:10.1083/jcb.114.1.111.
n A, Cabib E. The function of chitin synthases 2 and 3 in the Saccharomyces cerevisiae cell cycle[J]. Journal of Cell Biology, 1991, 114(1): 111-123. doi:10.1083/jcb.114.1.111.
[10] Liu Z Y, Zhang X P, Liu X, Fu C Y, Han X Y, Yin Y N, Ma Z H. The chitin synthase FgChs2 and other FgChss co-regulate vegetative development and virulence in F. graminearum[J]. Scientific Reports, 2016, 6: 34975. doi:10.1038/srep34975.
[11] Kong L A, Yang J, Li G T, Qi L L, Zhang Y J, Wang C F, Zhao W S, Xu J R, Peng Y L. Different chitin synthase genes are required for various developmental and plant infection processes in the rice blast fungus Magnaporthe oryzae[J]. PLoS Pathogens, 2012, 8(2): e1002526. doi:10.1371/journal.ppat.1002526.
[12] Ohm R A, Feau N, Henrissat B, Schoch C L, Horwitz B A, Barry K W, Condon B J, Copeland A C, Dhillon B, Glaser F, Hesse C N, Kosti I, Labutti K, Lindquist E A, Lucas S, Salamov A A, Bradshaw R E, Ciuffetti L, Hamelin R C, Kema G H J, Lawrence C, Scott J A, Spatafora J W, Turgeon B G, Wit P J, Zhong S B, Goodwin S B, Grigoriev I V. Diverse lifestyles and strategies of plant pathogenesis encoded in the genomes of eighteen Dothideomycetes fungi[J]. PLoS Pathogens, 2012, 8(12): e1003037. doi:10.1371/journal.ppat.1003037.
[13] Condon B J, Leng Y Q, Wu D L, Bushley K E, Ohm R A, Otillar R, Martin J, Schackwitz W, Grimwood J, MohdZainudin N, Xue C S, Wang R, Manning V A, Dhillon B, Tu Z J, Steffenson B J, Salamov A, Sun H, Lowry S, LaButti K, Han J, Copeland A, Lindquist E, Barrry K, Schmutz J, Baker S E, Ciuffetti L M, Grigoriev L V, Zhong S B, Turgeon B J. Comparative genome structure, secondary metabolite, and effector coding capacity across Cochliobolus pathogens[J]. PLoS Genetics, 2013, 9(1): e1003233. doi:10.1371/journal.pgen.1003233.
[14] 张晓雅, 李学然, 薛江芝, 王小敏, 刘玉卫, 谷守芹, 董金皋. 玉米大斑病菌StSLT2基因结构与表达模式分析[J]. 河北农业大学学报, 2019, 42(3): 32-37. doi:10.13320/j.cnki.jauh.2019.0052.
Zhang X Y, Li X R, Xue J Z, Wang X M, Liu Y W, Gu S Q, Dong J G.Gene structure and expression pattern analysis of StSLT2 in Setosphaeria turcica[J]. Journal of Hebei Agricultural University, 2019, 42(3): 32-37.
[15] Luo M, Ding J J, Li Y, Tang H P, Qi P F, Ma J, Wang J R, Chen G Y, Pu Z E, Li W, Li Z Y, Harwood W, Lan X J, Deng M, Lu Z X, Wei Y M, Zheng Y L, Jiang Q T. A single-base change at a splice site in Wx-A1 caused incorrect RNA splicing and gene inactivation in a wheat EMS mutant line[J]. Theoretical and Applied Genetics, 2019, 132(7): 2097-2109. doi:10.1007/s00122-019-03340-1.
[16] Qi Y Y, Xu W J, Xing T, Zhao M M, Li N N, Yan L, Xia G M, Wang M C. Synonymous codon usage bias in the plastid genome is unrelated to gene structure and shows evolutionary heterogeneity[J]. Evolutionary Bioinformatics, 2015, 11: 65-77. doi:10.4137/EBo.s22566.
[17] Fujiwara M, Ichinomiya M, Motoyama T, Horiuchi H, Ohta A, Takagi M. Evidence that the Aspergillus nidulans class Ⅰ and class Ⅱ chitin synthase genes, chsC and chsA, share critical roles in hyphal wall integrity and conidiophore development[J]. The Journal of Biochemistry, 2000, 127(3): 359-366. doi:10.1093/oxfordjournals.jbchem.a022616.
[18] Yamada E, Ichinomiya M, Ohta A,Horiuchi H. The class V chitin synthase gene csmA is crucial for the growth of the chsA chsC double mutant in Aspergillus nidulans[J]. Bioscience Biotechnology and Biochemistry, 2005, 69(1): 87-97. doi:10.1271/bbb.69.87.
[19] Yanai K, Kojima N, Takaya N, Horiuchi H, Ohta A, Takagi M. Isolation and characterization of two chitin synthase genes from Aspergillus nidulans[J]. Bioscience Biotechnology and Biochemistry, 1994, 58(10): 1828-1835. doi:10.1271/bbb.58.1828.
[20] Cheng W, Song X S, Li H P, Cao L H, Sun K, Qiu X L, Xu Y B, Yang P, Huang T, Zhang J B,Qu B,Liao Y C. Host-induced gene silencing of an essential chitin synthase gene confers durable resistance to Fusarium head blight and seedling blight in wheat[J]. Plant Biotechnology Journal, 2015, 13(9): 1335-1345. doi:10.1111/pbi.12352.
[21] Soulié M C, Perino C, Piffeteau A, Choquer M, Malfatti P, Cimerman A, Kunz C, Boccara M, Vidal-Cros A. Botrytis cinerea virulence is drastically reduced after disruption of chitin synthase class Ⅲ gene(Bcchs3a)[J]. Cellular Microbiology, 2006, 8(8): 1310-1321. doi:10.1111/j.1462-5822.2006.00711.x.
[22] Lee J I, Choi J H, Park B C, Park Y H, Lee M Y, Park H M, Maeng P J. Differential expression of the chitin synthase genes of Aspergillus nidulans, chsA, chsB and chsC, in response to developmental status and environmental factors[J]. Fungal Genetics and Biology, 2004, 41(6): 635-646. doi:10.1016/j.fgb.2004.01.009.
[23] 代猛, 裘娟萍, 汪琨. 几丁质合酶抑制剂作为抗真菌药物的研究进展[J]. 科技通报, 2017, 33(3): 71-76. doi:10.13774/j.cnki.kjtb.2017.03.013.
Dai M, Qiu J P, Wang K. Research on progress of chitin synthase inhibitors as anti-fungi drugs[J]. Bulletin of Science and Technology, 2017, 33(3): 71-76.
