Dof转录因子家族是植物中特有的一类转录因子,因其N端含有一个大概由52个保守氨基酸残基构成的C2-C2型单锌指结构域故而得名Dof(DNA binding with one finger)[1]。该家族属于单锌指蛋白超家族,一般由200~400个氨基酸构成。目前,已有多种植物Dof转录因子及蛋白被分离出来,如玉米[2]、小麦[3]、水稻[4]、马铃薯[5]等,并且在许多基因的启动子中发现了Dof蛋白结合元件[6-7]。研究表明,Dof转录因子在植物种子萌发、碳氮代谢、光响应、逆境胁迫等过程中发挥重要作用[8-12]。
植物在遭受非生物胁迫时,会有大量的转录因子参与基因的表达调控,其中就包括Dof转录因子[13-14]。在马铃薯中鉴定的35个Dof基因中,大多数能被盐、高温、干旱及激素诱导上调表达[15]。茶树中CsDof1和CsDof2能被低温及高温诱导上调表达[16]。而小麦的17个Dof转录因子中,多数则在干旱胁迫后下调表达[12]。尽管Dof转录因子能够响应非生物胁迫,但其作用机制仍然不十分明确,应用Dof转录因子改良植物的例子也十分少见。
大豆是一种重要的经济作物,但对大豆中Dof转录因子的研究目前仅限于种子发育等方面[17-18]。研究Dof转录因子对大豆应对非生物胁迫的影响对于改良大豆的抗逆性,提高其产量具有重要作用。本试验从大豆中选取3个Dof转录因子进行生物信息学分析,并对它们在高盐、干旱、低温及高温处理及叶、根、茎中的表达量进行检测,以期为大豆Dof转录因子抗逆机制研究打下基础。
1 材料和方法
1.1 植物材料
大豆品种北豆9号由齐齐哈尔大学植物分子育种研究室保存。
1.2 生物信息学分析
在大豆转录因子数据库PlantTFDB(http://www.plantgdb.org/GmGDB/)中搜索Dof转录因子基因。通过序列比对,选取其中3个序列相似性较低的Dof基因进行以下分析。用在线软件ExPASy(http://expasy.org/tools/pi_tool.html)预测蛋白的分子质量和等电点;用在线软件PSORT(https://wolfpsort.hgc.jp/)进行蛋白的亚细胞定位预测;用在线软件NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)对蛋白磷酸化位点进行分析;利用NCBI数据库(https://blast. ncbi. nlm. nih. gov/Blast. cgi)进行Dof同源基因的搜索;用DNAMAN软件进行蛋白序列比对。在GmGDB(http://www.plantgdb.org/GmGDB/)数据库中搜索Dof基因上游启动子序列。用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)数据库对启动子中的顺式作用元件进行预测分析。
1.3 非生物胁迫处理
使用Hoagland营养液,在16 h光照/8 h黑暗,25 ℃培养室内水培大豆幼苗。待大豆幼苗第1片三出复叶完全展开时进行以下处理。将大豆幼苗置于含150 mmol/L NaCl的营养液中进行高盐迫处理;将大豆幼苗置于含15% PEG8000的营养液中模拟旱胁迫处理;将大豆幼苗置于4 ℃培养箱中进行低温胁迫处理;将大豆幼苗置于42 ℃培养箱中进行高温胁迫处理。分别在处理的0,1,2,5,10,24,36 h取样,剪取0.1 g第1片三出复叶并迅速置于液氮中,同样剪取未处理的大豆根和茎。以上材料均在-80 ℃超低温冰箱中保存备用。
1.4 RNA提取及cNDA合成
使用TaKaRa RNAiso Plus分别提取上述1.3中各样本的总RNA,通过琼脂糖凝胶电泳和OD260/280值检测提取的RNA质量。使用Novoprotein cDNA反转录试剂盒合成第一链cDNA。
1.5 实时荧光定量PCR
分别根据3个Dof基因的cDNA序列设计实时荧光定量PCR引物,以大豆组成型表达基因β-Tubuin(GenBank登录号:GMU12286)为内参,4对引物序列如表1所示并送上海生工公司合成。在BIO-RAD CFX96 Real-time PCR仪上,对3个Dof基因在非生物胁迫及叶、根和茎中的表达量进行检测。实时荧光定量PCR反应体系如下:2×TB Green Premix Ex TaqⅡ(TaKaRa) 10 μL、cDNA 2 μL、上下游引物各0.8 μL,补水至总体积20 μL。反应程序如下:95 ℃预变性30 s;95 ℃ 5 s,58 ℃ 30 s,共循环40次。所有处理均做3次重复,采用2-ΔΔCt法计算基因的相对表达量。
表1 实时荧光定量PCR引物序列
Tab.1 Sequences of primers in Real-time fluorescent quantitative PCR
基因Gene上游引物Forward primer下游引物Reverse primerGmDof2.15′-GACAGTCACCACCATGAAGCAAG-3′5′-GATGCCATTCCAACTAGCTCTTCC-3′GmDof3.15′-CCACCAACACCAAGTTCTGCTAC-3′5′-TCTGCTTCCACCACCAACGG-3′GmDof4.65′-CTAGCTCCACCATCACCACAATG-3′5′-TCCTCCGCCTAACATTCCAGTC-3′β-Tubuin5′-GGAAGGCT TTCTTGCATTGGTA-3′5′-AGTGGCATCCTGGTACTGC-3′
2 结果与分析
2.1 三个大豆Dof基因的生物信息学分析
在大豆转录因子数据库PlantTFDB中共发现97条编码Dof转录因子蛋白的mRNA序列,选取序列相似性较低的GmDof2.1(GenBank登录号:XM003539689)、GmDof3.1(GenBank登录号:XM003543559)和GmDof4.6(GenBank登录号:XM006606352)进行后续分析。将3个大豆Dof基因编码蛋白序列输入NCBI数据库与其他物种的Dof蛋白进行同源性比对,发现GmDof2.1与赤豆VaDof2.1具有较高同源性(85.06%),GmDof3.1与木豆CcDof3.1具有较高同源性(79.45%),GmDof4.6与木豆CcDof4.6具有较高的同源性(78.9%)。且它们都具有1个保守的Dof结构域(图1)。

下划线代表Dof结构域。GenBank登录号:GmDof2.1.XP003539737;GmDof3.1.XP003543607; GmDof4.6.XP006606415; CcDof3.1.XP020223973;CcDof4.6.XP020238897;VaDof2.1.XP017431457。 The Dof domain was underlined. GenBank accession number:GmDof2.1.XP003539737; GmDof3.1.XP003543607; GmDof4.6.XP006606415; CcDof3.1.XP020223973; CcDof4.6.XP020238897; VaDof2.1.XP017431457.
图1 三个大豆Dof蛋白与其他物种Dof蛋白氨基酸序列比对
Fig.1 Alignment of amino acid sequences of three soybean Dof proteins and Dof proteins from other plants
蛋白质生化属性分析发现(表2),3个大豆Dof基因分别位于12,13,20号染色体上,编码的蛋白序列长度分别为305,212,301个氨基酸残基,等电点分别为7.57,8.70,8.75。亚细胞定位预测结果显示(表2),3个Dof蛋白均定位于细胞核中。磷酸化位点预测分析显示(表2),3个Dof蛋白均含有不同数量的丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)磷酸化位点,这些氨基酸可能在维持蛋白的空间结构、活性及信号转导等方面发挥作用。
2.2 三个大豆Dof基因启动子序列分析
在大豆基因组数据库中搜索3个大豆Dof基因起始密码子上游1 500 bp的启动子序列。对启动子序列中的顺式作用元件进行预测分析。结果如表3所示,GmDof2.1启动子序列中含有3种与逆境和激素响应相关的元件(ARE、DRE1和MBS),GmDof3.1启动子序列中含有6种与逆境和激素响应相关的元件(ABRE、ARE、CGTCA-motif、TGACG-motif、W-box和WUN-motif),GmDof4.6启动子序列中含有7种与逆境和激素响应相关的元件(ABRE、ARE、CGTCA-motif、GARE-motif、MBS、TGACG-motif和WUN-motif)。此外3个Dof启动子序列中还含有多种与光应答相关的元件。预测结果表明,3个大豆Dof基因均可能响应多种激素的诱导并广泛参与大豆对多种胁迫的应答。
表2 三个大豆Dof蛋白的鉴定及特性
Tab.2 Identification and feature of the three Dof proteins in soybean
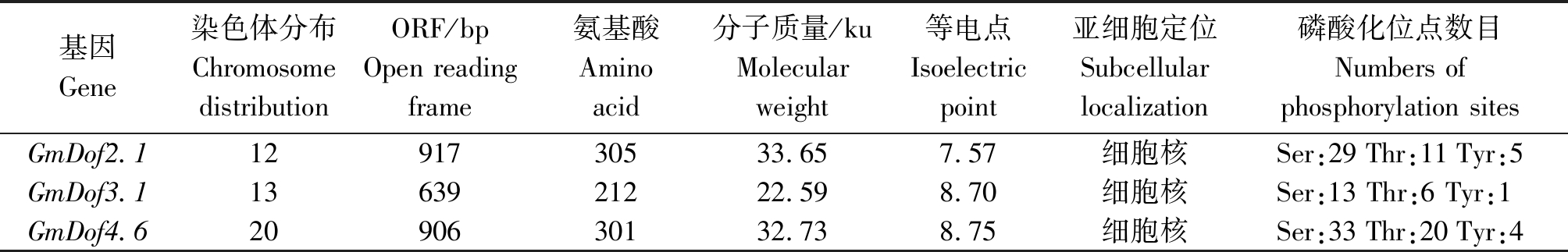
基因Gene染色体分布Chromosome distributionORF/bpOpen reading frame氨基酸Aminoacid分子质量/kuMolecularweight等电点Isoelectric point亚细胞定位Subcellular localization磷酸化位点数目Numbers of phosphorylation sitesGmDof2.11291730533.657.57细胞核Ser:29 Thr:11 Tyr:5GmDof3.11363921222.598.70细胞核Ser:13 Thr:6 Tyr:1GmDof4.62090630132.738.75细胞核Ser:33 Thr:20 Tyr:4
表3 三个大豆Dof基因启动子顺式作用元件预测
Tab.3 Prediction of cis-elements in the three Dof promoters in soybean
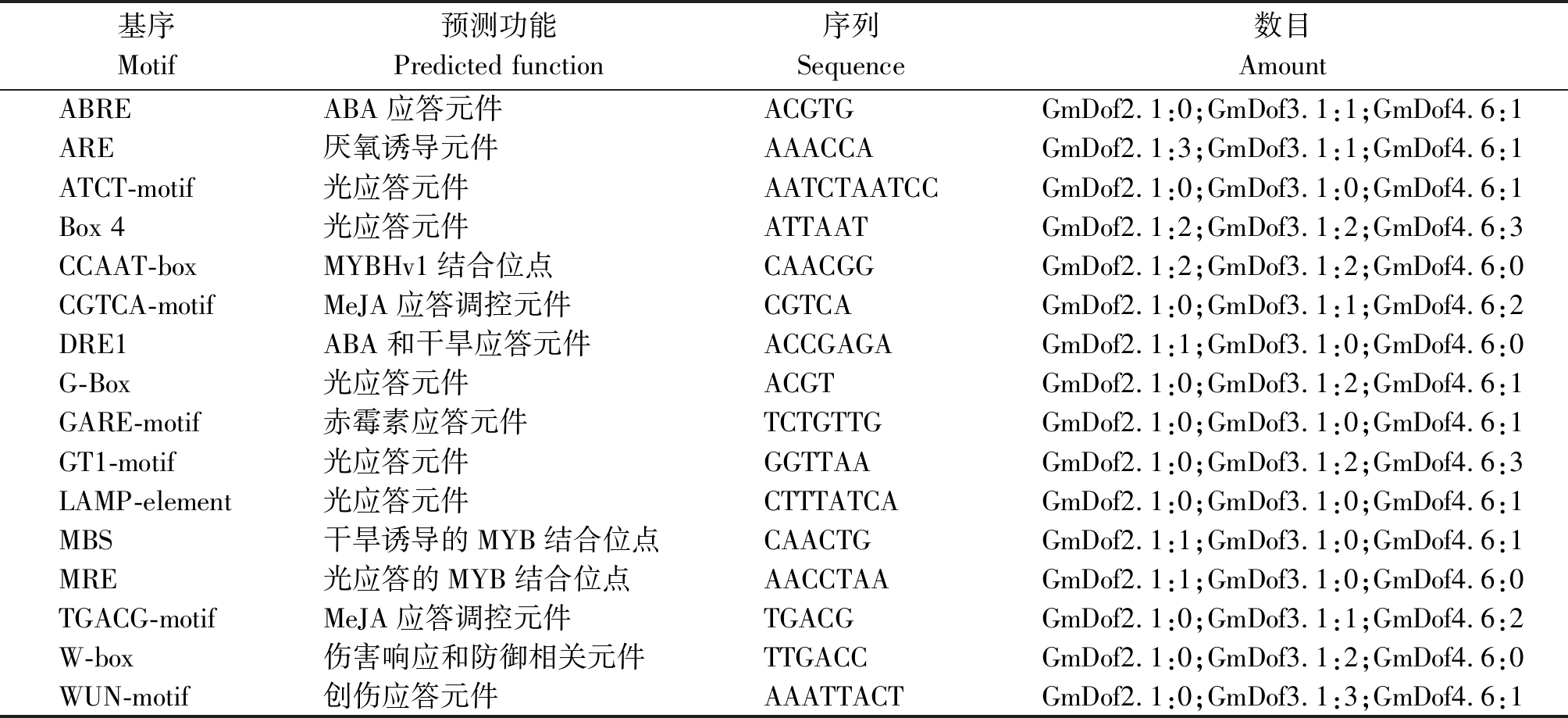
基序Motif预测功能Predicted function序列 Sequence数目AmountABREABA应答元件ACGTGGmDof2.1:0;GmDof3.1:1;GmDof4.6:1ARE厌氧诱导元件AAACCAGmDof2.1:3;GmDof3.1:1;GmDof4.6:1ATCT-motif光应答元件AATCTAATCCGmDof2.1:0;GmDof3.1:0;GmDof4.6:1Box 4光应答元件ATTAATGmDof2.1:2;GmDof3.1:2;GmDof4.6:3CCAAT-boxMYBHv1结合位点CAACGGGmDof2.1:2;GmDof3.1:2;GmDof4.6:0CGTCA-motifMeJA应答调控元件CGTCAGmDof2.1:0;GmDof3.1:1;GmDof4.6:2DRE1ABA和干旱应答元件ACCGAGAGmDof2.1:1;GmDof3.1:0;GmDof4.6:0G-Box光应答元件ACGTGmDof2.1:0;GmDof3.1:2;GmDof4.6:1GARE-motif赤霉素应答元件TCTGTTGGmDof2.1:0;GmDof3.1:0;GmDof4.6:1GT1-motif光应答元件GGTTAAGmDof2.1:0;GmDof3.1:2;GmDof4.6:3LAMP-element光应答元件CTTTATCAGmDof2.1:0;GmDof3.1:0;GmDof4.6:1MBS干旱诱导的MYB结合位点CAACTGGmDof2.1:1;GmDof3.1:0;GmDof4.6:1MRE光应答的MYB结合位点AACCTAAGmDof2.1:1;GmDof3.1:0;GmDof4.6:0TGACG-motifMeJA应答调控元件TGACGGmDof2.1:0;GmDof3.1:1;GmDof4.6:2W-box伤害响应和防御相关元件TTGACCGmDof2.1:0;GmDof3.1:2;GmDof4.6:0WUN-motif创伤应答元件AAATTACTGmDof2.1:0;GmDof3.1:3;GmDof4.6:1
2.3 三个大豆Dof基因在非生物胁迫下表达
大豆幼苗经NaCl、PEG、低温和高温处理后,利用实时荧光定量PCR对3个大豆Dof基因的表达动态进行检测。结果显示(图2),高盐处理后,GmDof2.1表达量先升高后低于对照,GmDof3.1和GmDof4.6表达量升高,分别在处理5,24 h达到最大值;干旱处理后结果与高盐处理类似,GmDof2.1表达量先升高后低于对照,GmDof3.1和GmDof4.6表达量升高,均在处理2 h达到最大值;低温处理后,GmDof2.1表达量先降低后升高,GmDof3.1和GmDof4.6表达量升高,分别在处理5,36 h达到最大值;高温处理后,3个Dof基因的表达量均持续升高,均在处理10 h达到最大值,且GmDof3.1和GmDof4.6表达量升高幅度较其他处理明显。由此推测3个Dof转录因子在大豆中均可不同程度的响应非生物胁迫。
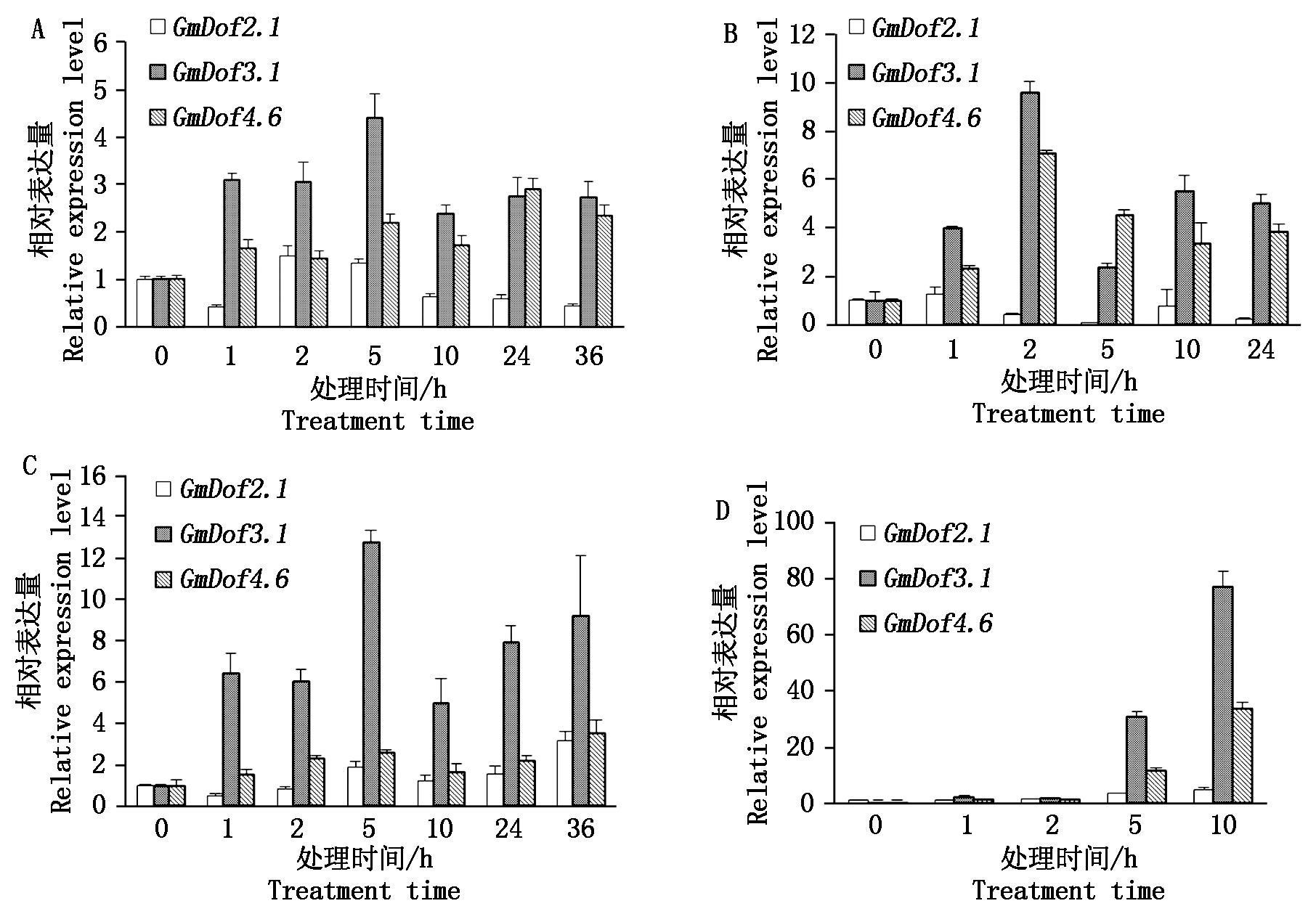
图2 三个Dof基因在高盐(A)、干旱(B)、低温(C)和高温(D)处理下的表达
Fig.2 Expression of the three Dof genes under high salt(A), drought(B), low temperature(C) and high temperature(D) treatments
2.4 三个大豆Dof基因在叶、根和茎中的表达
实时荧光定量PCR检测结果显示(图3),3个Dof基因在大豆叶、根和茎中均有表达,不具有组织特异性。其中GmDof2.1和GmDof3.1在大豆根中的表达量最高,GmDof4.6在大豆茎中的表达量最高。
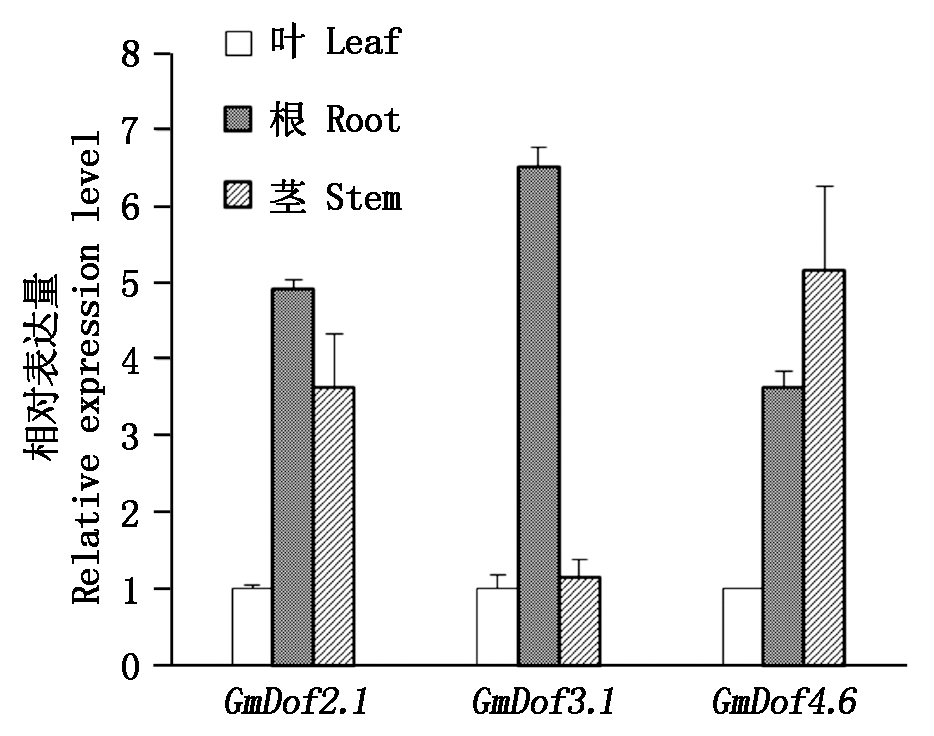
图3 三个Dof基因在大豆叶、根和茎中的表达
Fig.3 Expression of the three Dof genes in leaf, root and stem in soybean
3 结论与讨论
盐碱、干旱、低温和高温等是作物生长过程中最常见的非生物胁迫,对作物的生长发育和产量影响很大。转录因子在植物应对非生物胁迫时调控机制复杂,并且同一家族不同转录因子成员之间对非生物胁迫也存在相互调控的关系[19-20]。关于Dof转录因子的抗逆机制,有研究表明,Dof基因能够调控类黄酮的积累[21],而有些Dof基因则能响应水杨酸信号[22]。类黄酮和水杨酸都能在植物抵御逆境时发挥作用,例如,糖基化的类黄酮能帮助植物抵御紫外线的伤害,而水杨酸在植物进行防御反应时则作为信号分子[23]。
本研究利用生物信息学方法,对3个大豆Dof转录因子的基因及蛋白结构、理化性质、启动子中顺式作用元件等进行预测,为系统研究大豆Dof基因的功能提供了信息及参考依据。3个Dof基因编码的蛋白长度从212~305个氨基酸,变化较大,但它们都具有1个保守的Dof结构域,因此都属于Dof基因家族成员。根据其蛋白长度变化较大,推测大豆中Dof基因的起源和进化模式比较复杂,基因在功能上可能具有多样性。
基因在不同组织中的表达可能暗示该基因在相应表达部位的生物学功能。在禾本科植物水稻、玉米和小麦中,大部分Dof基因在各个组织或器官中都有不同程度的表达,但部分基因在某些组织和器官中具优势表达特征,它们可能在植物生长发育的某一个或某几个发育阶段发挥特殊功能[24]。同样,本试验中的3个Dof基因在大豆叶、根和茎中的表达情况也不同,可能预示其功能存在多样化。
研究表明,Dof转录因子作为转录激活子或抑制子可参与调节植物的防御反应[25]。在小麦的17个响应干旱胁迫的Dof基因中,除TaDof14和TaDof15的表达量明显上调外,其余 15个均下调,表明多数小麦Dof基因可能负向调控植物的干旱适应性[12]。但在水稻的30个Dof基因中,仅有3个基因的表达在PEG处理后受到显著抑制,而其他基因的表达则明显上调,它们可能正向调控植物的干旱适应性[26]。本试验对高盐、干旱、低温和高温处理下3个大豆Dof基因的表达量进行了检测。3个Dof基因的表达虽然总体趋势相似,均有上调,但在响应时间和响应强度上存在差异。表明这3个大豆Dof基因均参与大豆对非生物胁迫的响应,但它们是否与植物的抗逆性相关,调控机制如何等问题仍有待进一步深入研究。
[1] Yanagisawa S.Dof domain proteins:plant-specific transcription factors associated with diverse phenomena unique to plants[J].Plant & Cell Physiology,2004,45(4):386-391. doi:10.1093/pcp/pch055.
[2] Yanagisawa S, Izui K. Molecular cloning of two DNA-binding proteins of maize that are structurally different but interact with the same sequence motif[J]. The Journal of Biological Chemistry, 1993, 268(21):16028-16036.
[3] Kumar R, Taware R, Gaur V S, Guru S K, Kumar A.Influence of nitrogen on the expression of TaDof1 transcription factor in wheat and its relationship with photo synthetic and ammonium assimilating efficiency[J].Molecular Biology Reports,2009,36(8):2209-2220.doi:10.1007/s11033-008-9436-8.
[4] Li D J, Yang C H, Li X B, Gan Q, Zhao X F, Zhu L H.Functional characterization of rice OsDof12[J].Planta,2009,229(6):1159-1169.doi:10.1007/s00425-009-0893-7.
[5] Venkatesh J, Park S W.Genome-wide analysis and expression profiling of DNA-binding with one zinc finger(Dof) transcription factor family in potato[J].Plant Physiology and Biochemistry,2015,94:73-85.doi:10.1016/j.plaphy.2015.05.010.
[6] 杨桂燕, 赵震, 赵玉琳, 张凤娇, 高彩球. ThMYB3和ThDof2对ThVHAc1基因表达的调控[J]. 北京林业大学学报, 2015, 37(12):1-6. doi:10.13332/j.1000-1522.20140486.
Yang G Y, Zhao Z, Zhao Y L, Zhang F J, Gao C Q. Upstream regulators of ThVHAc1 [J]. Journal of Beijing Forestry University, 2015,37(12):1-6.
[7] 卫晓彬, 王亚楠, 张超, 单丽伟, 唐如春, 范三红. 小麦WPBF与高分子量谷蛋白基因上游Prolamin-Like box的特异结合[J]. 中国农业科学, 2012, 45(1):7-15. doi:10.3864/j.issn.0578-1752.2012.01.002.
Wei X B, Wang Y N, Zhang C, Shan L W, Tang R C, Fan S H. Specific binding of wheat WPBF and Prolamin-Like box in upstream of HMW-GS genes [J]. Scientia Agricultura Sinica, 2012,45(1):7-15.
[8] Gualberti G, Papi M, Bellucci L, Ricci L, Bouchez D, Camilleri C, Costantino P, Vittorioso P.Mutations in the Dof zinc finger genes DAG2 and DAG1 influence with opposite effects the germination of Arabidopsis seeds[J].The Plant Cell,2002,14(6):1253-1263.doi:10.1105/tpc.010491.
[9] Yanagisawa S, Akiyama A, Kisaka H, Uchimiya H, Miwa T.Metabolic engineering with Dof1 transcription factor in plants:improved nitrogen assimilation and growth under low-nitrogen conditions[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(20):7833-7838.doi:10.1073/pnas.0402267101.
[10] Huang W, Huang Y, Li M Y, Wang F, Xu Z S, Xiong A S.Dof transcription factors in carrot:genome-wide analysis and their response to abiotic stress[J].Biotechnology Letters,2016,38(1):145-155.doi:10.1007/s10529-015-1966-2.
[11] Gupta S, Malviya N, Kushwaha H, Nasim J, Bisht N C, Singh V K, Yadav D.Insights into structural and functional diversity of Dof(DNA binding with one finger)transcription factor[J].Planta,2015,241(3):549-562.doi:10.1007/s00425-014-2239-3.
[12] Shaw L M, Mcintyre C L, Gresshoff P M, Xue G P.Members of the Dof transcription factor family in Triticum aestivum are associated with light-mediated gene regulation[J].Functional & Integrative Genomics,2009,9(4):485-498.doi:10.1007/s10142-009-0130-2.
[13] Ma J, Li M Y, Wang F, Tang J, Xiong A S.Genome-wide analysis of Dof family transcription factors and their responses to abiotic stresses in Chinese cabbage[J].BMC Genomics,2015,16:33.doi:10.1186/s12864-015-1242-9.
[14] Corrales A R, Nebauer S G, Carrillo L, Fernández-Nohales P, Marqués J, Renau-Morata B, Granell A, Pollmann S, Vicente-carbajosa J, Molina R V, Medina J.Characterization of tomato cycling Dof factors reveals conserved and new functions in the control of flowering time and abiotic stress responses[J].Journal of Experimental Botany,2014,65(4):995-1012.doi:10.1093/jxb/ert451.
[15] 吴智明, 张圣旭, 梁关生. 马铃薯基因组中Dof转录因子家族的鉴定与表达特征分析[J]. 核农学报, 2015, 29(7):1260-1270. doi:10.11869/j.issn.100-8551.2015.07.1260.
Wu Z M, Zhang S X, Liang G S. Genome-wide identification and expression analysis of the Dof transcription factor family in potato(Solanum tuberosum L.) [J]. Journal of Nuclear Agricultural Sciences, 2015,29(7):1260-1270.
[16] 李辉, 黄蔚, 刘志薇, 王永鑫, 吴致君, 庄静. 茶树两个Dof转录因子的分离及其在温度胁迫中的响应分析[J]. 茶叶科学, 2016, 36(3):312-322.doi:10.13305/j.cnki.jts.2016.03.011.
Li H, Huang W, Liu Z W, Wang Y X, Wu Z J, Zhuang J. Isolation and expression analysis of two temperature responsive Dof genes from Camellia sinensis[J]. Journal of Tea Science, 2016,36(3):312-322.
[17] Zhang J H, Hao Q, Bai L L, Xu J, Yin W B, Song L Y, Xu L, Guo X J, Fan C M, Chen Y H,Ruan J,Hao S T,Li Y G,Wang R R C,Hu Z M.Overexpression of the soybean transcription factor GmDof4 significantly enhances the lipid content of Chlorella ellipsoidea[J].Biotechnology for Biofuels,2014,7(1):10. doi:10.1186/s13068-014-0128-4.
[18] Wang H W, Zhang B, Hao Y J, Huang J, Tian A G, Liao Y, Zhang J S, Chen S Y.The soybean Dof-type transcription factor genes, GmDof4 and GmDof11, enhance lipid content in the seeds of transgenic Arabidopsis plants[J].The Plant Journal:for Cell and Molecular Biology,2007,52(4):716-729.doi:10.1111/j.1365-313X.2007.03268.x.
[19] Zhao T J, Sun S, Liu Y, Liu J M, Liu Q, Yan Y B, Zhou H M. Regulating the drought-responsive element(DRE)-mediated signaling pathway by synergic functions of trans-active and trans-inactive DRE binding factors in Brassica napus[J].Journal of Biological Chemistry,2006,281(16):10752-10759. doi:10.1074/jbc.M510535200.
[20] Zhuang J, Jiang H H, Wang F, Peng R H, Yao Q H, Xiong A S.A rice OsAP23, functioning as an AP2/ERF transcription factor, reduces salt tolerance in transgenic Arabidopsis[J].Plant Molecular Biology Reporter,2013,31(6):1336-1345.doi:10.1007/s11105-013-0610-3.
[21] Skirycz A, Jozefczuk S, Stobiecki M, Muth D,Zanor M I, Witt I, Roeber B M.Transcription factor AtDOF4;2 affects phenylpropanoid metabolism in Arabidopsis thaliana[J].New Phytologist,2007,175(3):425-438. doi:10.1111/j.1469-8137.2007.02129.x.
[22] Kang H G, Singh K B.Characterization of salicylic acid-responsive, Arabidopsis Dof domain proteins:overexpression of OBP3 leads to growth defects[J].The Plant Journal,2000,21(4):329-339.doi:10.1046/j.1365-313x.2000.00678.x.
[23] 蔡晓锋, 张余洋, 张俊红, 李汉霞, 叶志彪. 植物Dof基因家族功能研究进展[J]. 植物生理学报, 2013, 49(1):1-12.doi:10.13592/j.cnki.ppj.2013.01.012.
Cai X F, Zhang Y Y, Zhang J H, Li H X, Ye Z B. Advances in research on function of the Dof gene family in plant [J]. Acta Phytophysiologica Sinica, 2013,49(1):1-12.
[24] 刘文婷, 马田田, 周春菊, 藏潇, 李浪金, 张宝军, 杜威, 张炜丽, 陈坤明. 禾本科植物单锌指家族基因对逆境应答的研究进展[J]. 生物工程学报, 2016, 32(5):541-553. doi:10.13345/j.cjb.150351.
Liu W T, Ma T T, Zhou C J, Zang X, Li L J, Zhang B J, Du W, Zhang H L, Chen K M. Advances in stress response of DNA binding with one finger transcription factor family genes in graminaceous plants [J]. Chinese Journal of Biotechnology, 2016,32(5):541-553.
[25] Shinozaki K, Yamaguchi-Shinozaki K, Seki M.Regulatory network of gene expression in the drought and cold stress responses[J].Current Opinion in Plant Biology,2003,6(5):410-417.doi:10.1016/S1369-5266(03)00092-X.
[26] 周淑芬, 颜静宛, 刘华清, 林智敏, 陈睿, 杨绍华, 王锋. 水稻Dof基因家族的组织表达谱及胁迫诱导表达特征分析[J]. 分子植物育种, 2012, 10(6):635-643. doi:10.3969/mpb.010.000635.
Zhou S F, Yan J W, Liu H Q, Lin Z M, Chen R, Yang S H, Wang F. Transcriptional profiling analysis of OsDof gene family in various rice tissues and their expression characteristics under different stresses [J]. Molecular Plant Breeding, 2012,10(6):635-643.
