植物谷氨酸受体(Glutamate receptors-like receptors,GLRs)最早是Lam等[1]于1998年在拟南芥中发现的,通过序列的同源性分析获得20个与动物离子型谷氨酸受体(Ionotropic glutamate receptors, iGLuRs)同源的基因,命名为AtGLRs。随后在多个不同植物中有关于谷氨酸受体基因家族的报道。在水稻基因组中都发现有同拟南芥谷氨酸受体高度同源的24个OsGLRs基因[2],Aouini等[3]鉴定出番茄基因组中包含13个SlGLRs基因,苹果基因组中鉴定出32个MdGLRs基因[4]。
目前,已有多个植物GLRs基因的分子生理功能获得解析,作为模式植物拟南芥的AtGLRs基因研究的相对较多[5],其他植物也有报道。根部的AtGLR3.2和AtGLR3.4主要在韧皮部表达,AtGLR3.2和AtGLR3.4缺失突变体表现出有大量侧根原基出现,推测和这2个基因相关的离子通道可能通过韧皮部的Ca2+信号调控侧根的生长[6]。Kang等[7]研究表明,AtGLR1.1参与拟南芥的ABA生物合成和C/N代谢以此调控种子的萌发,AtGLR1.1还参与了拟南芥的水分调节[8]。Zheng等[9]研究证实,AtGLR1.2和AtGLR1.3通过调控茉莉酸信号途径增强拟南芥的耐寒性。Kong等[10]的研究结果表明,在种子萌发过程中AtGLR3.5上调表达促进胞质Ca2+浓度的增加,从而抑制ABI4的表达,达到促进种子萌发的作用。Cho等[11]发现AtGLR3.1在叶片的保卫细胞中表达,超表达AtGLR3.1基因将影响保卫细胞对Ca2+信号的接收或转导。AtGLR3.4基因在植株受到外界机械损伤刺激时表达量会增加3~6倍,细胞质中酸中毒也可以诱导该基因的上调表达[12],这些研究结果表明,AtGLR3.4基因可能参与了植物的抗逆反应。Li等[13]研究报道,OsGLR3.1在维持水稻根尖分生组织细胞活力的过程中发挥重要作用。小萝卜RsGLuR基因在拟南芥中超表达,可以提高植株抗真菌感染的能力[14]。
大豆是全世界种植面积最大的豆科植物,根瘤菌与大豆共生的生物固氮作用是世界农业非常重要的组成部分,此外大豆还是世界范围内动物饲料蛋白和食用油的重要植物来源。2010年,大豆栽培品种美国的Williams 82(Glycine_max_v2.0) 基因组序列公布[15],并成为当前参考应用最多的大豆基因组序列。最近,我国科学家对国审大豆品种中黄13的基因组(Gmax_ZH13) 进行从头组装,最终得到1.025 Gb的基因组序列,包含20条染色体和1条叶绿体[16]。这些大豆基因组测序工作的完成为大豆基因的基础性研究提供了很好的参考条件,很多大豆的基因家族在基因组中被鉴定和发掘,但是还没有关于大豆GmGLRs基因相关信息的研究报道。本研究也是参考Williams 82(Glycine_max_v2.0) 基因组序列,在全基因组层面鉴定出GmGLRs基因,并就这些基因从生物信息学角度进行分析,同时也研究了它们的组织表达模式,为深入研究该家族成员的分子生物学功能奠定基础。
1 材料和方法
1.1 GmGLRs基因的鉴定及序列分析
在TAIR(http://www.arabidopsis.org)获取拟南芥AtGLRs的基因序列,以拟南芥的序列为探针在Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html#!infoalias=Org_Gmax)大豆基因组(Wm82.a2.v1)Blast,搜索获得E值小于e-10的相似性序列,将获得的序列(大豆和拟南芥)在InterProscan5 (http://www.ebi.ac.uk/Tools/pfa/iprscan/)在线分析功能结构域存在情况,最终确定目标序列。序列的转录名称、编码区长度、CDS序列长度、外显子个数均在Phytozome获得;利用ProtParam tool(http://web.expasy.org/protparam/)在线分析获得GmGLRs基因蛋白氨基酸序列的分子质量、等电点等基本信息。
1.2 GmGLRs基因染色体定位
通过大豆基因组数据库Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)获得GmGLRs基因在大豆染色体上的位置信息,用Map Inspect软件绘制基因的染色体物理分布图。
1.3 GmGLRs基因构建系统进化树
GmGLRs蛋白质氨基酸序列在Phytozome获得,拟南芥AtGLRs蛋白序列在TAIR上获取,用Mega 6 软件采用Neighbor-Joining构建大豆GmGLRs和拟南芥AtGLRs蛋白的系统进化树,校验参数Bootstrap=1 000。
1.4 GmGLRs基因结构和跨膜结构域预测
采用GSDS(Gene Structure Dispely Server,http://gsds.cbi.pku.edu.cn/)在线绘制大豆GmGLRs基因结构图[17];大豆GmGLRs蛋白序列的跨膜结构域预测在TMHMM Server v. 2.0;http://www.cbs.dtu.dk/services/TMHMM在线进行。
1.5 GmGLRs基因组织表达模式分析
GmGLRs基因在花、叶、根瘤、豆荚、根、根毛、种子、顶端生长点、茎等9个组织部位表达量数据(FPKM)来自Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)[18],将获得数据log2均一化处理后,用HemI 1.0软件绘制GmGLRs基因表达热图[19]。
2 结果与分析
2.1 GmGLRs基因鉴定及序列基本信息
利用拟南芥AtGLRs基因序列在Phytozome大豆基因组(Wm82.a2.v1)Blast,共鉴定出18个GmGLRs候选基因。将这18个候选基因在InterProScan上鉴别功能结构域,因4个结构域IPR001638、IPR001828、IPR001320和IPR017103在拟南芥AtGLRs基因中都存在(表1),所以最后确定17个同时具有这4个结构域的GmGLRs基因为GmGLR基因家族基因。
根据这17个GmGLRs基因在染色体上出现的先后顺序分别命名为GmGLR1~GmGLR17(表2),这些基因编码区平均长度约为4 727 bp,最长的是GmGLR15的5 491 bp,最短的是GmGLR16的3 404 bp;CDS平均长度为2 748 bp,最长的是GmGLR7、 GmGLR13和GmGLR14基因的2 844 bp,最短的是GmGLR6基因的2 409 bp;这些基因的外显子个数除了GmGLR8基因有7个,其他都有6个外显子。蛋白质理化性质分析表明,17个GmGLRs基因的蛋白质氨基酸序列个数平均为915个氨基酸,平均分子质量是102.086 4 ku, 最大的是105.822 ku(GmGLR13),最小的是91.405 ku(GmGLR5);理论等电点最大的是GmGLR14的8.41,最小的是GmGLR8的6.17。只有GmGLR8的等电点小于7,说明该蛋白编码弱酸性蛋白,将在亚细胞环境为酸性的条件下发挥功能[19]。
表1 InterPro 结构域在大豆和拟南芥GLRs蛋白中的分布
Tab.1 InterPro domains found in GmGLRs and AtGLRs

InterPro结构域InterPro domains表述DescriptionGmGLRs 蛋白GmGLRs proteinsAtGLRs蛋白AtGLRs proteinsIPR001638溶质结合蛋白家族3/MltF N-末端结构域1720IPR001828细胞外配体结合受体1720IPR001320离子型谷氨酸受体1720IPR017103植物离子型谷氨酸受体1720IPR000337GPCR, 家族3GmGLR2AtGLR2.5/AtGLR3.5
表2 GmGLRs基因家族基本信息
Tab.2 Basic information of GmGLRs gene family of soybean
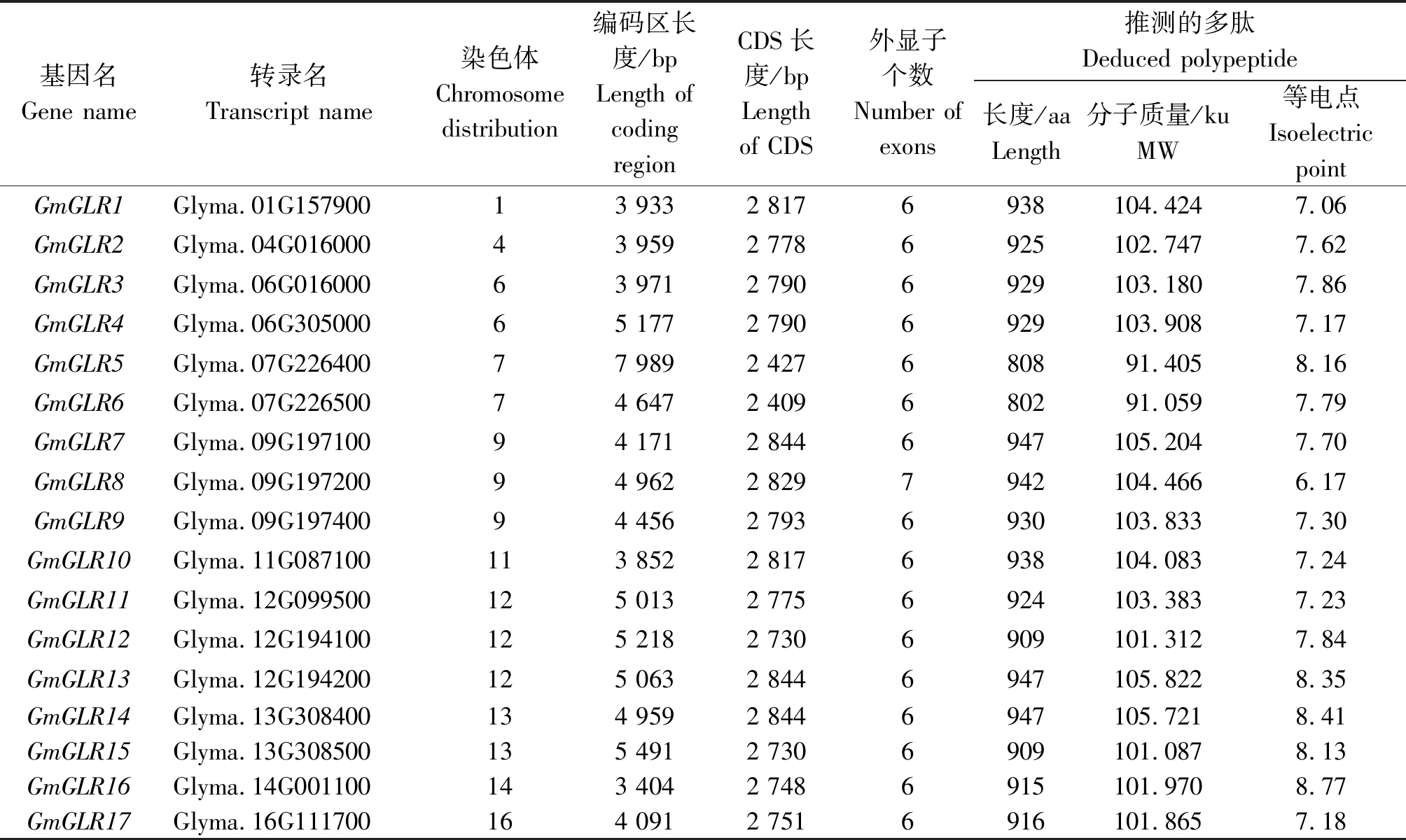
基因名Gene name转录名Transcript name染色体Chromosomedistribution编码区长度/bpLength ofcodingregionCDS长度/bpLengthof CDS 外显子个数Number ofexons推测的多肽Deduced polypeptide长度/aaLength分子质量/kuMW等电点IsoelectricpointGmGLR1Glyma.01G15790013 9332 8176938104.4247.06GmGLR2Glyma.04G01600043 9592 7786925102.7477.62GmGLR3Glyma.06G01600063 9712 7906929103.1807.86GmGLR4Glyma.06G30500065 1772 7906929103.9087.17GmGLR5Glyma.07G22640077 9892 427680891.4058.16GmGLR6Glyma.07G22650074 6472 409680291.0597.79GmGLR7Glyma.09G19710094 1712 8446947105.2047.70GmGLR8Glyma.09G19720094 9622 8297942104.4666.17GmGLR9Glyma.09G19740094 4562 7936930103.8337.30GmGLR10Glyma.11G087100113 8522 8176938104.0837.24GmGLR11Glyma.12G099500125 0132 7756924103.3837.23GmGLR12Glyma.12G194100125 2182 7306909101.3127.84GmGLR13Glyma.12G194200125 0632 8446947105.8228.35GmGLR14Glyma.13G308400134 9592 8446947105.7218.41GmGLR15Glyma.13G308500135 4912 7306909101.0878.13GmGLR16Glyma.14G001100143 4042 7486915101.9708.77GmGLR17Glyma.16G111700164 0912 7516916101.8657.18
2.2 GmGLRs基因染色体定位分析
根据基因位置信息,将鉴定出的17个GmGLRs基因定位在大豆的10条染色体上,定位结果如图1所示。这些基因在染色体上呈现不均匀分布,第9号和第12号染色体上各有3个GmGLRs基因,第6号、第7号和第13号染色体上各有2个GmGLRs基因,第1号、第4号、第11号、第14号和第16号染色体上都只有1个GmGLR基因。其中,第7号、第9号、第12号和第13号染色体的GmGLRs基因呈簇存在。
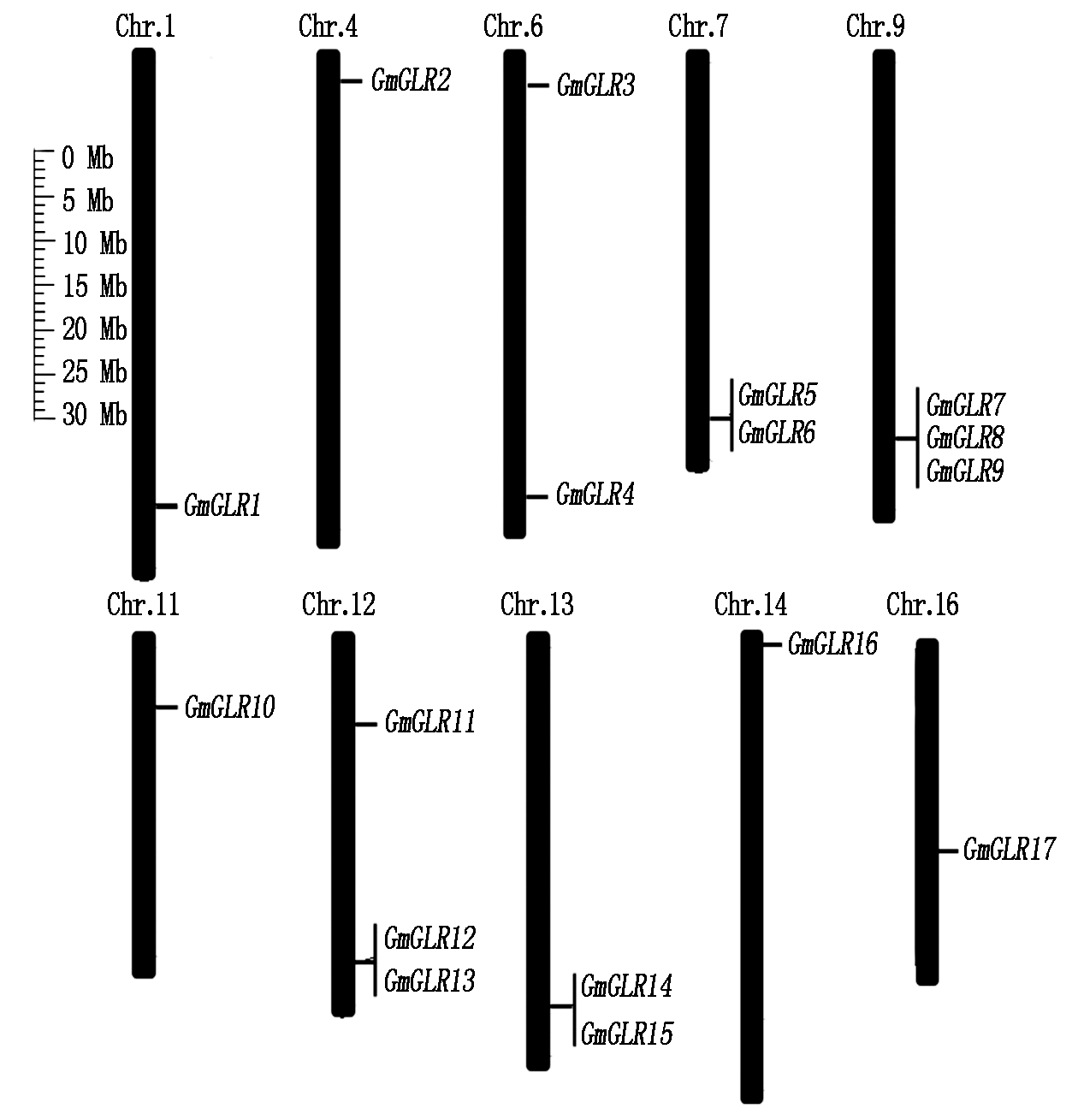
图1 GmGLRs基因的染色体定位
Fig.1 The chromosomal positions of GLRs gene in soybean
2.3 GmGLRs基因系统进化分析
为了分析GmGLRs基因的系统进化关系,利用GmGLRs蛋白质序列同拟南芥的AtGLRs蛋白质序列构建系统进化树,结果如图2所示。拟南芥AtGLRs基因分成3个亚家族,第一亚家族4个基因,第二亚家族和第三亚家族分别是9,7个基因。GmGLRs有GmGLR5和GmGLR6 2个基因同拟南芥第二亚家族聚在一起,其他的15个GmGLRs基因同拟南芥第三亚家族聚在一起,没有基因与拟南芥第一亚家族聚在一起。从进化树末端聚类可以看出,17个GmGLRs基因存在8对同源基因,只有GmGLR16单独一个分支。
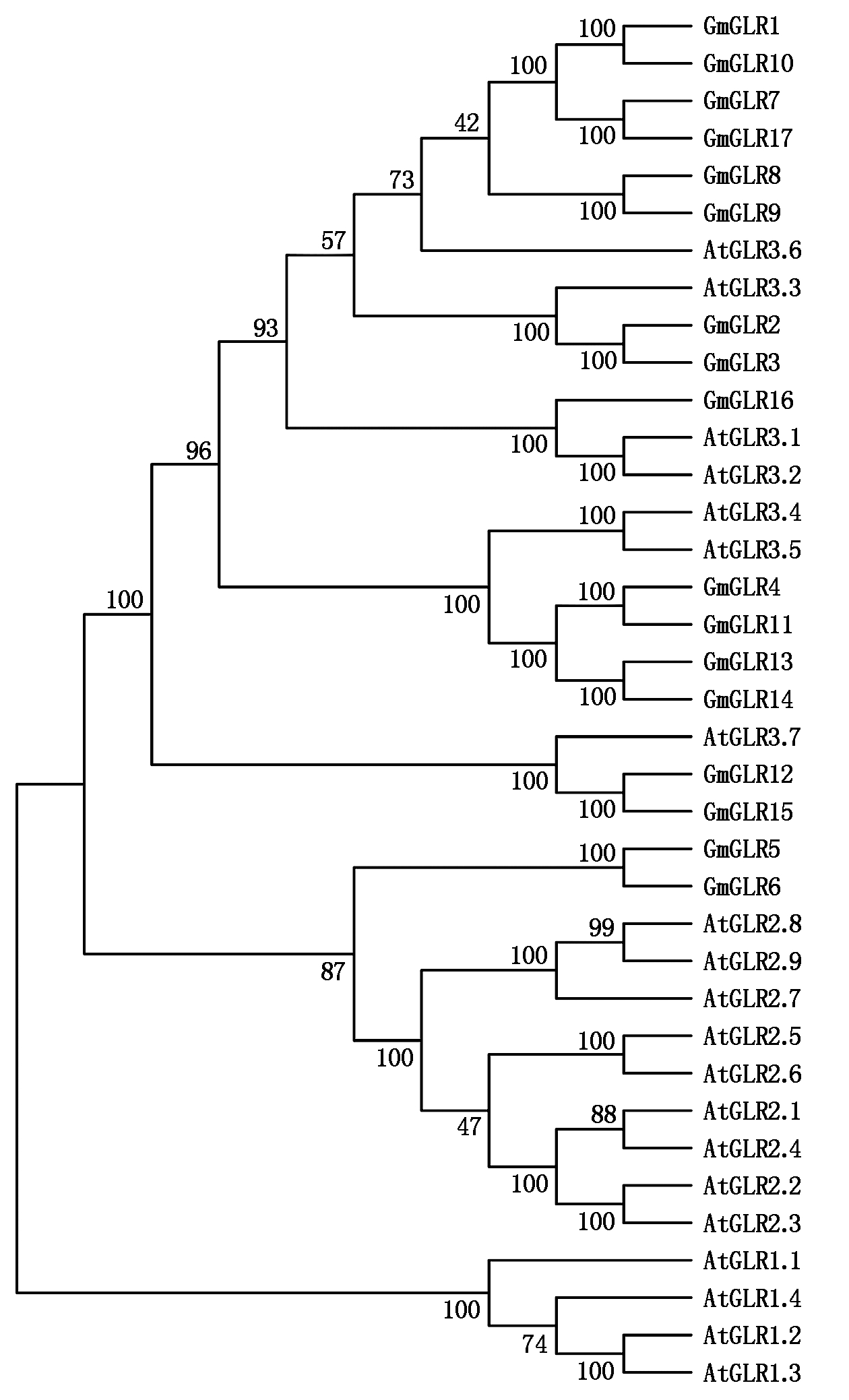
图2 大豆与拟南芥GLRs蛋白序列的系统进化树
Fig.2 The phylogenetic tree of GLRs protein in soybean and Arabidopsis
2.4 GmGLRs基因结构分析
根据搜索鉴定获得17个GmGLRs基因的基因组编码区序列和CDS序列,用GSDS 2.0软件在线绘制GmGLRs基因结构图,同时分析这17个GmGLRs基因的外显子和内含子情况。总体来看,除GmGLR8基因,其他16个GmGLRs基因都包含6个外显子和5个内含子(图3)。从外显子的长度分析保守性,保守性较高的外显子依次为E4、E3、E5,E4长度为32 bp的基因有14个,E3长度为278 bp的基因有13个,E5长度为407 bp的基因有9个(表3)。同源基因在基因结构上也表现出高度一致,其中GmGLR1和GmGLR10、GmGLR12和GmGLR15、GmGLR13和GmGLR14,所对应的外显子长度都一样;GmGLR2和GmGLR3、GmGLR7和GmGLR17,除E6以外的其他外显子长度是一样的;GmGLR4和GmGLR11,除E1和E6以外的外显子长度是一样的;同源基因长度不同的外显子差异也不大,所对应的内含子的长度差别也不大(表3)。GmGLR8基因结构差异的原因,可能是在进化过程中,第一个外显子插入一个内含子造成的。
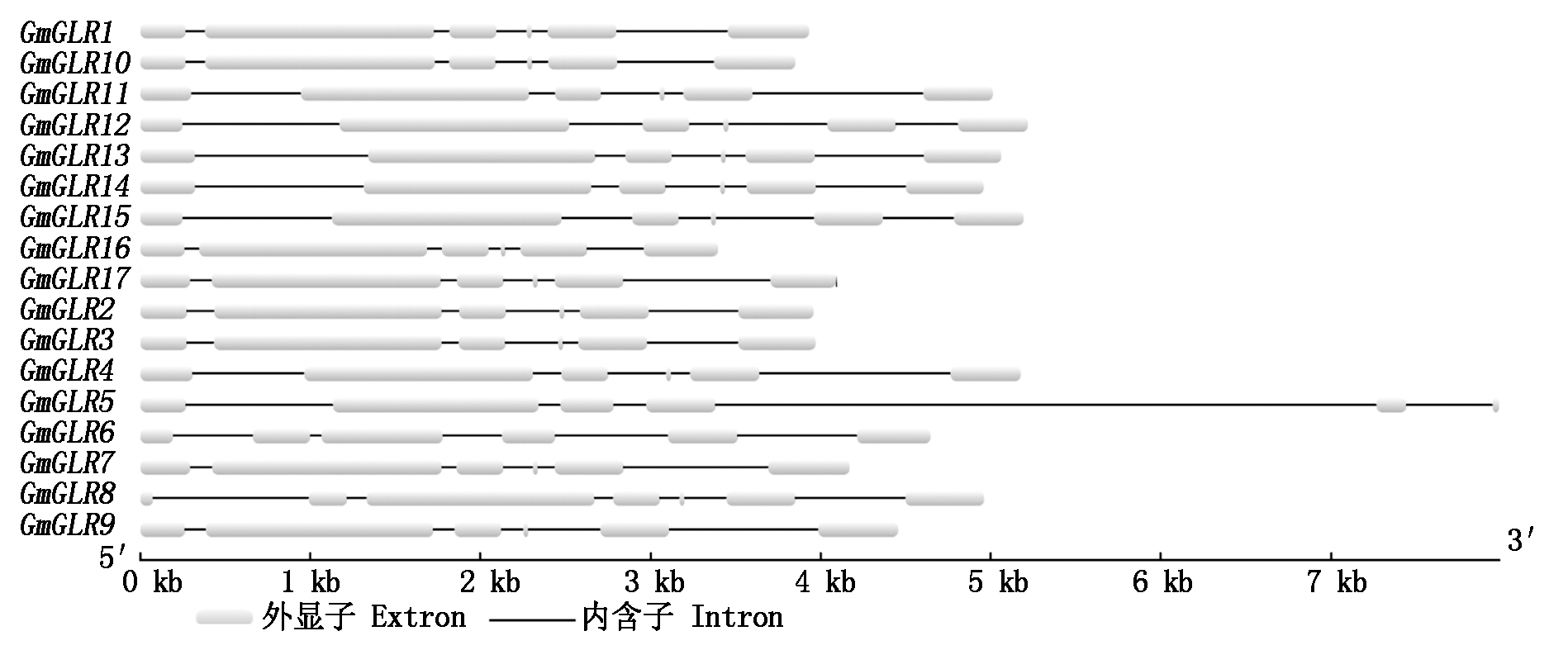
图3 GmGLRs基因外显子-内含子结构示意图
Fig.3 The diagram of extron and intron structure within GmGLRs
表3 GmGLRs基因外显子和内含子的比较
Tab.3 Comparation of exons and introns in GmGLRs genes bp
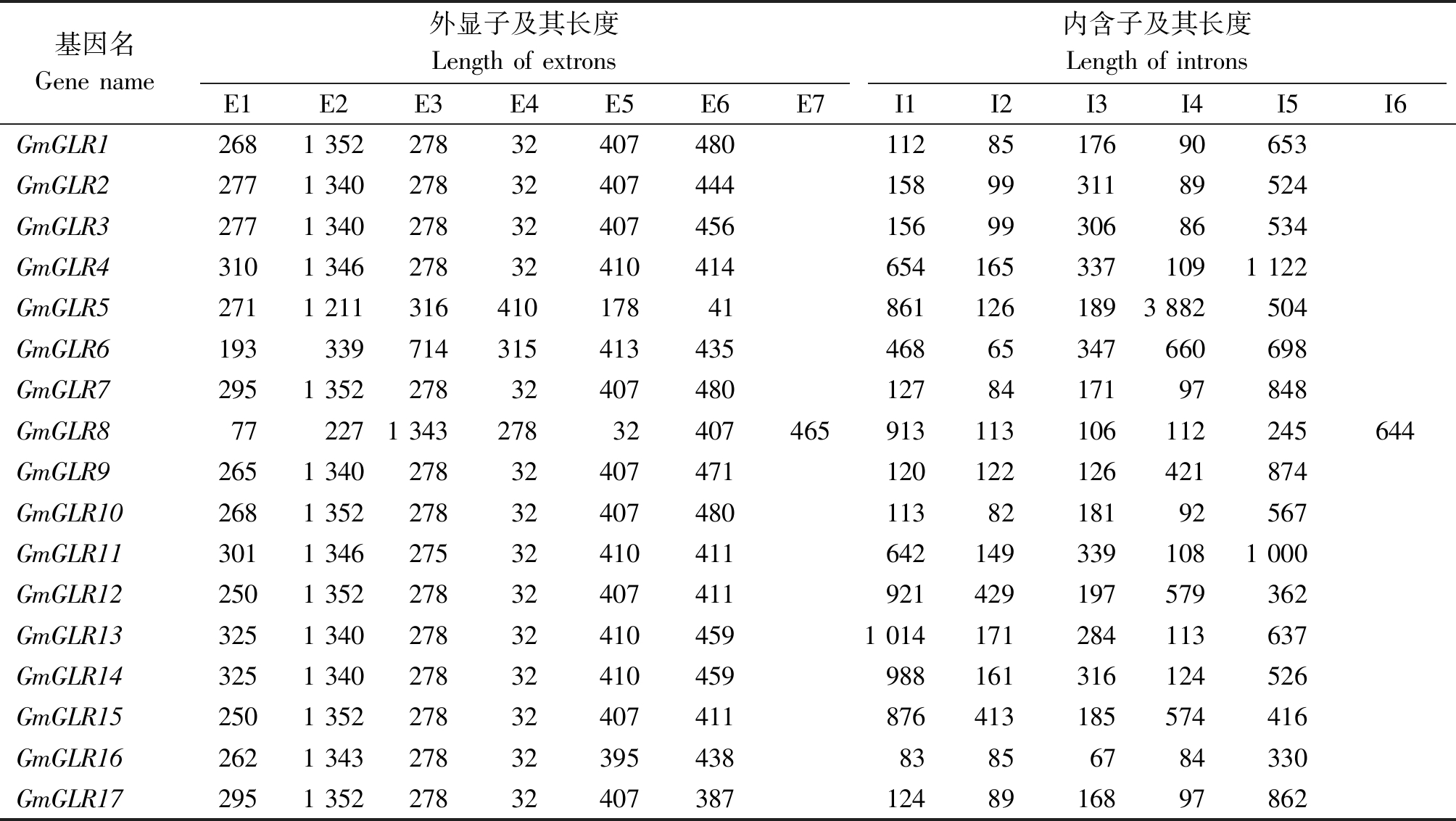
基因名Gene name外显子及其长度Length of extrons内含子及其长度Length of intronsE1E2E3E4E5E6E7I1I2I3I4I5I6GmGLR12681 352278324074801128517690653GmGLR22771 340278324074441589931189524GmGLR32771 340278324074561569930686534GmGLR43101 346278324104146541653371091 122GmGLR52711 211316410178418611261893 882504GmGLR619333971431541343546865347660698GmGLR72951 352278324074801278417197848GmGLR8772271 34327832407465913113106112245644GmGLR92651 34027832407471120122126421874GmGLR102681 352278324074801138218192567GmGLR113011 346275324104116421493391081 000GmGLR122501 35227832407411921429197579362GmGLR133251 340278324104591 014171284113637GmGLR143251 34027832410459988161316124526GmGLR152501 35227832407411876413185574416GmGLR162621 3432783239543883856784330GmGLR172951 352278324073871248916897862
2.5 GmGLRs跨膜结构域预测
利用TMHMM Server v.2.0 在线预测GmGLRs 蛋白跨膜结构域,如表4所示,10 个GmGLRs蛋白有3个跨膜结构域,4个GmGLRs蛋白有4 个跨膜结构域,2 个GmGLRs蛋白有5个跨膜结构域,1个GmGLRs蛋白有2 个跨膜结构域。同源基因的跨膜结构域在个数和位置上都表现高度一致,GmGLR2和GmGLR3、GmGLR7和GmGLR17,这3对等位基因跨膜结构域的位置都一致;GmGLR1和GmGLR10、GmGLR12和GmGLR15,这2对等位基因对应跨膜结构域的位置只有几个氨基酸的差异。GmGLRs 的跨膜位置多位于580-870 个氨基酸,该区域也是保守结构域相对集中的区域,由此可见,跨膜结构域在GmGLRs的生理功能上发挥重要作用。
2.6 GmGLRs基因组织表达模式分析
利用Phytozome 数据库中GmGLRs基因的组织表达量数据(FPKM),对GmGLRs基因在花、叶、根瘤、豆荚、根、根毛、种子、顶端生长点、茎等9个组织部位的表达模式进行分析,结果如图4所示。17个GmGLRs基因的表达都没有表现出组织特异性的差异,但是在表达丰度上存在显著差异,有8个基因高丰度表达,5个基因中丰度表达,4个基因低丰度表达。同源基因具有相似的表达趋势,GmGLR7和GmGLR17、GmGLR4和GmGLR11、GmGLR12和GmGLR15、GmGLR5和GmGLR6,这些同源基因在表达丰度和不同组织部位表达量上趋于一致。从不同组织部位表达高低的角度来看,GmGLR1在叶中表达量最高,GmGLR7在种子中表达量最高,GmGLR15在根中表达量最高,GmGLR12在顶端生长点中表达量最高,GmGLR8在花、叶和根中表达量最低,GmGLR6在根瘤、种子和顶端生长点中表达量最低。
表4 GmGLRs蛋白跨膜域预测
Tab.4 Prediction of the transmembrane regions of GmGLRs proteins

蛋白名Protein name跨膜位置Transmembrane positions跨膜数Transmembrane numberGmGLR1590-612,651-673,834-8563GmGLR23-25,589-611,623-640,655-677,833-8555GmGLR33-25,589-611,623-640,655-677,833-8555GmGLR47-24,602-624,661-683,842-8644GmGLR5545-567,611-6332GmGLR6467-489,533-555,717-7393GmGLR7599-621,660-682,843-8623GmGLR8599-621,660-682,844-8633GmGLR9585-607,646-668,829-8483GmGLR10594-612,649-671,834-8563GmGLR11598-620,657-679,838-8603GmGLR12584-606,651-673,830-8523GmGLR1312-34,605-627,664-686,847-8694GmGLR1412-34,605-627,664-686,847-8694GmGLR15584-606,651-673,827-8493GmGLR165-24,585-607,646-668,824-8464GmGLR17599-621,660-682,844-8633

F.花;L.叶;N.根瘤;P.豆荚;R.根;RH.根毛;Se.种子;SAM.顶端生长点;St.茎。 F.Flower;L.Leaves;N.Nodules;P.Pod;R.Root;RH.Root hair;Se.Seed;SAM.Shoot apical meristem;St.Stem.
图4 GmGLRs基因组织表达模式
Fig.4 The different tissues expression profile of GmGLRs
3 结论与讨论
结构域是生物大分子中具有特异结构和独立功能的区域,也是蛋白质功能单元[20],具有相同结构域的蛋白质分子在理论上具有相同生物学功能的可能性最大。拟南芥AtGLRs所有的成员都具有IPR001638、IPR001828、IPR001320和IPR017103这4个结构域,因此,在筛选其他植物基因组中GLRs的时候,也应以此为参考[3],所以本研究所最终确定的17个GmGLRs成员也是同时包含这4个结构域。
拟南芥的AtGLRs基因根据系统进化的分析结果分成3个亚家族[21],水稻的OsGLRs基因根据系统进化的结果分成4个亚家族,都有基因同拟南芥的3个亚家族聚类到一起,还有一个分支的7个基因有别于这3个亚家族[2];番茄SlGLRs基因也分成3个亚家族,其中有2个亚家族同拟南芥的第二和第三亚家族聚类到一起,另一个亚家族单独聚到一个分支[3];苹果的MdGLRs基因同拟南芥的系统进化结果一致,分成3个亚家族[4]。本研究结果显示,大豆的GmGLRs基因只包含同拟南芥AtGLRs基因第二和第三亚家族聚类到一起基因,缺少同拟南芥AtGLRs基因第一亚家族一致的基因,而且17个GmGLRs基因有15个同拟南芥AtGLRs基因第三亚家族聚类在一起,表明大豆的GmGLRs基因在生理功能上可能更趋同于拟南芥AtGLRs基因第三亚家族。
先前的研究表明,植物GLRs多拷贝基因通常成串地排列在同一条染色体上[2,3,22],大豆的GmGLRs基因也表现出了类似的特性。大豆基因组在进化过程中发生过大规模复制事件,使得大豆的很多基因表现出成对出现的分布模式[15,23-24],大豆的17个GmGLRs基因有8对基因成对出现,也表现出了大豆基因的这一普遍规律。大豆的17个GmGLRs基因中的同源基因,不管是在基因内含子-外显子结构上,还是跨膜结构域的存在形式上,以及基因组织表达模式上都表现出了高度一致性,表明这些基因的分子生物学功能也可能存在重复。
[1] Lam H M, Chiu J, Hsieh M H, Meisel L, Oliveira I C, Shin M, Coruzzi G. Glutamate-receptor genes in plants[J]. Nature, 1998, 396(6707): 125-126. doi:10.1038/24066.
[2] Singh A, Kanwar P, Yadav A K, Mishra M, Jha S K, Baranwal V, Pandey A, Kappor S, Tyagi A K, Pandey G K. Genome-wide expressional and functional analysis of calcium transport elements during abiotic stress and development in rice[J]. The FEBS Journal, 2014, 281(3): 894-915. doi:10.1111/febs.12656.
[3] Aouini A, Matsukura C, Ezura H, Asamizu E. Characterization of 13 glutamate receptor-like genes encoded in the tomato genome by structure phylogeny and expression profiles[J]. Gene, 2012, 493(1): 36-43. doi:10.1016/j.gene.2011.11.037.
[4] 罗华, 胡大刚, 张连忠, 郝玉金. 苹果MdGLRs家族基因生物信息学鉴定和表达分析[J]. 园艺学报, 2012, 39(3): 425-435. doi:10.16420/j.issn.0513-353x.2012.03.006.
Luo H, Hu D G, Zhang L Z, Hao Y J. Bioinformatics and expression analysis of apple MdGLRs genes family[J]. Acta Horticulturae Sinica, 2012, 39(3): 425-435.
[5] 何明洁, 孙伊辰, 程晓园, 时冬雪, 李迪秦, 陈益银, 冯永坤, 刘璐, 范腾飞, 杨超, 曹凤秋, 刘来华. 植物谷氨酸受体研究进展[J]. 植物学报, 2016, 51(6): 827-840. doi:10.11983/CBB15212.
He M J, Sun Y C, Cheng X Y, Shi D X, Li D Q, Chen Y Y, Feng Y K, Liu L, Fan T F, Yang C, Cao F Q, Liu L H. Current research advances on glutamate receptors(GLRs) in plants[J]. Chinese Bulletin of Botany, 2016, 51(6): 827-840.
[6] Vincill E D, Clarin A E, Molenda J N, Spalding E P. Interacting glutamate receptor-like proteins in phloem regulate lateral root initiation in Arabidopsis[J].The Plant Cell, 2013, 25(4): 1304-1313. doi:10.1105/tpc.113.110668.
[7] Kang J M, Turano F J. The putative glutamate receptor1.1(AtGLR1.1) functions as a regulator of carbon and nitrogen metabolism in Arabidopsis thaliana[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(11): 6872-6877. doi:10.1073/pnas.1030961100.
[8] Kang J M, Mehta S, Turano F J. The putative glutamate receptor 1.1(AtGLR1.1) in Arabidopsis thaliana regulates abscisic acid biosynthesis and signaling to control development and water loss[J]. Plant and Cell Physiology, 2004, 45(10): 1380-1389. doi:10.1093/pcp/pch159.
[9] Zheng Y, Luo L D, Wei J J, Chen Q, Yang Y P, Hu X Y, Kong X X. The glutamate receptors AtGLR1.2 and AtGLR1.3 increase cold tolerance by regulating jasmonate signaling in Arabidopsis thaliana[J]. Biochemical and Biophysical Research Communications, 2018, 506(4): 895-900. doi:10.1016/j.bbrc.2018.10.153.
[10] Kong D D, Ju C L, Parihar A, Kim S, Cho D, Kwak J M. Arabidopsis glutamate receptor homolog atglr3.5 modulates cytosolic Ca2+ level to counteract effect of abscisic acid in seed germination[J]. Plant Physiology, 2015,167(4): 1630-1642. doi:10.1104/pp.114.251298.
[11] Cho D, Kim S A, Murata Y, Lee S, Jae S K, Nam H G, Kwak J M. De-regulated expression of the plant glutamate receptor homolog AtGLR3.1 impairs long-term Ca2+ programmed stomatal closure[J].The Plant Journal, 2009, 58(3): 437-449. doi:10.1111/j.1365-313X.2009.03789.x.
[12] Meyerhoff O, Müller K, Roelfsema M R G, Latz A, Lacombe B, Hedrich R, Dietrich P, Becker D. AtGLR3.4, a glutamate receptor channel-like gene is sensitive to touch and cold[J]. Planta, 2005, 222(3): 418-427. doi:10.1007/s00425-005-1551-3.
[13] Li J, Zhu S H, Song X W, Shen Y, Chen H M, Yu J, Yi K, Liu Y F, Karplus V J, Wu P, Deng X W. A rice glutamate receptor-like gene is critical for the division and survival of individual cells in the root apical meristem[J]. The Plant Cell, 2006, 18(2): 340-349. doi:10.1105/tpc.105.037713.
[14] Kang S, Kim H B, Lee H, Choi J Y, Heu S, Oh C J, Kwon S I, An C S. Overexpression in Arabidopsis of a plasma membrane-targeting glutamate receptor from small radish increases glutamate-mediated Ca2+ influx and delays fungal infection[J]. Molecular Cell, 2006, 21(3): 418-427.
[15] Schmutz J, Cannon S B, Schlueter J, Ma J X, Mitros T, Nelson W, Hyten D L, Song Q J, Thelen J J, Cheng J L, Xu D, Hellsten U, May G D, Yu Y, Sakurai T, Umezawa T, Bhattacharyya M K, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S Q, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J C, Tian Z X, Zhu L C, Gill N, Joshi T, Libault M, Sethuraman A, Zhang X C, Shinozaki K, Nguyen H T, Wing R A, Cregan P, Specht J, Grimwood J, Rokhsar D, Stacey G, Shoemaker R C, Jackson S A.Erratum: genome sequence of the palaeopolyploid soybean[J]. Nature, 2010, 465(7278): 178-183. doi:10.1038/nature08957.
[16] Shen Y T, Liu J, Geng H Y, Zhang J X, Liu Y C, Zhang H K, Xing S L, Du J C, Ma S S, Tian Z X. De novo assembly of a Chinese soybean genome [J]. Science China Life Sciences, 2018, 61(8): 871-884. doi:10.1007/s11427-018-9360-0.
[17] Hu B, Jin J P, Guo A Y, Zhang H, Luo J C, Gao G. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(8): 1296-1297. doi:10.1093/bioinformatics/btu817.
[18] 才晓溪, 沈阳, 周伍红, 贾博为, 孙明哲, 王金玉, 杨珺凯, 李建伟, 孙晓丽. 大豆CHX基因家族全基因组鉴定与生物信息学分析[J]. 基因组学与应用生物学, 2018, 37(12): 5360-5369. doi:10.13417/j.gab.037.005360.
Cai X X, Shen Y, Zhou W H, Jia B W, Sun M Z, Wang J Y, Yang J K, Li J W, Sun X L. Genome-wide identification and bioinformatics analysis of soybean CHX gene family[J]. Genomics and Applied Biology, 2018, 37(12): 5360-5369.
[19] 郭栋, 杜媚, 周宝元, 刘颖慧, 赵明. 玉米SAUR基因家族的鉴定与生物信息学分析[J]. 植物遗传资源学报, 2019, 20(1): 90-99. doi:10.13430/j.cnki.jpgr.20180707001.
Guo D,Du M,Zhou B Y,Liu Y H,Zhao M. Identification and bioinformatics analysis of maize SAUR gene family[J]. Journal of Plant Genetic Resources, 2019, 20(1): 90-99.
[20] 李志强, 廖隆理.生皮化学与组织学[M]. 北京: 中国轻工业出版社, 2010: 34.
Li Z Q, Liao L L. Skin chemistry and histology[M].Beijing: China Light Industry Press, 2010: 34.
[21] Lacombe B, Becker D, Hedrich R, DeSalle R, Hollmann M, Kwak J M, Schroeder J I, Le Novère N, Nam H G, Spalding E P, Tester M, Turano F J, Chiu J, Coruzzi G. The identity of plant glutamate recaptors[J]. Science, 2001, 292(5521): 1486-1487. doi:10.1126/science.292.5521.1486b.
[22] Chiu J C, Brenner E D, Desalle R, Nitabach M N, Holmes T C, Coruzzi G M. Phylogenetic and expression analysis of the glutamate-receptor-like gene family in Arabidopsis thaliana[J]. Molecular Biology and Evolution, 2002, 19(7): 1066-1082. doi:10.1093/oxfordjournals.molbev.a004165.
[23] Li X Y, Dhaubhadel S. Soybean 14-3-3 gene family: identification and molecular characterization[J]. Planta, 2011, 233(3): 569-582. doi:10.1007/s00425-010-1315-6.
[24] 常玮, 王娟, 于洋, 陈吉宝. 大豆NUDX基因家族全基因组分析[J]. 西北农林科技大学学报(自然科学版), 2018, 46(9): 27-34. doi:10.13207/j.cnki.jnwafu.2018.09.004.
Chang W, Wang J, Yu Y, Chen J B. Genome-wide analysis of soybean NUDX gene family[J]. Journal of Northwest A&F University(Natural Science Edition), 2018, 46(9): 27-34.
