根际是土壤-植物生态系统物质交换的活跃界面。根际存在着许多对植物有益的微生物群落,包括生防菌、能生产植物生长激素及固氮的菌种等[1]。内生真菌(Endophytic fungi)是一类在其生活史中至少有一个阶段生活在植物组织内,但不会对植物造成显著不良影响的真菌[2]。内生菌可以与植物宿主相互作用,互惠共生,不仅可以提高植物抗生物逆境(抗病、抗虫及其他食草动物)和抗非生物逆境能力,而且可以帮助植物吸收磷、氮和硫及微量元素[3]。据报道,从大豆根瘤中分离到的内生菌株DD287具有抗盐碱性,并对大豆生长具有促进作用[4];野生辣木(Moringa peregrina)中分离到的菌株LK12和LK13可以产生赤霉素(GAs),显著促进GAs-缺陷型突变体水稻和正常水稻苗的生长[5];野生大麦(Hordeum murinum ssp. Murinum)中分离到菌株可以在营养缺乏条件下增加大麦的产量[2],并显著降低大麦种子感染病原菌的概率[6];暗隔内生真菌(DSE)结合丛枝菌根真菌(AFM)可以显著促进黄瓜生长,提高产量,增强抗根结线虫病害的能力[7]。综上所述,根际或内生真菌具有防病促生长作用,而且安全、高效。因此,利用生防菌控制作物病害已成为目前植物病害防治研究热点之一。
韭菜(Allium tuberosum Rottler ex Sprengel)是我国最常见的一种普通蔬菜。在前期研究中发现香蕉与韭菜轮/间作能显著降低香蕉枯萎病的发病率,减轻病害程度[8],也能显著降低番茄、黄瓜等蔬菜的根结线虫病害[9]。据此,推测韭菜中可能含有某些活性物质,能有效杀死病原菌及根结线虫,随后前人也分离到一些杀菌物质[10]和杀线虫物质[11]。进一步研究发现,韭菜处理能影响香蕉根际微生物种群的结构[12]。因此,推测韭菜中除了含有活性物质外,也可能存在一些能杀死病原菌或者根结线虫的内生菌或者根际微生物。随后,通过16S rRNA分析了韭菜根际和内生细菌[13],得出了一些有益的结论。但是韭菜根际和植物组织中还含有大量的真菌,这些真菌也具有生防菌的潜力,有必要对这些真菌的种群结构及多样性进行分析。因此,在前期的研究基础上,本研究拟通过Illumina技术对韭菜根际土壤、叶片和根系中的18S rRNA序列进行分析,对根际真菌及内生真菌(叶片和根系)群落进行比较,以期为进一步从韭菜中分离鉴定高效防控植物病原菌及根结线虫病害的生防真菌提供参考,也对推进植物真菌病害及根结线虫病害的防控具有积极意义。
1 材料和方法
1.1 样品收集
试验中所用的韭菜为大金钩品种,取自青岛城阳区惜福镇种植3年的田地。将带土的完整植株放入无菌袋带回实验室。抖动根系除去多余土壤,然后用毛刷收集黏在根上的土壤,过2 mm筛后作为根际土壤,用于分析根际真菌。然后将植株用水冲洗后,将叶片和根系分离,切成2 cm小段,75%乙醇中浸泡2 min,在3%次氯酸钠中浸泡5 min,用无菌水冲洗5次。将最后的洗涤液涂布在PDA平板上,置于28 ℃培养7 d,以7 d后无菌生长的为合格样品。根际土壤、叶片和根系分别设置3个重复,共9个样品,用于分析叶片和根系中的内生真菌。
1.2 PCR扩增及Illumina测序
将土壤和消毒后的叶片和根系提取总DNA,用OD260/280及琼脂糖凝胶电泳检验质量及完整性。用PCR扩增仪器GeneAmp PCR System 9700(Life Technologies,Carlsbad,CA,USA)对微生物的18S基因进行扩增,引物为EF4(GGAAGGGRTGTATTTATTAG)和NS2(GGCTGCTGGCACCAGACTTGC)[14-15]。使用KAPA HiFi HotStart ReadyMix PCR Kit试剂盒扩增。扩增体系为25 μL:12.5 μL 2× KAPA HiFi HotStart ReadyMix,引物各0.25 μmol/L,10 ng模板DNA。PCR扩增程序为:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,25个循环;72 ℃ 5 min。1%琼脂糖胶电泳检测PCR产物,纯化回收,用Agilent2100 Bioanalyzer System(Santa Clara,CA,USA)进行定量,等量混合。由北京源宜基因科技股份有限公司构建文库,使用Illumina Miseq平台进行测序。
1.3 统计及生物信息学分析
使用MOTHUR软件[16]除去初始序列中低质量(得分≤20)序列、去除含N序列、去除同聚体过长序列、去除引物错配过多及引物序列、去除过短(≤200 bp)和过长(≥500 bp)序列、去除嵌合体,最终得到高质量的序列。根据序列相似度,使用USEARCH[17]将序列聚类为操作分类单元(Operational Taxonomic Units,OTU),相似度阈值为97%;使用RDP分类器[18]以SILVA数据库[19]为参比,对OTU 序列进行系统分类;使用MOTHUR[16]计算Alpha多样性指数(Chao、Shannon、Simpson);使用PyNAST[20]将OTU序列比对到SILVA数据库[19],用FastTree[21]构建系统发育树,用UniFrac[22]生成群落间距离矩阵,对UniFrac矩阵用custom R scripts制作Heatmap,Canoco 4.5制作主成分分析(PCA);使用Metastats[23]检验组间物种相对丰度的差异。数据的差异显著性分析用SAS 8.0软件中最小极差法(LSD)进行。
2 结果与分析
2.1 Illumine测序结果
利用MOTHUR软件对拼接后的序列进行过滤后,从根际土壤、叶片和根系组织的9个样品中总共得到348 484条高质量的序列。单个样品中的高质量序列数为20 717~45 531。将这些序列用USEARCH软件以97%的相似度阈值进行聚类,共得到123个OTU(以OTU为基础进行多样性分析)。根际真菌和内生真菌的覆盖率指数(Coverage)为0.92~1.00,并没有显著差异(表1),说明测序深度涵盖的大部分是微生物,所得的序列可以进行进一步的分析。
2.2 根际真菌和内生真菌群落组成
根际土壤真菌群落和内生真菌群落显著不同(图1)。在根际土壤中,共鉴定到5个菌门。其中含量最高的2个菌门为子囊菌门(Ascomycota)和担子菌门(Basidiomycota),含量分别为67.20%和24.44%,占总含量的91.64%。叶片中共检测到了4个菌门,其中含量最高的2个菌门子囊菌门(91.39%)和担子菌门(8.07%)总计为总含量的99.46%。而根系中检测到2个菌门:子囊菌门(96.60%)和担子菌门(2.52%),总计为总含量的99.12%。将鉴定到的5个菌门进行聚类发现,含量最高的2个菌门子囊菌门和担子菌门分别聚为一类,而另外3个含量很低的菌门聚为一类(图2)。
在检测的9个样品中共检测到17个菌纲、29个菌目和35个菌科,其中含量最高的3个菌纲在根际土壤、叶片和根系中含量分别为71.70%,93.86%,97.17%;3个菌目分别为55.84%,91.20%,97.17%;3个菌科分别为53.62%,90.28%,96.29%。而且根际真菌和内生真菌在纲、目、科3个分类级别上都具有显著差异(表2)。
在属的水平上,根际土壤中共检测到35个菌属。35个菌属中含量最高的3个菌属含量总计为53.62%;叶片中共鉴定到20个菌属,含量最高的3个属总计为总含量的90.28%;而根系中只检测到7个菌属,含量最高的3个属含量总计为96.29%。统计分析表明,在属的水平上,根际土壤真菌和内生真菌存在显著差异(表2)。
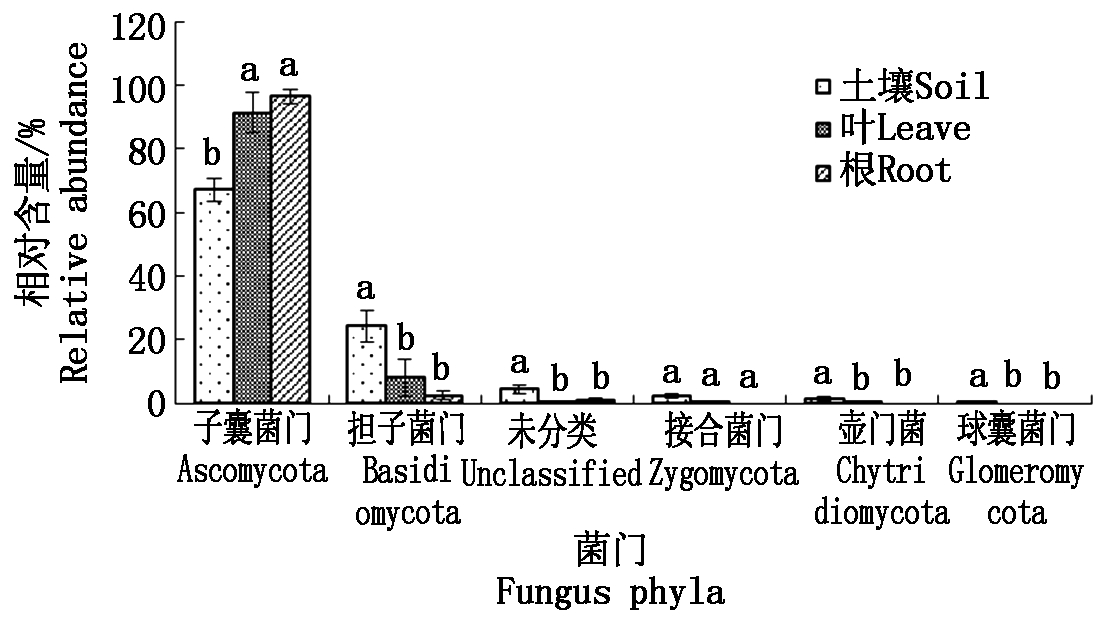
图1 根际土壤、叶片和根系中不同菌门真菌类群相对丰度图
Fig.1 The relative abundance of different phyla in rhizosphere soil,leaf and root fungal communities of Chinese leek
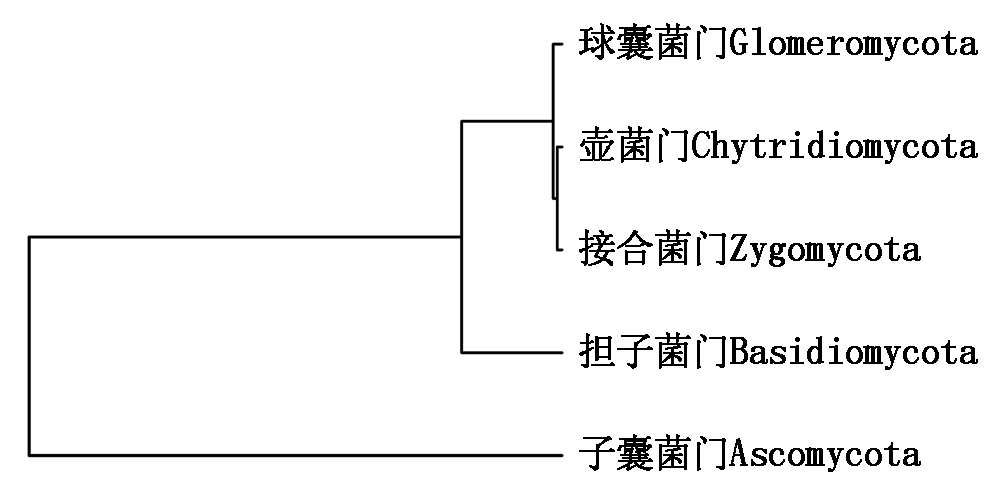
图2 韭菜根际土壤、叶片和根系中真菌在门水平上的聚类
Fig.2 Hierarchical cluster tree of fungal communities from the rhizosphere,leaf and root of Chinese leekat the phylum level
2.3 根际真菌和内生真菌多样性比较
α-多样性参数中(表1),根际真菌的OTUs显著高于叶片和根系,分别为叶片和根系的5.63,14.65倍;在根际真菌中,Chao指数分别为叶片和根系的3.71,9.21倍;ACE指数也分别为叶片和根系2.02,3.85倍,这些数据说明根际真菌的丰富度明显高于内生真菌。根际真菌的Shannon指数分别是叶片和根系的1.98,3.26倍,而Simpson指数仅为叶片和根系的0.26,0.18倍,这些数据说明根际真菌的多样性显著高于内生真菌。
2.4 根际真菌和内生真菌的比较
试验结果表明:根际真菌和内生真菌具有显著的差异。在门的水平上,叶片和根系中的子囊菌门的含量分别为根际土壤中的1.36,1.44倍。除此之外,其他各个菌门含量均低于根际土壤(图1)。在纲、目、科的水平上存在类似的含量关系。在属的水平上,在检测到的35个菌属中,有14个菌属在根际真菌和内生真菌中的含量存在显著差异。叶片和根系中含量最高的菌属分别为根际真菌的2.63,3.53倍,其余菌属含量均显著低于根际真菌。而叶片和根系相比,在门、纲、目、科、属的水平上都存在一些差异,但是差异并不显著。这说明根际真菌和内生真菌的含量存在显著差异,但叶片和根系中内生真菌含量基本相似(表2)。
表1 韭菜根际土壤、叶片和根系真菌的α-多样性指数
Tab.1 α- diversity index of fungal communities from the rhizosphere,leaf and root of Chinese leek
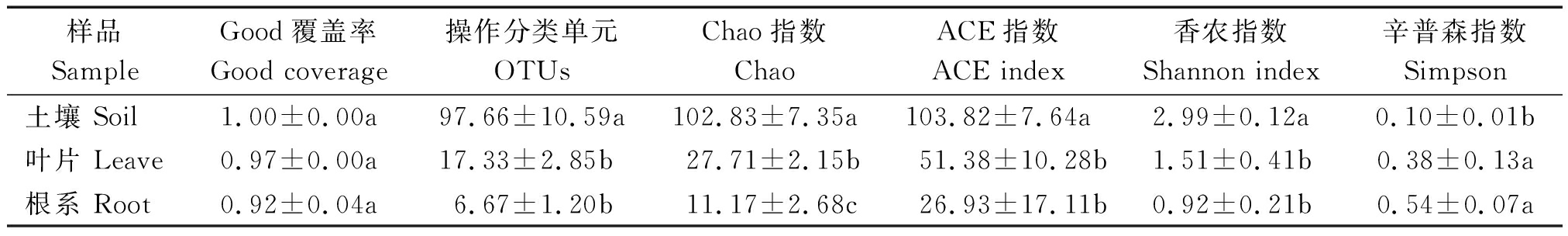
样品SampleGood覆盖率Good coverage操作分类单元OTUsChao指数ChaoACE指数ACE index香农指数Shannon index辛普森指数Simpson土壤 Soil1.00±0.00a97.66±10.59a102.83±7.35a103.82±7.64a2.99±0.12a0.10±0.01b叶片 Leave0.97±0.00a17.33±2.85b27.71±2.15b51.38±10.28b1.51±0.41b0.38±0.13a根系 Root0.92±0.04a6.67±1.20b11.17±2.68c26.93±17.11b0.92±0.21b0.54±0.07a
表2 韭菜根际和内生真菌中具有显著差异的纲、目、科、属
Tab.2 The class,order,family and genera showing significant differences between rhizpsphere or endophytic fungal communities
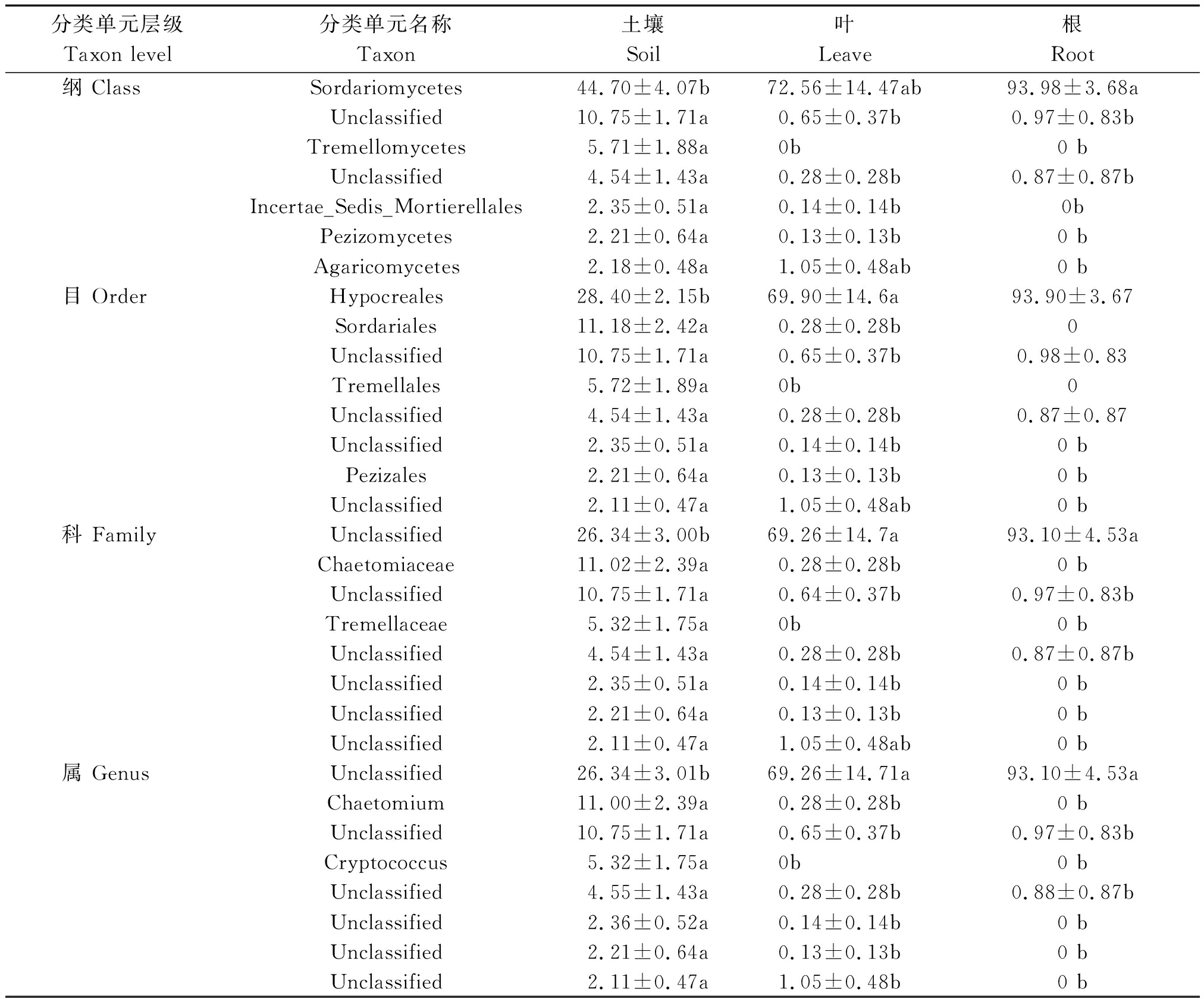
分类单元层级Taxon level分类单元名称Taxon土壤Soil叶Leave根Root纲 ClassSordariomycetes44.70±4.07b72.56±14.47ab93.98±3.68aUnclassified10.75±1.71a0.65±0.37b0.97±0.83bTremellomycetes5.71±1.88a0b0 bUnclassified4.54±1.43a0.28±0.28b0.87±0.87bIncertae_Sedis_Mortierellales2.35±0.51a0.14±0.14b0bPezizomycetes2.21±0.64a0.13±0.13b0 bAgaricomycetes2.18±0.48a1.05±0.48ab0 b目 OrderHypocreales28.40±2.15b69.90±14.6a93.90±3.67Sordariales11.18±2.42a0.28±0.28b0Unclassified10.75±1.71a0.65±0.37b0.98±0.83Tremellales5.72±1.89a0b0Unclassified4.54±1.43a0.28±0.28b0.87±0.87Unclassified2.35±0.51a0.14±0.14b0 bPezizales2.21±0.64a0.13±0.13b0 bUnclassified2.11±0.47a1.05±0.48ab0 b科 FamilyUnclassified26.34±3.00b69.26±14.7a93.10±4.53aChaetomiaceae11.02±2.39a0.28±0.28b0 bUnclassified10.75±1.71a0.64±0.37b0.97±0.83bTremellaceae5.32±1.75a0b0 bUnclassified4.54±1.43a0.28±0.28b0.87±0.87bUnclassified2.35±0.51a0.14±0.14b0 bUnclassified2.21±0.64a0.13±0.13b0 bUnclassified2.11±0.47a1.05±0.48ab0 b属 GenusUnclassified26.34±3.01b69.26±14.71a93.10±4.53aChaetomium11.00±2.39a0.28±0.28b0 bUnclassified10.75±1.71a0.65±0.37b0.97±0.83bCryptococcus5.32±1.75a0b0 bUnclassified4.55±1.43a0.28±0.28b0.88±0.87bUnclassified2.36±0.52a0.14±0.14b0 bUnclassified2.21±0.64a0.13±0.13b0 bUnclassified2.11±0.47a1.05±0.48b0 b
注:表中仅列出根际真菌中含量>1%的纲、目、科和属。
Note:Only the fungal class,order,family and genera in rhizosphere soil that the amounts were higher than 1% was listed in the table.
为了分析根际真菌和内生真菌的关系,用PyNAST软件将OTU序列比对到SILVA数据库,用FastTree构建系统发育树,用UniFrac生成群落间矩阵。UniFrac距离矩阵分为2种:Unweighted UniFrac 和Weighted UniFrac。其中Unweighted UniFrac聚类热图显示(图3-A),根际土壤的3个样品聚为一类,而叶片和根系的6个样品聚为一类。在Unweighted UniFrac PCA中,第一主成分将根际真菌和内生真菌区分开来,而且第二主成分可以将叶片和根系的内生真菌区分开来(图4-A); 但是Weighted UniFrac聚类热图显示(图3-B),根际土壤的2个样品聚为一类,而叶片和根系的6个样品和根际土壤中的1个样品聚为一类。在Unweighted UniFrac PCA中,第一主成分和第二主成分不能将各个样品明确地区分开来,但是也可以明确地看出根际真菌和内生真菌分为两大类(图4-B),说明根际真菌和内生真菌种群结构组成存在显著差异。
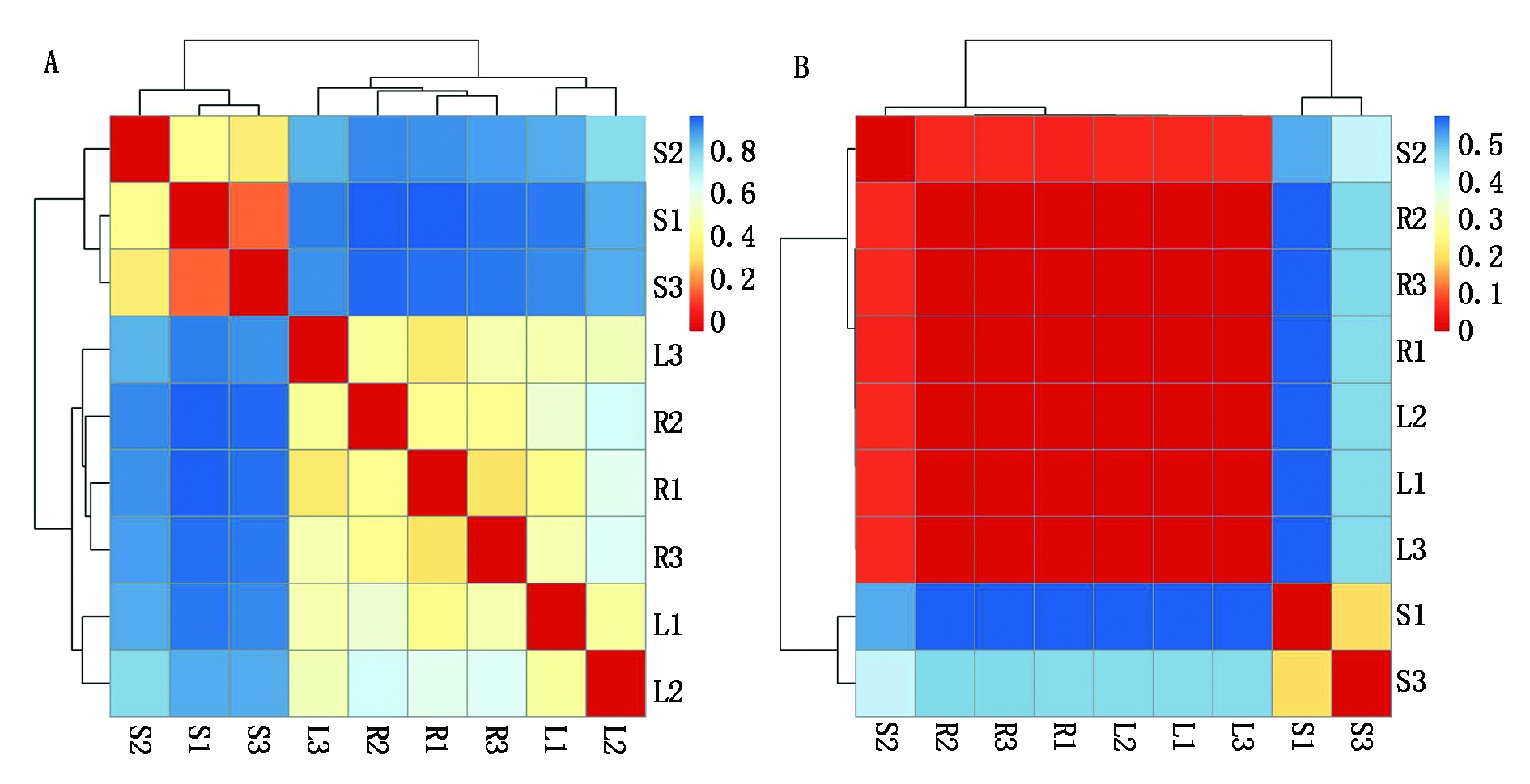
颜色从蓝色渐变到红色表示样品间相似度增高;R.根际土壤;L.叶;R.根。
The color gradient from blue to red indicated increasing similarity;R.Rhizosphere soil; L.Leaf; R.Root.
图3 韭菜根际真菌和内生真菌的Unweighted(A)和Weighted(B)UniFrac距离热图
Fig.3 Heatmap of the rhizosphere and endophytic fungal communities of Chinese leek based on of Unweighted(A) and Weighted(B)UniFrac distance
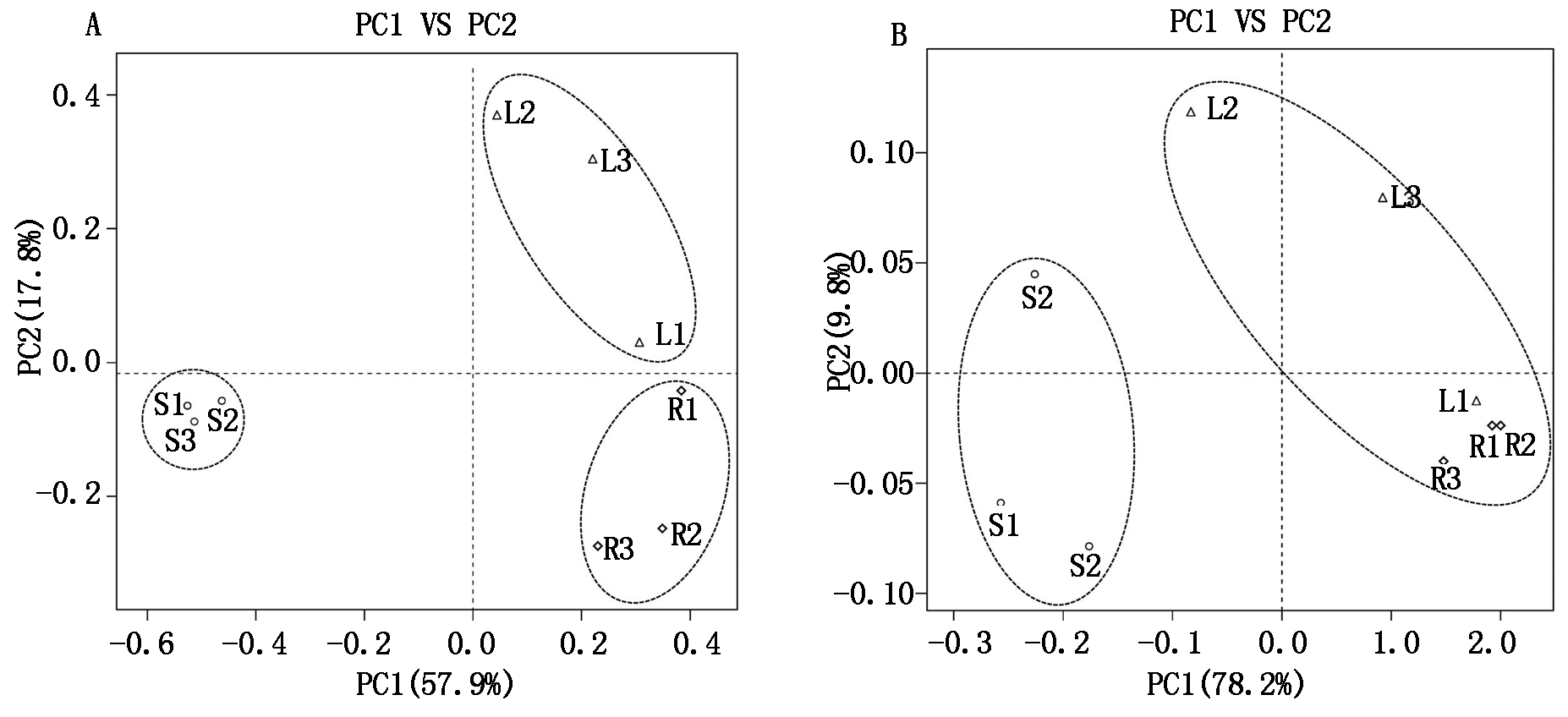
S.根际土壤;L.叶;R.根。
S.Rhizosphere soil; L.Leaf; R.Root.
图4 韭菜根际真菌和内生真菌的Unweighted(A)和Weighted(B)UniFrac主成分分析
Fig.4 Principal component analysis(PCA)of the rhizosphere and endophytic fungal communities of Chinese leek ased on of Unweighted(A) and Weighted(B)UniFrac
3 讨论
通过高通量测序技术分析微生物种群多样性已经在藻类[24]、昆虫[25]、动物[26-27]、水体[28]和土壤[29-31]等多方面得到应用。在本研究中,采用该技术对韭菜根际真菌(根际土壤)和内生真菌(叶片和根系)种群做了分析,一方面对两类真菌的种群结构进行深入的了解,并明确了两类菌群的差异,另一方面挖掘了大量的未分类菌群,丰富了菌种资源,该研究对韭菜内生真菌和根际真菌的开发利用提供了参考。
从种群结构上来看,根际真菌中含有已分类的5个菌门,而在叶片和根系中含有4个菌门和2个菌门。从数量上看,在鉴定到的种群中,子囊菌门在根际真菌、叶片和根系中分别为67.20%,91.39%,96.60%,形成了绝对的优势菌群。在香蕉根际土壤中, 子囊菌门含量为42%,是优势种群[32];在豌豆(Pisum sativum)根际土壤中,子囊菌门为55.6%,同样是优势种群[33]。在α-多样性指数(Chao、ACE、Shannon等)的分析中,所有指数显著高于叶片和根系,说明根际真菌的多样性、丰富度均显著高于内生真菌。而叶片和根系相比,叶片的OTUs、Chao、ACE、Shannon等参数分别为根系的2.60,2.48,1.90,41.65倍,但是统计分析表明差异不显著。根际作为植物体内和体外环境联系的场所,在根际经常发生包括根系与根系、根系与昆虫、根系与微生物之间的互作[34]。而这些复杂的交互作用导致了根际微生物的多样性。相对根际区域,植物体内相对稳定单一的环境,造就了相对单一的微生物种群。也就是说,根际真菌群落多样化和内生菌群落的相对单一化都是对各自环境及功能适应的结果。
在根际真菌和内生真菌的β-多样性分析中,建立了Unweighted UniFrac 和Weighted UniFrac 2种UniFrac距离矩阵。Unweighted UniFrac只考虑OTU组成,不考虑其丰度,而Weighted UniFrac 则考虑其丰度。通过2种矩阵建立的聚类热图和PCA结果均不相同。按照Unweighted UniFrac建立的聚类热图和PCA能确切地将根际真菌和内生真菌区分开来,而且可以将叶片和根系的内生真菌也区分开来,而Weighted UniFrac的区分效果不是很理想。这说明内生真菌和根际真菌在种群结构上是有显著差别的,而当考虑丰度时,各个样品之间的差异就不如单纯比较种群结构组成明显。这可能是由于大田土壤中真菌分布情况、韭菜本身生长情况以及试验误差等种种客观和主观原因造成了真菌丰度检测结果的差异,导致了Unweighted UniFrac和Weighted UniFrac 2种矩阵分析上出现了一些偏差。这种情况在前人研究中也出现过[35]。虽然2种矩阵建立的聚类热图和PCA结果稍有差异,但是都能清晰地反映内生真菌和根际真菌种群结构上的差异。
在以往的研究中,通常用传统的培养方法研究微生物群落[36-37],但是绝大多数的微生物不能用传统方法进行培养,而DNA测序的方法可以挖掘到新的微生物[38]。与传统的培养方法相比,通过高通测序技术可以检测到大量的微量微生物群落,其中许多在现存的库中不存在(即未分类序列/菌门/菌属等)。在本研究中,在门的水平上,未分类菌门占4.54%,而在鉴定的35个菌属中,有24个为未分类菌,占总含量的76.75%。前人通过高通量测序技术研究土壤微生物时,也发现了大量的未分类菌群:在香蕉根际土壤中,89 332(58%)为分类序列[32];施用氯苯嘧啶醇后,草坪根际土壤真菌中有19%为未分类菌门,对照中有34%为未分类菌门[39];在犬粪便微生物中,4个样品中的未分类菌属分别为39.36%,52.29%,37.34%和25.66%[40];烤烟-小麦和烤烟-油菜连作以及烤烟-小麦-玉米和烤烟-油菜-玉米轮作4种种植模式连续种植10后,根际土壤中未分类菌属分别为24.95%,20.88%,11.40%和14.03%[31]。本研究及前人研究中发现的大量的未分类菌群,一方面说明了高通量测序在鉴定微生物,尤其微量、稀有及不能培养的种群时具有明显的优势,另一方面也说明了韭菜根际及内生真菌中有大量的未知种类有待开发,而这些新发现的未分类种群为生防菌的开发和利用提供了新的资源。
在本研究中,韭菜根际土壤、叶片和根系的18S rRNA 高通量测序结果表明,韭菜根际真菌和内生真菌(叶片和根系)的种群结构有显著差异。韭菜根际真菌的多样性、丰富度均显著高于内生真菌。韭菜根际真菌群落主要由子囊菌门和担子菌门2个菌门组成;而内生真菌主要由子囊菌门单独一个菌门组成。叶片和根系中内生真菌稍有不同,但是差异并不显著。该研究为分离鉴定韭菜中抑制植物病原菌及致死根结线虫的生防真菌提供参考。
[1] 陆雅海,张福锁. 根际微生物研究进展[J]. 土壤,2006,38(2):113-121.
[2] Murphy B R,Doohan F M,Hodkinson T R. Fungal root endophytes of a wild barley species increase yield in a nutrient-stressed barley cultivar[J]. Symbiosis,2015,65(1):1-7.
[3] 姜道宏. 植物内生真菌及其展望[J]. 中国生物防治学报,2015,31(5):742-749.
[4] 赵龙飞,徐亚军,常佳丽. 轮纹病菌拮抗性大豆根瘤内生菌的筛选、抗性和促生作用[J]. 中国生物防治学报,2016,32(3):396-405.
[5] Khan A L,Waqas M,Hussain J,et al. Endophytes Aspergillus caespitosus LK12 and Phoma sp.LK13 of Moringa peregrina produce gibberellins and improve rice plant growth[J]. Journal of Plant Interactions,2014,9(1):731-737.
[6] Murphy B R,Doohan F M,Hodkinson T R. Persistent fungal root endophytes isolated from a wild barley species suppress seed-borne infections in a barley cultivar[J]. BioControl,2015,60(2):281-292.
[7] 高春梅,李 敏,刘润进. AMF和DSE组合菌剂促生防线虫病效应[J]. 菌物学报,2016,35(10):1208-1217.
[8] Huang Y H,Wang R C,Li C H,et al. Control of fusarium wilt in banana with Chinese leek[J]. European Journal of Plant Pathology,2012,134(1):87-95.
[9] Huang Y H, Mao Z C, Xie B Y. Chinese leek(Allium tuberosum Rottler ex Sprengel)reduced disease symptom caused by root-knot nematode[J]. Journal of Integrative Agriculture,2016,15(2):364-372.
[10] Zuo C W,Li C Y,Li B,et al. The toxic mechanism and bioactive components of Chinese leek root exudates acting against Fusarium oxysporum f. sp cubense tropical race 4[J]. European Journal of Plant Pathology,2015,143(3):447-460.
[11] 黄永红.韭菜对根结线虫的防控作用及其活性成分分析研究[D].北京:中国农业科学院, 2015.
[12] 黄永红,吕 顺,李春雨,等. 香蕉枯萎病菌4号生理小种对香蕉根际土壤微生物及酶活性的影响[J]. 湖南农业大学学报:自然科学版,2012,38(2):173-176.
[13] Huang Y H. Comparison of rhizosphere and endophytic microbial communities of Chinese leek through high-throughput 16S rRNA gene Illumina sequencing[J]. Journal of Integrative Agriculture,2018,17(2):359-367.
[14] Tasumi E,Kanazawa S,Fukuda S. Degradation of a herbicide,beflubutamid,in upland soils and isolation of its degrading microbes[J]. Soil Science & Plant Nutrition,2007,53(3):236-245.
[15] Smit E,Leeflang P,Glandorf B,et al. Analysis of fungal diversity in the wheat rhizosphere by sequencing of cloned PCR-amplified genes encoding 18S rRNA and temperature gradient gel electrophoresis[J]. Applied & Environmental Microbiology,1999,65(6):2614-2621.
[16] Schloss P D,Westcott S L,Ryabin T,et al. Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology,2009,75(23):7537-7541.
[17] Edgar R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods,2013,10(10):996-998.
[18] Wang Q,Garrity G M,Tiedje J M,et al. Na?ve bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied & Environmental Microbiology,2007,73(16):5261-5267.
[19] Pruesse E,Quast C,Knittel K,et al. SILVA:a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB[J]. Nucleic Acids Research,2007,35(21):7188-7196.
[20] Caporaso J G,Bittinger K,Bushman F D,et al. PyNAST:a flexible tool for aligning sequences to a template alignment[J]. Bioinformatics,2010,26(2):266-267.
[21] Price M N,Dehal P S,Arkin A P. FastTree:computing large minimum evolution trees with profiles instead of a distance matrix[J]. Molecular Biology and Evolution,2009,26(7):1641-1650.
[22] Lozupone C,Knight R. UniFrac:a new phylogenetic method for comparing microbial communities[J]. Applied and Environmental Microbiology,2005,71(12):8228-8235.
[23] White J R,Nagarajan N,Pop M. Statistical methods for detecting differentially abundant features in clinical metagenomic samples[J]. PLoS Computational Biology,2009,5(4):e1000352.
[24] 宋 伦,刘卫东,吴 景,等. 有害甲藻Stoeckeria algicida在辽东湾的时空分布[J]. 生态学报,2017,37(4):1339-1345.
[25] 王雪庆,于淑惠,孙 涛,等. 白蜡虫蜡花微生物多样性分析[J]. 昆虫学报,2016,59(10):1086-1092.
[26] Jia H R,Geng L L,Li Y H,et al. The effects of Bt Cry1le toxin on bacterial diversity in the midgut of Apis mellifera ligustica(Hymenoptera:Apidae)[J]. Scientific Reports,2016,6:24664).
[27] Guo J J,Fu X L,Liao H D,et al. Potential use of bacterial community succession for estimating post-mortem interval as revealed by high-throughput sequencing[J]. Scientific Reports,2016,6:24197.
[28] Jiao S,Liu Z S,Lin Y B,et al. Bacterial communities in oil contaminated soils:Biogeography and co-occurrence patterns[J]. Soil Biology & Biochemistry,2016,98:64-73.
[29] Wang J,Xue C,Song Y,et al. Wheat and rice growth stages and fertilization regimes alter Soil bacterial community structure,but not diversity[J]. Frontiers in Microbiology,2016,7:1207.
[30] Fang H,Lian J J,Wang H F,et al. Exploring bacterial community structure and function associated with atrazine biodegradation in repeatedly treated soils[J]. Journal of Hazardous Materials,2015,286:457-465.
[31] 陈丹梅,陈晓明,梁永江,等. 种植模式对土壤酶活性和真菌群落的影响[J]. 草业学报,2015,24(2):77-84.
[32] Shen Z Z,Ruan Y Z,Chao X E,et al. Rhizosphere microbial community manipulated by 2 years of consecutive biofertilizer application associated with banana Fusarium wilt disease suppression[J]. Biology and Fertility of Soils,2015,51(5):553-562.
[33] Yu L,Nicolaisen M,Larsen J,et al. Molecular characterization of root-associated fungal communities in relation to health status of Pisum sativum using barcoded pyrosequencing[J]. Plant and Soil,2012,357(1-2):395-405.
[34] Bais H P,Weir T L,Perry L G,et al. The role of root exudates in rhizosphere interactions with plants and other organisms[J]. Annual Review of Plant Biology,2006,57:233-266.
[35] Shen Z Z,Wang D S,Ruan Y Z,et al. Deep 16S rRNA pyrosequencing reveals a bacterial community associated with banana fusarium wilt disease suppression induced by Bio-Organic fertilizer application[J]. PLoS One,2014,9(5):e98420.
[36] Koukol O,Kolarik M,Kolarova Z,et al. Diversity of foliar endophytes in wind-fallen Picea abies trees[J]. Fungal Diversity,2012,54(1):69-77.
[37] Compant S,Mitter B,Gualberto Colli-Mull J A,et al. Endophytes of grapevine flowers,berries,and seeds:identification of cultivable bacteria,comparison with other plant parts,and visualization of niches of colonization[J]. Microbial Ecology,2011,62(1):188-197.
[38] Masuko K,Murata M,Yudoh K,et al. Ion torrent PGM as Tool for fungal community analysis:a case study of endophytes in eucalyptus grandis reveals high taxonomic diversity[J]. PLoS One,2013,8(12):e81718.
[39] 赵 爽,张 宁,刘东阳,等. 氯苯嘧啶醇施用对草坪斑枯病致病菌及根际土壤真菌的影响[J]. 南京农业大学学报,2015,38(4):590-595.
[40] Wu X,Zhang H,Chen J,et al. Comparison of the fecal microbiota of dholes high-throughput Illumina sequencing of the V3-V4 region of the 16S rRNA gene[J]. Applied Microbiology and Biotechnology,2016,100(8):3577-3586.