中国是家禽遗传资源非常丰富的国家,养鹅的历史也比较悠久,因此,形成了众多的各有特色的地方品种。随着鹅产业的快速发展,市场过分追求经济利益最大化的性状,许多生产性能低下的地方品种受到了一定程度的冲击,有被淘汰、灭绝的趋势。为了保持鹅种遗传资源的多样性,需要对一些重要的地方品种进行保护。目前,活体保种主要有原种场原地保种和基因库异地保种等方法。太湖鹅作为江苏地区比较重要的一个地方鹅种,以肉质细腻、高产、适应性强等特点闻名于世,并被列入国家级保护名录。由于受到外来品种的冲击,以及近亲交配导致的品种衰退,太湖鹅数量不断下降。目前,保种群体主要存在于原种场和国家水禽基因库。
由于不同时期分子生物学技术的发展,研究遗传多样性所采用的分子方法有着较大的差异。最初采用随机扩增多态性DNA(RFLP)、RAPD等第一代分子标记对基因组DNA进行分析。但该方法不能明确具体DNA序列,随后被第二代分子标记微卫星重复序列标记和第三代分子标记单核苷酸多态性标记取代。随着测序成本的降低,基于酶切的简化基因组测序技术开始用于群体遗传多样性标记的分析[1-6]。与早期的分子标记相比,简化基因组在全基因组水平研究分子标记的多样性,标记数量足够丰富;与全基因组重测序相比,成本较低,所以是非常理想的用于遗传多样性研究的方法。
为了全面评估太湖鹅原种场保种和基因库保种2种方法的保种效果,本试验利用基于酶切的简化基因组测序方法,对太湖鹅3种群体(原始群体TS、原种场群体TC和基因库群体TK)进行遗传多样性分析,比较了原种场群体和基因库群体的保种效果。研究结果将为下一步太湖鹅保种方法的改进提供理论依据。
1 材料和方法
1.1 试验动物
为了比较太湖鹅群体在原种场和国家水禽基因库分别采用不同保种方法导致的保种效果,采集3个太湖鹅群体TS(原始群体)、TC(原种场群体)和TK(基因库群体)血样。3个群体血统都来自于苏州太湖鹅原种场,其中TS群体血样为2007年在太湖鹅原种场所采血样,TC群体和TK群体血样分别为2017年在太湖鹅原种场和在国家水禽基因库采集。TS群体可视为TC群体和TK的祖先群体,通过TC群体和TK群体分别于与TS群体进行比较,分析太湖鹅原种场群体和国家水禽基因库群体的保种效果。
1.2 试验方法
1.2.1 ddRAD简化基因组测序 常规酚-氯仿法提取基因组DNA,采用ddRAD建库的方式构建长度为400~600 bp的pair-end文库,进行ddRAD简化基因组测序。
1.2.2 SNP检测 SNP检测主要使用GATK[7]软件工具包实现。根据Clean Reads在参考基因组的定位结果,使用samtools[8]进行去重复(Mark duplicates)、GATK进行局部重比对(Local realignment)、碱基质量校正(Base recalibration)等预处理,以保证检测得到的SNP结果准确性。质控条件分为2步,第1步为Q20(正确率99%的碱基数目比例)> 95%,ddRAD depth(双酶切基因组测序的覆盖深度)>60%,SNP call rate(在所有群体中单核苷酸多态性检出率)>70%,MAF<0.05;第2步在第1步质控的基础上,SNP call rate>90%(在单个群体中的检出率)。
1.3 数据处理
平均杂合度(Ho)、近交系数(FIS)和群体分化指数(FST)计算应用PopGen32[9]软件。群体结构分析采用admixture软件[10],选择信号采用PLINK软件,以100 kb为窗口,10 kb为步长在基因组上滑动计算区域内的FST和π值。
2 结果与分析
2.1 测序数据质量统计
3个群体测得的总reads数都达到了106级别,GC含量百分比均在41.5%左右(表1),属于正常范围。Q30百分比均在90%以上,Q20百分比达到97%,可以进行后续分析。
表1 三个群体测序数据质量统计
Tab.1 Quality statistics of sequencing data of three groups

群体 Group测序总reads数Read sum测序总碱基数Base sumGC含量百分比/%GC percentQ30百分比/%Q30 percentQ20百分比/%Q20 percent原始群体TS4.16×1061.20×10941.3191.73697.034原种场群体TC4.28×1061.23×10941.4191.95397.128基因库群体TK3.98×1061.15×10941.6991.54396.943
2.2 基因组SNP鉴定
经过2步数据质控,在原始群体、原种场群体和基因库群体3个太湖鹅群体中分别鉴定出SNP数目为3.56×105,4.00×105,3.44×105个(表2)。从基因组水平看,属于转换的SNP数量是属于颠换的SNP数量的2.4倍左右,说明SNP突变大多为转换型。
表2 三个群体SNP分布统计结果
Tab.2 The SNPs distribution of three groups
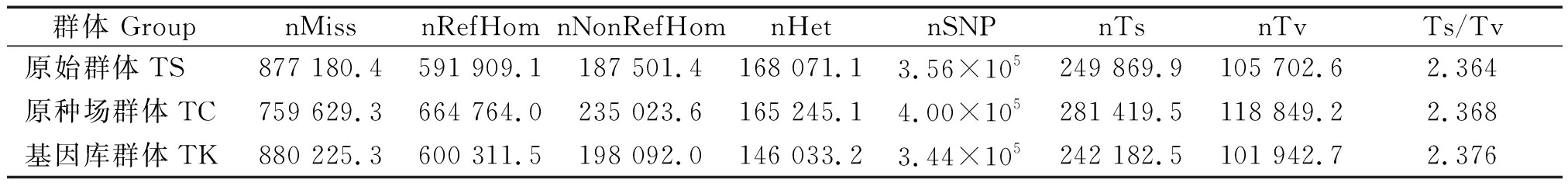
群体 GroupnMissnRefHomnNonRefHomnHetnSNPnTsnTvTs/Tv原始群体TS877 180.4591 909.1187 501.4168 071.13.56×105249 869.9105 702.62.364原种场群体TC759 629.3664 764.0235 023.6165 245.14.00×105281 419.5118 849.22.368基因库群体TK880 225.3600 311.5198 092.0146 033.23.44×105242 182.5101 942.72.376
注:nMiss.群体SNP中缺失位点数目;nRefHom.和参考序列上基因型一致的纯合位点数目;nNonRefHom.和参考序列上基因型不一致的纯合位点数目;nHet.杂合的位点数目;nSNP.和参考序列比总的SNP位点数目;nTs.转换类型的SNP数量;nTv.颠换类型的SNP数量;Ts/Tv.转换与颠换类型的SNP数量比值。
Note:nMiss.Number of missing SNP sites;nRefHom.Number of homozygous loci consistent with reference sequences; nNonRefHom.Number of homozygous loci not consistent with reference sequences; nHet.Number of heterozygous loci; nSNP.Total number of SNP sites compared to reference sequence; nTs.Number of SNPs belong to transition type; nTv.Number of SNPs belong to transvertion type; Ts/Tv.The number ration of transition SNPs to transversion SNPs.
2.3 群体内遗传多样性分析
根据变异位点对群体遗传学参数进行统计分析,结果见表3。TS群体、TC群体和TK群体的平均杂合度分别为0.295 5,0.266 2,0.258 2。TC群体和TK群体的平均杂合度明显低于TS群体,核苷酸多样性也低于TS群体。与TS群体相比,TC群体和TK群体的近交系数(FIS)明显上升。
表3 三个群体杂合度和近交系数统计
Tab.3 The heterozygosity and inbreeding coefficient of three populations of geese
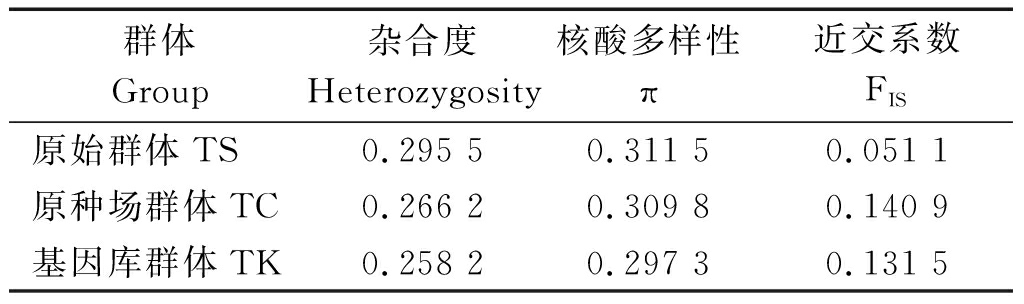
群体Group杂合度Heterozygosity核酸多样性π近交系数FIS原始群体TS0.295 50.311 50.051 1原种场群体TC0.266 20.309 80.140 9基因库群体TK0.258 20.297 30.131 5
2.4 群体间遗传分化分析
通过滑窗分析,根据各个位点的遗传分化系数(FST)计算不同群体间平均FST值。其中TC群体和TK群体之间的FST值为0.043 8,TC群体和TS群体之间的FST值为0.017 7,TK群体和TS群体之间的FST值为0.048 0。群体间FST值越高,说明群体间的序列差异越大。TK群体与另外2个群体的FST值均大于TS群体与TC群体的FST值,表明TK群体与其他2个群体的差异较大。
群体结构聚类(图1)表明,TC群体与TS群体共享更多的血统,TK群体与TS群体之间的共有血统较少。主成分分析结果(图2)表明,TC群体和TS群体距离较近,TK群体单独聚在一处。这与遗传分化指数的结果均表明,与TC群体相比,TK群体经过时间的推移有明显分化的趋势。
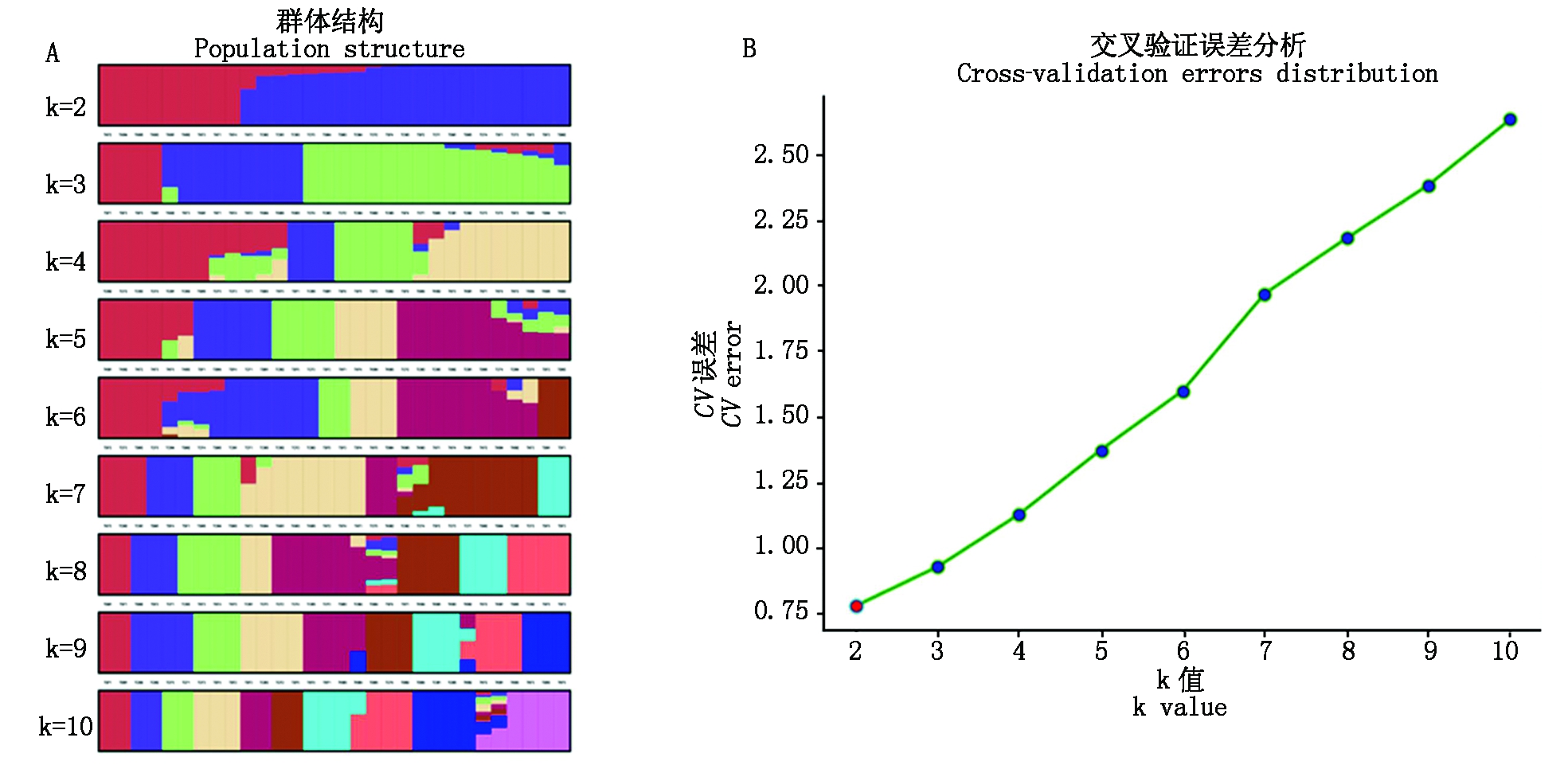
A.展示了原始群体(TS)、原种场群体(TC)和基因库群体(TK)在不同k值下的群体结构图,每种颜色代表一个分组;B.不同k值下群体结构的CV值曲线图。CV误差越小,越接近于群体本身的结构状态;本图说明k=2时较符合实际情况,即3个群体分为明显的两类,原始群体和基因库群体分别分为一类,而原种场群体具有较多的原始群体血统,以及少部分基因库群体血统。
A.Each different color tile denotes a sectionalization in population structure clustering graph with different k value of TS,TC and TK populations;B.Curve graph of CV error responding to different k value,the smaller CV error indicates more close to a real structural state of populations;This graph showed when k is equal 2,it is more in line with the actual situation,that is to say,three populations were classified to two groups,and TS and TK contain completely different kinships,while TC population with TS kinship mixed a bit of TK kinships.
图1 群体结构聚类图
Fig.1 The picture of population structure clustering
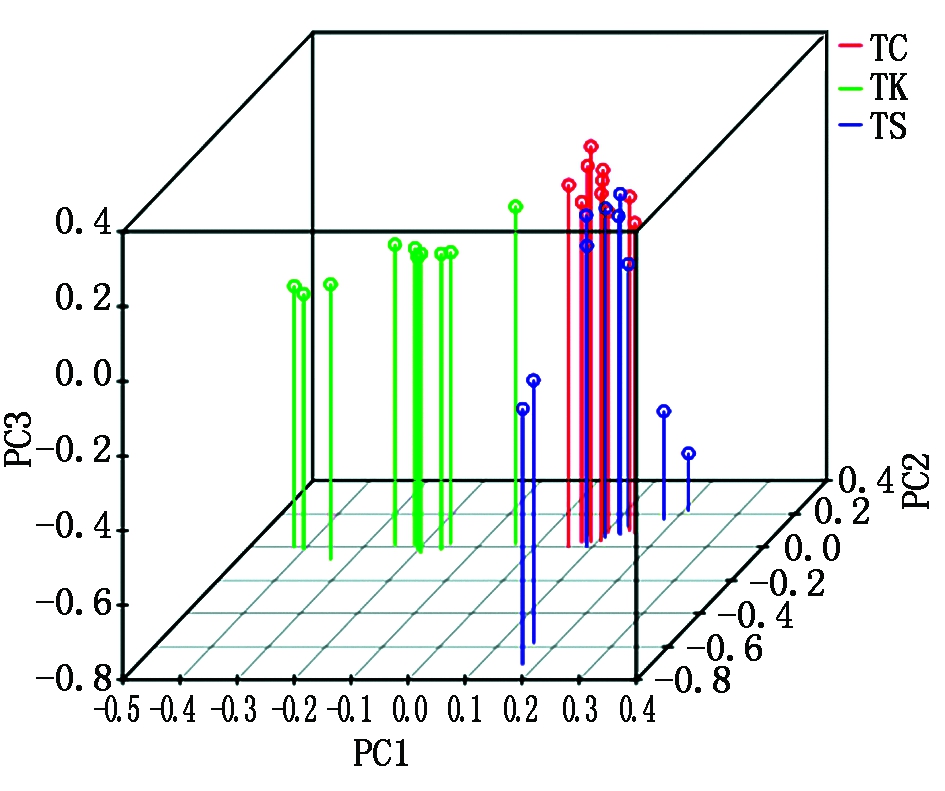
图2 三个群体主成分分析
Fig.2 PCA analysis of three populations
2.5 选择信号分析
结合核苷酸多样度θπ比率和遗传分化指数FST选择前5%的区域(图3)。TC群体与TS群体比较得到140个受选区域,TK群体与TS群体比较得到200个受选区域。在这些受选区域中鉴定受选基因,TC群体与TS群体比较得到431个受选基因,TK群体与TS群体比较得到633个受选基因,其中共有的受选基因79个。
GO分析结果表明,TC群体与TS群体相比,受选择基因主要富集在磷脂分解通路(GO:0009395,phospholipid catabolic process,corrected P-value=6.36×10-8)中。TK群体与TS群体相比,受选择基因没有显著富集到任何GO条目。
KEGG富集结果表明,TC群体与TS群体相比,受选基因主要富集在脂类代谢通路中(表4)。TK群体与TS群体相比,受选基因没有显著富集到任何KEGG通路。
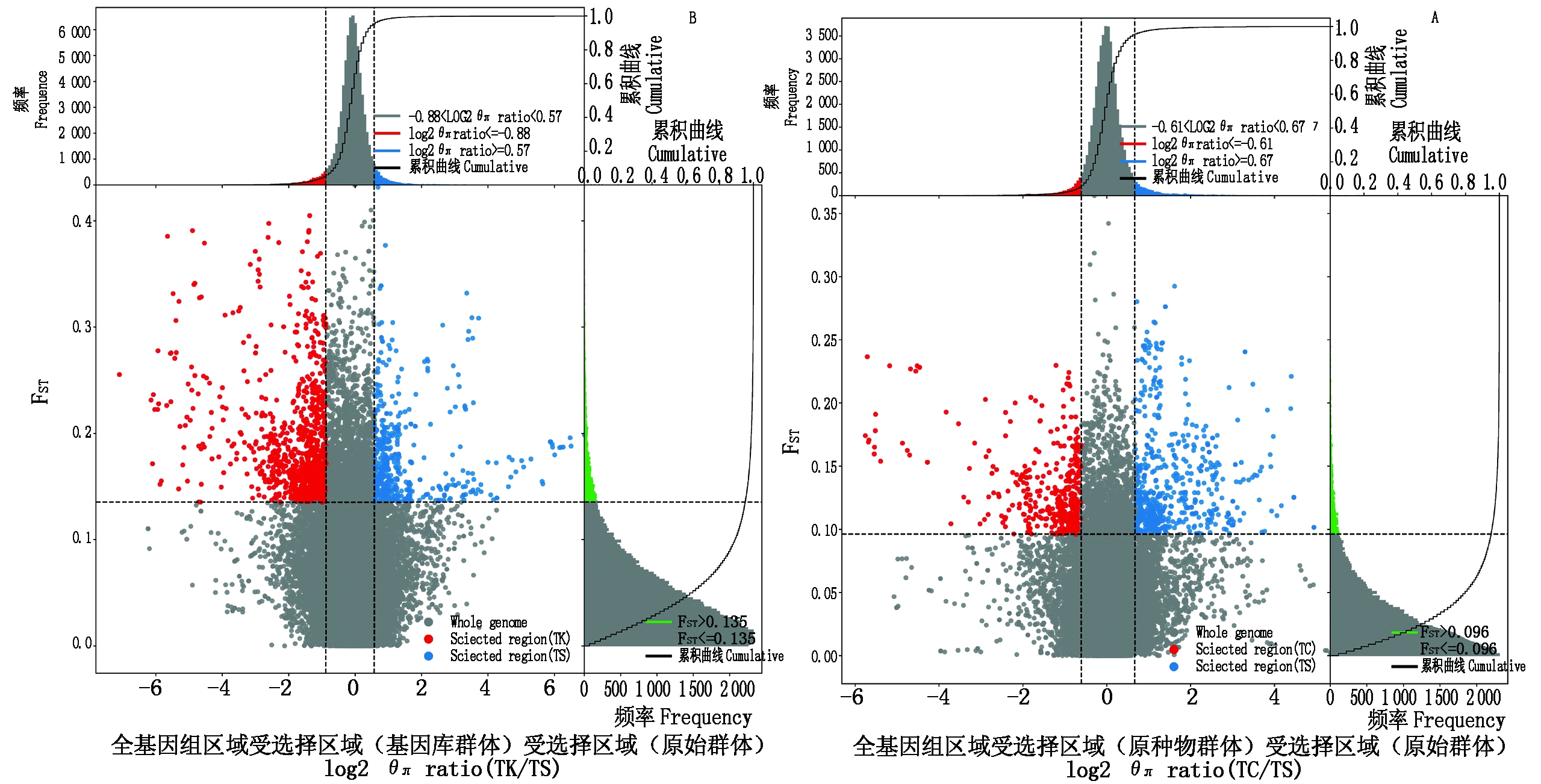
A.TC群体和TS群体之间比较选择信号;B.TK群体和TS群体之间比较选择信号;A图和B图中的红色点分别表示TC群体和TK群体的前5%受选区域,蓝色便表示TS群体的前5%受选区域。
A.The selection signal of TC population vs TS population; B.The selection signal of TK population vs TS population; The red points in A and B
indicate the selection regions of TC and TK populations respectively,and the blue points denote the selection regions of TS population.
图3 种群分化选择区域分布图
Fig.3 The regional distribution of population differentiation
表4 TC群体与TS群体比较的受选基因KEGG通路
Tab.4 KEGG pathway of selected genes between TC and TS populations
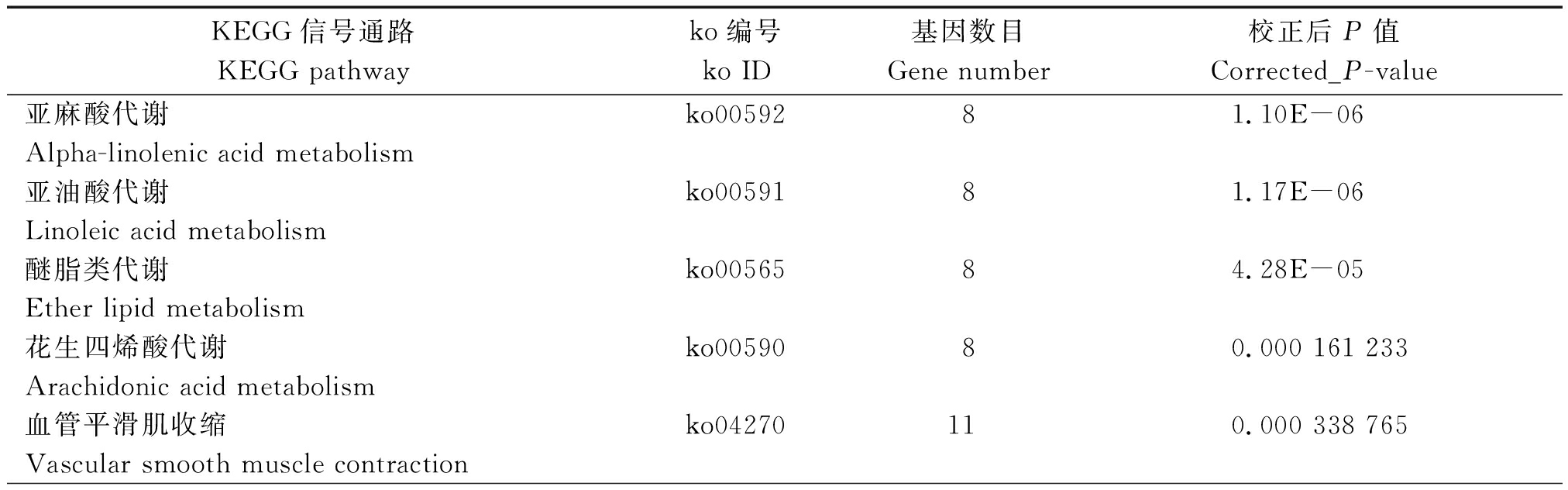
KEGG信号通路KEGG pathwayko编号ko ID基因数目Gene number校正后P值Corrected_P-value亚麻酸代谢ko0059281.10E-06Alpha-linolenic acid metabolism亚油酸代谢ko0059181.17E-06Linoleic acid metabolism醚脂类代谢ko0056584.28E-05Ether lipid metabolism花生四烯酸代谢ko0059080.000 161 233Arachidonic acid metabolism血管平滑肌收缩ko04270110.000 338 765Vascular smooth muscle contraction
表4(续)
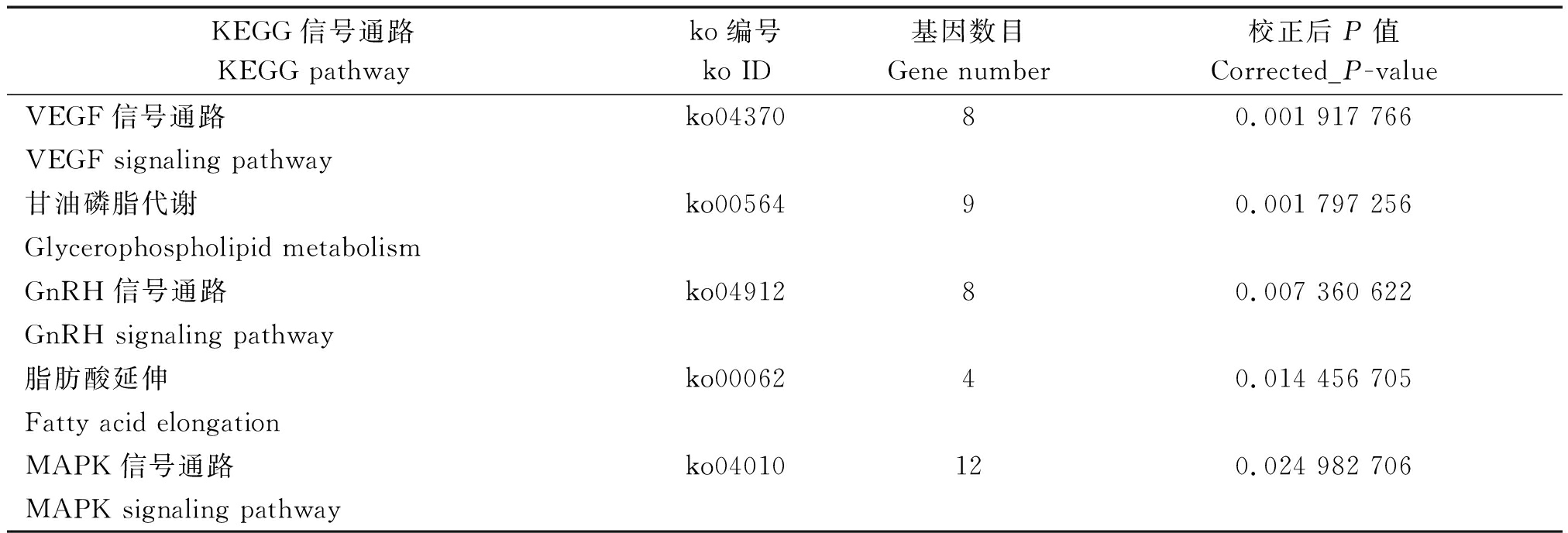
KEGG信号通路KEGG pathwayko编号ko ID基因数目Gene number校正后P值Corrected_P-valueVEGF信号通路ko0437080.001 917 766VEGF signaling pathway甘油磷脂代谢ko0056490.001 797 256Glycerophospholipid metabolismGnRH信号通路ko0491280.007 360 622GnRH signaling pathway脂肪酸延伸ko0006240.014 456 705Fatty acid elongationMAPK信号通路ko04010120.024 982 706MAPK signaling pathway
3 讨论与结论
动物保护的关键是维持物种的遗传多样性,即长期维持群体的遗传性和适应性的相对稳定,因此,种群遗传多样性的研究是保护生物学研究的核心内容之一[11]。切实有效的保护策略必须建立在对物种遗传多样性充分了解的基础上,而基于DNA序列变异的群体遗传多样性参数分析则成为衡量群体遗传结构和监测保种效果的主要指标。
为全面了解太湖鹅品种在原种场和国家水禽基因库的保种效果,本试验利用简化基因组测序方法研究全基因组范围内的遗传变异情况,分析各群体的杂合度、核酸多样性以及群体近交系数,并根据群体分化系数FST和群体之间核酸多样性π比值等选择信号联合筛选出群体之间遗传多样性具有显著差异的区域以及相关基因。GO和KEFF富集分析表明,TC群体与TS群体相比,脂类代谢相关基因受到了选择。
群体的遗传杂合度和核酸多样性能够正确反映群体遗传多样性。遗传杂合度(H)是度量遗传变异的一个最适参数,反映了各个群体在多个位点上的遗传变异[12]。以往多是根据线粒体DNA或微卫星等分子标记进行遗传多样性研究[13-16]。利用微卫星标记对家鸭细胞核DNA进行遗传多样性检测,发现家鸭的平均杂合度均大于0.5[17-20]。李慧芳等 [14]用微卫星标记研究我国重点保护的地方鹅品种的遗传多样性,发现平均遗传杂合度为0.577 4。汤青萍等 [21]、段修军等 [22]用微卫星标记得到太湖鹅的平均杂合度分别为0.520 6和0.653 0,远高于本研究通过简化基因组测序方法得到的3个太湖鹅群体平均杂合度(0.26~0.30左右)。这与王洪志等 [23]用简化基因组测序方法得到鹿苑鸡的遗传杂合度(0.20)明显低于微卫星得到的结果(0.593~0.737)[24-26]类似。这可能有两方面的原因:一是简化基因组测序等高通量方法费用较高,其所使用的样本量要明显少于微卫星标记使用的数量;二是经过优化的微卫星标记位点多态性高且分布相对集中于PCR扩增的区域[23]。显然,不同分子标记检测方法得到的结果没有可比性。但整体而言,简化基因组测序涵盖全基因组范围,覆盖度广、数量大,虽然得到的群体杂合度较低,但可以更全面地反映整个基因组的多态位点情况,结果更为可靠。
与原始群体相比,原种场群体和基因库群体的遗传杂合度均有降低,近交系数也明显增加,所以必须采取科学有效的措施来避免近交系数上升过快。
根据Wright [27]理论,FST值为0~0.05,说明各亚群间不存在分化;FST值为0.05~0.15,各亚群间中度分化;FST值为0.15~0.25,说明各亚群间已高度分化。本研究发现,TK群体与TS群体、TK群体与TC群体的FST值分别为0.043 8和0.048 0,接近于0.05,表明TK群体与其他2个群体的差异已经接近中度分化。而TC群体和TS群体的FST值为0.017 7,表明2个群体基本不存在分化,即TC群体基本保持了太湖鹅原始种群的特性。TK群体与TS群体产生分化可能由于环境的影响,保种环境的饲养条件、营养水平和疾病控制等因素可能存在差异,从而使保护的种群发生相应的变化。适当增加保种群的大小,以及适当引入原种场的个体[28],在一定程度上可以减少差异,保持原有种群特性的稳定,从而达到更好的保种效果。
[1] Pan Y E,Wang X E,Sun G L,et al.Application of RAD sequencing for evaluating the genetic diversity of domesticated panax notoginseng (araliaceae)[J].PLoS One,2016,11(11):0166419.
[2] Valdisser P R,Pappas G J,Menezes I D,et al.SNP discovery in common bean by restriction-associated DNA(RAD)sequencing for genetic diversity and population structure analysis[J].Molecular Genetics and Genomics,2016,291(3):1277-1291.
[3] 王洋坤,胡 艳,张天真.RAD-seq技术在基因组研究中的现状及展望[J].遗传,2014,36(1):41-49.
[4] 赵纪萍.基于RAD测序技术的6个中国家兔地方品种的遗传地位研究[D].杨凌:西北农林科技大学,2017.
[5] 翟正晓.基于RAD简化基因组测序技术的13种中国地方优良鸡品种SNPs多态性图谱构建及群体遗传学分析[D].上海:上海交通大学,2014.
[6] 段修军,董 飚,孙国波,等.基于酶切的简化基因组测序在水禽品种进化关系研究中的应用[J].西北农业学报,2015,24(1):13-17.
[7] Mckenna A,Hanna M,Banks E,et al.The genome analysis toolkit:a MapReduce framework for analyzing next-generation DNA sequencing data[J].Genome Research,2010,20(9):1297-1303.
[8] Li H,Handsaker B,Wysoker A,et al.The sequence alignment/map format and SAMtools[J].Bioinformatics,2009,25(16):2078-2079.
[9] See L M, Hassan R, Tan S G, et al. POPGENE, the user-friendly shareware for population genetic analysis[J].Biotechnology, 2006, 7(2): 104-110.
[10] Alexander D H,Novembre J,Lange K.Fast model-based estimation of ancestry in unrelated individuals[J].Genome Research,2009,19(9):1655-1664.
[11] 薛 辉,吴孝兵,晏 鹏.微卫星标记在分子生态学中的应用及其位点的分离策略[J].应用生态学报,2005,16(2):385-389.
[12] 陈红菊,岳永生,樊新忠,等.山东地方鸡种遗传距离与聚类分析方法比较研究[J].畜牧兽医学报,2004,35(1):33-36.
[13] 朱文奇,李慧芳,宋卫涛,等.基于线粒体DNA D-loop区序列的我国灰鹅遗传多样性和起源分析[J].生态学杂志,2010,29(3):549-553.
[14] 李慧芳,屠云洁,汤青萍,等.6个中国重点保护地方鹅品种的遗传多样性[J].四川农业大学学报,2005,23(4):466-469.
[15] Randi E,Lucchini V.Organization and evolution of the mitochondrial DNA control region in the avian Genus alectoris[J].Journal of Molecular Evolution,1998,47(4):449-462.
[16] 宋春红,陈红菊,马月辉,等.中国6个地方鸡品种的母系起源[J].畜牧兽医学报,2007,38(7):735-740.
[17] 龚道清,张 红,张 军,等.运用微卫星标记分析11个鸭种(群)的亲缘关系[J].畜牧兽医学报,2005,36(12):1256-1260.
[18] 吉文林,钱 凯,李慧芳,等.国家水禽基因库七个家鸭群体遗传多样性检测[J].中国家禽,2006,28(24):72-74.
[19] 汤青萍,李慧芳,屠云洁,等.中国重点保护地方鸭品种资源的多样性分析[J].西北农林科技大学学报:自然科学版,2007,35(2):47-52.
[20] 刘宏祥,王宝维,宋卫涛,等.马踏湖鸭群体遗传结构探讨[J].中国家禽,2015,37(3):57-59.
[21] 汤青萍,章双杰,郭 军,等.太湖鹅群体遗传多样性研究[J].家畜生态学报,2010,31(1):30-33.
[22] 段修军,董 飚,王日君,等.太湖鹅微卫星标记与体重的相关性[J].江苏农业学报,2011,27(3):597-601.
[23] 王洪志,李国辉,张 贤,等.基于RAD-seq简化基因组测序评价鹿苑鸡不同保种群保种现状[J].畜牧兽医学报,2017,48(5):818-825.
[24] 王得前,陈国宏,吴信生,等.运用微卫星技术分析中国地方鸡品种的亲缘关系[J].扬州大学学报:农业与生命科学版,2003,24(2):1-6.
[25] 李慧芳,陈宽维,汤青萍,等.利用微卫星标记分析云南6个地方鸡品种的遗传多样性[J].江苏农业学报,2006,26(1):33-37.
[26] 吴兆林,高玉时,童海兵.鹿苑鸡微卫星和AFLP指纹分析[J].中国家禽,2005,9(S):83-85.
[27] Wright S.Evolution and the genetics of populations,Volume 4:variability within and among natural population[M].Chicago:University of Chicago Press,1978.
[28] 赵振华,黎寿丰,吴兆林,等.不同保种方法对地方鸡种的保种效果分析[J].中国家禽,2009,31(14):23-25.