马鹿(Cervus elaphus)隶属偶蹄目、鹿科、鹿亚科、鹿属。马鹿种类多、分布广,国内外关于马鹿起源进化及分类的研究报道较多,盛和林[1]从形态特征、王宗仁等[2]从染色体组型、Emerson等[3]从血液蛋白电泳多态性、Feulner等[4]以微卫星标记多样性、苏莹[5]以Y染色体基因为标记(父系遗传)、李明等[6]、张丽等[7]以线粒体DNA为标记(母系遗传)进行了研究。前人的研究为探明我国马鹿分类与进化积累了大量宝贵资料。现行分类学把我国马鹿分为8个亚种,它们是阿拉善亚种、甘肃亚种、天山亚种、阿勒泰亚种、塔里木亚种、东北亚种四川亚种和西藏亚种,但对我国马鹿亚种的系统进化关系还存在一定的分歧。
线粒体DNA控制区是线粒体基因组非编码区,其进化速率较快,鹿科动物控制区进化速率约为编码区2倍[8],线粒体DNA标记特别适应于亲缘关系较近物种(种内或种间)的遗传结构和系统进化等方面研究。本研究通过测定阿尔泰马鹿、天山马鹿、塔里木马鹿、阿拉善马鹿及甘肃马鹿共5个亚种计146个体线粒体DNA控制区序列,分析各亚种遗传多样性、种群遗传结构,构建单倍型系统进化树,旨在了解我国马鹿群体遗传资源状况和进化地位,为有效保护及合理利用马鹿遗传资源提供参考资料。
1 材料和方法
1.1 试验材料
所有样品均为血样,样品信息详见表1。
表1 所用样品信息和单倍型数据
Tab.1 Sample information and data of haplotype
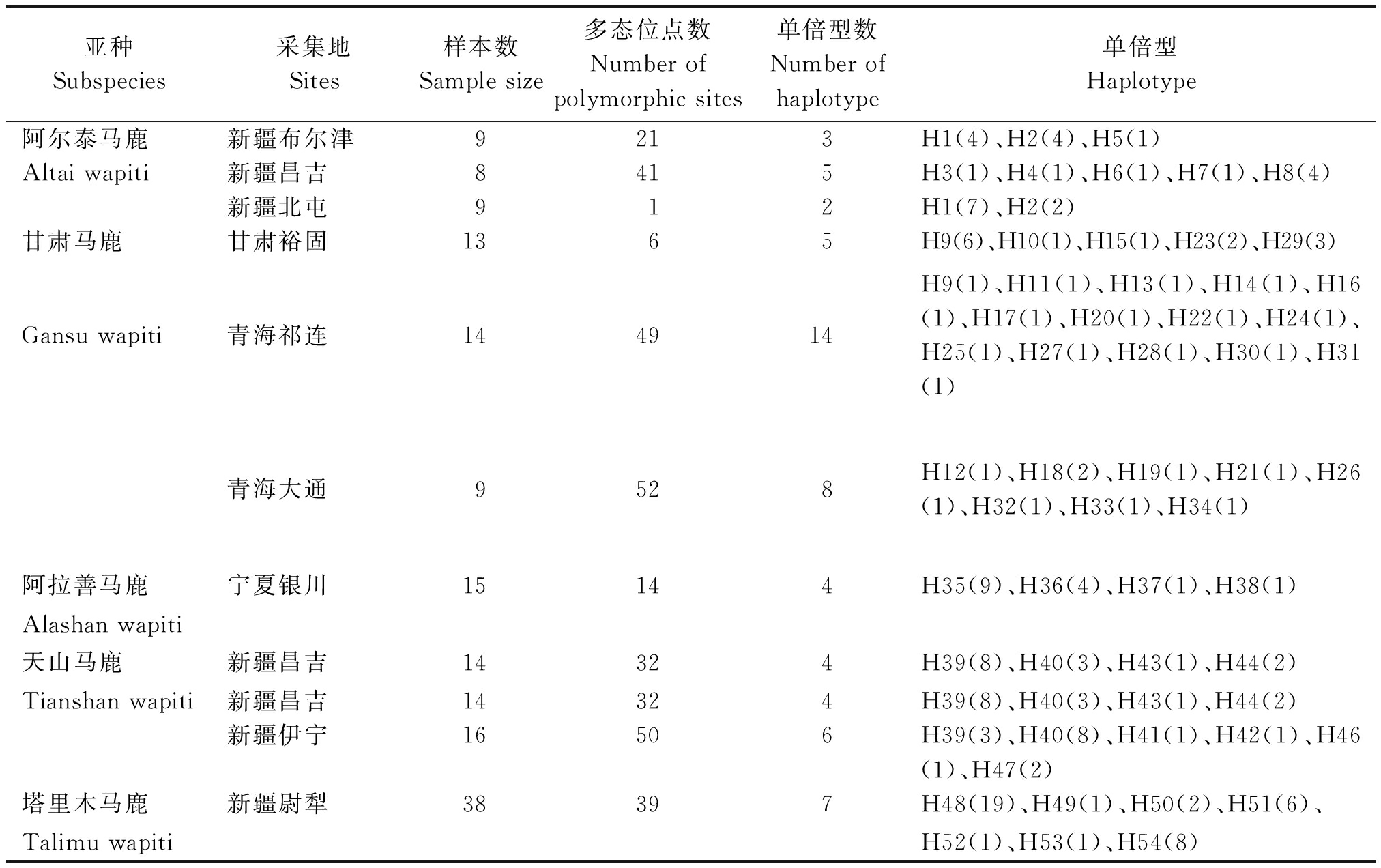
亚种Subspecies采集地Sites样本数Sample size多态位点数Number ofpolymorphic sites单倍型数Number ofhaplotype单倍型Haplotype阿尔泰马鹿新疆布尔津9213H1(4)、H2(4)、H5(1)Altai wapiti新疆昌吉8415H3(1)、H4(1)、H6(1)、H7(1)、H8(4)新疆北屯912H1(7)、H2(2)甘肃马鹿甘肃裕固1365H9(6)、H10(1)、H15(1)、H23(2)、H29(3)Gansu wapiti青海祁连144914H9(1)、H11(1)、H13(1)、H14(1)、H16(1)、H17(1)、H20(1)、H22(1)、H24(1)、H25(1)、H27(1)、H28(1)、H30(1)、H31(1)青海大通9528H12(1)、H18(2)、H19(1)、H21(1)、H26(1)、H32(1)、H33(1)、H34(1)阿拉善马鹿宁夏银川15144H35(9)、H36(4)、H37(1)、H38(1)Alashan wapiti天山马鹿新疆昌吉14324H39(8)、H40(3)、H43(1)、H44(2)Tianshan wapiti新疆昌吉14324H39(8)、H40(3)、H43(1)、H44(2)新疆伊宁16506H39(3)、H40(8)、H41(1)、H42(1)、H46(1)、H47(2)塔里木马鹿新疆尉犁38397H48(19)、H49(1)、H50(2)、H51(6)、Talimu wapitiH52(1)、H53(1)、H54(8)
1.2 基因组DNA提取
采用基因组DNA提取试剂盒提取总DNA,琼脂糖凝胶电泳检测提取效果,于-20 ℃保存。
1.3 引物设计和PCR扩增
在马鹿线粒体控制区序列两端保守位置设计1对引物,上游引物5′-CACCCAAAGCTGAAGTTCTAT-3′,下游引物5′-CTCATCTAGGCATTTTCAGTG-3′,引物由上海生工合成。PCR反应体系总体积为25 μL:模板DNA 100 ng、dNTP(2.5 mol/L)2 μL、10×Buffer 2.5 μL、上下游引物(10 μmol/μL)各1 μL、0.5 U Taq DNA聚合酶,加纯水补足。扩增条件:94 ℃预变性 5 min;94 ℃变性30 s,55 ℃复性30 s,72 ℃延伸1 min,30个循环;72 ℃延伸7 min。
1.4 测序和拼接
电泳检测RCR产物,均为单一清晰条带,送上海生工完成测序工作。用Chromas 2.22校对测序峰图,DNAMAN 6.0完成序列比对和线粒体控制区全长序列拼接。
1.5 序列分析
用DNAStar 6.0统计碱基含量及格式转换;ClustalX1.8序列比对;DnaSP 6.0[9]统计单倍型数、计算核苷酸多样性(Nucleotide diversity)、单倍型多样性(Haplotype diversity,h)、种群之间的FST值和基因流(Nm);Arlequin3.1[10]计算Tajima′s D值和Fu′s Fs值;Network 5.0[11]构建单倍型网状树;Mega 7.0[12]统计单倍型多态位点信息,并基于Kimura-2-Parameter双参数模型、SPR法构建最大简约树(Bootstrap test 1000)。MrBayes v3.04b[13]基于 HKY+I+G模型,构建贝叶斯树(运行300万代,每100代取样一次)。
2 结果和分析
2.1 序列特征及种群遗传多样性
146只马鹿线粒体DNA控制区序列全长916~995 bp,序列组成中碱基T含量最高,碱基G含量最低,A+T数量明显高于C+G。序列比对共检测到167个多态位点(图1),定义54个单倍型,其中阿尔泰马鹿8个(H1~H8)、甘肃马鹿26个(H9~H34)、阿拉善马鹿4个(H35~H38)、天山马鹿9个(H39~H47)和塔里木马鹿7个(H48~H54),亚种内不同种群间存在共享单倍型,亚种间无共享单倍型(表1)。5个亚种单倍型多样度h值为0.600~0.959、核苷酸多样度π值为0.006 3~0.014 4,甘肃马鹿核苷酸多样度和单倍型多样度明显高于其他马鹿,阿拉善马鹿核苷酸多样度和单倍型多样度最低(表2)。
2.2 种群遗传结构
5个亚种种群内中性检验,塔里木马鹿、天山马鹿、阿拉善马鹿和阿尔泰马鹿Tajima′s D值和Fu′s Fs值显著性检验P>0.10,差异不显著,甘肃马鹿中性检验Tajima′s D值和Fu′s Fs值呈现较大负值,显著性检验P<0.05,显著偏离中性突变(表2)。亚种间的遗传分化指数FST值均在0.5以上,塔里木马鹿与其他马鹿的遗传分化程度最大,FST值高于0.8,显著性检验P< 0.01,差异极显著。亚种间的基因流Nm值均低于0.5(表3)。
表2 马鹿种群遗传多样性及中性检验
Tab.2 Diversity and neutral test on wapiti population
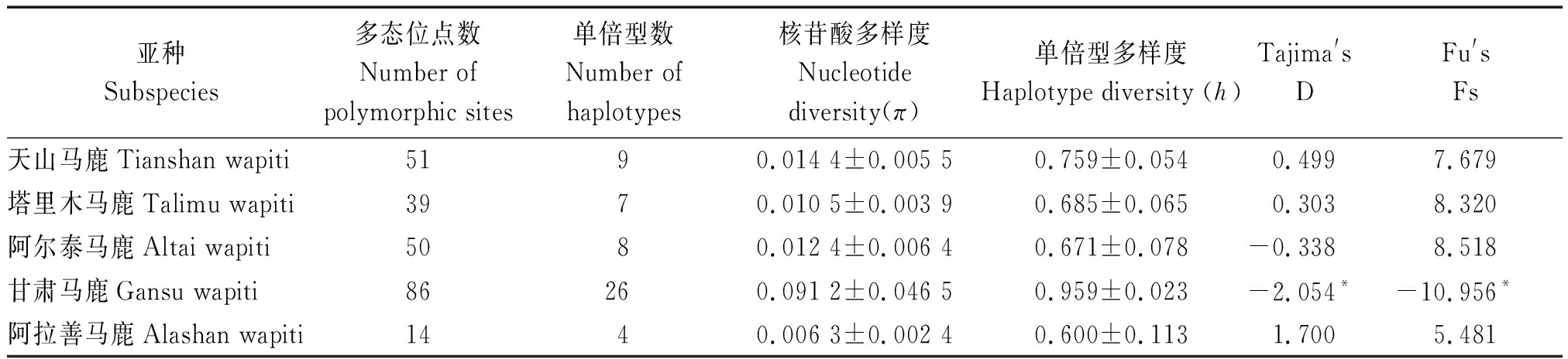
亚种Subspecies多态位点数Number ofpolymorphic sites单倍型数Number ofhaplotypes核苷酸多样度Nucleotidediversity(π)单倍型多样度Haplotype diversity (h)Tajima'sDFu'sFs天山马鹿Tianshan wapiti5190.014 4±0.005 50.759±0.0540.4997.679塔里木马鹿Talimu wapiti3970.010 5±0.003 90.685±0.0650.3038.320阿尔泰马鹿Altai wapiti5080.012 4±0.006 40.671±0.078-0.3388.518甘肃马鹿Gansu wapiti86260.091 2±0.046 50.959±0.023-2.054*-10.956*阿拉善马鹿Alashan wapiti1440.006 3±0.002 40.600±0.1131.7005.481
注:*.P<0.05,差异显著。
Note:*.P<0.05,Significant difference.
表3 马鹿亚种间的遗传分化指数(下三角)及基因流(上三角)
Tab.3 Genetic differentiation coefficient(FST)and gene flow(Nm) among wapiti
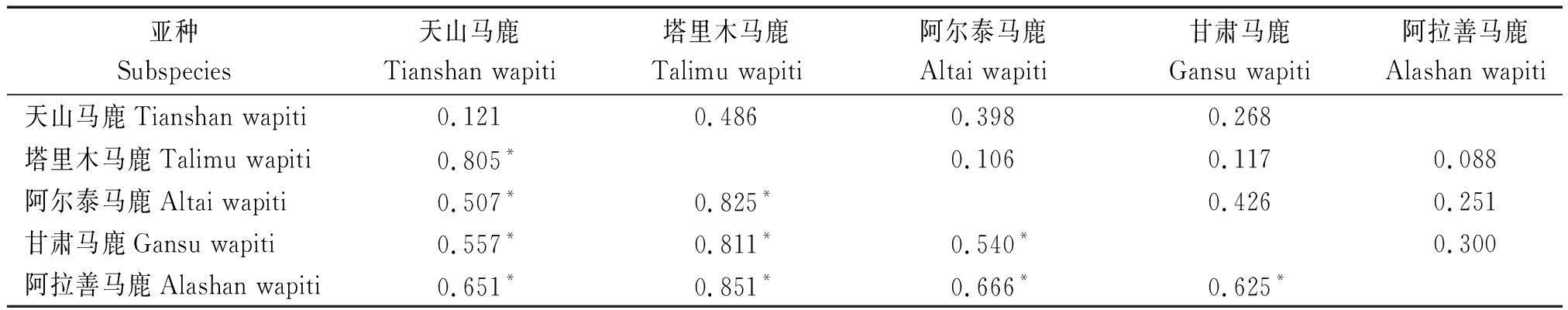
亚种Subspecies天山马鹿Tianshan wapiti塔里木马鹿Talimu wapiti阿尔泰马鹿Altai wapiti甘肃马鹿Gansu wapiti阿拉善马鹿Alashan wapiti天山马鹿Tianshan wapiti0.1210.4860.3980.268塔里木马鹿Talimu wapiti0.805*0.1060.1170.088阿尔泰马鹿Altai wapiti0.507*0.825*0.4260.251甘肃马鹿Gansu wapiti0.557*0.811*0.540*0.300阿拉善马鹿Alashan wapiti0.651*0.851*0.666*0.625*
注:*.P<0.01,差异极显著。
Note:*.P<0.01,highly significant difference.
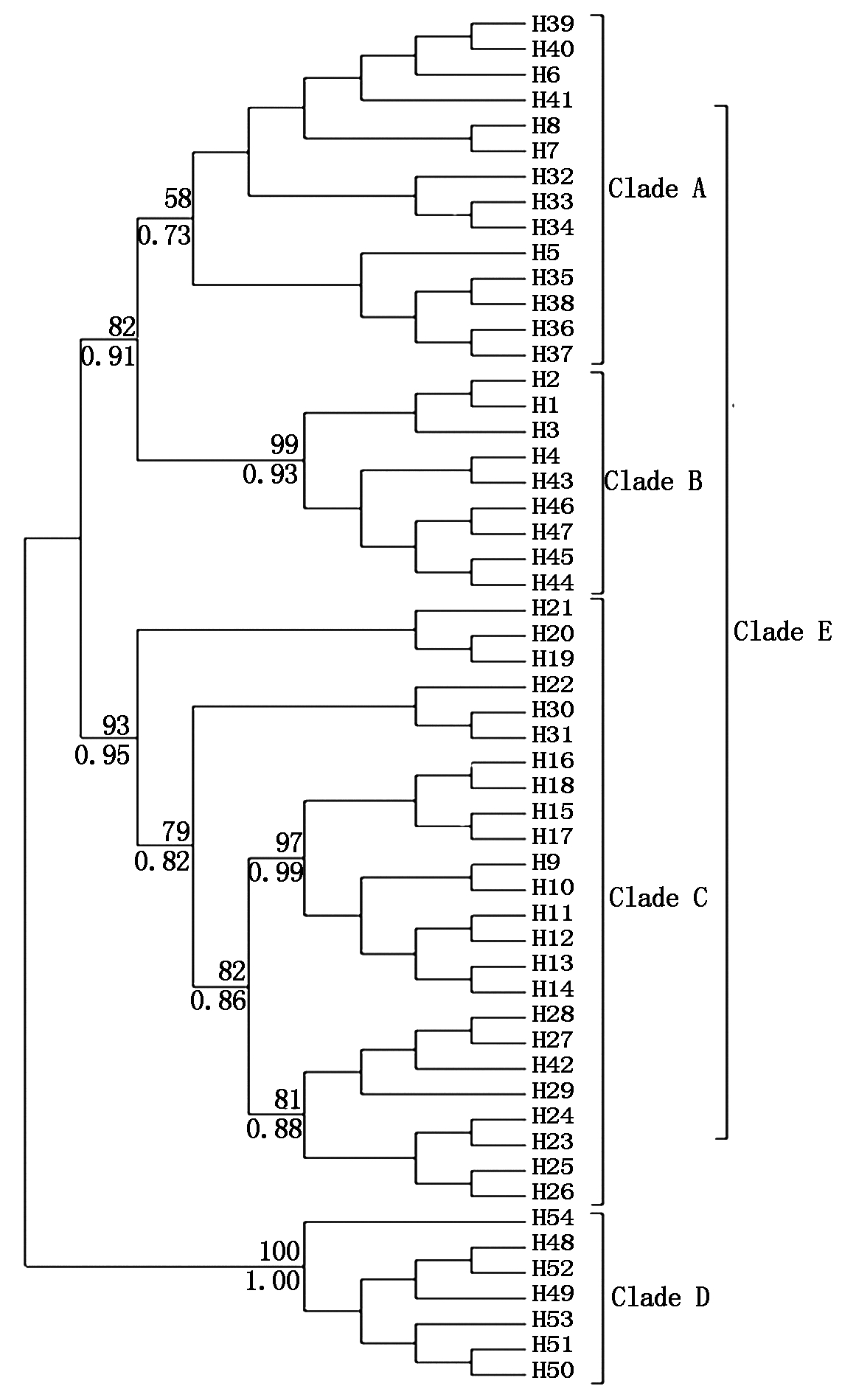
2.3 系统进化树
基于线粒体DNA控制区序列单倍型,构建最大简约树和贝叶斯树,2种进化树的拓扑结构基本一致(图2)。进化树结果显示,54个单倍型分为两大进化枝(D、E),进化枝D由塔里木马鹿单倍型组成,其他马鹿组成进化枝E;进化枝E又细分为A枝、B枝、C枝3个姐妹枝,进化枝A构成较为复杂,包含阿尔泰马鹿、甘肃马鹿、天山马鹿以及阿拉善马鹿单倍型,进化枝B由阿尔泰马鹿和天山马鹿单倍型组成,进化枝C由甘肃马鹿和天山马鹿单倍型组成。基于单倍型构建的中介网络图(图3)清晰地识别2个进化枝,与系统进化树的结果相一致。
3 讨论
3.1 遗传多样性和种群遗传结构
核苷酸多样性(π)和单倍型多样性(h)常用于衡量群体遗传多样性程度[14-16]。本研究中马鹿5个亚种呈现较高的单倍型多样性(h>0.600),中等程度的核苷酸多样性(π>0.006 3),表明遗传多样性较为丰富,尤其是甘肃马鹿遗传多样性最为丰富,单倍型多样性和核苷酸多样性最高,在36个体中检测到86个多态位点和26个单倍型,塔里木马鹿和阿拉善马鹿遗传多样性相对贫乏。从单倍型分布上,所有亚种没有共享单倍型,亚种内的不同种群间共享单倍型也较少。5个亚种群体中性检验仅发现甘肃马鹿Tajima′s D值和Fu′s Fs值呈现较大负值,显著偏离中性突变,这说明甘肃马鹿受到自然选择的作用,历史上可能发生过群体扩张事件;其他4个亚种中性检验Tajima′s D值和Fu′s Fs值差异不显著(P>0.10),推断是平衡选择的结果。
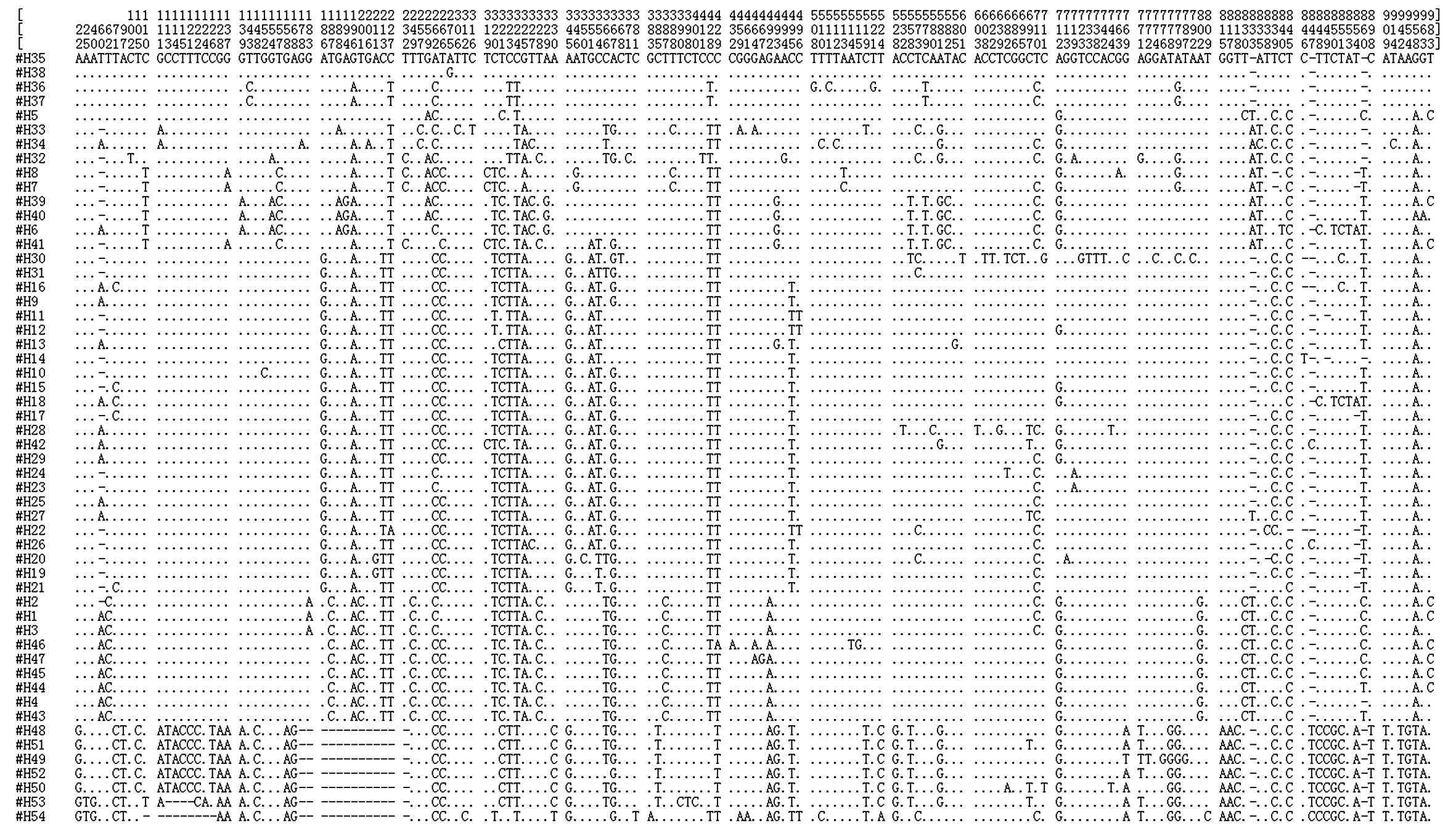
图1 西北马鹿单倍型多态位点信息
Fig.1 Information of polymorphic sites for haplotypes of wapiti
进化枝上和枝下的数字分别代表MP
树支持率和贝叶斯树后验概率。
Number above the branches are bootstrap proportions derived from MP tree and below those the branches are Bayesian posterior probabilities
图2 基于马鹿线粒体DNA控制区单倍型构建的MP树和贝叶斯树
Fig.2 MP and Bayesian trees of haplotypes based on mtDNA control region of wapiti
5个亚种遗传分化指数Fst值均>0.5,显著性检验P<0.01,可推断各亚种产生了显著的遗传分化,特别是塔里木马鹿与其他4个亚种的Fst值均在0.8以上;基因流检测至各亚种间基因流Nm值均<0.5,表明群体之间遗传变异程度高,基因交流有限,群体间会发生遗传分化。从单倍型上分析,所有亚种没有共享单倍型,亚种内的不同种群间共享单倍型数量也不多,也表明亚种间已经发生明显的遗传分化。
3.2 系统进化
关于马鹿的系统进化关系仍存在一定分歧,大多数观点认为马鹿分为东、西2个进化群体,西部群体包括欧洲种群和我国新疆南部塔里木种群,东部种群包括北美种群、亚洲及我国新疆北部的种群组成[17-19]。本研究基于单倍型构建的我国马鹿5个亚种系统进化树显示塔里木马鹿和其他4个马鹿亚种来源于共同祖先的2个不同进化枝,支持马鹿的进化分为东部种群和西部种群;我国的塔里木马鹿较早地从祖先中分化出来,在一个封闭的地理环境下独立进化,产生了“瓶颈效应”而有别于其他马鹿种群。阿尔泰马鹿、甘肃马鹿、天山马鹿及阿拉善马鹿起源于同一个进化枝,但4个亚种的进化关系错综复杂,进化树显示它们在进化枝上相互交错聚集;姐妹枝A、B、C均有天山马鹿单倍型,阿拉善马鹿聚类于枝系A,枝系A构成最复杂,4个亚种均有单倍型聚于此枝系,可能的原因是它们存在共同祖先,它们从祖先分化出来后在各自的地理条件下进化,经历了“创立者效应”,也不能排除生产中为了提高鹿茸产量,种群间的基因交流所致,邓铸疆等[20]认为马鹿亚种间存在基因交流情况。为此,还需要增加马鹿样本种类、数量或更多的遗传标记深入研究。
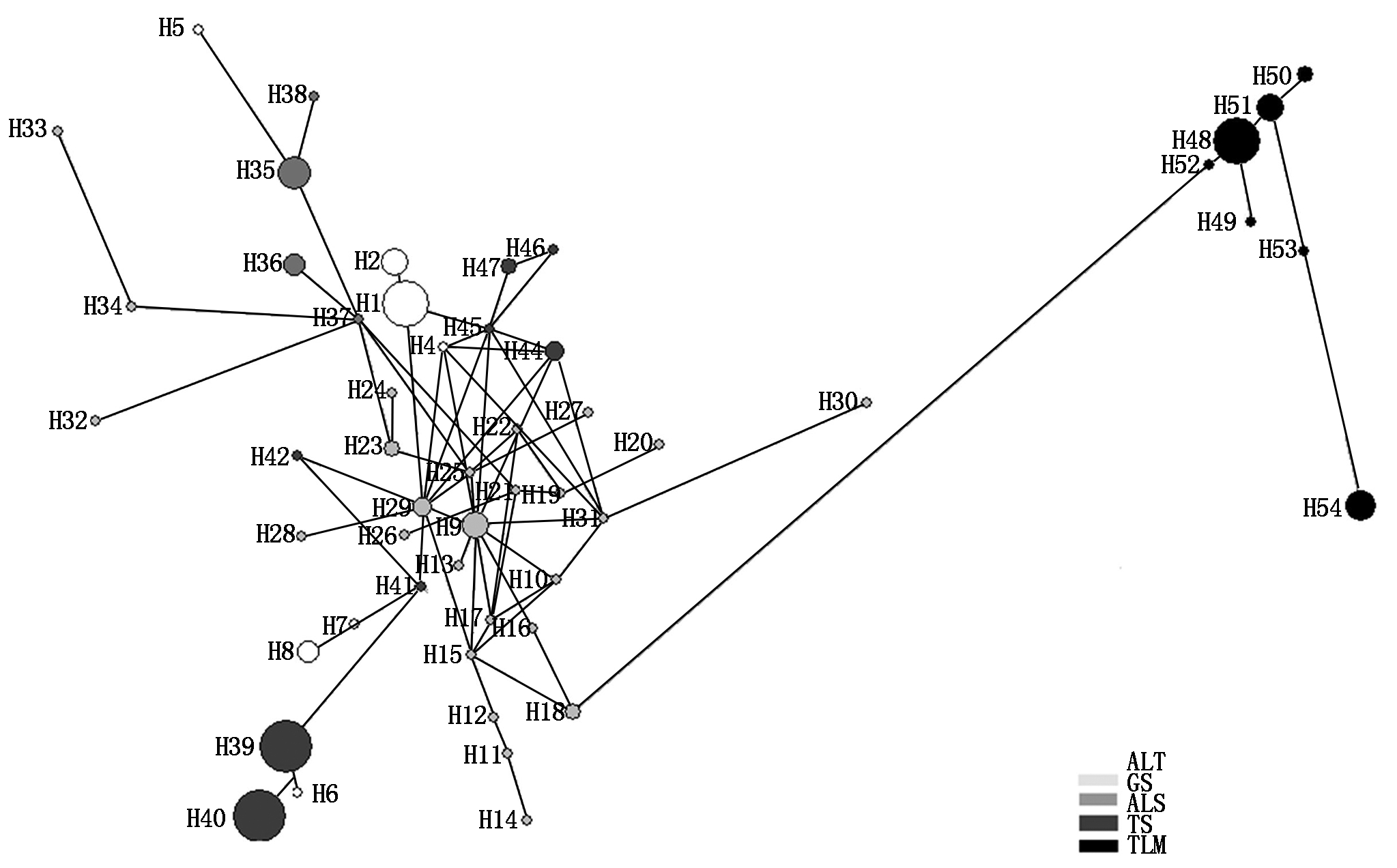
图3 基于马鹿线粒体DNA控制区单倍型构建的中介网络图
Fig.3 Median-joining network based on haplotypes of mtDNA control region of wapiti
[1] 盛和林. 中国鹿类动物[M]. 上海:华东师范大学出版社,1992.
[2] 王宗仁,杜若甫. 鹿的核型与染色体进化[M]. 北京:科学出版社,1988.
[3] Emerson B C,Tate M L. Genetic analysis of evolutionary relationships among deer(subfamily Cervinae)[J].J Hered,1993,84(4):266-273.
[4] Feulner P G,Bielfeldt W,Zachos F E,et al. Mitochondrial DNA and microsatellite analyses of the genetic status of the presumed subspecies Cervus elaphus montanus(Carpathian red deer)[J]. Heredity,2004,93(3):299-306.
[5] 苏 莹. 马鹿群体Y染色体相关基因遗传多样性分析[D]. 长春:吉林农业大学,2016:1-2.
[6] 李 明,王小明,盛和林,等. 马鹿四个亚种的起源和遗传分化研究[J]. 动物学研究,1998,19(3):2-3,5-8.
[7] 张 丽,滚双宝,雷天云,等. 应用mtDNA Cytb基因全序列分析中国5个马鹿群体的遗传多样性和系统发育[J]. 华北农学报,2010,25(4):12-16.
[8] Cronin A M. Mitochondrlal DNA phylogeny of deer(Cervidae)[J]. J Mamm,1991,72(3):553-565.
[9] Rozas J,Ferrer-Mata A,Sanchez-DelBarrio J C,et al. DnaSP 6:DNA sequence polymouphism analysis of large datasets[J]. Molecular Biology and Evolution,2017,34:3299-3302.
[10] Excoffier L,Lischer H E. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources,2010,10(3):564-567.
[11] Network 4.6.1.1 user guide[OL].[2014-10-29],http://www.fluxs-engineering.com.
[12] Tamura K,Stecher G,Peterson D,et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution,2013,30(12):2725-2729.
[13] Ronquist F,Huelsenbeck J P. MrBayes 3:bayesian phylogenetic inference under mixed models[J]. Bioinformatics,2003,19(12):1572-1574.
[14] 彭银辉,周于娜,刘旭佳,等. 基于线粒体控制区序列的光裸方格星虫群体遗传多样性分析[J]. 水产学报,2017,41(10):1542-1551.
[15] 杨天燕,孟 玮,海 萨,等. 基于线粒体Cyt b序列对新疆额尔齐斯河贝加尔雅罗鱼遗传结构的分析[J]. 动物学杂志,2017,52(2):304-313.
[16] 成述儒,曾玉峰,王欣荣,等. 甘肃境内6个牦牛群体mtDNA-环序列遗传多样性与聚类分析[J]. 华北农学报,2014,29(3):16-21.
[17] Mahmut Halik,Masuda Ryuichi,Onuma Manabu,et al. Molecular phylogeography of the red deer(Cervus elaphus)populations in Xinjiang of China:Comparison with other Asian ,European and North American populations [J] .Zoological Science ,2002,19:485-495.
[18] Ludt C J,Schroeder W,Rottmann O,et al. Mitochondrial DNA phylogeography of red deer(Cervus elaphus)[J]. Molecular Phylogenetics and Evolution,2004,31(3):1064-1083.
[19] Polziehn R O,Strobeck C. A phylogenetic comparison of red deer and wapiti using mitochondrial DNA[J]. Molecular Phylogenetics and Evolution,2002,22(3):342-356.
[20] 邓铸疆,任战军,熊建杰,等. 西北马鹿群体遗传多样性及系统地位[J]. 西北农林科技大学学报:自然科学版,2010,38(9):42-46,52.