牛肉中蛋白质含量十分丰富,特别是氨基酸、脂肪酸等组成比例比猪肉、鸡肉等更倾向于人体需要,更重要的是牛肉容易吸收、提升机体免疫力,深受消费者的喜爱[1-2]。自从20世纪90年代以来,转录组学在生物学前沿的研究中诞生并逐渐成为领域研究重点,中国地方牛种相关的转录组研究由此也有了很大的进展[3-4],其主要原因一是蛋白质组学需要转录组结果的辅助并加以验证和补充;二是随着非编码RNA研究的深入,使转录组学的研究探索更为宽泛;三是RNA-seq技术使得转录组技术获得了大量的数据,拓展了其研究领域[5-8]。目前,关于牛组织转录组研究有了许多成果,2012年Mccabe等[9]运用RNA-seq分析,得出泌乳期奶牛肝脏产生的负调控与脂肪代谢相关;2016年Bai等[10]对中国荷斯坦奶牛进行全血RNA-seq数据分析,探索免疫系统与产奶量的关系。现已认识到基因组的动态状态,包括DNA修饰和RNA定量和定性变化[11],其特征在于从简单模型生物到人类,这个进步则通过各种基因组测量技术和全面的转录组学研究实现的[12-14]。
虽然转录组研究在地方牛种上取得了很大的进展,但目前对肉牛肌肉生长发育机制的研究相对较少,本试验拟通过Illumina 2500测序平台对生长性能不同的布莱凯特黑牛和鲁西黄牛的背最长肌转录组文库进行高通量测序和生物信息学分析,将结果与参考基因组比对后分析其差异基因的表达情况并进行基因注释,为下一步发现筛选肌肉调控基因提供依据,为后续了解基因功能、各基因互作效应以及开发分子育种新途径奠定基础。
1 材料和方法
1.1 试验材料
为保证试验的准确性,所用动物布莱凯特黑牛和鲁西黄牛选自山东布莱凯特黑牛科技股份有限公司和大地黄牛。选择饲养条件一致、体况相近、健康的12月龄黑牛和黄牛各3头,为得到布莱凯特黑牛和鲁西黄牛骨骼肌全部的转录信息,此次试验对样本进行高通量双末端测序。
1.2 主要试验试剂及仪器
TRNzolTrans、 DNA MarkerⅡTransSCript ONE STEP gDNA Removal和cDNA Synthes-is SuperMix均购自天根生化科技(北京)有限公司;无水乙醇、DEPC、PBS、RNAase-free water、1 000 bp DNA Ladder Mark均购自青岛赛尚科贸有限公司;高通量测序仪Hiseq-2500购自美国 Illumina 公司。
1.3 RNA的提取、测序及cDNA的合成
选取黑牛和黄牛背最长肌肌肉组织样品各60 mg,加入1 mL的TRNzol(Roche)试剂粉碎匀浆后抽提总RNA。测定其浓度和纯度,将检验合格的RNA,使用反转录试剂盒(Roche)逆转录成cDNA。
1.4 cDNA文库的制备及RNA-Seq文库测序
根据布莱凯特黑牛和鲁西黄牛肌肉组织RNA反转录建立cDNA文库,并进行RNA-Seq测序。RNA-seq测序获得原始数据后,需要对数据进行去除接头序列、去低质量reads和核糖体RNA的过滤处理,再对测序质量进行评估,重复检验获得高质量数据,即Clean Data[15]。将Clean Data与参考基因组进行比对,然后通过一定的分析与筛选,得出样品的类型,并根据样品的类型制定相应的分析。
1.5 比对序列的构建和定量
利用TOPHAT软件将Clean reads与参考基因组进行比对,根据参考基因组的注释文件对Mapped reads进行转录组的数据分析。
1.6 差异基因筛选
本试验差异基因筛选主要采用的是T检验和方差分析与倍数分析法,以鲁西黄牛为对照组,利用布莱凯特黑牛和鲁西黄牛结果数据T检验得出的P值和差异倍数Fold change进行筛选,将差异倍数(|log2(Fold chang-e)|>1)和显著水平(P-value<0.05)2个水平作为筛选标准,筛选出样品间的表达差异基因。
差异基因功能分析:本试验利用Blast2GO软件对差异表达基因进行GO功能注释,选择所有基因作为背景基因,使用生物学统计方法计算P值,以P<0.01为阈值得到高频注释,对应分析显著联系的生物学功能[16];对基因的KEGG生物通路注释,使用Fisher Exact Test计算P值,以0.05为阈值获得有统计意义的信号转导及通路[17];利用STRING在线软件构建蛋白互作网络图。
2 结果与分析
2.1 数据测序质量评估及差异基因筛选
原始数据质量分布图(图1)可以直观表现每个read各位置碱基的测序质量,reads质量分数是中间略高两侧偏低;根据每个碱基的质量分布图,呈J型曲线,说明在0~30分值的碱基量少,而大多数碱基在30~49的分值范围,每个样品都具有良好的质量,表明测序的错误率大部分都低于0.1%。

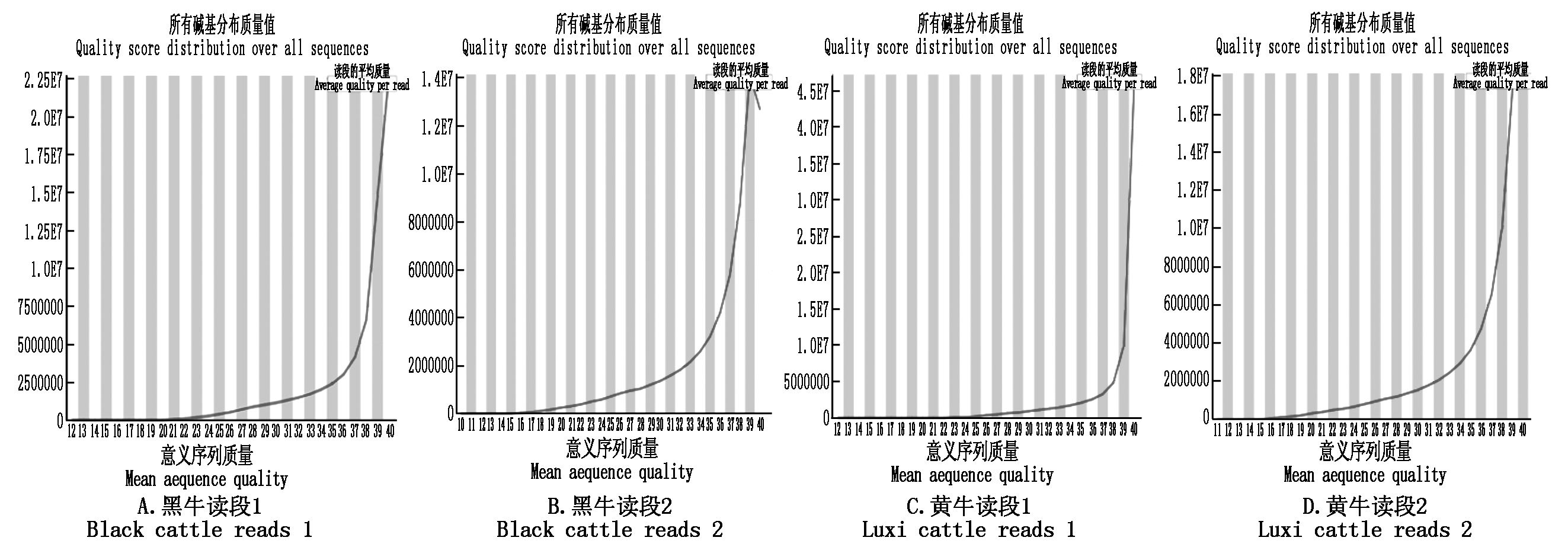
图1 原始测序数据质量分布
Fig.1 Original sequencing data quality map
2.2 测序序列与参考基因的比对统计
由表1可知,布莱凯特黑牛和鲁西黄牛文库中分别约有85.38%和87.88%的原始序列可以比对到参考基因组,其中唯一映射分别达到79.40%和80.71%,说明2个文库的比对准确度较高。黄牛和黑牛2个转录组文库干净序列分别为90.06%,88.79%,cleanQ20与Q30比率均高于85%。
布莱凯特黑牛和鲁西黄牛2个文库分别获得13 242,13 350个基因,共计13 758个基因。5 500个基因表达量相同,约占总参考基因的39.98%,8 258个基因表达量有所不同,占比60.02%。根据FDR≤0.001且|log2Ratio|≥1获得554个差异显著表达基因(图2),其中296个基因表达上调,258个基因表达下调。进一步发现差异表达基因中有36 个基因编码产生肌钙蛋白、肌红蛋白和肌球蛋白,13个基因只在布莱凯特黑牛中表达,6个基因只在鲁西黄牛中表达,其表达量均较低。
表1 测序序列与参考基因的比对统计
Tab.1 Statistics of sequencing reads aligned to reference genes
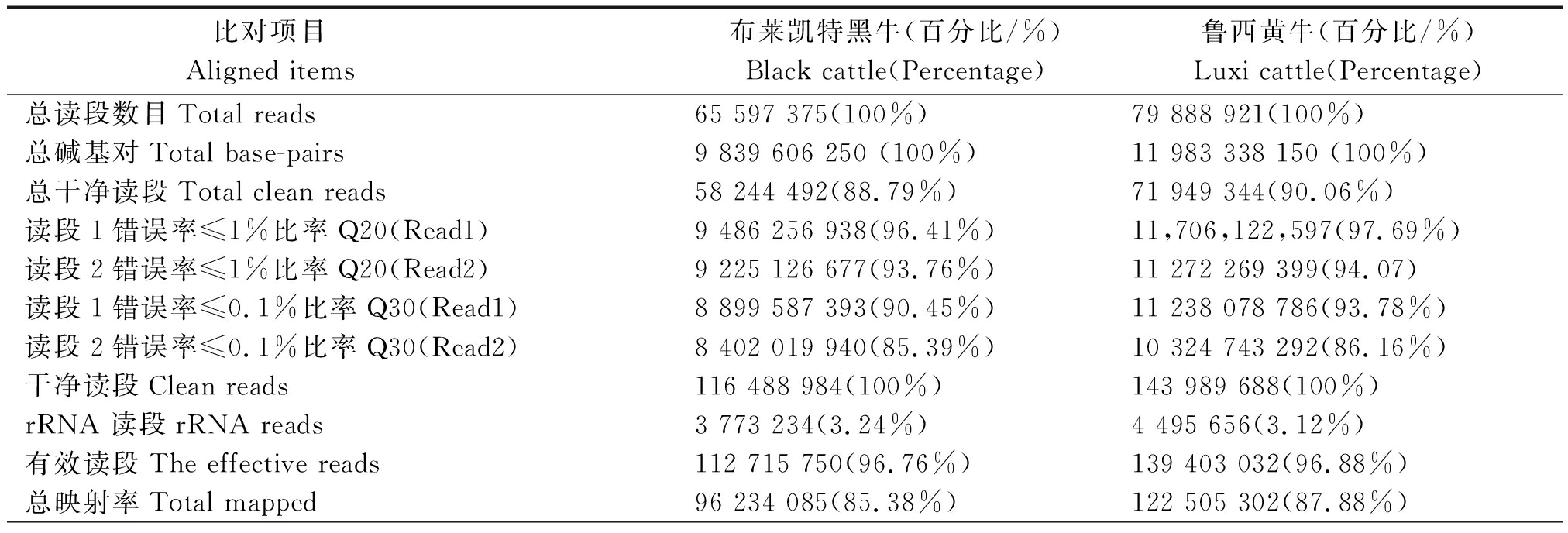
比对项目 Aligned items 布莱凯特黑牛(百分比/%)Black cattle(Percentage)鲁西黄牛(百分比/%)Luxi cattle(Percentage)总读段数目Total reads65 597 375(100%)79 888 921(100%)总碱基对Total base-pairs9 839 606 250 (100%)11 983 338 150 (100%)总干净读段Total clean reads58 244 492(88.79%)71 949 344(90.06%)读段1错误率≤1%比率Q20(Read1)9 486 256 938(96.41%)11,706,122,597(97.69%)读段2错误率≤1%比率Q20(Read2)9 225 126 677(93.76%)11 272 269 399(94.07)读段1错误率≤0.1%比率Q30(Read1)8 899 587 393(90.45%)11 238 078 786(93.78%)读段2错误率≤0.1%比率Q30(Read2)8 402 019 940(85.39%)10 324 743 292(86.16%)干净读段Clean reads116 488 984(100%)143 989 688(100%)rRNA 读段rRNA reads3 773 234(3.24%)4 495 656(3.12%)有效读段The effective reads112 715 750(96.76%)139 403 032(96.88%)总映射率Total mapped96 234 085(85.38%)122 505 302(87.88%)
表1(续)
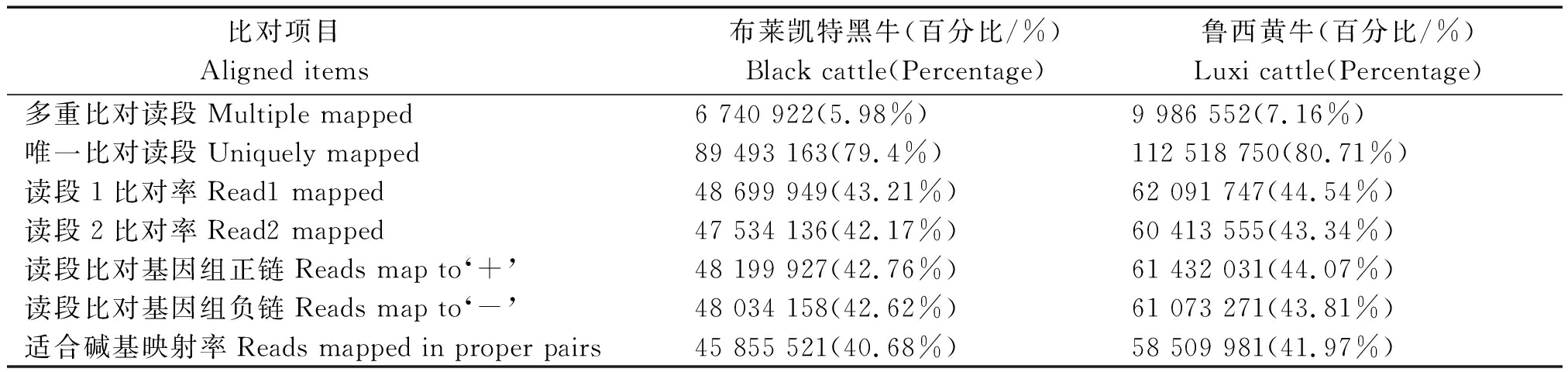
比对项目 Aligned items 布莱凯特黑牛(百分比/%)Black cattle(Percentage)鲁西黄牛(百分比/%)Luxi cattle(Percentage)多重比对读段Multiple mapped6 740 922(5.98%)9 986 552(7.16%)唯一比对读段Uniquely mapped89 493 163(79.4%)112 518 750(80.71%)读段1比对率Read1 mapped48 699 949(43.21%)62 091 747(44.54%)读段2比对率Read2 mapped47 534 136(42.17%)60 413 555(43.34%)读段比对基因组正链Reads map to‘+’48 199 927(42.76%)61 432 031(44.07%)读段比对基因组负链Reads map to‘-’48 034 158(42.62%)61 073 271(43.81%)适合碱基映射率Reads mapped in proper pairs45 855 521(40.68%)58 509 981(41.97%)

黑牛与黄牛差异基因表达分析,其中A代表黑牛基因表达量上调,B代表表达量没有变化,C代表表达量下调。
Analysis of differential gene expression between black cattle and yellow cattle, among them,A represents the up-regulation of black cattle gene expression,B is no change,C is down-regulated.
图2 不同牛种差异表达基因的聚类分析
Fig.2 Cluster analysis of differentially expressed genes in different breeds of cattle
2.3 差异基因的功能分析
利用Blast2GO软件对差异表达基因进行GO功能注释(图3),注释分为生物学过程、细胞组分和分子功能三大类,分别包括22 641,7 720和4 414个基因。其中细胞过程有334个基因,为生物学过程分类中基因最多的条目,其次单一生物过程有300个基因;细胞和细胞内组分分别占有348,328个基因;在分子功能分类中,270个基因的基因绑定条目占据最高。进一步分析发现,生物学过程与新陈代谢过程中相关的基因有280个,与生物学调控相关的有219个基因。在差异表达基因GO功能注释包括25个二级分类中,有10个二级分类共29个基因与肌细胞的发育和分化有关。

图3 差异表达基因的GO分类
Fig.3 GO classification of differentially expressed genes
差异表达基因的KEGG分类图可以直观展示差异表达基因参与的主要生命代谢的活动。如图4,本试验将差异基因聚类为细胞过程、人类疾病、环境信息、遗传信息、代谢和生物系统六大类, 21个二级分类。其中富集最多的一类是人类疾病,慢性进行性舞蹈病(25个基因)为其条目中占据基因最多的二级分类,最少的一类是生物学过程(8个基因)。将554个差异表达基因与KEGG数据库进行比对(表2),发现共有554个基因被注释到了KEGG中的226个通路中。其中61(26.99%)个基因参与了新陈代谢通路(bta01100)、25(11.06%)个基因参与慢性进行性舞蹈病(bta05016)、7(3.98%)个基因参与肌动蛋白细胞骨架通路(ko04810),6个基因参与了ECM受体干扰通路。骨骼肌表达差异基因主要富集到Wnt、CMC、TGF-β、DCM、ROAC、VSMC 6条肌肉生长发育相关通路(表2)。

图4 差异表达基因的KEGG分类图
Fig. 4 KEGG classification of differentially expressed genes
表2 骨骼肌表达差异基因 KEGG 代谢途径分类
Tab.2 KEGG classification of skeletal muscle differential expression genes
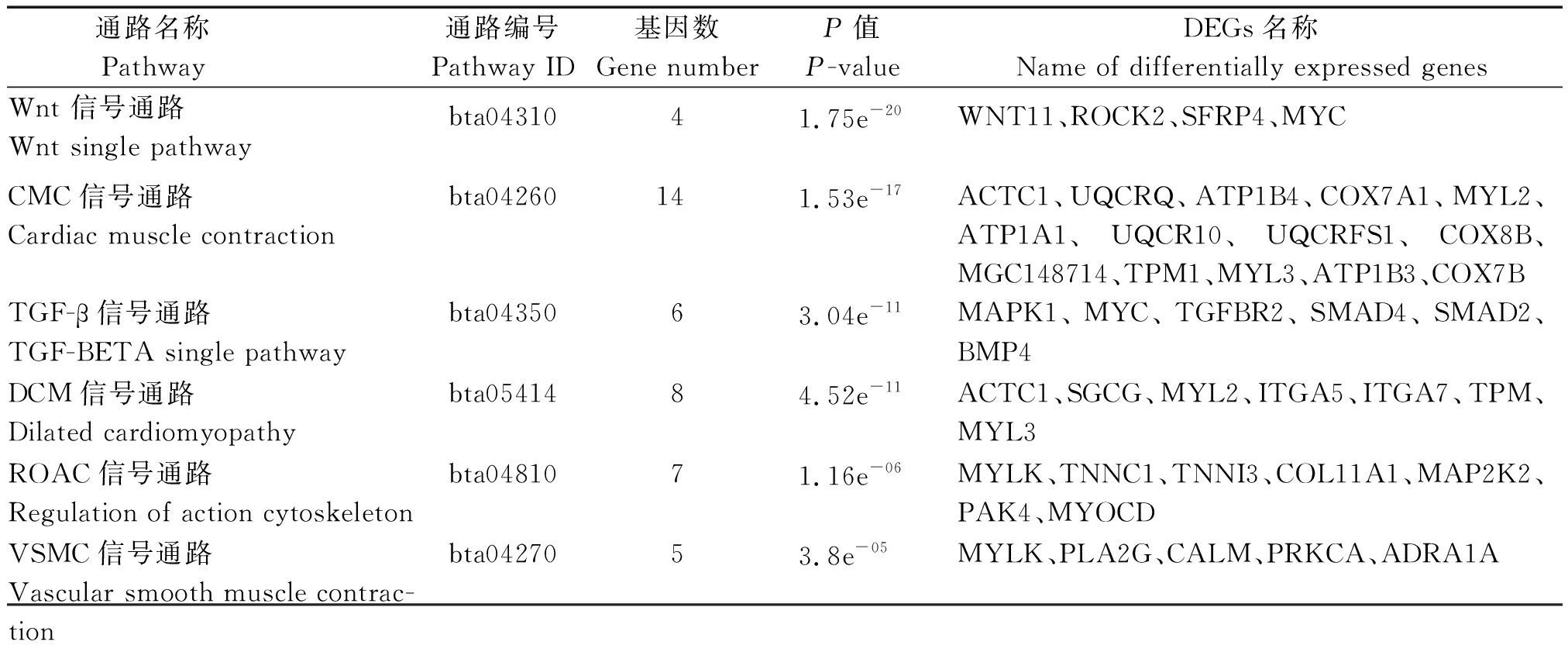
通路名称 Pathway 通路编号Pathway ID基因数Gene numberP值P-valueDEGs名称Name of differentially expressed genesWnt 信号通路 Wnt single pathwaybta0431041.75e-20WNT11、ROCK2、SFRP4、MYCCMC信号通路Cardiac muscle contractionbta04260141.53e-17ACTC1、UQCRQ、ATP1B4、COX7A1、MYL2、ATP1A1、UQCR10、UQCRFS1、COX8B、MGC148714、TPM1、MYL3、ATP1B3、COX7BTGF-β信号通路TGF-BETA single pathwaybta0435063.04e-11MAPK1、MYC、TGFBR2、SMAD4、SMAD2、BMP4DCM信号通路Dilated cardiomyopathybta0541484.52e-11ACTC1、SGCG、MYL2、ITGA5、ITGA7、TPM、MYL3ROAC信号通路Regulation of action cytoskeletonbta0481071.16e-06MYLK、TNNC1、TNNI3、COL11A1、MAP2K2、PAK4、MYOCDVSMC信号通路Vascular smooth muscle contrac-tionbta0427053.8e-05MYLK、PLA2G、CALM、PRKCA、ADRA1A
在差异表达基因蛋白质互作网络图5中,圆圈(Node)代表蛋白质,共542个蛋白质,差异表达基因蛋白质网络中互作最多的蛋白质是HMCN1,与5 588 aa互作,连通性最好;最少的是ATP5E和TOMM5,均互作51 aa。整合蛋白(ITGA7/1 137 aa、ITGA5/1 055 aa)和胶原蛋白(COL12A1/3 126 aa、COL15A1/1 386 aa)与其他蛋白间的互作最多,其次是肌球蛋白(MYL2/166 aa、MYL3/199 aa、MYLPF/170 aa),肌钙蛋白以及一些与生长和发育密切相关的调节因子互作较少。

图5 差异表达基因蛋白质互作网络图
Fig.5 The network of differential expression in gene protein interaction
3 讨论与结论
3.1 与参考基因对比分析
运用RNA-Seq测序法对肉牛的肌肉转录组进行分析,全面揭示不同生长性能肉牛肌肉间基因的表达差异,将有助于发现调控肌肉生长发育的相关基因,为后续了解基因功能以及开发分子育种新途径奠定基础[18-20]。本试验对转录组数据进行质量评估,结果发现碱基质量分值均高于30,错误率大部分都低于0.1%,表明测序碱基质量良好;文库中分别有85.38%和87.88%的原始序列可以比对到参考基因组,其中唯一映射分别达到79.40%和80.71%,文库覆盖率高,表明转录组测序在建库时样品代表性高,数据准确可靠,可用于后续试验分析。布莱凯特黑牛和鲁西黄牛转录组文库分别存在13 242和13 350个基因,其中5 500个基因的表达量相同,8 258个基因的表达量不同。依据FDR≤0.001且|log2Ratio|≥1筛选出554个差异显著的基因作为差异表达基因,其中296个基因表达上调,258个基因表达下调。进一步筛选发现差异表达基因中有36 个基因编码产生肌钙蛋白、肌红蛋白和肌球蛋白,认为这些基因可能为骨骼肌生长中的保守基因,该结果与小尾寒羊上的研究结果一致[21]。
3.2 差异基因筛选
对差异表达基因进行GO功能注释,结果分为生物学过程、细胞组分和分子功能三大类,生物学过程占据基因数最多。进一步分析发现生物学过程与新陈代谢过程中相关基因有280个,与生物学调控相关的基因219个,这些结果与先前在猪上的研究一致[22],说明这些差异表达基因之间相互作用,共同执行生物学功能。另外发现有多个基因参与了肌肉组织发育、肌细胞分化、肌肉组织形态发生等肌细胞和肌肉组织的形成过程。在GO功能注释包括25个二级分类中,其中10个二级分类共包括29个基因与肌细胞的发育和分化有关,分别是整合蛋白(ITGA3、ITGA7 和 ITGA8)、胶原蛋白(COL1A1、COL4A1和 COL4A4)、肌球蛋白(MYL2、MYH6、MYOCD、MYL6B、MYLPF和MYL3)、肌钙蛋白(TNNI1、TNNI3和 TNNC1)和一些调控因子,说明上述基因可能在肌细胞和肌肉组织的形成过程中挥发一定的作用。其中MYH6、MYL2、MYL3、MYLPF、TNNC1、TNNI3、COL4A4、 ITGA7和 ITGA8在鲁西黄牛文库中表达下调,ITGA3、MYOCD、COL4A1和COL1A1表达上调。 KEGG通路注释结果发现共有554个基因被注释到KEGG中的226个通路中,其中61个基因参与了新陈代谢通路、9个基因参与肌动蛋白细胞骨架通路,6个基因参与了ECM受体干扰通路。根据文库的差异表达基因,筛选出6条与骨骼肌生长发育相关的通路(Wnt、CMC、TGF-BETA、DCM、ROAC、VSMC),说明这些信号通路与肌细胞的增殖分化与骨骼肌的生长发育过程密切相关。MYL2、MYL3、MYLPF均是 Wnt、DMC和TGF-β 信号通路中的成员,这些通路参与了肌细胞和肌肉组织的形成过程。
STRING是收录多个物种预测的和试验验证的蛋白质-蛋白质互作的数据库,结合差异表达分析结果和数据库收录的互作关系,构建差异表达基因互作网络。本试验对554个差异表达基因编码的蛋白进行互作分析,结果发现,细胞外基质中的整合蛋白和胶原蛋白与其他蛋白有大量的互作,其中整合蛋白(ITGA7/1 137 aa、ITGA5/1 055 aa)和胶原蛋白(COL12A1/3 126 aa、COL15A1/1 386 aa)与其他蛋白间的互作最多,其次是肌球蛋白(MYL2/166 aa、MYL3/199 aa、MYLPF/170 aa),肌钙蛋白以及一些与生长和发育密切相关的调节因子互作则相对较少,该结果揭示细胞外基质、肌球蛋白、肌钙蛋白参与了肌肉生物学过程,这一发现与人的研究结果一致[23]。MYL2、MYL3和 MYLPF与其他蛋白间互作良好,其均是 Wnt、DMC和TGF-β 信号通路中的成员,这些通路参与了肌细胞和肌肉组织的形成的过程[24-26]。从本研究转录组数据推测,MYL2、MYL3和 MYLPF基因可能与骨骼肌的生长和发育的调控密切相关,筛选作为骨骼肌候选基因,但具体功能尚不清楚,需要在本研究基础上进一步应用分子以及生化手段进行揭示。
3.3 骨骼肌候选基因
肌球蛋白轻链2(Myosin light chain 2,MLC2或MYL2)是肌球蛋白轻链家族的一员,为调节性轻链,是由167个氨基酸组成的小分子蛋白,位于牛的17号染色体[27]。早期研究MYL2主要集中在心肌组织中,基因突变可导致心肌增生[28]。张素珍等[29]发现MYL2基因在成熟的小鼠骨骼肌中表达而在体外培养的C2C12成肌细胞分化过程中不表达,敲除该基因后,小鼠的生长出现严重异常,甚至有致命性的表现[30];在鼠的非肌肉成纤维细胞中,Ⅱ型肌纤维的完整性主要靠MYL2基因来维持[31],对Ⅱ型肌纤维的活性起调控作用,使肌纤维的类型发生转化,进而影响肌肉的生长[32]。
肌球蛋白轻链3(Myosin light chain 3,MLC1v或MYL3)属于肌浆球蛋白家族,编码199个氨基酸,位于牛的22号染色体[33]。早期研究发现,MYL3结合钙离子促进肌肉发育,并参与横纹肌的收缩,进一步研究发现MYL3似乎参与了力量发育和神经调节肌肉收缩的过程[34-35],最新研究发现人类卫星细胞在缺氧环境中培养时,MYL3基因表达增加,促进其分化成成肌细胞[36]。使用Northern印迹法研究发现成年小鼠心肌中MYL3表达水平高于骨骼肌[37]。
快肌肌浆球蛋白可调节磷酸化轻链(Myosin light chain,phosphorylatable,fast skeletal muscle,HUMMLC2B或MYLPF)编码160个氨基酸,位于牛的25号染色体[38]。Xu 等[39]研究MYLPF基因含有一个强启动子,能在体细胞和非肌肉的肌肉中激活[40]。Lee等[41]研究发现,MYLPF基因的SNP位点对不同基因型的猪肉品质化学组成、滴水损失和剪切力具有显著相关性,推测MYLPF基因的SNP位点可以作为肉质性状选择的标记辅助选择位点。Ryan等[42]最近研究MYLPF基因启动子区域SNPs时同样发现与肉质性状相关。
本试验通过 RNA-Seq 得到了两转录组文库质量可靠的大数据均达到5G左右,通过生物信息学分析筛选得到骨骼肌差异基因MYL2、MYL3和MYLPF;因筛选的基因是Wnt、DMC和TGF-β 信号通路中的成员,参与了肌细胞和肌肉组织的形成的过程,推测MYL2、MYL3和 MYLPF基因可能与骨骼肌的生长和发育的调控密切相关,需要进一步分析验证。
[1] 王国刚,王明利,杨 春. 中国肉牛产业发展的阶段识别及时空分异特征[J]. 经济地理,2014,34(10):131-136,170.
[2] 侯仕农. 中国肉牛产业可持续发展探讨[J]. 当代畜禽养殖业,2016(8):50.
[3] Birney E,Stamatoyannopoulos J A,Dutta,et al. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project[J]. Nature,2007,447(7146):799-816.
[4] Mortazavi A,Williams B A,Mccue K,et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods,2008,5(7):621-628.
[5] 纪 岭,李小金. 转录组测序(RNA-seq)技术及其应用[J]. 农技服务,2015,32(7):171-172.
[6] Zhang J,Wu K,Zeng S,et al.Transcriptome analysis of cymbidium sinense and its application to the identification of genes associated with floral development[J]. BMC Genomics,2013,14(1):279.
[7] Donadio C,Tognotti D,Caponi L,et al. β-trace protein is highly removed during haemodialysis with high-flux and super high-flux membranes[J]. BMC Nephrology,2017,18(1):68.
[8] Zheng Y,Zhao L,Gao J,et al. Assembler:a package for de novo assembly of Roche-454/Sanger transcriptome sequences[J]. BMC Bioinformatics,2011,12(1):453.
[9] Mccabe M,Waters S,Morris D,et al. RNA-seq analysis of differential gene expression in liver from lactating dairy cows divergent in negative energy balance[J]. BMC Genomics,2012,13(1):193.
[10] Bai X E,Zheng Z Q,Liu B,et al. Whole blood transcriptional profiling comparison between different milk yield of Chinese Holstein cows using RNA-seq data[J]. BMC Genomics,2016,17(7):512.
[11] 张文力. 高通量测序数据分析现状与挑战[J]. 集成技术,2012,1(3):20-24.
[12] Clark T A,Sugnet C W,Ares M,et al. Genomewide analysis of m RNA processing in yeast using splicing-specific microarrays[J]. Science,2002,296(5569):907-910.
[13] Okoniewski M J,Miller C J. Hybridization interactions between probesets in short oligo microarrays lead to spurious correlations[J]. BMC Bioinformatics,2006,7(1):276.
[14] Royce T E,Rozowsky J S,Gerstein M B. Toward a universal microarray:prediction of gene expression through nearest-neighbor probe sequence identification[J]. Nucleic Acids Research,2007,35(15):e99.
[15] 王海波,邹竹荣,龚 明. 基于RNA-seq数据筛选的小桐子抗冷相关基因的表达模式及其聚类分析[J]. 基因组学与应用生物学,2014,33(6):1196-1205.
[16] Brenner S,Johnson M,Bridgham J,et al.Gene expression analysis by massively parallel signature sequencing(MPSS)on microbead arrays[J]. Nature Biotechnology,2000,18(6):630-634.
[17] Schrooten C,Dassonneville R,Ducrocq V,et al. Error rate for imputation from the Illumina BovineSNP50 chip to the Illumina BovineHD chip[J]. Genetics Selection Evolution,2014,46(1):10.
[18] Gómez J,Reguero J R,Morís C,et al. Mutation analysis of the main hypertrophic cardiomyopathy genes using multiplex amplification and semiconductor next-generation sequencing[J]. Circulation Journal,2014,78(12):2963-2971.
[19] Kodzius R,Kojima M,Nishiyori H,et al. Cap analysis of gene expression[J]. Nature Methods,2006,3(3):211-222.
[20] Nakamura M,Carninci P.Cap analysis gene expression:CAGE[J].Tanpakushitsu Kakusan Koso,2004,49(17S):2688-2693.
[21] Zhang C L. Studies on bimodular transcriptome and myosin light chain gene family structure of small tail Han sheep and dorper[J]. Gene,2015,569(1):51-59.
[22] Damon M,Wyszynska-Koko J,Vincent A,et al. Comparison of muscle transcriptome between pigs with divergent meat quality phenotypes identifies genes related to muscle metabolism and structure[J]. PLoS One,2012,7(3):e33763.
[23] Wu G,Feng X,Stein L. A human functional protein interaction network and its application to cancer data analysis[J]. Genome Biology,2010,11(5):R53.
[24] Lee E C,Lotz M M,Steele G D,et al.The integrin alpha 6 beta 4 is a laminin receptor[J]. Cell Biol, 1992, 117(3): 671-678.
[25] Massague J. How cells read TGF-beta signals[J]. Nature Reviews Molecular Cell Biology,2000,1(3):169-178.
[26] Legant W R,Choi C K,Miller J S,et al. Multidimensional traction force microscopy reveals out-of-plane rotational moments about focal adhesions[J]. Proceedings of the National Academy of Sciences of the United States of America,2013,110(3):881-886.
[27] Kang P B,Griggs R C. Advances in muscular dystrophies[J]. JAMA Neurology,2015,72(7):741-742.
[28] Weterman M A J,Barth P G,Spaendonck-Zwarts S K. Y.Recessive MYL2 mutations cause infantile type I muscle fibre disease and cardiomyopathy[J]. Brain,2013,136(Pt1):282-293.
[29] 张素珍,解慧琪,徐 永,等. 肌球蛋白轻链在肌肉再生中作用的体外实验研究[J]. 中国修复重建外科杂志,2008,22(6):753-758.
[30] Park I,Han C,Jin S,et al. Myosin regulatory light chains are required to maintain the stability of myosin II and cellular integrity[J]. Biochemical Journal,2011,434(1):171-180.
[31] Zhang S Z,Xu Y,Xie H Q,et al. The possible role of Myosin light chain in myoblast proliferation[J]. Biological Research,2009,42(1):121-132.
[32] Vicente-Manzanares M,Ma X,Adelstein R S,et al. Non-muscle myosin II takes centre stage in cell adhesion and migration[J]. Nature Reviews Molecular Cell Biology,2009,10(11):778-790.
[33] Timson D J ,Trayer H R, Smith K J,et al. Size and charge requirements for kinetic modulation and actin binding by alkali 1-type myosin essential light chains[J]. Biological Chemistry, 1999, 274(26):18271-18277.
[34] Moran I. Tuning smooth muscle contraction by molecular motors[J]. Molecular Medicine,2003,81(8):481-487.
[35] Meder B,Laufer C,Hassel D,et al. A single serine in the carboxyl terminus of cardiac essential myosin light chain-1 controls cardiomyocyte contractility in vivo[J]. Circulation Research,2009,104(5):650-U175.
[36] Koning M,Werker P M,Luyn van M J,et al.Hypoxia promotes proliferation of humanmyogenic satellite cells:a potential benefactor in tissue engineering of skeletal muscle[J]. Tissue Eng Part A,2011,17(13):1747-1758.
[37] Komurcu-Bayrak E,Ozsait B,Erginel-Unaltuna N, et al. Isolation and analysis of genes mainly expressed in adult mouse heart using subtractive hybridization cDNA library[J]. Molecular Biology Reports, 2012, 39(8): 8065-8074.
[38] Blumenthal D K. Stull J T.Activation of skeletal musclemyosin light chain kinase by Calcium(2+)and calmodulin[J]. Biochemistry,1980,19(24):5608-5614.
[39] Xu Y,He J,Tian H L,et al. Fast skeletal muscle-specific expression of a zebrafishmyosin light chain 2 gene and characterization of its promoter bydirect injection into skeletal muscle[J]. DNA and Cell Biology,1999,18(1):85-95.
[40] Ju B,Chong S W,He J,et al. Recapitulation of fast skeletal muscle development in zebrafish by transgenic expression of GFP underthe MYLZ2 promoter[J]. Developmental Dynamics,2003,227(1):14-26.
[41] Lee Y H,Kwon E J,Cho E S,et al. Association analysis of polymorphism in KIAA1717,HUMMLC2B,DECR1 and FTO genes with meat quality traits of the Berkshire breed[J]. African Journal of Biotechnology,2011,10(25):5068-5074.
[42] Ryan M T,O'halloran A M,Hamill R M,et al. Polymorphisms in the regulatory region of the porcine MYLPF gene are related to meat quality traits in the Large White breed[J]. Meat Science,2016(113):104-106.