RT-qPCR是一种在转录水平上对mRNA表达量进行评价的常用方法,具有定量准确、灵敏度高、重复性好、特异性强、成本低等特点[1-2]。但是,影响mRNA表达量的因素很多,如RNA提取的质量和纯度、反转录效率、扩增效率[3-4]。因此,需用内参基因对目的基因表达量进行校正和标准化,使实验数据更准确、可靠。GAPDH、ACTB、RPS18等内参基因常被用以标准化目的基因,但对其稳定性进行对比筛选的研究较少,由于内参基因在不同组织、生理状态以及不同试验条件下的表达水平不稳定,具有品种和组织特异性[3,5-7]。因此,为获得准确、可靠的试验数据,需要根据品种、组织、细胞类型以及试验条件选择适宜的稳定表达的内参基因对RT-qPCR数据进行校正和标准化[8-9]。
猪是提供肉类的重要经济型家畜,也常作为研究疾病的动物模型[10]。因此以猪为素材,利用RT-qPCR方法研究了大量基因的表达量,以提高猪肉产量,改善肉品质[11-19]。到目前为止,已有不少学者对猪脂肪组织、肌肉组织、软组织、外周血单核细胞、子宫内膜上皮、内脏器官组织、不同胚胎期样本,以及品种和杂交组合进行内参基因筛选[6,10,20-30]。大蒲莲猪是我国优质的地方猪种,首批入选中国畜禽遗传资源志,但是关于大蒲莲猪不同发育阶段体组织内参基因稳定性筛选的研究未见报道,为此本研究应用geNorm[3]和NormFinder[31]软件评价大蒲莲猪不同发育阶段肌肉、肠组织、内脏器官、血液中ACTB、B2M、GAPDH、RPL4、SDHA、TBP、YWHAZ、PPIA 8个内参基因的稳定性,筛选适用于校正和标准化目的基因表达量的稳定内参基因,为后续大蒲莲猪基因的研究提供参考依据。
1 材料和方法
1.1 组织样品采集
选择出生日期相近的健康大蒲莲猪24头,分别于1日龄、14日龄、1月龄、2月龄、4月龄、6月龄进行屠宰,每阶段屠宰4头。屠宰后取肌肉、心脏、肝脏、脾脏、肺脏、肾脏、肠组织(十二指肠、空肠、回肠)和血液,液氮中速冻后置-80 ℃冰箱中保存备用。
1.2 总RNA提取及逆转录
利用RNAiso Plus(TaKaRa,大连)试剂进行组织总RNA的提取。取0.3 g组织液氮研磨成粉末和0.4 mL血液加入1 mL RNAiso Plus中,充分振荡,参照说明书中步骤提取样本中的总RNA。使用Nanodrop 2000微量分光光度计(赛默飞,美国)进行RNA浓度与纯度的检测,而后用0.8%琼脂糖电泳检测RNA的完整性。取1 μg左右 RNA样品,根据PrimerScript RT reagent Kit with gDNA Eraser(TaKaRa,大连)试剂盒的操作步骤,进行反转录合成cDNA。
1.3 引物设计合成和荧光定量PCR
根据相关文献报道,选择ACTB、B2M、GAPDH、RPL4、SDHA、TBP、YWHAZ、PPIA作为候选内参基因进行表达稳定性分析。内参基因引物序列均参考文献[21-22,32]。内参基因引物见表1。
10倍倍比稀释cDNA样品,使用含有10-4,10-5,10-6,10-7,10-8,10-9,10-10倍cDNA样品作为反应模板,计算候选内参基因的扩增效率。通过LightCycler® 480 software release软件计算各候选内参基因Ct值,每个稀释梯度的起始模板浓度与Ct值间呈线性关系。根据Ct值在Excel中经对数拟合作图,绘制标准曲线。根据E=(10(-1/slope))×100计算扩增效率。8个内参基因RT-PCR扩增效率为89%~105%。
本研究使用LightCycler® 480 SYBR Green Ⅰ Master(Roche,瑞士)荧光定量试剂盒,Roche LightCycler® 480(Roche,瑞士)荧光定量PCR仪。反应体系为20 μL,包括:SYBR GreenⅠMaster 10 μL,上、下游引物(0.5 μmol/L)各1 μL,cDNA 1 μL,双蒸水7 μL。反应条件为:①预变性:95 ℃ 5 min;②扩增反应:95 ℃预变性10 s,60 ℃退火和延伸20 s,40个循环;③溶解曲线形成:95 ℃ 5 s,65 ℃ 1 min,97 ℃ 10 s。反应后根据溶解曲线确保PCR产物具有特异性,导出内参基因的Ct值。每个样本每对引物3个重复,每板设阴性对照。
1.4 geNorm分析程序
geNorm软件是Vandesompele等[3]编写的用于Excel平台的VBA宏程序,用于RT-qPCR检测候选内参基因稳定性评价和适宜内参基因数的确定。通过计算某一内参基因与其他所有基因两两比值的平均变异度(M),评价基因稳定性,M值越小,基因表达越稳定。同时计算引入 1 个新的内参基因后标准化因子的配对变异V值,根据 Vn/Vn+1确定所需适宜内参基因个数。默认V值为 0.15,如果Vn/Vn+1大于 0.15,则有必要引入第n+ 1 个内参基因,反之,则不必引入新的内参基因,最适内参基因数为n个。
1.5 NormFinder分析程序
NormFinder是通过评价内参基因的表达稳定性的程序[31]。NormFinder程序不但可以比较内参基因的表达差异,还可以计算样品组间变异。内参基因的稳定性值低,表示基因的表达变异小,反之,则表示基因的表达变异大,但是该程序只能筛选出一个最适宜的内参基因。
1.6 MIQE指南
内参基因表达量检测按照实时定量PCR实验研究发表最小信息指南进行[33]。
1.7 数据处理与分析
在 Excel 表格找到不同样本中某一参考基因最小 Ct 值,再计算其他样本中该参考基因Ct 值与最小Ct 值的差值ΔCt,样本内参基因的相对表达量则为 2-ΔCt(ΔCt=样本 Ct 值-最小 Ct 值),将数据导入 geNorm V3.5[3]和NormFinder V2.0[31],计算各候选内参基因稳定性值。
2 结果与分析
2.1 RNA质量检测和荧光定量PCR体系的建立
经1%的琼脂糖凝胶电泳,RNA的3条带(28S、18S和5S)清晰,证明其完整性较好。紫外分光光度计检测表明,各样品总RNA A260/280比值为1.93~2.01,浓度为194.7~371.5 ng/μL,表明RNA样品质量良好。
候选内参基因的扩增曲线呈典型的“S”形,融解曲线在79~91 ℃出现标准的单峰,而在其他位置未有任何峰出现。通过梯度稀释cDNA样本作为模板进行PCR扩增后绘制每个候选内参基因的标准曲线,通过计算得知,各内参基因的标准曲线的扩增效率变化在0.95~1.04,符合利用相对定量分析法进行表达量分析的要求。
2.2 利用geNorm 分析候选内参基因表达稳定性结果
2.2.1 不同日龄大蒲莲猪候内脏中内参基因表达稳定性分析 应用geNorm软件评价大蒲莲猪内脏中8个候选内参基因的稳定性(M值)(图1)。TBP和PPIA是心脏、肝脏和肺脏中表达最稳定的内参基因,M值分别为0.425,0.181和0.181。PPIA和YWHAZ是肾脏中表达最稳定的内参基因,M值为0.125。PPIA和RPL4是脾脏中表达最稳定的内参基因,M值为0.169。大蒲莲猪心脏中候选内参基因稳定性(M值)依次为TBP和PPIA(0.43)<YWHAZ(0.49)<RPL4(0.54)<GAPDH(0.59)<ACTB(0.70)<SDHA(0.78)<B2M(0.85),肝脏中候选内参基因稳定性(M值)依次为TBP和PPIA(0.181)<RPL4(0.250)<YWHAZ(0.326)<GAPDH(0.429)<SDHA(0.488)<ACTB(0.568)<B2M(0.713),肾脏中候选内参基因稳定性(M值)依次为PPIA和YWHAZ(0.125)<TBP(0.229)<RPL4(0.287)<ACTB(0.334)<GAPDH(0.372)<B2M(0.561)<SDHA(0.676),脾脏中候选内参基因稳定性(M值)依次为PPIA和RPL4(0.169)<TBP(0.232)<YWHAZ(0.369)<B2M(0.440)<ACTB(0.498)<GAPDH(0.653)<SDHA(0.802),肺脏中候选内参基因稳定性(M值)依次为TBP和PPIA(0.181)<RPL4(0.349)<ACTB(0.390)<YWHAZ(0.436)<GAPDH(0.611)<SDHA(0.801)<B2M(0.950)。使用geNorm程序对大蒲莲猪心脏、肝脏、肾脏、脾脏和肺脏组织样同时进行内参基因稳定性评价结果显示,最稳定内参基因为TBP和RPL4(0.443),其后依次为PPIA(0.550)、YWHAZ(0.651)、B2M(0.739)、ACTB(0.843)、SDHA(0.998)、GAPDH(1.191)。
Vandesompele等[3]提出使用最小数量的最稳定内参基因计算标准化因子(NF)。为得到可信赖的准确RT-qPCR结果,本试验还对最佳内参基因使用数量进行了分析。geNorm 程序建议Vn/Vn+1设置为0.15,这表明如果Vn/Vn+1小于0.15,则没必要引入第n + 1 个内参基因。大蒲莲猪心脏中内参基因的配对变异V2/3为0.161,肝脏为0.091,肾脏为0.092,脾脏为0.083,肺脏为0.141(图2)。只有心脏中内参基因的配对变异V2/3大于0.15,肝脏、肾脏、脾脏、肾脏、肺脏的V2/3均小于0.15,这就表明测定心脏基因的表达量时,则需要引入3个内参基因,V3/4为0.130。
使用geNorm程序对大蒲莲猪心脏、肝脏、肾脏、脾脏和肺脏组织样一起进行最佳内参基因使用数量分析显示,V2/3为0.190,虽然引入新的内参基因可使配对变异V值变小,但是V3/4、V4/5、V5/6、V6/7、V7/8值分别为0.168,0.157,0.156,0.206,0.200,均大于0.15。这表明本试验中大蒲莲猪心脏、肝脏、肾脏、脾脏和肺脏组织样适宜内参基因需分类选择。
2.2.2 不同日龄大蒲莲猪候血液中内参基因表达稳定性分析 应用geNorm软件评价大蒲莲猪血液中8个候选内参基因的稳定性(M值)(图1)。ACTB和YWHAZ是血液中最稳定表达内参基因,M值为0.114。候选内参基因稳定性(M值)依次为ACTB和YWHAZ(0.18)<GAPDH(0.29)<PPIA(0.34)<SDHA(0.41)<TBP(0.54)<B2M(0.59)<RPL4(0.72)。
geNorm软件对内参基因适宜数量分析显示(图2),V2/3为0.114,小于0.15,而且V3/4低于V2/3,这表明选择2个内参基因(ACTB和YWHAZ)标准化血液中目的基因RT-qPCR检测结果可以保证目的基因表达量的准确性,如果需要更准确、可靠的检测结果可选择3个内参基因标准化目的基因表达量。
2.2.3 不同日龄大蒲莲猪候肠组织中内参基因表达稳定性分析 应用geNorm软件评价大蒲莲猪肠组织中8个候选内参基因的稳定性(M值)。TBP和PPIA是肠组织中最稳定表达内参基因,M值为0.21。内参基因稳定性(M值)依次为TBP和PPIA(0.210)<YWHAZ(0.265)<GAPDH(0.307)<RPL4(0.367)<B2M(0.463)<ACTB(0.541)<SDHA(0.599)(图1)。GeNorm软件对内参基因适宜数量分析显示,V2/3为0.93,小于0.15,而且V3/4低于V2/3(图2),这表明选择2个内参基因(TBP和PPIA)标准化肠组织中目的基因表达量,可以保证RT-qPCR数据的准确性。如果需要更准确、可靠的RT-qPCR数据,可选择3个内参基因标准化目的基因表达量。

A、B、C、D、E、F、G、H分别为GeNorm软件计算不同日龄大蒲莲猪心脏、肝脏、肾脏、脾脏、肺脏、血液、肠组织、肌肉8个候选内参基因的
平均表达稳定性值M和2个最适宜内参基因。M值越低,内参基因表达越稳定。
The average expression stability(M)of eight candidate reference genes and the best combination of two genes were calculated for heart(A),liver(B),kindey(C),spleen(D),lung(E),blood(F),intestine(G),muscle(H). Lower M values indicate more stable expression.
图1 geNorm软件对8个候选内参基因稳定性分析
Fig.1 Ranking of eight candidate reference genes using geNorm program
2.2.4 不同日龄大蒲莲猪候肌肉中内参基因表达稳定性分析 geNorm软件分析评价大蒲莲猪肌肉中8个内参基因的稳定性(图1)。TBP和PPIA是大蒲莲猪肌肉中表达最稳定内参基因,M值分别为0.448。内参基因稳定性(M值)依次为TBP和PPIA(0.448)<YWHAZ(0.460)<GAPDH(0.516)<RPL4(0.537)<ACTB(0.576)<SDHA(0.608)<B2M(0.647)。geNorm软件对内参基因适宜数量分析显示,V2/3为0.136,小于0.15,而且V3/4低于V2/3(图2),这表明选择2个内参基因(TBP和PPIA)标准化肌肉中目的基因表达量可以保证RT-qPCR数据的准确性。如果需要更准确、可靠的RT-qPCR数据,可选择3个内参基因标准化目的基因表达量。

A、B、C、D、E、F、G、H分别为geNorm软件计算不同日龄大蒲莲猪心脏、肝脏、肾脏、脾脏、肺脏、血液、肠组织、肌肉适宜内参基因数。
The optimal number of reference genes for normalization were determinated for heart(A),liver(B),kindey(C),spleen(D),lung(E),blood(F),intestine(G),muscle(H).
图2 geNorm软件分析确定标准化的最佳参考基因数
Fig.2 Determination of the optimal number of reference genes for normalization using geNorm program
2.3 NormFinder分析
NormFinder根据内参基因表达量变化计算稳定值和标准误。NormFinder分析结果显示,TBP是大蒲莲猪肌肉、心、肝、脾、肺、肾和肠组织中最稳定的内参基因,稳定性值最低,血液中内参基因ACTB的稳定性值最低,即ACTB为血液中最稳定内参基因(表2)。大蒲莲猪肌肉中内参基因的稳定性依次为TBP(0.305)>PPIA(0.394)>YWHAZ(0.473)>SDHA(0.478)>RPL4(0.535)>ACTB(0.642)>GAPDH(0.653)>B2M(0.946),心脏中内参基因的稳定性依次为TBP(0.183)>PPIA(0.251)>YWHAZ(0.287)>GAPDH(0.305)>RPL4(0.338)>ACTB(0.583)>SDHA(0.622)>B2M(0.664),肝脏中内参基因的稳定性依次为TBP(0.091)>PPIA(0.112)>YWHAZ(0.188)>RPL4(0.275)>SDHA(0.327)>GAPDH(0.398)>ACTB(0.491)>B2M(0.747),肾脏中内参基因的稳定性依次为TBP(0.047)>PPIA(0.194)>YWHAZ(0.212)>RPL4(0.218)>ACTB(0.298)> GAPDH(0.311)>B2M(0.621)>SDHA(0.638),脾脏中内参基因的稳定性依次为TBP(0.061)> RPL4(0.088)>PPIA(0.286)>ACTB(0.418)>GAPDH(0.576)>B2M(0.775)>YWHAZ(0.891)>SDHA(5.110),肺脏中内参基因的稳定性依次为TBP(0.086)>RPL4(0.090)>PPIA(0.110)>ACTB(0.112)>YWHAZ(0.396)>GAPDH(0.673)>SDHA(0.845)>B2M(0.887),肠组织中内参基因的稳定性依次为TBP(0.056)>PPIA(0.062)>YWHAZ(0.306)>GAPDH(0.356)>ACTB(0.573)>RPL4(0.668)>B2M(0.943)>SDHA(1.485),血液中内参基因的稳定性依次为ACTB(0.062)>YWHAZ(0.214)>GAPDH(0.220)>PPIA(0.309)>SDHA(0.342)>B2M(0.408)>TBP(0.442)< RPL4(0.724)。
表1 候选内参基因详细信息
Tab.1 Detail information of selected candidate reference genes
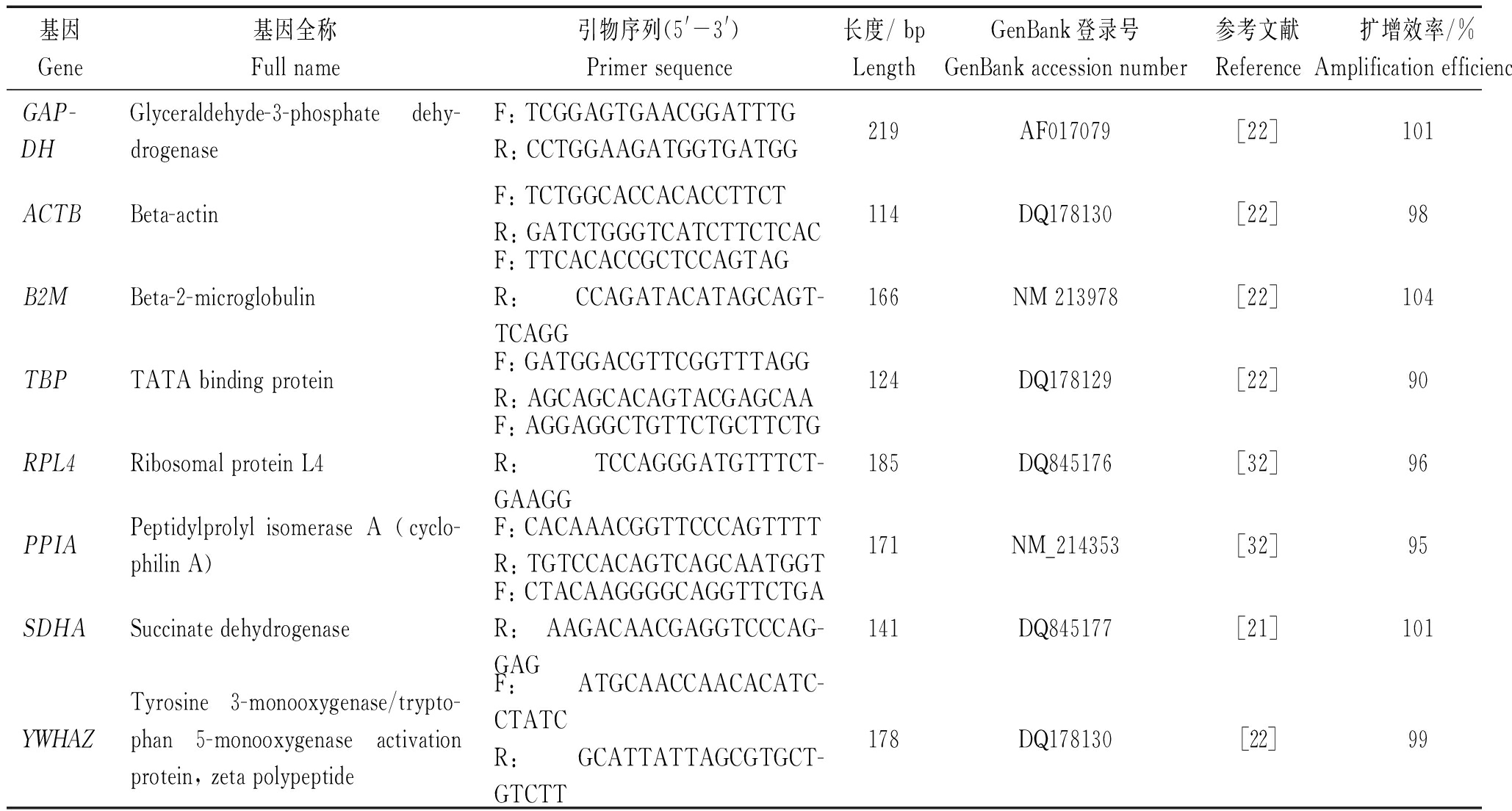
基因Gene基因全称Full name引物序列(5'-3')Primer sequence长度/ bpLengthGenBank登录号GenBank accession number参考文献Reference扩增效率/%Amplification efficiency回归系数R2GAP-DHGlyceraldehyde-3-phosphate dehy-drogenaseF: TCGGAGTGAACGGATTTGR: CCTGGAAGATGGTGATGG219AF017079[22]1010.993 4ACTBBeta-actinF: TCTGGCACCACACCTTCTR: GATCTGGGTCATCTTCTCAC114DQ178130[22]980.994 5B2MBeta-2-microglobulinF: TTCACACCGCTCCAGTAGR:CCAGATACATAGCAGT-TCAGG166NM 213978[22]1040.993 4TBPTATA binding proteinF: GATGGACGTTCGGTTTAGGR: AGCAGCACAGTACGAGCAA124DQ178129[22]900.992 8RPL4Ribosomal protein L4F: AGGAGGCTGTTCTGCTTCTGR: TCCAGGGATGTTTCT-GAAGG185DQ845176[32]960.995 7PPIAPeptidylprolyl isomerase A (cyclo-philin A)F: CACAAACGGTTCCCAGTTTTR: TGTCCACAGTCAGCAATGGT171NM_214353[32]950.994 2SDHASuccinate dehydrogenaseF: CTACAAGGGGCAGGTTCTGAR:AAGACAACGAGGTCCCAG-GAG141DQ845177[21]1010.992 4YWHAZTyrosine 3-monooxygenase/trypto-phan 5-monooxygenase activation protein, zeta polypeptideF: ATGCAACCAACACATC-CTATCR: GCATTATTAGCGTGCT-GTCTT178DQ178130[22]990.995 2
表2 NormFinder分析大蒲莲猪不同发育阶段体组织内参基因稳定性
Tab.2 Stability values of reference gene in Dapulian pigs in different tissue with different development periods calculated by NormFinder
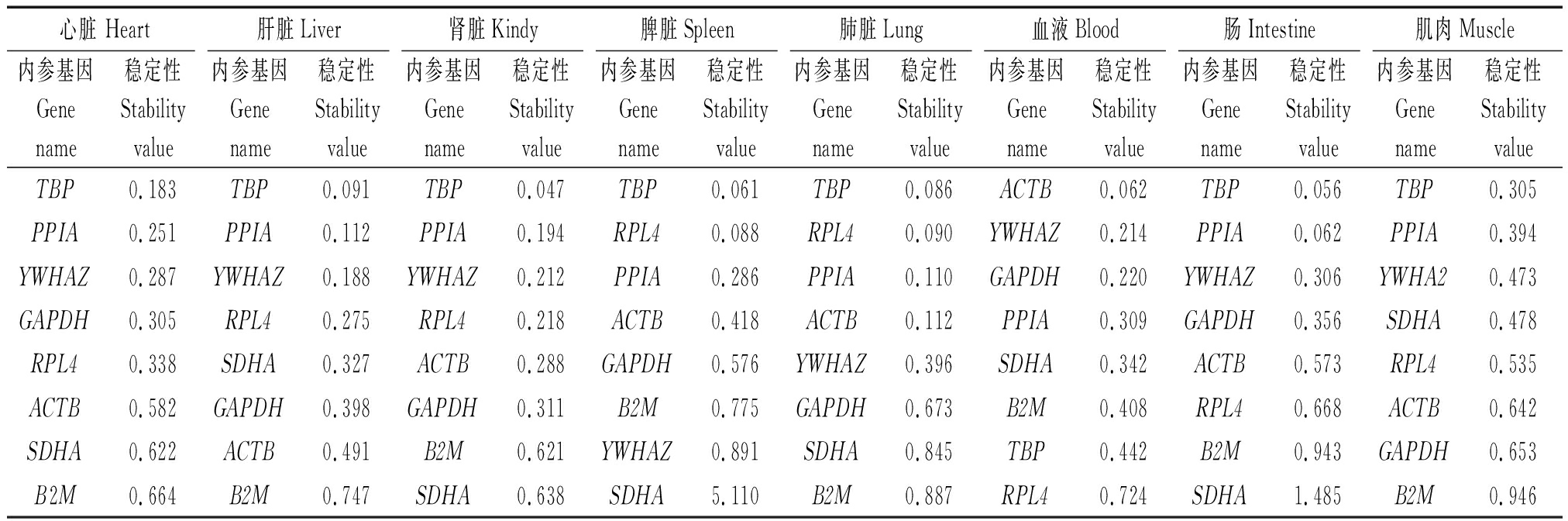
心脏 Heart肝脏Liver肾脏Kindy脾脏Spleen肺脏Lung血液Blood肠Intestine肌肉Muscle内参基因Genename稳定性Stabilityvalue内参基因Genename稳定性Stabilityvalue内参基因Genename稳定性Stability value内参基因Genename稳定性Stabilityvalue内参基因Genename稳定性Stabilityvalue内参基因Genename稳定性Stabilityvalue内参基因Genename稳定性Stabilityvalue内参基因Genename稳定性StabilityvalueTBP0.183TBP0.091TBP0.047TBP0.061TBP0.086ACTB0.062TBP0.056TBP0.305PPIA0.251PPIA0.112PPIA0.194RPL40.088RPL40.090YWHAZ0.214PPIA0.062PPIA0.394YWHAZ0.287YWHAZ0.188YWHAZ0.212PPIA0.286PPIA0.110GAPDH0.220YWHAZ0.306YWHA20.473GAPDH0.305RPL40.275RPL40.218ACTB0.418ACTB0.112PPIA0.309GAPDH0.356SDHA0.478RPL40.338SDHA0.327ACTB0.288GAPDH0.576YWHAZ0.396SDHA0.342ACTB0.573RPL40.535ACTB0.582GAPDH0.398GAPDH0.311B2M0.775GAPDH0.673B2M0.408RPL40.668ACTB0.642SDHA0.622ACTB0.491B2M0.621YWHAZ0.891SDHA0.845TBP0.442B2M0.943GAPDH0.653B2M0.664B2M0.747SDHA0.638SDHA5.110B2M0.887RPL40.724SDHA1.485B2M0.946
3 讨论与结论
基因表达分析是分子生物学研究中最常用的方法之一,由于RT-qPCR方法准确性、特异性高,成本低,已经成为量化基因表达的常用方法。但是,不同内参基因的归一化影响RT-qPCR数据的准确性。因此,选择适宜的内参基因是正确评价基因表达可靠性和重现性的关键。事实上,研究学者已经对不同品种、细胞、组织,以及不同试验条件下内参基因进行筛选。近年来为研究基因的功能,以及基因与肉品质、繁殖性能间的关联,应用RT-qPCR分析猪体组织、胚胎基因表达[11,34-39]。遗憾的是,这些研究均使用常用的内参基因,如GAPDH、ACTB和B2M对RT-qPCR数据进行标准化,而未对内参基因稳定性进行筛选,选择最稳定的内参基因对RT-qPCR数据进行标准化。Larsen等[36,40]根据前人研究选择使用内参基因GAPDH标准化Presenilin-1基因的mRNA表达,但对猪脑组织基因表达研究中分析了不同内参基因的表达稳定性。为获得准确的RT-qPCR数据,需要对猪不同组织、不同品种内参基因稳定性进行筛选。因此,本试验对大蒲莲猪不同发育阶段内脏、血液、肠组织、肌肉等组织中内参基因稳定性进行对比分析,选择适宜内参基因。
本试验采用geNorm、NormFinder软件分析不同日龄大蒲莲猪心脏、肝脏、脾脏、肺脏、肾脏、肠组织(大肠和小肠)、血液、肌肉中8个内参基因的稳定性,选择适宜的内参基因。结果表明,尽管geNorm、NormFinder软件对内参基因稳定性排列顺序不同,但是最稳定内参基因基本相同。由于geNorm、NormFinder软件的计算法则和分析程序不同,所以内参基因稳定性排列顺序会稍有差异。为选择最适宜、最稳定的内参基因,有必要使用这2种软件联合分析,为适宜内参基因选择提供依据。大蒲莲猪心脏中TBP、PPIA和YWHAZ为表达最稳定的3个内参基因,geNorm软件分析显示其稳定性M值相近。NormFinder与geNorm分析结果相同,这3个内参基因稳定性最佳,排在前3位,TBP的稳定性值最高,PPIA、YWHAZ的稳定性值相近,仅次于TBP。按照同样的方法,肝脏、肾脏、肠组织和肌肉中最稳定的内参基因均为TBP和PPIA,脾脏中最稳定内参基因为TBP和RPL4,肺脏中最稳定内参基因为TBP、RPL4、PPIA和ACTB,血液中最稳定内参基因为ACTB、YWHAZ和GAPDH。有趣的是,大蒲莲猪不同日龄心脏、肝脏、脾脏、肾脏、肺脏、肠组织和肌肉中TBP均被选为最适宜的内参基因,这与参考文献[10,21- 22,24,28,41-43]的研究结果一致。因此,TBP是标准化猪肌肉、内脏、肠等不同组织RT-qPCR数据的适宜内参基因。NormFinder与geNorm分析结果均显示,大蒲莲猪血液中最适宜的内参基因为ACTB,与Nygard等[21]研究结果一致。Nygard等研究结果发现ACTB是标准化17种猪体组织中高表达目的基因RT-qPCR数据的适宜内参基因[21]。大蒲莲猪血液中稳定性最差的内参基因为RPL4,其次是B2M。但是有研究表明,B2M为猪外周血、肺脏、肾脏等组织中稳定表达的内参基因[10,43-45]。 RPL4是巴马微型猪外周血液中最稳定的内参基因之一[28],这与我们的研究结果不一致。
因此,适宜内参基因的选择需要根据品种、试验条件、组织类型等具体条件进行筛选,这样才能获得准确可靠的RT-qPCR数据。本试验结果也表明,常用的内参基因SDHA、B2M并不是最稳定的,最适用于标准化目的基因的。由此可见,在使用RT-qPCR方法对目的基因表达量进行研究时,首先需要筛选稳定性高的适宜内参基因,以获得目的基因的准确RT-qPCR数据。
本试验中geNorm、NormFinder软件联合分析结果表明,大蒲莲猪心脏中最稳定的适宜内参基因为TBP、PPIA和YWHAZ,肝脏、肾脏、肠组织和肌肉中最稳定的适宜内参基因为TBP和PPIA,脾脏中最稳定适宜内参基因为TBP和RPL4,肺脏中最稳定的适宜内参基因为TBP、RPL4、PPIA和ACTB,血液中最稳定的适宜内参基因为ACTB、YWHAZ和GAPDH。
本研究使用geNorm、NormFinder软件对大蒲莲猪不同发育阶段组织RT-qPCR分析中适宜内参基因进行分析,结果表明,大蒲莲猪心脏、脾脏、肺脏中最适宜内参基因分别为TBP、PPIA、YWHAZ,TBP、RPL4,TBP、RPL4、PPIA、ACTB。肝脏、肾脏、肠组织和肌肉中最适宜内参基因为TBP和PPIA,血液中最适宜内参基因为ACTB、YWHAZ和GAPDH。不同日龄大蒲莲猪8种体组织中适宜内参基因数量和组合顺序的不同,同时发现常用内参基因SDHA和B2M稳定性基本最低。因此,在研究目的基因表达差异时首先应进行内参基因稳定性分析,选择适宜的内参基因标准化目的基因表达,为获得准确RT-qPCR数据提供必要条件。
[1] Valasek M A,Repa J J. The power of real-time PCR[J]. Adv Physiol Educ,2005,29:151-9.
[2] Mackay I M. Real-time PCR in the microbiology laboratory[J]. Clin Microbiol Infect,2004,10:190-212.
[3] Vandesompele J,De Preter K,Pattyn F,et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology,2002,3(7):RESEARCH0034.
[4] Park S J,Huh J W,Kim Y H,et al. Selection of internal reference genes for normalization of quantitative reverse transcription polymerase chain reaction(qRT-PCR)analysis in the canine brain and other organs[J]. Molecular Biotechnology,2013,54(1):47-57.
[5] Yperman J,De Visscher G,Holvoet P,et al. Beta-actin cannot be used as a control for gene expression in ovine interstitial cells derived from heart valves[J]. J Heart Valve Dis,2004,13:848-53.
[6] Maroufi A,Van Bockstaele E,De Loose M. Validation of reference genes for gene expression analysis in chicory(Cichorium intybus)using quantitative real-time PCR[J]. BMC Molecular Biology,2010,11:15.
[7] Beekman L,Tohver T,Dardari R,et al. Evaluation of suitable reference genes for gene expression studies in bronchoalveolar lavage cells from horses with inflammatory airway disease[J]. BMC Molecular Biology,2011,12:5.
[8] Ho-Pun-Cheung A,Cellier D,Lopez-Crapez E. Considerations for normalisation of RT-qPCR in oncology [J]. Ann Biol Clin(Paris),2008,66:121-129.
[9] Huggett J,Dheda K,Bustin S,et al. Real-time RT-PCR normalisation; strategies and considerations[J]. Genes and Immunity,2005,6(4):279-284.
[10] Uddin M J,Cinar M U,Tesfaye D,et al.Age-related changes in relative expression stability of commonly used housekeeping genes in selected porcine tissues[J]. BMC Res Notes,2011,4:441.
[11] Li Q G,Tao Z,Shi L H,et al. Expression and genome polymorphism of ACSL1 gene in different pig breeds[J]. Molecular Biology Reports,2012,39(9):8787-8792.
[12] Xiao Z Z,Wang C,Mo D L,et al. Promoter CpG methylation status in porcine Lyn is associated with its expression levels[J]. Gene,2012,511(1):73-78.
[13] Bruun C S,Jensen L K,Leifsson P S,et al. Functional characterization of a porcine emphysema model[J]. Lung,2013,191(6):669-675.
[14] 谢立兰,安 康,陈 力,等. 猪DDX21基因的克隆、序列分析及原核表达[J]. 华北农学报,2017,32(3):42-47.
[15] 张 丽,刘丽霞,农伟伦,等. 甘肃黑猪H-FABP基因多态性筛查及蛋白功能预测[J]. 华北农学报,2018,33:46-52.
[16] 张秀秀,黄万龙,郭云涛,等. ssc-miR-486靶基因预测及生物信息学分析[J]. 华北农学报,2016,31(5):62-70.
[17] 黄晓宇,袁军虎,杨巧丽,等. 烟台黑猪SLA-DQA基因编码区多态性及生物信息学分析[J]. 华北农学报,2016,31(5):86-93.
[18] 刘丽霞,张 丽,赵生国,等. 八眉猪 DRA 基因编码区多态性及生物信息学分析[J]. 华北农学报,2015,30(4):66-71.
[19] 马鲜平,朱 涛,龙安格,等. 荣昌猪骨形态发生蛋白15基因克隆与表达谱分析[J]. 华北农学报,2016,31(S1):458-462.
[20] Erkens T,Van Poucke M,Vandesompele J,et al. Development of a new set of reference genes for normalization of real-time RT-PCR data of porcine backfat and longissimus dorsi muscle,and evaluation with PPARGC1A[J]. BMC Biotechnol,2006,6:41.
[21] Nygard A B,Jorgensen C B,Cirera S A. Selection of reference genes for gene expression studies in pig tissues using SYBR green qPCR[J]. BMC Molecular Biology,2007,8:67.
[22] Martino A,Cabiati M,Campan M A,et al. Selection of reference genes for normalization of real-time PCR data in minipig heart failure model and evaluation of TNF-alpha mRNA expression[J]. Journal of Biotechnology,2011,153(3/4):92-99.
[23] Kuijk E W,Du Puy L,Van Tol H T,et al. Validation of reference genes for quantitative RT-PCR studies in porcine oocytes and preimplantation embryos[J]. BMC Developmental Biology,2007,7:58.
[24] Park S J,Kwon S G,Hwang J H,et al. Selection of appropriate reference genes for RT-qPCR analysis in Berkshire,Duroc,Landrace,and Yorkshire pigs[J]. Gene,2015,558(1):152-158.
[25] Wang S Q,Li J Q,Zhang A L,et al. Selection of reference genes for studies of porcine endometrial gene expression on gestational day 12[J]. Biochemical and Biophysical Research Communications,2011,408(2):265-268.
[26] McCulloch R S,Ashwell M S,O′Nan A T,et al. Identification of stable normalization genes for quantitative real-time PCR in porcine articular cartilage[J]. Journal of Animal Science and Biotechnology,2012(4):181-187.
[27] Li Q M,Domig K J,Ettle T,et al. Evaluation of potential reference genes for relative quantification by RT-qPCR in different porcine tissues derived from feeding studies[J]. International Journal of Molecular Sciences,2011,12(3):1727-1734.
[28] Ju X H,Yong Y H,Xu H J,et al. Selection of reference genes for gene expression studies in PBMC from Bama miniature pig under heat stress[J]. Veterinary Immunology and Immunopathology,2011,144(1/2):160-166.
[29] Gu Y R,Li M Z,Zhang K,et al. Evaluation of endogenous control genes for gene expression studies across multiple tissues and in the specific sets of fat-and muscle-type samples of the pig[J]. Journal of Animal Breeding and Genetics,2011,128(4):319-325.
[30] 台玉磊,韩立强,杨国庆,等. 仔猪组织基因表达中实时定量PCR内参基因的选择[J]. 农业生物技术学报,2010,18(04Roll/卷:18):732-736.
[31] Andersen C L,Jensen J L,Orntoft T F. Normalization of real-time quantitative reverse transcription-PCR data:A model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J]. Cancer Research,2004,64(15):5245-5250.
[32] Cinar M U,Islam M A,Uddin M J,et al. Evaluation of suitable reference genes for gene expression studies in porcine alveolar macrophages in response to LPS and LTA[J]. BMC Res Notes,2012,5:107.
[33] Bustin S A,Benes V,Garson J A,et al. The MIQE guidelines:minimum information for publication of quantitative Real-Time PCR experiments[J]. Clinical Chemistry,2009,55(4):611-622.
[34] Davoli R,Gandolfi G,Braglia S,et al. New SNP of the porcine Perilipin 2(PLIN2)gene,association with carcass traits and expression analysis in skeletal muscle[J]. Molecular Biology Reports,2011,38(3):1575-1583.
[35] Huang J,Ma G J,Zhu M J,et al. Molecular characterization of the porcine STAT4 and STAT6 genes[J]. Molecular Biology Reports,2012,39(6):6959-6965.
[36] Larsen K,Madsen L B,Bendixen C. Porcine dorfin:molecular cloning of the RNF19 gene,sequence comparison,mapping and expression analysis[J]. Molecular Biology Reports,2012,39(12):10053-10062.
[37] 罗宗刚,李 祥,李明洲,等. 脂联素(AdipoQ)及其受体基因(AdipoR1 and AdipoR2)在猪背最长肌和腰大肌中的表达差异[J]. 农业生物技术学报,2011,19(3):507-512.
[38] 宋子仪,史新娥,杨 浩,等. 基于一种新的天花板培养方法分析猪成熟脂肪细胞去分化过程中关键基因表达模式[J]. 农业生物技术学报,2013,21(4):379-387.
[39] 唐中林,李 勇,邓 宏,等. IGF2荧光定量PCR方法建立及其在中外猪胚胎骨骼肌中的表达[J]. 农业生物技术学报,2008,16(2):202-207.
[40] Madsen L B,Thomsen B,Larsen K,et al. Molecular characterization and temporal expression profiling of presenilins in the developing porcine brain[J]. BMC Neuroscience,2007,8:72.
[41] Mcbryan J,Hamill R M,Davey G,et al. Identification of suitable reference genes for gene expression analysis of pork meat quality and analysis of candidate genes associated with the trait drip loss[J]. Meat Science,2010,86(2):436-439.
[42] 彭馥芝,冉茂良,翁 波,等. 猪睾丸组织定量PCR分析中内参基因的选择[J]. 中国农业科学,2017(15):3033-3041.
[43] 齐 波,朱荣生,王怀中,等. 测定猪不同组织基因表达时RT-PCR内参基因的选择[J]. 家畜生态学报,2017(6):8-12.
[44] Wang J Y,Wang Y P,Wang H Z,et al. Selection of reference genes for gene expression studies in porcine whole blood and peripheral blood mononuclear cells under polyinosinic:polycytidylic acid stimulation[J]. Asian-Australasian Journal of Animal Sciences,2014,27(4):471-478.
[45] 王继英,王彦平,郭建凤,等. 仔猪外周血中内参基因的筛选及细胞因子和受体的表达水平[J]. 中国农业科学,2015(7):1437-1444.