机体可通过调节蛋白的合成来调节基因的快速、可逆及空间表达。翻译起始既是蛋白合成的第一步,也是限制蛋白质合成的关键步骤[1]。真核翻译起始因子2(Eukaryotic translation initiation factor 2,eIF2)可与GTP、tRNAiMet相互结合,形成三元复合物,随后该三元复合物与核糖体40S结合形成翻译起始前复合物,形成的翻译起始前复合物能够与mRNA的5′末端结合并且搜寻mRNA的翻译起始位点(通常为AUG密码子)[2],继而启动后续的翻译过程。
eIF2由3个序列高度保守的亚基(α、β、γ亚基)组成。α亚基是eIF2的调节亚基,eIF2上游的各种磷酸化激酶通过这一亚基上的第51位丝氨酸磷酸化位点调节eIF2蛋白的活性[3]。β亚基的主要功能是与eIF2下游的蛋白eIF5、eIF2B以及RNA结合,启动蛋白合成[4-5]。γ亚基是eIF2 3个亚基中最大的一个[6],同时也是eIF2的核心亚基[2],具有结合GTP以及tRNA的能力,还包含能够结合Zn2+的锌指结合基序[7-8]。
eIF2蛋白γ亚基由EIF2S3基因编码,由472个氨基酸组成,蛋白分子量约51 ku。研究发现,EIF2S3基因变异及表达量变化与多种疾病相关。如EIF2S3的第465位亮氨酸向丝氨酸的移码突变以及108位丝氨酸向精氨酸的错义突变会引起临床表现为精神发育迟滞、癫痫、性腺机能减退/先天性不育、小头和肥胖的MEHMO症[9-10];EIF2S3基因可能通过NDRG1基因间接抑制前列腺癌的转移[11];EIF2S3基因表达下调与结直肠癌[12-13]以及非小细胞肺癌[14]显著相关。此外,EIF2S3可能也通过影响eIF2的整体功能影响了动物体的其他性状,如Li等[15]研究发现,EIF2S3是与鸡胸肌初始pH值相关的一个候选基因。由此可见,EIF2S3在生物体内发挥着多种重要功能,如免疫以及肌肉品质。但是,目前有关EIF2S3基因的转录调控机制仍然还不清楚。本研究旨在初步分析猪EIF2S3基因的启动子活性以及转录调控元件,以期为进一步研究猪EIF2S3基因的表达机制奠定基础。
1 材料和方法
1.1 试验材料
试验用新生荣昌猪耳组织样由重庆市畜牧科学院安富猪场提供;DNA提取试剂盒购自QIAGEN公司;Q5超保真DNA聚合酶、Xho Ⅰ和Hind Ⅲ限制性内切酶购自NEB公司;pGL4.17骨架载体、内参载体pRL-TK、蛋白酶K和双荧光素酶报告基因系统试剂盒购自Promega公司;人293贴壁细胞购自重庆源于启生物技术有限公司;胎牛血清和DMEM培养基购自Gbico公司;脂质体LipofectamineTM 2000转染试剂购自Invitrogen公司。
1.2 EIF2S3转录调控区生物信息学分析
利用软件Neural Network Promoter Prediction(http://www.fruitfly.org/seq_tools/promoter.html)预测EIF2S3转录调控区核心启动子,MethPrimer软件预测CpG岛,软件JASPAR(http://jaspar.genereg.net/)预测转录因子结合位点。
1.3 EIF2S3候选启动子区及各5′端缺失片段的扩增
由于基因的启动子区域通常都在转录起始位点上游1 000~2 000 bp,因此,本研究选取转录起始位点上游约1.5 kb为候选启动子区。同时,由于有研究发现,如果克隆片段中只含有转录起始位点上游的区域可能会导致假阴性的试验结果,而若是克隆片段中包含转录起始位点、ATG或部分CDS区序列则会大大提高阳性率[16],因此,本研究克隆片段均包含转录起始位点下游200 bp的序列。
荣昌猪DNA的提取参照试剂盒说明书。浓度及OD值用Nanodrop 2000测定。随后参考NCBI数据库中的猪EIF2S3基因组序列,利用Primer 3.0软件设计EIF2S3基因候选启动子区及各5′端缺失片段扩增引物,同时利用DNAman软件分析候选启动子序列的酶切位点以筛选最适的多克隆位点,引物信息详见表1。以荣昌猪DNA为模板,采用PCR的方法,成功获得了转录起始位点上游1 563 bp的EIF2S3候选启动子区域。PCR扩增的50反应体系如下:5×Q5 Reaction Buffer 10 μL,2 mmol/L dNTP 5 μL,5×Q5 High GC Enhancer 10 μL,正向、反向引物各2.5 μL,Q5 High-fidelity DNA Polymerase 0.5 μL,ddH2O 19 μL,模板0.5 μL。反应程序为:98 ℃,30 s;98 ℃,10 s,60 ℃,30 s,72 ℃,1 min 30 s(具体视产物长度而定,通常1 kb长度延伸1 min),35 个循环;72 ℃,2 min。
1.4 EIF2S3基因候选启动子区各5′端缺失重组载体的构建
以先前扩增得到的1 563 bp片段为模板扩增另外5个5′端缺失片段,其大小分别为: 1 088,706,406,253,86 bp(由于上下游引物均带有酶切位点及保护碱基,并且扩增范围延长至下游200 bp,因此,实际产物分别为1 782,1 307,925,625,472,305 bp)。PCR产物进行纯化后用XhoⅠ和Hind Ⅲ进行双酶切,2%琼脂糖凝胶电泳后回收目的片段。最后,将所有片段与pGL4.17骨架载体进行连接,连接产物转化大肠杆菌后菌液PCR鉴定阳性克隆,并利用Xho Ⅰ和Hind Ⅲ双酶切以及测序的方法来验证重组载体构建成功与否。
表1 EIF2S3重组载体构建引物
Tab.1 Primers for the reporter construction of EIF2S3
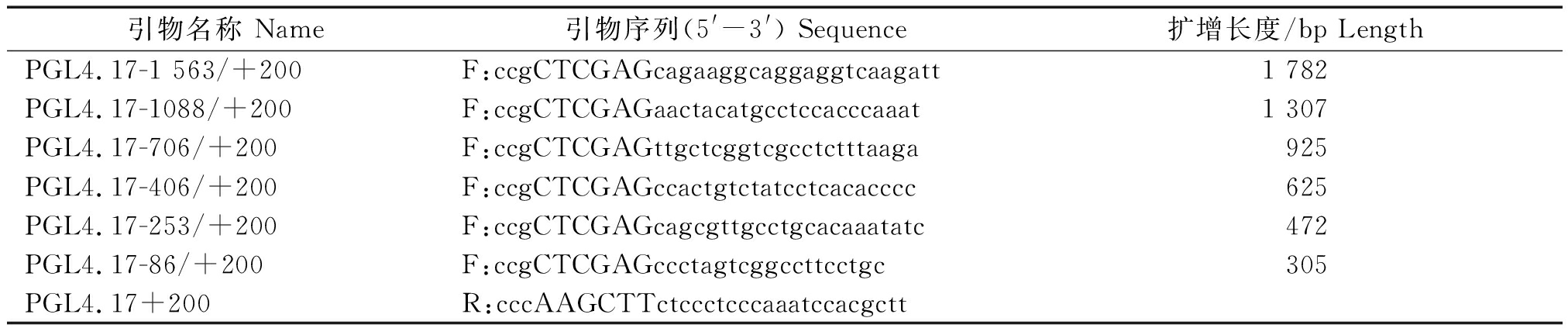
引物名称 Name引物序列(5'-3') Sequence扩增长度/bp LengthPGL4.17-1 563/+200F:ccgCTCGAGcagaaggcaggaggtcaagatt1 782PGL4.17-1088/+200F:ccgCTCGAGaactacatgcctccacccaaat1 307PGL4.17-706/+200F:ccgCTCGAGttgctcggtcgcctctttaaga925PGL4.17-406/+200F:ccgCTCGAGccactgtctatcctcacacccc625PGL4.17-253/+200F:ccgCTCGAGcagcgttgcctgcacaaatatc472PGL4.17-86/+200F:ccgCTCGAGccctagtcggccttcctgc305PGL4.17+200R:cccAAGCTTctccctcccaaatccacgctt
注:大写字母代表酶切位点,大写字母左侧小写字母为酶切位点保护碱基。
Note:Capital letters represent cleavage sites and lower case letters on the left side of capital letters are restriction sites.
1.5 人293细胞培养及细胞转染
将人293细胞按2.5×105个的密度接种至24孔板中,于含10%的胎牛血清的DMEM培养基、含5% CO2 的37 ℃培养箱中培养,待细胞密度达到90%时,采用LipofectamineTM 2000转染试剂(详细步骤参照试剂使用说明书)将构建好的重组载体和阴性对照pGL4.17分别与内参载体pRL-TK共转染到人293细胞内,48 h后收集细胞。每个重组载体设3个生物学重复。
1.6 EIF2S3转录调控区活性分析
利用Promega公司的双荧光素酶活性检测试剂盒及Thermo公司的荧光和化学发光检测仪(Thermo Scientific FLuoroskan Ascent FL)来测量分析,具体操作步骤见试剂盒说明书。以每个孔的海肾荧光素酶活性(pRL-TK)为内参校正萤火虫荧光素酶活性(重组载体)得到每个重组载体的转录活性值,再除以阴性对照pGL4.17的活性值获得每个重组载体的相对转录活性值。
1.7 统计分析
试验数据用Excel进行分析,利用方差分析进行差异显著性分析,以P<0.05为差异显著。
2 结果与分析
2.1 EIF2S3候选启动子区生物信息学分析
本试验以荣昌猪DNA为模板,采用巢式PCR的方法,成功扩增出转录起始位点上游1 563 bp的EIF2S3的启动子区域。把1 563 bp的碱基序列输入在线软件Neural Network Promoter Prediction预测发现3个核心的启动子,均位于转录起始位点上游651~794 bp(表2)。MethPrimer软件预测CpG岛结果揭示,从转录起始位点至转录起始位点上游1 563 bp这段序列中,存在2个CpG岛,分别是-596/-473(123 bp)和-392/-198(195 bp)(图1)。
表2 EIF2S3调控区域内核心启动子分布
Tab.2 The distribution of core promoters in EIF2S3 regulation area
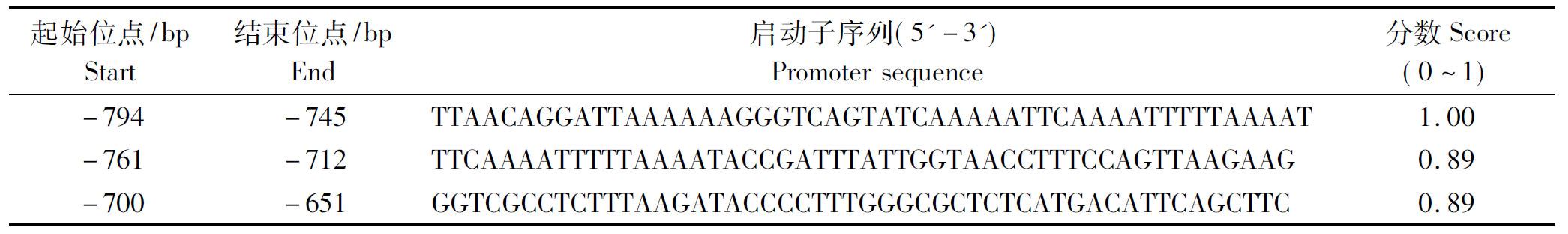
起始位点/bpStart结束位点/bpEnd启动子序列(5'-3')Promoter sequence分数Score(0~1)-794-745TTAACAGGATTAAAAAAGGGTCAGTAT-CAAAAATTCAAAATTTTTAAAAT1.00-761-712TTCAAAATTTTTAAAATACCGATTTATTG-GTAACCTTTCCAGTTAAGAAG0.89-700-651GGTCGCCTCTTTAAGATACCCCTTT-GGGCGCTCTCATGACATTCAGCTTC0.89
2.2 EIF2S3基因启动子区各片段的获取
经PCR扩增和1%琼脂糖凝胶电泳检测的EIF2S3基因启动子区各片段大小与预期的大小一致(图2)。
2.3 EIF2S3基因转录调控区各5′端缺失重组载体的构建
本研究共构建了6个不同长度的重组载体:pGL4.17-1 563、pGL4.17-1088、pGL4.17-706、pGL4.17-406、pGL4.17-253、pGL4.17-86。菌液PCR和Xho Ⅰ与Hind Ⅲ双酶切鉴定结果均表明,试验所扩增的片段长度大小与预期一致(图3)。测序和序列比对则进一步确定扩增产物为目的片段。
2.4 EIF2S3基因启动子活性分析
利用双荧光素酶报告系统分析各重组质粒的活性,结果表明,所构建的6个不同长度的重组质粒的启动子活性均显著高于pGL4.17对照载体,但是不同长度的启动子片段在人293细胞中具有不同的活性。其中,pGL4.17-1563、pGL4.17-1088、pGL4.17-706片段的启动子活性虽然差异不显著,但却是所有片段中活性最高的,其次是pGL4.17-406、pGL4.17-253、pGL4.17-86,因而可以推测,EIF2S3基因的核心启动子区域为-706~+200,且-1 563~-706不存在任何调控基因启动子活性的调控元件(图4);从图4可以发现,pGL4.17-706的荧光素酶活性显著高于pGL4.17-406,表明,-706~-406可能存在正向调控元件;pGL4.17-406荧光素酶活性显著高于pGL4.17-253表明-406~-253也可能存在正向调控元件,由于pGL4.17-706与pGL4.17-253之间的活性变化甚至高达5倍,pGL4.17-253与pGL4.17-86之间的活性变化虽然还呈现显著变化,但是在变化倍数上并不明显,因此可以推测,-706~-253的序列对于EIF2S3的正向转录调控应该最为重要;pGL4.17-253荧光素酶活性显著高于pGL4.17-86也表明,-253~-86存在正向调控元件,但两者的荧光素酶活性均已不是很高,因此可以推测,-253~-86 bp 以及-86~200 bp的序列对EIF2S3的正向调控作用并不是很关键。
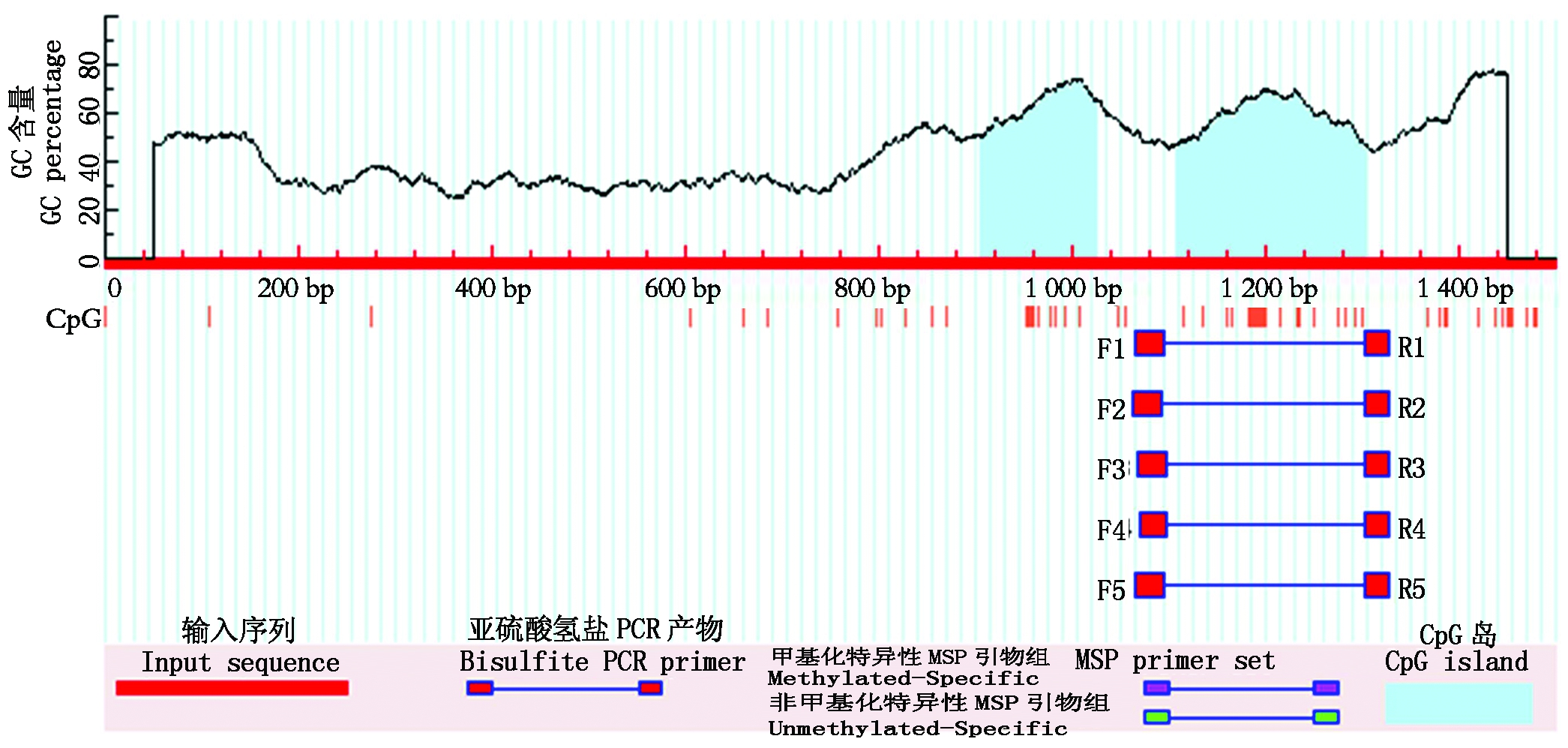
图1 EIF2S3基因启动子区CpG岛的预测
Fig.1 CpG island prediction of EIF2S3 gene promoter
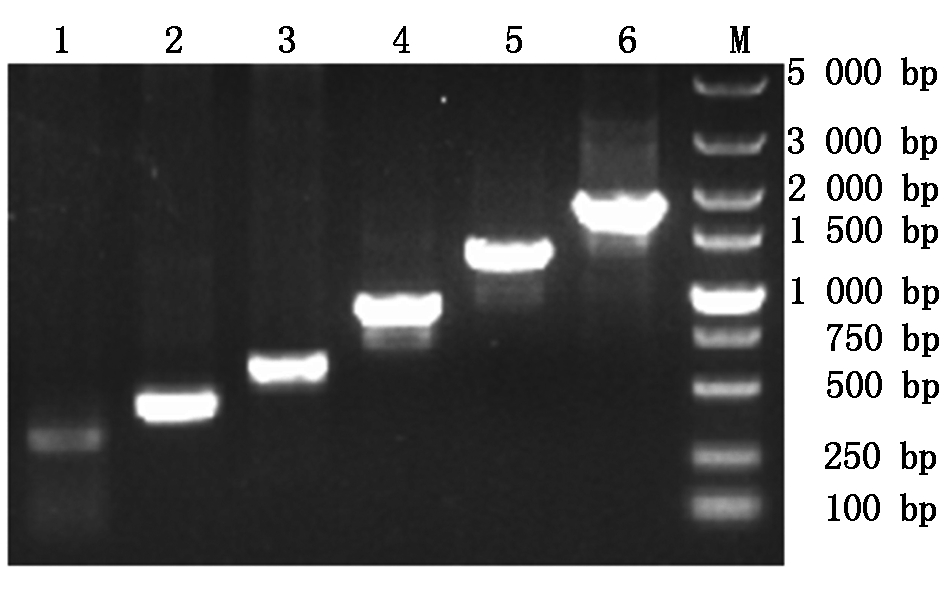
1.305 bp;2.472 bp;3.625 bp;4.925 bp;5.1 307 bp;6.1 782 bp;M.DL5000 Marker。
图2 EIF2S3基因启动子区各片段扩增的琼脂糖凝胶电泳
Fig.2 Agarose gel electrophoresis of amplified fragments of the EIF2S3 gene promoter region
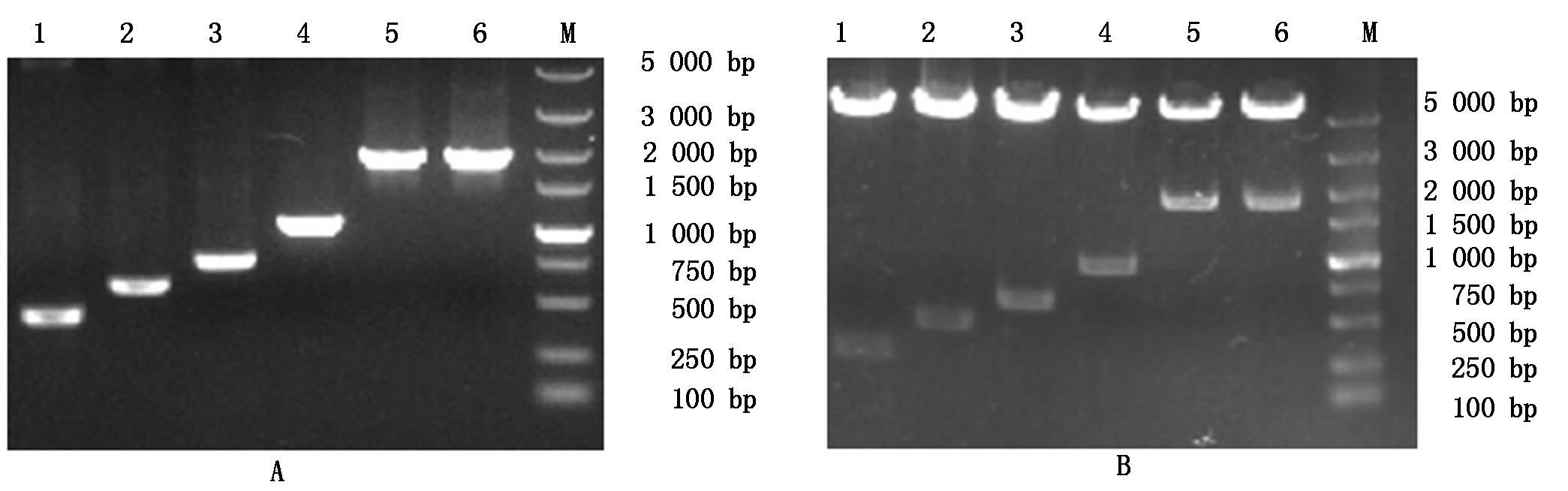
图3 重组载体PCR(A)及双酶切鉴定(B)
Fig.3 PCR (A) and double enzyme identification (B) of recombinant vector
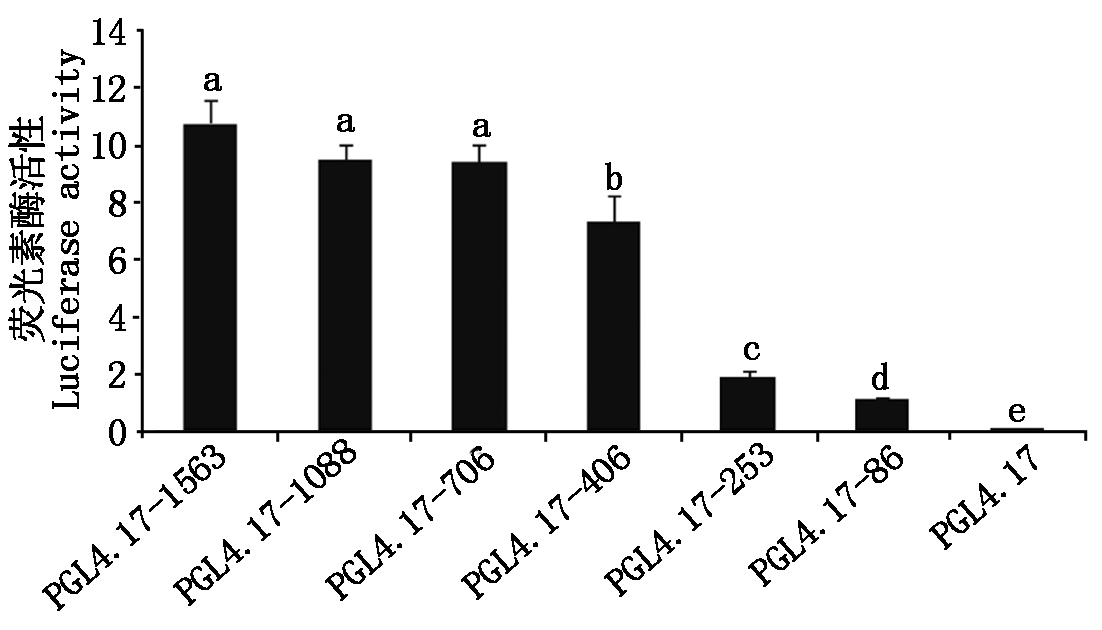
相同字母代表差异不显著(P>0.05);不同字母代表差异显著(P<0.05)。
The same letter indicates that the difference is not significantly(P>0.05),and the different letters represents the difference is significantly(P<0.05).
图4 不同长度片段荧光素酶活性分析
Fig.4 Luciferase activity analysis of different length fragments
2.5 EIF2S3基因核心启动子区转录因子的分析
鉴于-706~-253的序列对于EIF2S3的转录调控应该最为重要,本试验运用JASPAR软件对这一区域中潜在的转录因子及结合位点进行了预测,relative profile score 阈值采用默认设置,即0.8。总共预测出440个转录因子结合位点,如SP1、KLF4、MyoD1、MyoG、NFKB1等的结合位点。由于数量过多,因此,只列出阈值为0.95以上的转录因子并按分数由高到低排序,如表3。结果发现,在-706~-253共存在9个relative profile score 阈值为0.95以上的转录因子结合位点,-706 ~-406的转录因子有6个,分别为HINFP、NFKB1、Gfi1、HOXA5、NFE2L1∷MafG以及Pdx1;-406~-253的转录因子有3个,分别CEBPB、THAP1和Hltf(表3)。此外,在核心启动子区域也存在许多重要的转录因子结合位点。如SP1、KLF4、MyoD1、MyoG、Gata1、GATA2、GATA3、Gata4、NFKB1等。
表3 转录因子及其结合位点预测
Tab.3 Prediction of transcription factors and its putative binding sites
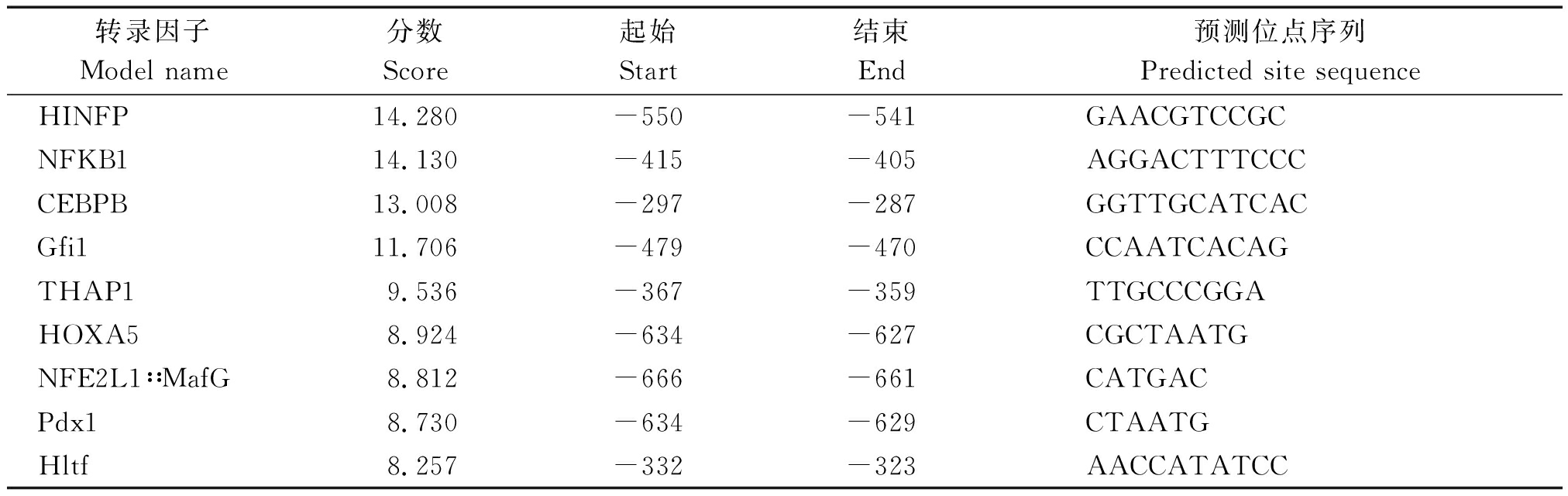
转录因子Model name分数Score起始Start结束End预测位点序列Predicted site sequenceHINFP14.280-550-541GAACGTCCGCNFKB114.130-415-405AGGACTTTCCCCEBPB13.008-297-287GGTTGCATCACGfi111.706-479-470CCAATCACAGTHAP19.536-367-359TTGCCCGGAHOXA58.924-634-627CGCTAATGNFE2L1∶∶MafG8.812-666-661CATGACPdx18.730-634-629CTAATGHltf8.257-332-323AACCATATCC
3 讨论与结论
目前,有关EIF2S3基因的转录调控机制仍然还不清楚。本研究首先利用分析软件对EIF2S3候选启动子区进行了生物信息学分析,随后以荣昌猪DNA为模板构建EIF2S3基因的启动子区各5′端缺失重组载体,同时转染人293细胞,利用双荧光素酶报告系统检测各重组质粒的荧光素酶活性,继而找出EIF2S3基因的核心启动子区,为进一步研究猪EIF2S3基因的表达机制奠定了基础。
本试验利用在线软件Neural Network Promoter Prediction对候选启动子区的1 563 bp序列进行预测发现3个核心的启动子,均位于转录起始位点上游651 ~794 bp,这符合大多数基因启动子的分布。同时,本研究利用双荧光素酶报告基因载体试验证实,EIF2S3基因的核心启动子区域为-706~+200,因此,预测结果和双荧光素酶活性试验验证结果基本一致。CpG岛是DNA甲基化的发生区域,而DNA甲基化通常可通过影响DNA的构象[17-18]、DNA与转录因子的结合[19]以及染色质的结构[20]来调节基因的表达。因此,CpG岛的预测更有利于明确启动子区域的位置。本研究利用MethPrimer软件预测CpG岛结果揭示候选启动子区中存在2个CpG岛,分别是-596/-473(123 bp)和-392/-198(195 bp),正好与本研究的双荧光素酶活性分析的核心启动子位置的结果相互佐证。
本研究共构建了6个5′端缺失重组载体,其中,双荧光素酶活性最高的片段是pGL4.17-1563、pGL4.17-1088、pGL4.17-706,但随着插入片段5′端缺失,EIF2S3调控区域的转录活性发生了显著变化。pGL4.17-706与pGL4.17-406、pGL4.17-406与pGL4.17-253之间的活性均出现了显著下降,说明-706~-406区域和-406~-253区域均是EIF2S3的正向调控区。且pGL4.17-706与pGL4.17-253之间的活性变化甚至高达5倍,而pGL4.17-253与pGL4.17-86之间的活性变化虽然还呈现显著变化,但是在变化倍数上并不明显,说明-706~-253区域可能就是EIF2S3基因最核心的启动子区域,这一区域中的转录调控元件对EIF2S3基因的启动子调控最为重要。
对-706~-253区域进行转录因子及结合位点的预测,总共发现了440个转录因子结合位点,许多转录因子在这一区域都有多个结合位点,如SP1、KLF4、MyoD1、MyoG、Gata1、GATA2、GATA3、Gata4、NFKB1等。KLF4是一种可在多种组织中广泛表达的转录因子,参与调控了细胞的增殖、分化、胚胎发育等重要生命过程[21]。同时,KLF4也是一个具有双重功能转录因子,根据不同的靶基因,通过不同的机制,既可以发挥激活又可以发挥抑制转录的作用[22]。转录因子Sp1是一种反式激活子,可以通过增强转录调控区域GC盒的亲和力而参与细胞增殖、分化及凋亡过程[23],其结合位点在转录激活的结构域中分布非常广泛[24-25],本研究在EIF2S3基因核心启动子区域发现多个Sp1的结合位点,这正好与前人的研究结果一致。MyoD1是一种生肌决定因子,主要通过诱导细胞周期阻滞来调节肌细胞的分化,在整个分化过程中主要作为肌细胞特异性基因转录的转录活化因子[26]。MyoG是一种成肌分化因子,主要作为肌肉特异性靶基因的转录活化子在肌肉分化、细胞周期退出以及肌肉萎缩中发挥作用[27-28]。Li 等[15]研究发现,EIF2S3是鸡胸肌初始pH值的一个候选基因,本研究在EIF2S3基因核心启动子区域发现MyoD和MyoG的结合位点,这更有利于理解EIF2S3在胸肌初始pH中的功能。NFκB是一种几乎存在于所有细胞类型中的多效转录因子,主要参与炎症、免疫、细胞生长和分化、肿瘤发生以及细胞凋亡等生物过程。其与启动子区域结合的主要结构域则是NFKB1,鉴于之前许多研究均报道EIF2S3基因与癌症相关[11-14],并且EIF2S3基因关键转录调控区域正好包含多个NFKB1结合位点,可以推测,EIF2S3基因在癌症中的表达变化可能是受转录因子NFκB调控。Gata1、GATA2、GATA3、Gata4均属于锌指转录因子家族,除Gata1既具有转录激活又具有转录抑制功能外,其余3个转录因子均具有转录激活功能[29-30]。在9个阈值为0.95以上的转录因子中,既有转录活化子,如HINFP、Pdx1和CEBPB,也有转录抑制子,如Gfi1,还有同时具有激活和抑制2种功能的转录因子,如NFkB1和Hltf。综上,可以发现,在EIF2S3基因的核心转录区域中包含多种转录因子结合位点,但转录激活子明显多于转录抑制子以及兼具激活和抑制2种功能的转录因子,这些转录因子通过激活和抑制协同作用,共同调节了EIF2S3基因的转录。
本试验利用生物信息学分析、PCR扩增、基因克隆、细胞转染、双荧光素酶活性分析等方法,获得了EIF2S3基因启动子区域的序列特征,确定了-706~+200 bp为EIF2S3基因的核心启动子区域、-706~-253 bp 为EIF2S3基因的核心启动子区域以及关键的调控区域,并且预测了关键调控区域的转录因子及其结合位点。研究结果为进一步研究猪EIF2S3基因的表达机制奠定了基础。
[1] Jackson R J,Hellen C U,Pestova T V.The mechanism of eukaryotic translation initiation and principles of its regulation[J].Nature Reviews Molecular Cell Biology,2010,11(2):113-127.
[2] Yatime L,Mechulam Y,Blanquet S,et al.Structure of an archaeal heterotrimeric initiation factor 2 reveals a nucleotide state between the GTP and the GDP states[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(47):18445-18450.
[3] Samuel C E.Mechanism of interferon action:phosphorylation of protein synthesis initiation factor eIF-2 in interferon-treated human cells by a ribosome-associated kinase processing site specificity similar to hemin-regulated rabbit reticulocyte kinase[J].Proceedings of the National Academy of Sciences of the United States of America,1979,76(2):600-604.
[4] Hershey J W.Protein phosphorylation controls translation rates[J].The Journal of Biological Chemistry,1989,264(35):20823-20826.
[5] Kimball S R,Jefferson L S.Amino acids as regulators of gene expression[J].Nutrition & Metabolism,2004,1(1):3.
[6] Gaspar N J,Kinzy T G,Scherer B J,et al.Translation initiation factor eIF-2.cloning and expression of the human cDNA encoding the gamma-subunit[J].The Journal of Biological Chemistry,1994,269(5):3415-3422.
[7] Roll-Mecak A,Alone P,Cao C,et al.X-ray structure of translation initiation factor eIF2 gamma:implications for tRNA and eIF2 alpha binding[J].The Journal of Biological Chemistry,2004,279(11):10634-10642.
[8] Ito T,Marintchev A,Wagner G.Solution structure of human initiation factor eIF2 alpha reveals homology to the elongation factor eEF1B[J].Structure,2004,12(9):1693-1704.
[9] Moortgat S,Désir J,Benoit V,et al.Two novel EIF2S3 mutations associated with syndromic intellectual disability with severe microcephaly,growth retardation,and epilepsy[J].American Journal of Medical Genetics Part a,2016,170(11):2927-2933.
[10] Skopkova M,Hennig F,Shin B S,et al.EIF2S3 mutations associated with severe X-Linked intellectual disability syndrome MEHMO[J].Human Mutation,2017,38(4):409-425.
[11] Tu L C,Yan X,Hood L,et al.Proteomics analysis of the interactome of N-mycdownstream regulated gene 1 and its interactions with the androgen response program in prostatecancer cells[J].Molecular & Cellular Proteomics,2007,6(4):575-588.
[12] Quyun C,Ye Z,Lin S C,et al.Recent patents and advances in genomic biomarker discovery for colorectal cancers[J].Recent Patents on DNA & Gene Sequences,2010,4(2):86-93.
[13] Chang Y T,Huang C S,Yao C T,et al.Gene expression profile of peripheral blood in colorectal cancer[J].World Journal of Gastroenterology,2014,20(39):14463-14471.
[14] Chian C F,Hwang Y T,Terng H J,et al.Panels of tumor-derived RNA markers in peripheral blood of patients with non-small cell lung cancer:their dependence on age,gender and clinical stages[J].Oncotarget,2016,7(31):50582-50595.
[15] Li X,Liu X,Nadaf J,et al.Using targeted resequencing for identification of candidate genes and SNPs for a QTL affecting the pH value of chicken meat[J].G3 (Bethesda,Md.),2015,5(10):2085-2089.
[16] 侯德富,关勇军,关 瑞,等.人NPCEDRG基因启动子的克隆及CCAAT/NFY结合位点初步分析[J].生物化学与生物物理进展,2011,38(8):713-723.
[17] Fang J,Cheng J,Wang J,et al.Hemi-methylated DNA opens a closed conformation of UHRF1 to facilitate its histone recognition[J].Nature Communications,2016,7(7):11197.
[18] Choy J S,Wei S,Lee J Y,et al.DNA methylation increases nucleosome compaction and rigidity[J].Journal of the American Chemical Society,2010,132(6):1782-1783.
[19] Banovich N E,Lan X,Mcvicker G,et al.Methylation QTLs are associated with coordinated changes in transcription factor binding,histone modifications,and gene expression levels[J].PLOS Genetics,2014,10(9):e1004663.
[20] Wachter E,Quante T,Merusi C,et al.Synthetic CpG islands reveal DNA sequence determinants of chromatin structure[J].eLife,2014,3:e03397.
[21] Elemento O.KLF4:A new player in plasma cell development[J].Cell Cycle,2016,15(20):2691-2692.
[22] 吴 钊.KLF4在正常生理活动与肿瘤中的作用[J].癌变·畸变·突变,2010,22(3):246-248.
[23] Emili A,Greenblatt J,Ingles C J.Species-specific interaction of the glutamine-rich activation domains of Sp1 with the TATA box-binding protein[J].Molecular and Cellular Biology,1994,14(3):1582-1593.
[24] 胡慧艳,贾 青,侯胜奎,等.猪DKK1基因启动子区的克隆及其活性分析[J].畜牧兽医学报,2017,48(6):1150-1157.
[25] 李丽莎,彭永东,郑晓宁,等.山羊PMEL基因启动子活性及转录调控元件分析[J].畜牧兽医学报,2017,48(5):826-835.
[26] 刘丑生,赵兴波,李 宁,等.动物肌肉生长发育调控的功能基因研究进展[J].中国畜牧杂志,2003,39(5):48-49.
[27] Wright W E,Sassoon D A,Lin V K.Myogenin,a factor regulating myogenesis,has a domain homologous to MyoD[J].Cell,1989,56(4):607-617.
[28] 栾兆进,曲绪仙,贺建宁,等.家畜MyoG基因结构和功能研究进展[J].家畜生态学报,2015,36(7):1-4.
[29] Fujiwara T.GATA transcription factors:basic principles and related human disorders[J].The Tohoku Journal of Experimental Medicine,2017,242(2):83-91.
[30] Yang Y Q,Gharibeh L,Li R G,et al.GATA4 loss-of-function mutations underlie familial tetralogy of fallot[J].Human Mutation,2013,34(12):1662-1671.