粳稻栽培品种中淀粉合成相关基因的等位变异及群体结构分析
张善磊1,2,岳红亮2,赵春芳2,陈 涛2,张亚东2,周丽慧2,赵 凌2,梁文化2,王才林1,2
(1.南京农业大学 农学院,江苏省现代作物生产协同创新中心,江苏 南京 210095; 2.江苏省农业科学院 粮食作物研究所,江苏省优质水稻工程技术研究中心,国家水稻改良中心南京分中心,江苏 南京 210014)
摘要:为了明确粳稻栽培品种中淀粉合成相关基因的等位变异及群体结构和亲缘关系状况,以国内外181份粳稻栽培品种为试验材料,利用34个淀粉合成相关基因的分子标记进行遗传多样性分析,并结合185个SSR/Indel标记分析其群体结构及亲缘关系。34个淀粉合成相关基因分子标记共检测到74个等位基因,变幅为2~4个,平均每个标记检测到的等位基因数为2.176个,基因多样性指数变幅为0.011~0.494,平均0.148;多态信息含量(PIC)变幅为0.011~0.399,平均0.128,中度多态位点(0.25<PIC<0.50)有6个,无高度多态位点(PIC>0.50),表明粳稻中淀粉合成相关基因的遗传多样性不够丰富。群体结构分析及基于Nei′s遗传距离的UPGMA聚类分析表明,供试材料分为3个亚群(POP1~POP3)和1个混合群(Mixed),地理位置相近的品种大都聚于同一亚群中。可为水稻食味品质性状的遗传改良提供依据,为后续淀粉合成相关基因与淀粉品质性状的关联分析奠定基础。
关键词:粳稻;遗传多样性;群体结构;淀粉合成相关基因
水稻是我国最重要的粮食作物,其产量和品质指标直接关系到商品价值和种植推广[1-2]。丰富优异的种质资源是新品种选育和遗传改良工作的基础,而种质资源亲缘关系的远近和遗传多样性的高低则是育成品种产量、品质和抗性突破的关键[3]。近年来,随着分子生物学和基因组学研究的深入,利用分子标记从DNA水平对水稻群体进行遗传结构与遗传多样性研究较多。邓宏中等[4]发现,选育品种的等位基因数及稀有等位基因数显著低于地方品种。陈锦文等[5]揭示云南哈尼族当前栽培水稻的遗传基础较狭窄,多样性较低。遗传多样性和群体遗传结构分析,可揭示不同材料的遗传状况,规避遗传背景相似的亲本组合,降低育种工作量,拓宽育成品种间的遗传距离,同时也是重要农艺性状的优异等位基因关联挖掘工作的基础。
胚乳中淀粉的品质是稻米蒸煮食味品质的主要决定因素,其生物合成的遗传调控系统非常复杂,涉及的基因有20多个,包括ADP-葡萄糖焦磷酸化酶大小亚基的编码基因AGPlar、AGPsma、AGPis;颗粒结合淀粉合成酶的编码基因Wx、GBSSⅡ;可溶性淀粉合成酶的编码基因SSⅠ、SSⅡa、SSⅡb、SSⅡc、SSⅢa、SSⅢb、SSⅣa、SSⅣb;淀粉分支酶的编码基因SBE1、SBE3、SBE4及脱分支酶的编码基因ISA、PUL等[6-7]。已有研究表明,每个基因都存在数目不一的复等位基因变异,这些复等位基因对稻米的外观品质,尤其是蒸煮食味品质具有显著影响。王才林等[8]将水稻蜡质基因Wx的等位变异型Wx-mq在水稻育种中进行广泛应用,育成一系列食味品质优良的粳稻品种南粳46、南粳5055、南粳9108等,在江苏及周边地域进行了大面积推广。水稻特别是栽培品种中淀粉合成相关基因优异变异位点的发掘,对水稻优质育种中食味品质性状的改良具有重要意义。
本研究利用来源于18个淀粉合成相关基因的34个分子标记对国内外181份粳稻栽培品种的等位基因变异进行检测,并结合185个水稻基因组上均匀分布的SSR/Indel标记,共219个分子标记,对群体结构及亲缘关系进行了详细分析,以明确淀粉合成相关基因的遗传多样性及当前粳稻栽培品种特别是江苏省内品种的遗传结构现状,为粳稻新品种选育过程中亲本的选配、优质基因的充分利用提供理论依据。
1 材料和方法
1.1 供试材料
供试材料181份粳稻,包括:江苏地区粳稻品种76份;辽宁地区粳稻品种12份;山东地区粳稻品种10份;黑龙江地区粳稻品种9份;浙江地区粳稻品种8份;天津地区粳稻品种7份;河南、宁夏和云南地区粳稻品种各6份;吉林地区粳稻品种5份;新疆地区粳稻品种4份;贵州地区粳稻品种2份;北京、河北、陕西、上海地区粳稻品种各1份;国外粳稻品种26份,含23份日本粳稻、2份韩国粳稻和1份苏联粳稻。供试材料均种植于江苏省农业科学院粮食作物研究所试验田。2016年5月13日播种,6月13日移栽,每个品种种植4行,每行12株,株行距13.3 cm×26.7 cm,常规栽培方式管理。
1.2 DNA提取和标记检测
水稻分蘖盛期采集各品种幼嫩的新鲜叶片,采用卢扬江等[9]的CTAB法并略作修改提取基因组DNA。选取均匀分布于水稻12条染色体上的185个多态性SSR/Indel标记和34个淀粉合成相关基因分子标记(强新涛等[10])对供试材料进行遗传分析。引物由上海英潍捷基贸易有限公司合成。
1.3 PCR扩增和电泳检测
10 μL的PCR反应总体系包含:ddH2O 7.2 μL,模板DNA(20 ng/μL)1 μL,10×Buffer(25.0 mmol/L)1 μL,dNTP(2.5 mmol/L)0.2 μL,正反向引物(10 μmol/L)0.4 μL,Taq DNA聚合酶(2 U/μL)0.2 μL。PCR扩增反应条件:94 ℃预变性4 min;94 ℃变性30 s,46~58 ℃复性30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。
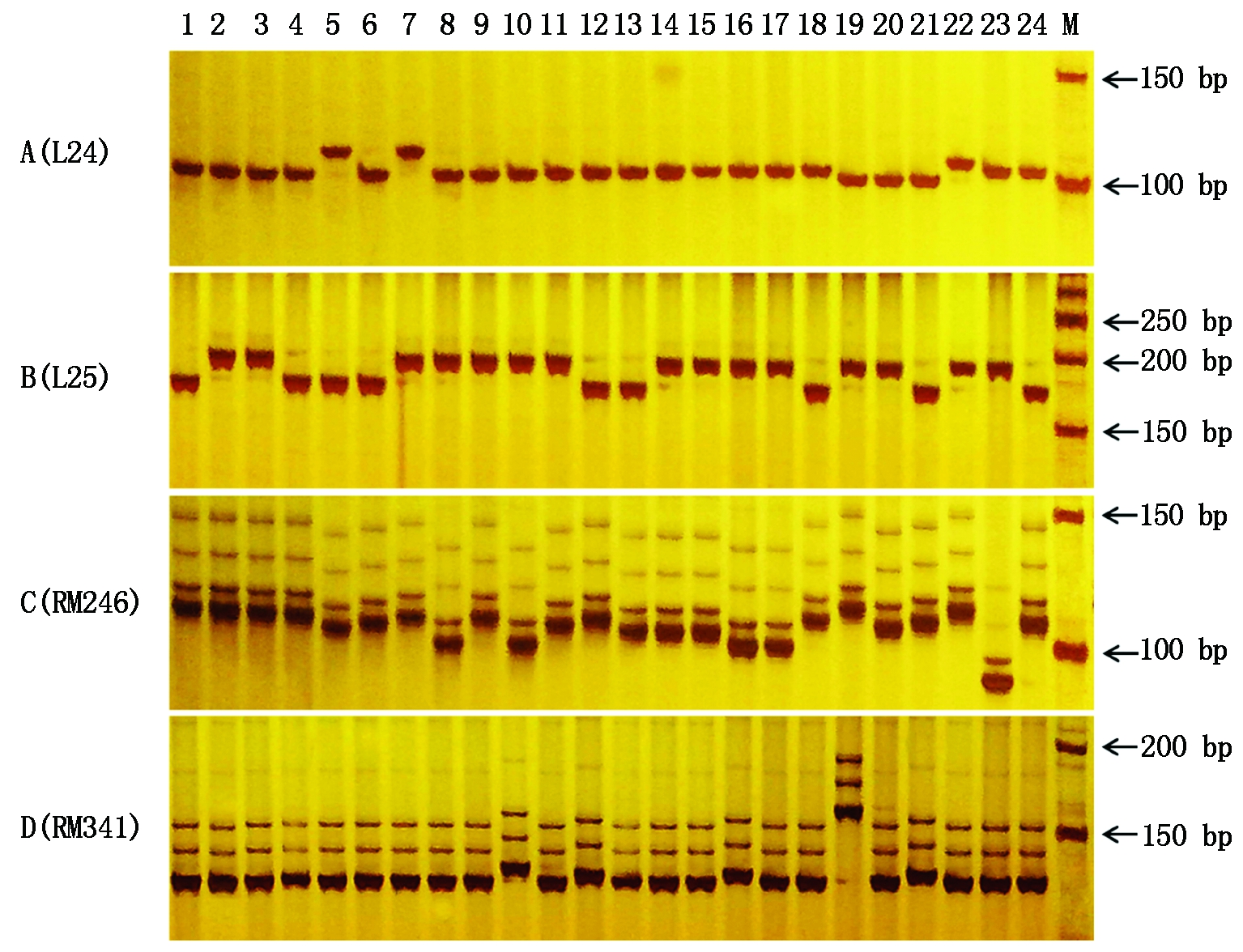
SSR/Indel引物的扩增产物在9%的聚丙烯酰胺凝胶上电泳分离,银染后在胶片观察灯上拍照、统计多态性条带;CAPS标记的扩增产物经酶切后在2%~3%浓度的琼脂糖凝胶上电泳分离,DuRed核酸燃料染色后,在紫外凝胶成像仪上观察记载。
1.4 统计分析
根据PCR扩增图谱,按多态性条带迁移位置不同次序编号(如“1/1”、“2/2”等),无条带记为“?/?”。标记统计结果使用PowerMarker v3.25软件[11]进行遗传多样性分析,分别计算等位基因数、基因多样性指数、多态信息含量(PIC)及品种间的Nei′s遗传距离,采用Nei′s遗传距离按非加权配对法(UPGMA)进行聚类分析[11-12],使用NTSYSpc version 2.1软件[13]对181份粳稻栽培品种进行基于Nei′s遗传距离的主成分分析。
使用Structure 2.3.4软件[14]对181份粳稻栽培品种进行遗传结构分析,粳稻群体亚群数K值设为1~15,将软件运行时的参数迭代(Length of burn-in period)设为50 000,蒙特卡罗迭代(Marko chain monte carlo, MCMC)设为100 000,每个K值重复运行5次,依据似然值最大的原则,分析群体最佳亚群数K值。计算所有品种在各个亚群中的Q值,以Q值大于0.5作为亚群归类标准,若某一品种在所有亚群中的Q值均小于0.5,表明该品种的遗传组分较丰富,应归类到混合亚群中[15]。
基于219个分子标记的检测结果,使用SPADiGe version 1.4c软件[16]对181份粳稻栽培品种进行亲缘关系分析。亲缘关系矩阵中的负值表明,这2份品种间的亲缘关系小于其他任意2份品种,故统计时将其值设为0[15]。
2 结果与分析
2.1 分子标记检测及淀粉合成相关基因标记的多态性分析
利用219个分子标记对181份粳稻栽培品种进行多态性检测,结果表明,所有分子标记在供试材料中均可检测到多态性(图1)。219个标记共检测到665个等位变异,平均为3.037个。基因多样性指数变幅为0.011~0.766,平均为0.264。多态信息含量(PIC)变幅为0.011~0.727,平均为0.235,中度多态位点(0.25<PIC<0.50)有73个,高度多态位点(PIC>0.50)有20个。
群体内34个淀粉合成相关基因分子标记共检测到74个等位变异,变幅为2~4个,平均每个标记检测到的等位基因数为2.176个,在Wx基因第一内含子剪切点上游的(CT)n微卫星序列的标记L05中,可检测到4个等位基因。L10、L11等29个标记均只检测到2个等位基因。基因多样性指数变幅为0.011~0.494,平均为0.148,Wx、SSSⅢ-2、PUL基因变异率较高,而SSSⅠ基因的等位变异率最低。多态信息含量(PIC)变幅为0.011~0.399,平均为0.128,中度多态位点(0.25<PIC<0.50)有6个,无高度多态位点(PIC>0.50)(表1)。
2.2 粳稻栽培品种群体遗传结构分析
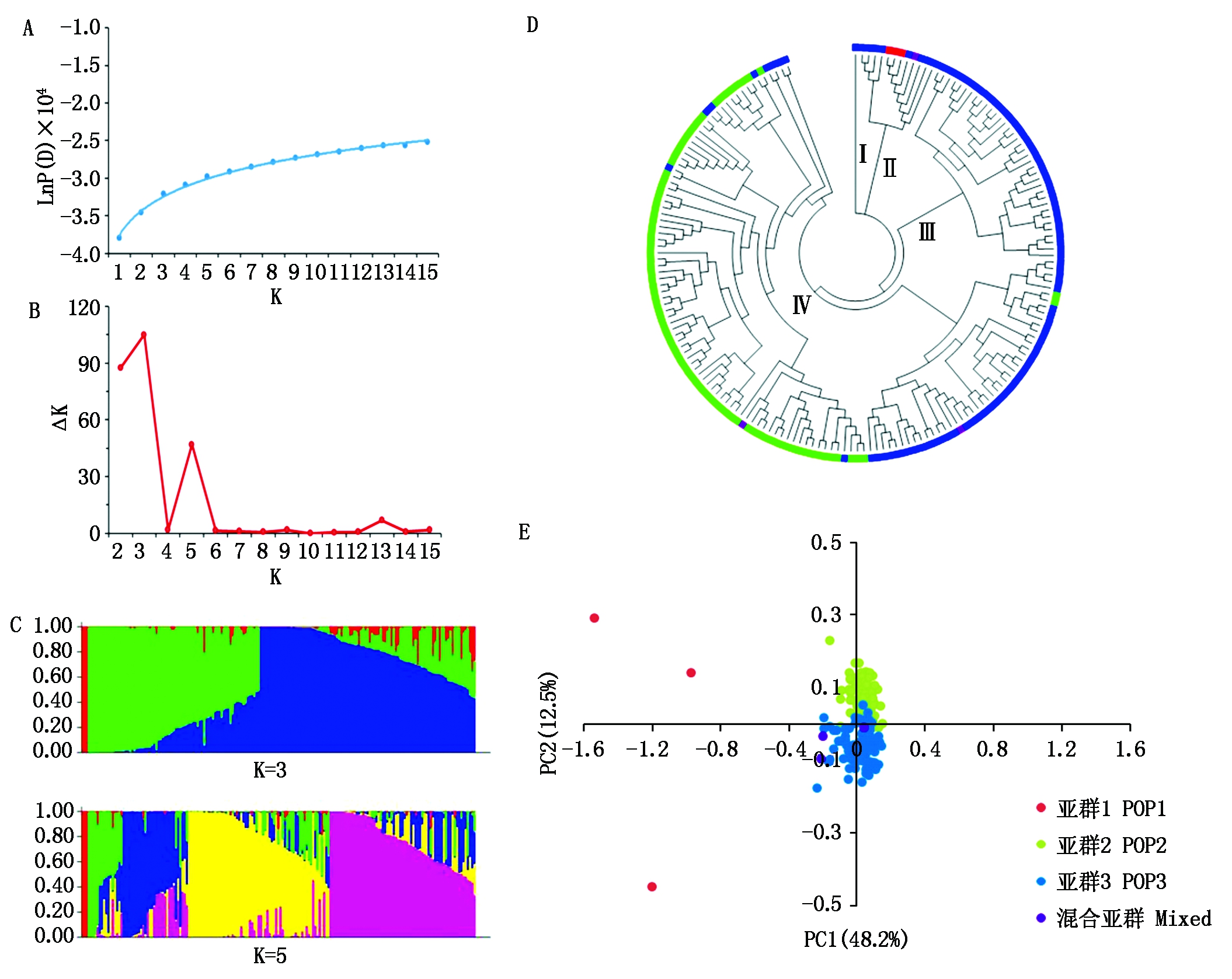
基于219个分子标记检测结果,对181份粳稻栽培品种进行群体遗传结构分析。发现LnP(D)值随着K=1~15持续增大,无明显峰值,不能确定最佳亚群数K(图2-A)。根据Evanno等[17]方法,当K=3和5时,ΔK分别取得第1峰值和第2峰值,即181份粳稻品种可分成3或5个亚群(图2-B)。根据各品种在不同亚群中的Q值,以遗传成分值大于0.5作为亚群归类标准,结果表明,98.34%的品种遗传结构比较单一,分别归类到3个相应的亚群中,1.66%的品种遗传背景比较复杂,被归类到一个混合亚群中(图2-C)。在亚群分布上,归类于第1亚群(POP1)的粳稻品种有中国91(日本)、农林229(日本)和一品(韩国),占供试材料的1.66%。归类于第2亚群的粳稻品种有79份,占供试材料的43.65%,主要来源于长江中下游地区和贵州;归类于第3亚群的粳稻品种有96份,占供试材料的53.04%,除贵州外,来源地区相对广泛。归类于混合亚群的品种有TD70(江苏)、农林24(日本)和香血糯(江苏),占供试材料的1.66%。
表1 粳稻栽培品种中淀粉合成相关基因分子标记的遗传变异
Tab.1 Genetic variation of molecular markers of starch synthesis-related genes in Japonica rice cultivars
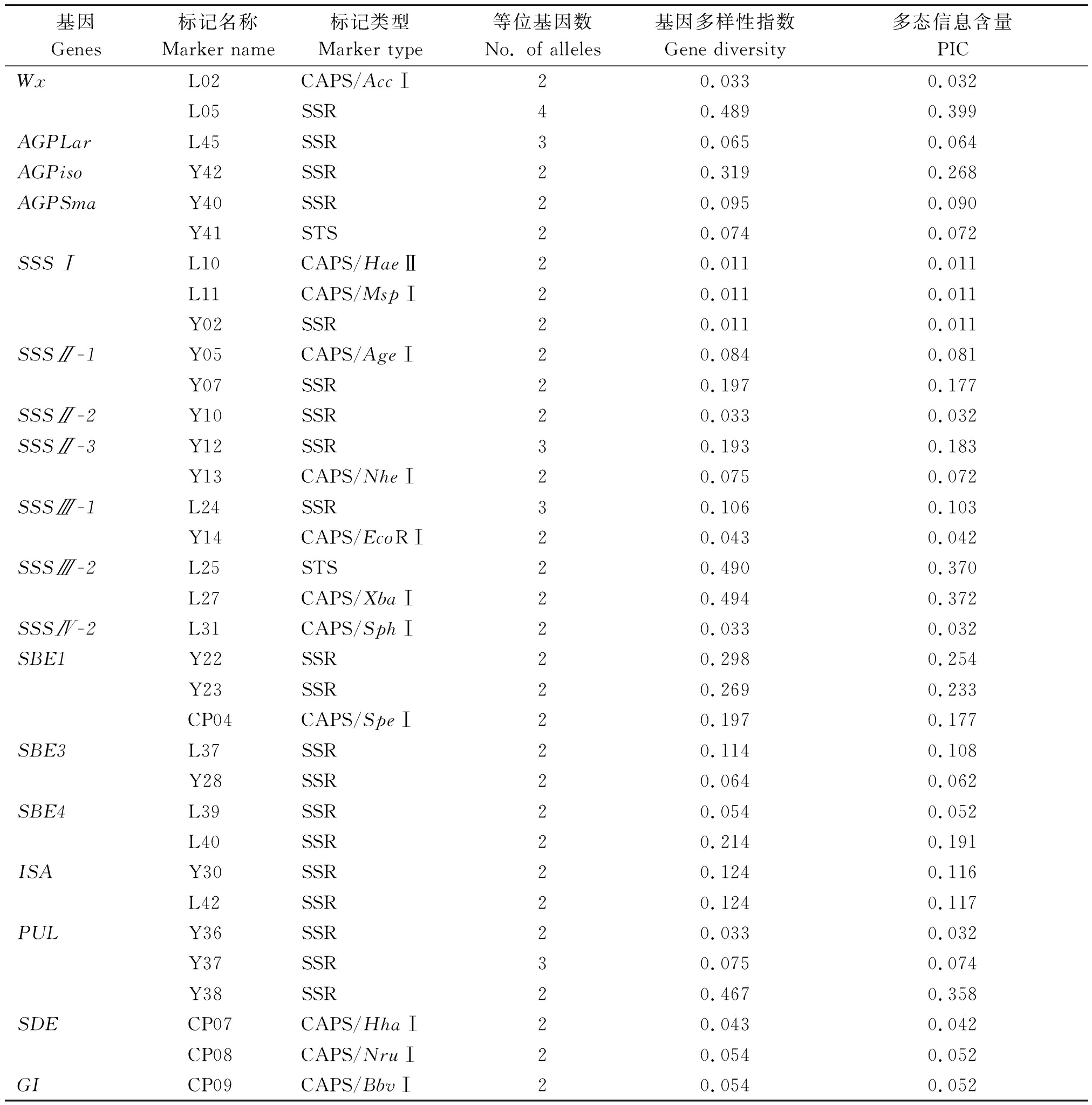
基因Genes标记名称Marker name标记类型Marker type等位基因数No. of alleles基因多样性指数Gene diversity多态信息含量PICWxL02CAPS/AccⅠ20.0330.032L05SSR40.4890.399AGPLarL45SSR30.0650.064AGPisoY42SSR20.3190.268AGPSmaY40SSR20.0950.090Y41STS20.0740.072SSSⅠL10CAPS/HaeⅡ20.0110.011L11CAPS/MspⅠ20.0110.011Y02SSR20.0110.011SSSⅡ-1Y05CAPS/AgeⅠ20.0840.081Y07SSR20.1970.177SSSⅡ-2Y10SSR20.0330.032SSSⅡ-3Y12SSR30.1930.183Y13CAPS/NheⅠ20.0750.072SSSⅢ-1L24SSR30.1060.103Y14CAPS/EcoRⅠ20.0430.042SSSⅢ-2L25STS20.4900.370L27CAPS/XbaⅠ20.4940.372SSSⅣ-2L31CAPS/SphⅠ20.0330.032SBE1Y22SSR20.2980.254Y23SSR20.2690.233 CP04CAPS/SpeⅠ20.1970.177SBE3L37SSR20.1140.108Y28SSR20.0640.062SBE4L39SSR20.0540.052L40SSR20.2140.191ISAY30SSR20.1240.116L42SSR20.1240.117PULY36SSR20.0330.032Y37SSR30.0750.074Y38SSR20.4670.358SDE CP07CAPS/HhaⅠ20.0430.042 CP08CAPS/NruⅠ20.0540.052GI CP09CAPS/BbvⅠ20.0540.052
2.3 粳稻栽培品种聚类和主成分分析
基于Nei′s遗传距离的聚类分析表明,粳稻品种可被分成离散程度较好的4大类。来自云南的楚粳27单独聚为Ⅰ类;类群Ⅱ包含12份粳稻品种,占供试材料的6.63%,其中,63.9%的品种来源于东北和日本;类群Ⅲ由龙粳24号、松辽1号、盐粳48号等36份品种组成,占供试材料的19.89%,包含22份国内品种、13份日本品种和1份韩国品种;类群Ⅳ由132份品种组成,占供试材料的72.93%,其中,长江中下游地区(江苏、浙江、山东等)粳稻品种大多聚于此类群的左侧分支中,而辽宁、天津及日本等地区的粳稻品种大多聚于此类群的右侧分支中。结合Structure分析,同一亚群的粳稻品种大多聚集在同一类群分支上,而混合亚群的粳稻品种分布较为分散(图2-D)。因此,181份粳稻品种可分成3个亚群和一个混合群,即K=3为该粳稻群体的最佳亚群数。
主成分分析表明,181份粳稻栽培品种可分成3个亚群(POP1、POP2、POP3)和一个混合群(Mixed),主成分1(PC1)和主成分2(PC2)分别解释了48.2%和12.5%的遗传变异,与群体聚类结果基本一致,进一步对群体遗传结构中亚群的划分进行了验证(图2-E)。
2.4 粳稻栽培品种亲缘关系分析
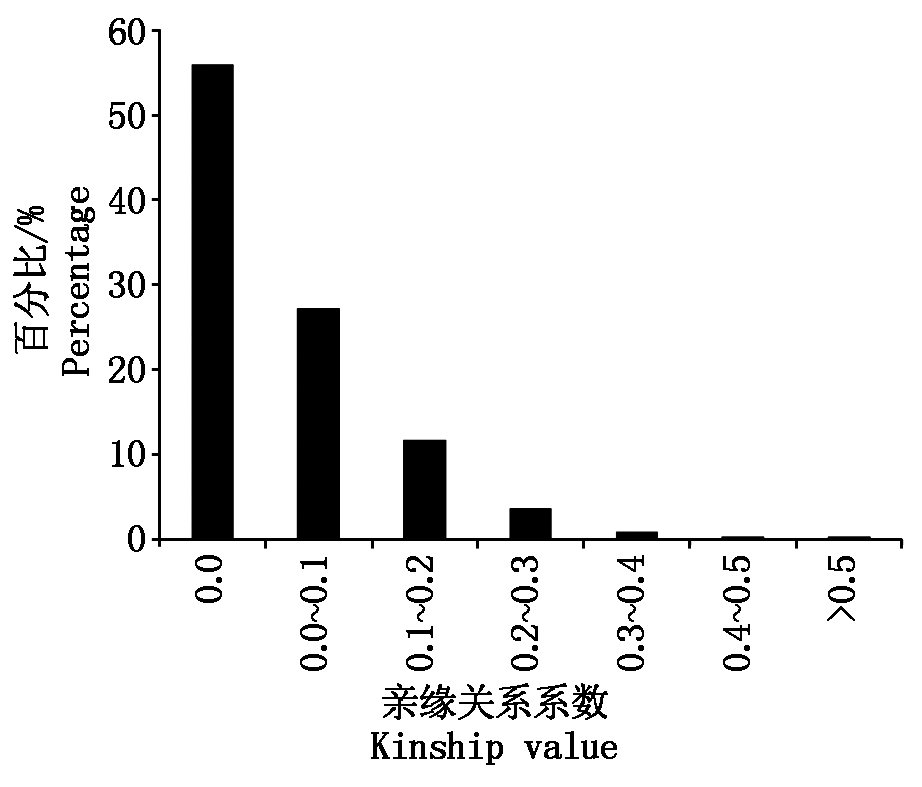
亲缘关系分析表明,品种间亲缘关系系数小于0.5的占99.7%,多数系数在0.1以下。其中,品种间亲缘关系系数为0的占56.0%;亲缘关系系数为0.0~0.1的占27.2%;亲缘关系系数为0.1~0.2的占11.7%;亲缘关系系数为0.2~0.3的占3.6%;亲缘关系系数为0.3~0.4的占0.9%;亲缘关系系数为0.4~0.5的占0.3%;亲缘关系系数大于0.5的仅占0.3%(图3)。表明供试材料来源较广,群体内个体间的亲缘关系较远。
3 讨论
不同品种间的遗传多样性差异和亲缘关系远近是评价作物种质资源状况、培育突破性品种的重要依据。前人对粳稻遗传多样性已有较多研究,张科等[18]利用62个SSR标记分析黑龙江省近年审定的73个水稻品种,遗传相似性均值为0.759。罗兵等[19]利用24个SSR标记分析太湖地区42份粳稻品种,遗传相似性系数均值为0.610。樊传章等[20]利用77个SSR标记分析云南及不同地理分布的粳稻,多态信息含量均值为0.62。本研究中,219个分子标记共检测到665个等位变异,基因多样性指数均值为0.264,多样性较低。多态信息含量均值为0.235,其中,中度以上的多态位点有93个。
34个淀粉合成相关基因的分子标记共检测到74个等位变异,基因多样性指数均值为0.148,多态信息含量均值为0.128,低于强新涛等[10]基于籼稻栽培品种的研究结果,可见粳稻栽培品种中淀粉合成相关基因的遗传多样性明显小于籼稻。Wx基因的CT重复序列可在基因表达水平上影响胚乳中直链淀粉的合成,而SSSⅡ-3和PUL基因对胚乳支链淀粉的合成及分支链延伸起重要作用,其中SSSⅡ-3是决定糊化温度的主效基因。本研究中,这3个基因位点均表现出较高的等位变异率,在Wx的(CT)n位点检测到4个等位基因,在SSSⅡ-3(Y12)和PUL(Y37)位点上均检测到3个等位基因,高于敖雁等[21]的检测结果。Wx(CT)n位点在盐糯12、南粳9108、日本晴、一品等江苏和日本粳稻品种中的变异率较其他地区高。SSSⅡ-3(Y12)位点的稀有变异主要存在于南粳46、镇稻16号、武运粳19号等江苏育成品种中。PUL(Y37)位点的稀有变异主要存在于农林229、辽粳10号、龙粳24号等具有日本粳稻血缘的品种中。后期将通过分析这些位点与供试材料中的稻米食味品质性状的关联性,从而评估这些多态位点的育种利用价值。
通过群体遗传结构、聚类和主成分分析,181份粳稻栽培品种的群体分类结果是相似的,呈现明显的地域倾向性,地理位置相近的品种大都聚于同一大类群中,地理位置相同或处在同一积温带的品种大都聚于同一类群的亚类群中,如类群Ⅱ包含的品种63.9%来源于东北和日本,类群Ⅳ包含的品种主要来源于辽宁、天津和长江中下游地区,类群内左侧亚类群的粳稻品种大多来源于长江中下游地区(江苏、浙江、山东等),而右侧亚类群以东北、天津和日本粳稻为主。较其他品种,归类于混合亚群(Mixed)的TD70(江苏)、农林24(日本)和香血糯(江苏)具有更为丰富的遗传多样性,后期可作育种亲本加以利用。
受自然环境和生产需求的影响,不同地区的育种目标有所不同,导致育成品种在农艺性状、生理生化和遗传基础上存在较大差异[22]。马作斌等[23]发现,水稻品种之间的遗传差异与纬度和地理距离有很大关系,纬度相近的国家或地区的水稻品种之间的遗传距离较近,而纬度较远的国家或地区的水稻品种之间的遗传距离较远。徐振华等[22]认为,地理位置相对较近的材料间,具有较近亲缘关系,反之,亲缘关系较远。本研究中亲缘关系系数大于0.5的仅占0.3%,其中,江苏育成品种间亲缘关系系数大于0.5的占0.6%,可见,粳稻群体内大部分品种间存在较大的遗传差异,亲缘关系较远,而少数来源于同一地区或地理分布上临近的品种,亲缘关系较近,同一育种单位育成品种间的亲缘关系系数会更高,如江苏的镇稻14号与镇稻16号,盐粳9号与淮稻5号,其亲缘关系系数值均大于0.7,这可能与骨干亲本来源有关。因此,针对省内品种遗传基础狭窄性,要强化省外、国外等地理位置较远地区粳稻品种的引进和利用,尤其注意发掘和利用淀粉合成相关基因的优异等位变异,在粳稻育种过程中,考虑育种材料目标性状的同时,应尽量拓宽亲本材料间的遗传基础,培育出遗传多样性丰富的优质高产多抗广适水稻新品种。
参考文献:
[1] 周 娟, 陈平平, 李志斌, 等. 几个杂交早稻品种的产量及光合特性比较[J]. 作物研究, 2015, 29(5): 461-471, 481.
[2] 何秀英, 廖耀平, 程永盛, 等. 水稻品质研究进展与展望[J]. 广东农业科学, 2009(1): 11-16.
[3] 李红宇, 侯昱铭, 陈英华, 等. 用SSR标记评估东北三省水稻推广品种的遗传多样性[J]. 中国水稻科学, 2009, 23(4): 383-390.
[4] 邓宏中, 王彩红, 徐 群, 等. 中国水稻地方品种与选育品种的遗传多样性比较分析[J]. 植物遗传资源学报, 2015, 16(3): 433-442.
[5] 陈锦文, 谢旺有, 王天生, 等. 基于SSR标记的水稻光温敏不育系遗传多样性分析[J]. 江西农业学报, 2017, 29(3): 1-6.
[6] Nakamura Y. Towards a better understanding of the metabolic system for amylopectin biosynthesis in plants: rice endosperm as a model tissue[J]. Plant & Cell Physiology, 2002, 43(7): 718-725.
[7] 刘燕清, 强新涛, 赵春芳, 等. 水稻淀粉合成相关基因分子标记的筛选与利用[J]. 江苏农业学报, 2015, 31(3): 471-476.
[8] 王才林, 陈 涛, 张亚东, 等. 通过分子标记辅助选择培育优良食味水稻新品种[J]. 中国水稻科学, 2009, 23(1): 25-30.
[9] 卢扬江, 郑康乐. 提取水稻DNA的一种简易方法[J]. 中国水稻科学, 1992, 6(1): 47-48.
[10] 强新涛, 赵春芳, 赵 凌, 等. 籼稻栽培品种中淀粉合成相关基因的遗传变异和群体结构分析[J]. 江苏农业学报, 2016, 32(2): 241-249.
[11] Liu K, Muse S V. PowerMarker: an integrated analysis environment for genetic marker analysis[J]. Bioinformatics, 2005, 21(9): 2128-2129.
[12] Nei M. Genetic distance between populations[J]. American Naturalist, 1972, 106(949): 283-292.
[13] Rohlf F J. Numerical taxonomy and multivariate analysis system, version 2.1[CP].New York: Exeter Software,2000.
[14] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945-959.
[15] Lu Q, Zhang M, Niu X, et al. Genetic variation and association mapping for 12 agronomic traits in indica rice[J]. BMC Genomics, 2015, 16(1): 1067.
[16] Hardy O J, Vekemans X. SPAGeDi: a versatile computer program to analyse spatial genetic structure at the individual or population levels[J]. Molecular Ecology Notes, 2002, 2(4): 618-620.
[17] Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Molecular Ecology, 2005, 14(8): 2611-2620.
[18] 张 科, 魏海锋, 卓大龙, 等. 黑龙江省近年审定水稻品种基于SSR标记的遗传多样性分析[J]. 植物遗传资源学报, 2016, 17(3): 447-454.
[19] 罗 兵, 徐港明, 孙海燕, 等. 利用简单重复序列(SSR)标记分析太湖稻区现代粳稻品种的遗传多样性[J]. 农业生物技术学报, 2014, 22(12): 1502-1513.
[20] 樊传章, 李 娟, 伍腾飞, 等. 云南及不同地理分布粳稻的SSR遗传多样性分析[J]. 分子植物育种, 2016, 14(12): 3577-3587.
[21] 敖 雁, 王 安, 崔小芬, 等. 太湖地区水稻地方品种资源品质基因及性状的遗传评价[J]. 江苏农业科学, 2016, 44(7): 98-103.
[22] 徐振华, 金正勋, 张忠臣, 等. 黑龙江省粳稻种质资源遗传结构分析[J]. 作物杂志, 2014(1): 38-44.
[23] 马作斌, 王昌华, 王 辉, 等. 不同国家水稻品种的遗传多样性分析[J]. 植物遗传资源学报, 2014, 15(3): 540-545.
Allelic Variation of Starch Synthesis Related Genes and Population Structure in Japonica Rice Cultivars
ZHANG Shanlei1,2, YUE Hongliang2, ZHAO Chunfang2, CHEN Tao2, ZHANG Yadong2, ZHOU Lihui2, ZHAO Ling2, LIANG Wenhua2, WANG Cailin1,2
(1.College of Agriculture, Nanjing Agricultural University, Jiangsu Collaborative Innovation Center for Modern Crop Production, Nanjing 210095, China;2.Institute of Food Crops, Jiangsu Academy of Agricultural Sciences, Jiangsu High Quality Rice R & D Center, Nanjing Branch of China National Center for Rice Improvement, Nanjing 210014, China)
Abstract: To evaluate the allelic variation of starch synthesis related genes inJaponica rice cultivars, their population structure and genetic relationship, 34 molecular markers of starch synthesis related genes were used to detect the genetic diversity of 181Japonica rice cultivars and their population structure and genetic relationship were analyzed combine with 185 SSR/Indel markers. A total of 74 alleles were detected by 34 molecular markers of starch synthesis related genes, ranging from 2 to 4, with an average value of 2.176 alleles per marker. Genetic diversity index ranged from 0.011 to 0.494 with an average value of 0.148. The polymorphism information content (PIC) ranged from 0.011 to 0.399 with an average value of 0.128. Six moderately polymorphic loci (0.25<PIC<0.50) and zero highly polymorphic locus (PIC>0.50) were detected, indicating that the genetic diversity of starch synthesis related genes inJaponica rice cultivars was not enough at present. Population structure analysis and UPGMA cluster analysis based on Nei′s genetic distance divided the populations into three main groups (POP1-POP3) and one mixed group (Mixed). Most of the cultivars derived from the neighboring regions were clustered into one large group. It could provide a basis for genetic improvement of eating quality traits of rice, and lay a foundation for the subsequent association analysis of starch synthesis-related genes and starch quality traits.
Key words: Japonica rice; Genetic diversity; Population structure; Starch synthesis related genes
中图分类号:S511.03;Q78
文献标识码:A
文章编号:1000-7091(2018)06-0116-07
doi:10.7668/hbnxb.2018.06.016
收稿日期:2018-07-28
基金项目:国家科技支撑计划项目(2015BAD01B02);江苏省重点研发项目(BF2016370);江苏省自主创新资金(SCX(17)1063);现代农业产业技术体系(CARS-01-62)
作者简介:张善磊(1990-),男,安徽六安人,在读硕士,主要从事水稻遗传育种研究。
通讯作者:王才林(1959-),男,江苏无锡人,研究员,博士,博士生导师,主要从事水稻遗传育种研究。