表1 两个F2群体及其亲本分蘖数分析
Tab.1 Phenotypic data of tiller number in two F2 populations
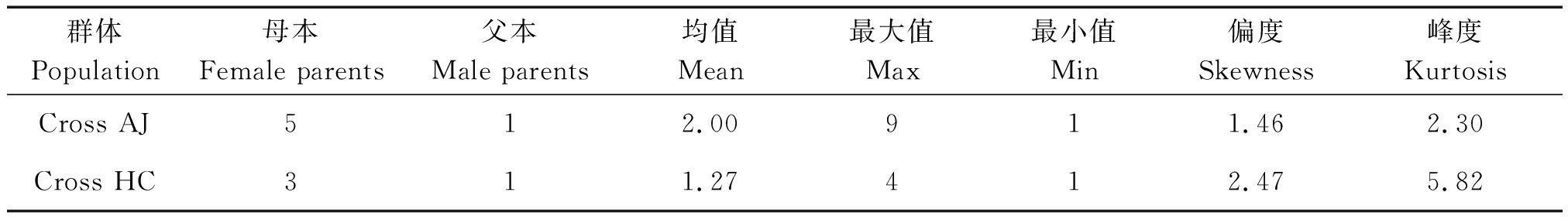
群体Population 母本Female parents父本Male parents 均值Mean 最大值Max最小值Min偏度Skewness 峰度KurtosisCross AJ512.00911.462.30 Cross HC311.27412.475.82
对于许多主要农作物(如小麦、水稻、大麦和高粱等)来说,分蘖是与产量直接相关的重要农艺性状之一[1]。一方面,分蘖会影响整个植株的株型,导致植株的光吸收能力、开花和结实率发生变化,最终使产量发生改变[2];另一方面,分蘖可以产生不定根,这使得分蘖株能够在没有主茎的情况下正常生长,从而维持一定的群体产量[1,3]。
分蘖是由主茎基部的节发生的分枝,其形成是许多因子相互作用的结果,这些因子包括环境信号、激素途径和基因调控网络[4-5]。前人已经对一些主要禾谷类作物的分蘖遗传机制进行了广泛研究。水稻的研究结果表明,光、温、水、肥等环境因素[6-7],生长素、细胞分裂素、独脚金内酯[8]等激素都会影响水稻分蘖的形成和生长发育。同时,水稻中相继克隆了11个控制分蘖的基因,包括水稻分蘖过程中的关键调控基因MOC1[9],其余几个为多蘖矮秆基因(D3、D17、D10等)[10-18],这些基因与激素共同调控分蘖形成的相关网络也得到解析[19-20]。除此之外,分蘖通常还受数量性状位点(QTL)控制。根据Gramene网站(http://archive.gramene.org/qtl)统计结果,目前水稻在不同群体中已经鉴定分蘖相关QTL共计213个,高粱中共计19个。小麦中也有一些控制分蘖主效位点的研究报道 [21-23]。近年来,谷子中分蘖相关QTL的定位研究也有相继报道。Doust等[24-25]利用F2∶3群体分别在不同温度和不同密度种植条件下共鉴定了41个谷子分蘖相关QTL;Jia等[26]利用全基因组关联方法鉴定了5个环境条件下共计57个与分蘖相关的位点;Mauro-Herrera等[3]利用重组自交系群体分别在温室和大田条件下,检测到谷子苗期、开花期和成熟期共42个控制分蘖相关QTL;Zhang等[27]利用重组自交系群体分别在长日照和短日照条件下共检测到6个控制分蘖相关QTL。尽管如此,有关环境因素和激素因子如何影响谷子分蘖的形成和生长发育,其机制还不清楚。
与其他主要农作物相比,谷子农艺性状基因或QTL发掘研究还比较落后。随着谷子参考基因组的公布[28-29],谷子中许多遗传研究相继开展,尤其是多种类型的分子标记被开发并应用于基因精细定位、基因克隆和分子标记辅助选择。Sato等[30]和Masumoto等[31]利用SSR(Simple sequence repeat)和TD(Transposon display)标记完成了谷子stb1和NEKODE1基因的精细定位;利用SNP(Single nucleotide polymorphism)、CAPS(Cleaved-amplified polymorphic sequence)和InDel(Insertion-deletion)等不同分子标记,谷子SiDWARF2、SiDWARF3、SiYGL1、SiAGO1b和Loose panicle1等基因相继精细定位和克隆[26, 32-37]。随着下一代测序技术的发展,涌现出了不少新的快速对目标基因或QTL进行定位的方法,其中包括简化基因组测序技术-RAD-seq,该技术利用限制性内切酶对基因组进行酶切,产生一定大小的片段,构建测序文库,对酶切后产生的RAD标记进行高通量测序,具有通量高、准确性高、性价比高、试验周期短和不受基因组序列限制等优点[38],已经广泛应用于SNP标记开发、高密度遗传图谱构建、植物重要性状的QTL定位等研究领域。谷子中也有利用RAD-seq技术对株高、米色等重要农艺性状进行QTL定位的研究报道[39-40]。
本研究采用简化基因组测序技术(RAD-seq),以2个谷子分蘖型农家种(母本)和2个不分蘖育成种(父本),为亲本杂交获得F1,F1各自自交后获得的2个F2群体为主要研究对象, 对谷子分蘖相关QTL进行了分析;同时,新开发了与分蘖QTL(qAJTN7-3和qHCTN7)紧密连锁的分子标记,旨在为谷子分蘖基因的克隆和解析分蘖遗传机制奠定基础。
本研究分别以2个F2分离群体为主要研究对象,其中,矮宁黄×晋谷21号杂交组合,以分蘖农家种矮宁黄为母本、不分蘖育成种晋谷21号为父本,命名为Cross AJ;红苗粘谷×长农35号杂交组合,以分蘖农家种红苗粘谷为母本、不分蘖育成种长农35号为父本,命名为Cross HC(图1)。于2014年获得2个杂交组合的F1单株,2015年将收获的F1单株及其亲本种植于山西省农业科学院谷子研究所试验田,通过2个杂交组合F1单株分别自交,获得了2个不同的F2遗传群体(Cross AJ、Cross HC)。为便于调查,亲本及群体均采用宽窄行种植模式,宽行行距0.48 m,窄行行距0.18 m,行长3 m。于谷子分蘖期调查分蘖数(含主茎),亲本取5株调查,取其平均数作为分蘖数值,F2群体逐株挂牌调查。另外,在本研究中,用于验证InDel标记和分蘖数间相关性的160份谷子种质(部分种质由中国农业科学院国家农作物种质资源库提供)的种植模式、调查方式与F2群体及其亲本相同。
1.2.1 DNA样品准备和提取 DNA取样:待幼苗长至三叶一心时,剪取适量叶片放入2 mL离心管内,然后迅速放入-80 ℃超低温冰箱保存备用。谷子基因组DNA提取采用CTAB法[41]。
1.2.2 连锁图谱构建和QTL分析 RAD-seq试验方法及步骤参照Baird等[42]描述进行,参考基因组版本Setaria_italica_v2.0(https://www.ncbi.nlm.nih.gov/genome/?term=foxtail+millet),选取限制性内切酶EcoRⅠ(Cross AJ)和TaqⅠ(Cross HC)分别酶切基因组DNA用于构建RAD-seq DNA文库,使用Illumina HiSeq X Ten平台进行测序。根据识别标签序列得到每个个体的测序reads,使用BWA(Burrows-Wheeler Aligner)[43]软件将个体reads与参考基因组进行序列比对。比对结果进行格式转换后使用GATK(Genome Analysis ToolKit)[44]软件和SAMtools[45]软件用于检测SNP和过滤SNP,SNP鉴定和基因分型方法参照Wang等[39]描述。使用MSTmap[46]软件用于分子标记排序(主要参数:作图函数为Haldane、P值为0.000 000 1、缺失阈值为0.3、目标函数为最大似然法)和连锁图谱构建(取最小阈值LOD 5.0对所有标记进行分组,每个连锁群上标记顺序通过两两标记之间的最小重组频率计算)。
利用WinQTLCart 2.5[47]软件中复合区间作图法(CIM)进行分蘖数QTL定位。进行CIM分析时,选用步长(Walking speed)1 cM和滑窗(Scanning window)10 cM,按照模型6,选取1 000次排列测验,显著水平设置为0.05,判断是否存在QTL。QTL的命名方法为:q加群体简称(AJ、HC),后面加性状简写(TN),最后加染色体名称(当同一条染色有多个QTL时加-1,-2)。
1.2.3 分子标记开发 本研究利用GATK[44]软件搜索InDel,InDel长度设定在1~10 bp。根据选定InDel位点的侧翼序列(上游250 bp,下游250 bp)设计引物进行验证,引物设计采用Primer 3.0(http://primer3.ut.ee/)在线设计,引物由天一辉远(北京)有限公司合成,本研究用到引物序列为MRI5F:5′-GTAGTCGTCCGTCCAAGC-3′,MRI5R:5′-CACCAC CATCAAACAAAGG-3′;MRI322F:5′-GGCAATCCCAA GTATGTGCA-3′,MRI322R:5′-AAGGGAGGAAGTGAA GGTGG-3′;MRI348F:5′-TCTGGTCACCTGTCGTTCTC-3′,MRI348R:5′-AAGTAAGCCACCGTAGCCTT-3′;MRI351F:5′-AAGTTGCCCTTAACCCACCT-3′,MRI351R:5′-GACATTGCCTCGCCGTAAAA-3′;MRI381F:5′-AACC TCCACCATGAAACCCT-3′,MRI381R:5′-CTGGGAGG AAAGAGGGAGTG-3′。
PCR反应体系为10 μL,包括:1 μL 10×PCR Buffer,1 μL 双向引物(2 μmol/L),0.8 μL dNTP(2.5 mmol/L),0.1 μLTaq DNA聚合酶,1 μL 模板DNA(50 ng/μL),灭菌水 6.1 μL。PCR反应程序为:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,35个循环;最后72 ℃终延伸5 min。
PCR扩增产物在8%非变性聚丙烯酰胺凝胶进行电泳,根据Marklund等[48]描述方法银染显色,拍照后统计存档。
亲本和F2群体的表型分析、分子标记分型与160份谷子种质(包括112不分蘖种质和48份分蘖种质)表型相关性分析采用统计分析软件SPSS 17.0。
矮宁黄×晋谷21号群体(Cross AJ)共获得543个F2单株,红苗粘谷×长农35号群体(Cross HC)共获得131个F2单株。亲本表型如图1所示,2个F2群体及其亲本分蘖数统计如表1所示。对于Cross AJ,亲本之间有较大差异,其F2群体变异范围为1~9;对于Cross HC,亲本差异较Cross AJ群体的亲本略小,F2群体变异范围为1~4。
表1 两个F2群体及其亲本分蘖数分析
Tab.1 Phenotypic data of tiller number in two F2 populations

群体Population 母本Female parents父本Male parents 均值Mean 最大值Max最小值Min偏度Skewness 峰度KurtosisCross AJ512.00911.462.30 Cross HC311.27412.475.82

图1 两个F2群体亲本的表型
Fig.1 The phenotype of four parents in two F2 populations
本研究利用RAD-seq技术,对Cross AJ的亲本和543个F2单株、Cross HC的亲本和131个F2单株进行测序。结果表明,Cross AJ共获得88.33 Gb的原始数据,对原始数据初步分析后,统计出数据的平均测序深度为4.55×,平均上图率为89.77%;Cross HC共获得872 Mb的原始数据,平均测序深度3.12×,平均上图率89.37%(表2)。
通过SNP鉴定后,筛选亲本间可信度高的SNP多态性标记,然后对群体个体进行基因型检测,最后利用MSTmap软件绘制遗传连锁图谱。Cross AJ共获得48 790个上图SNP标记,总图距为1 461 cM;Cross HC共获得9 968个上图SNP标记,总图距为1 648.8 cM(表2)。
表2 两个F2群体的RAD-seq统计结果
Tab.2 Summary of RAD-seq in two F2 populations
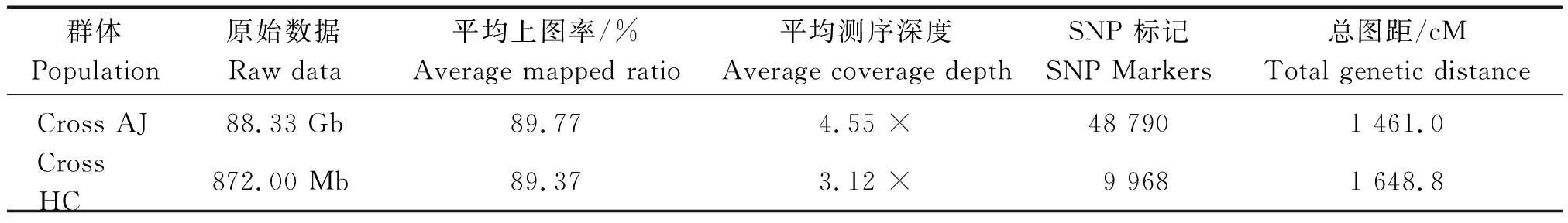
群体Population原始数据Raw data 平均上图率/%Average mapped ratio平均测序深度Average coverage depthSNP 标记SNP Markers总图距/cMTotal genetic distanceCross AJ88.33 Gb89.774.55 ×48 7901 461.0Cross HC872.00 Mb89.373.12 ×9 9681 648.8
结合2个F2群体的表型数据和SNP标记分型结果,利用WinQTLCart 2.5软件进行分蘖相关QTL定位。本研究共鉴定出8个QTL(表3)。在Cross AJ群体中,共鉴定了6个QTL,分别为qAJTN1、qAJTN5、qAJTN7-1、qAJTN7-2、qAJTN7-3和qAJTN9,解释表型变异0.7%~9.8%;在Cross HC群体中,共鉴定了2个QTL,分别为qHCTN5和qHCTN7,解释表型变异1.4%~8.3%。在这些QTL中,效应较大的有qAJTN5、qAJTN7-3和qHCTN7,分别解释表型变异的9.8%,9.3%和8.3%,其加性效应值分别为0.58,0.45和0.43,说明其增效等位基因来自母本矮宁黄或红苗粘谷。
表3 分蘖相关QTL分析
Tab.3 QTL analysis of tillering in two F2 populations of foxtail millet
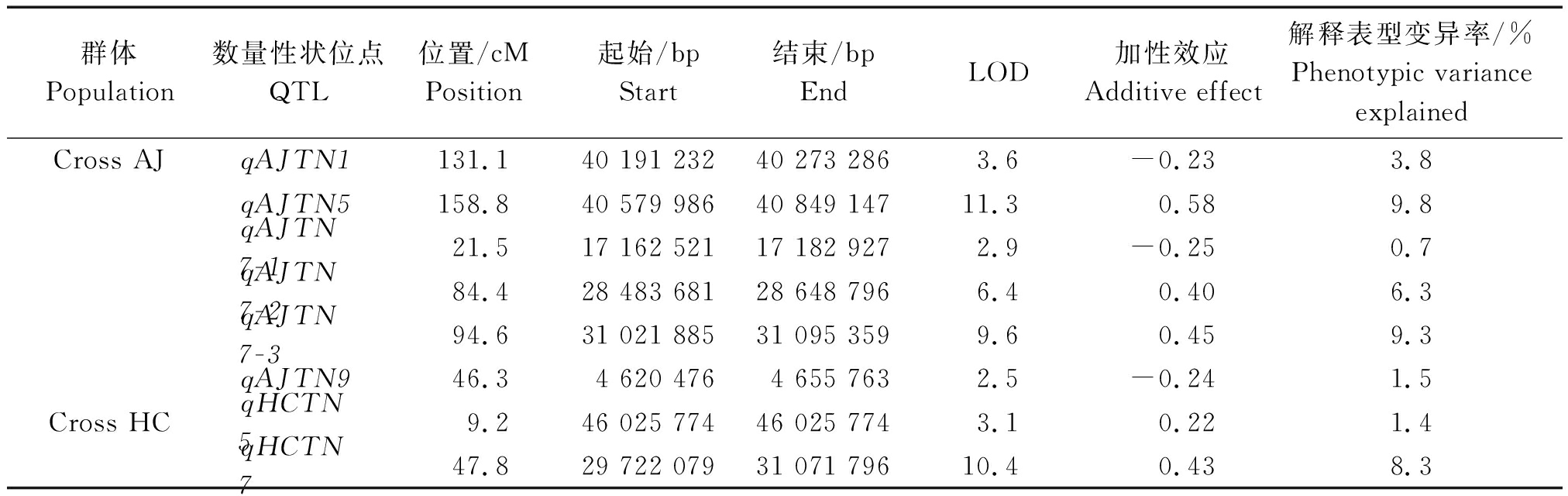
群体Population数量性状位点QTL位置/cMPosition起始/bpStart结束/bpEndLOD加性效应Additive effect解释表型变异率/%Phenotypic varianceexplainedCross AJ qAJTN1131.140 191 23240 273 2863.6-0.233.8qAJTN5158.840 579 98640 849 14711.30.589.8qAJTN7-121.517 162 52117 182 9272.9-0.250.7qAJTN7-284.428 483 68128 648 7966.40.406.3qAJTN7-394.631 021 88531 095 3599.60.459.3qAJTN946.34 620 4764 655 7632.5-0.241.5Cross HCqHCTN59.246 025 77446 025 7743.10.221.4qHCTN747.829 722 07931 071 79610.40.438.3
在2个群体中各选择了1个效应较大的QTL(qAJTN7-3和qHCTN7),并在其定位区间内开发了新的紧密连锁分子标记,以有利于分蘖性状的分子标记辅助选择。首先,将亲本基因组测序结果与豫谷1号参考基因组进行序列比对,搜索插入缺失位点,设计引物进行PCR扩增,扩增片段进行聚丙烯酰胺凝胶电泳验证,筛选在亲本之间具有多态性的标记,进一步获得在F2后代中能够良好分型的InDel标记(图2-A)。其次,利用MSTmap软件对新标记进行遗传连锁分析。连锁分析结果发现,标记MRI351和MRI322定位在qAJTN7-3区间内;MRI351、MRI5、MRI348和MRI381定位在qHCTN7区间内(图2-B)。
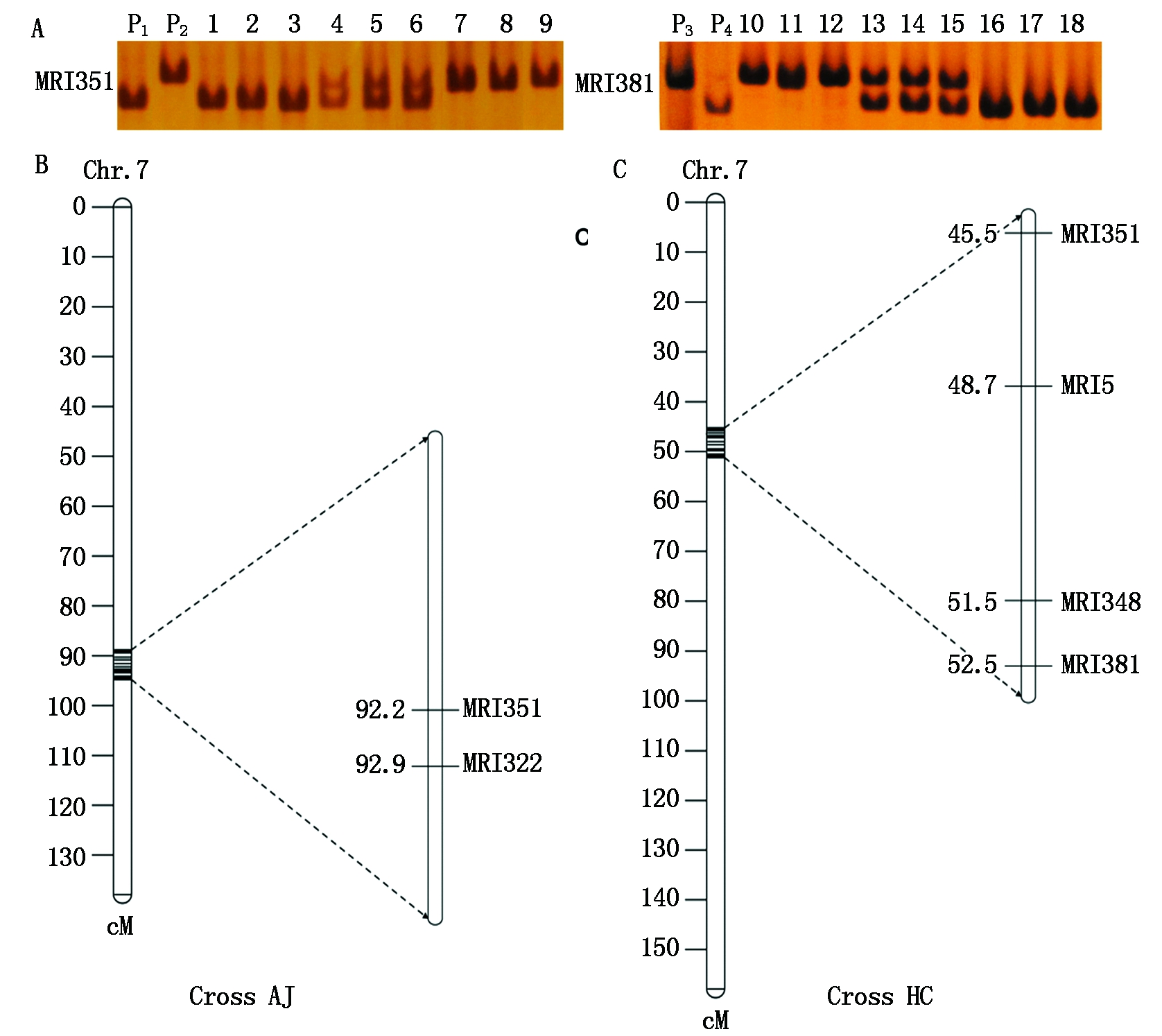
A.标记MRI351和MRI381在亲本和2个F2群体部分单株中的聚丙烯酰胺凝胶电泳结果;B、C.标记MRI5、MRI348、MRI322、MRI351和MRI381在各自染色体上的位置。P1.矮宁黄,P2.晋谷21,P3.红苗粘谷,P4.长农35;1-9为Cross AJ的部分F2单株,其中,1-3单株表型为分蘖,4-9表型为不分蘖;10-18为Cross HC的部分F2单株,其中,10-12单株表型为分蘖,13-18表型为不分蘖。
A. The PCR fragments of MRI351 and MRI381 in PAGE; B,C.The genetic positions of MRI5,MRI348,MRI322,MRI351 and MRI381 on Chr.7 of Cross AJ and Cross HC. P1. Aininghuang, P2. Jingu 21, P3. Hongmiaozhangu, P4. Changnong 35; 1-9. F2 individuals (partial) in Cross AJ, of which the phenotype of 1-3 individuals were tillering, 4-9 individuals were no-tillering; 10-18. F2individuals (partial) in Cross HC, of which the phenotype of 10-12 individuals were tillering, 13-18 individuals were no-tillering.
图2 新开发的分子标记及其在染色体上的位置
Fig.2 Newly developed markers and their genetic positions on chromosomes
为了进一步确认InDel标记和分蘖数间的相关性,选取了2个F2群体中的标记MRI351和MRI381,对160份谷子种质进行了基因分型和相关性分析(表4)。根据分子标记检测结果,将与分蘖亲本带型一致的基因型标记为“1”,与不分蘖亲本带型一致的基因型标记为“0”,与双亲带型均不一致的基因型标记为“2”,没有扩增结果的标记为“-”。利用统计分析软件SPSS 17.0的Pearson′s双侧检验,得到相关系数R分别为0.280(标记MRI351)和0.242(标记MRI381),且差异达到极显著水平(表5)。上述结果表明,MRI351和MRI381与分蘖性状紧密连锁,可应用于分蘖型谷子分子标记辅助育种。
表4 160份谷子种质和标记MRI351、MRI381基因分型结果
Tab.4 The genotype of MRI351 and MRI381 in the 160 foxtail millet accessions
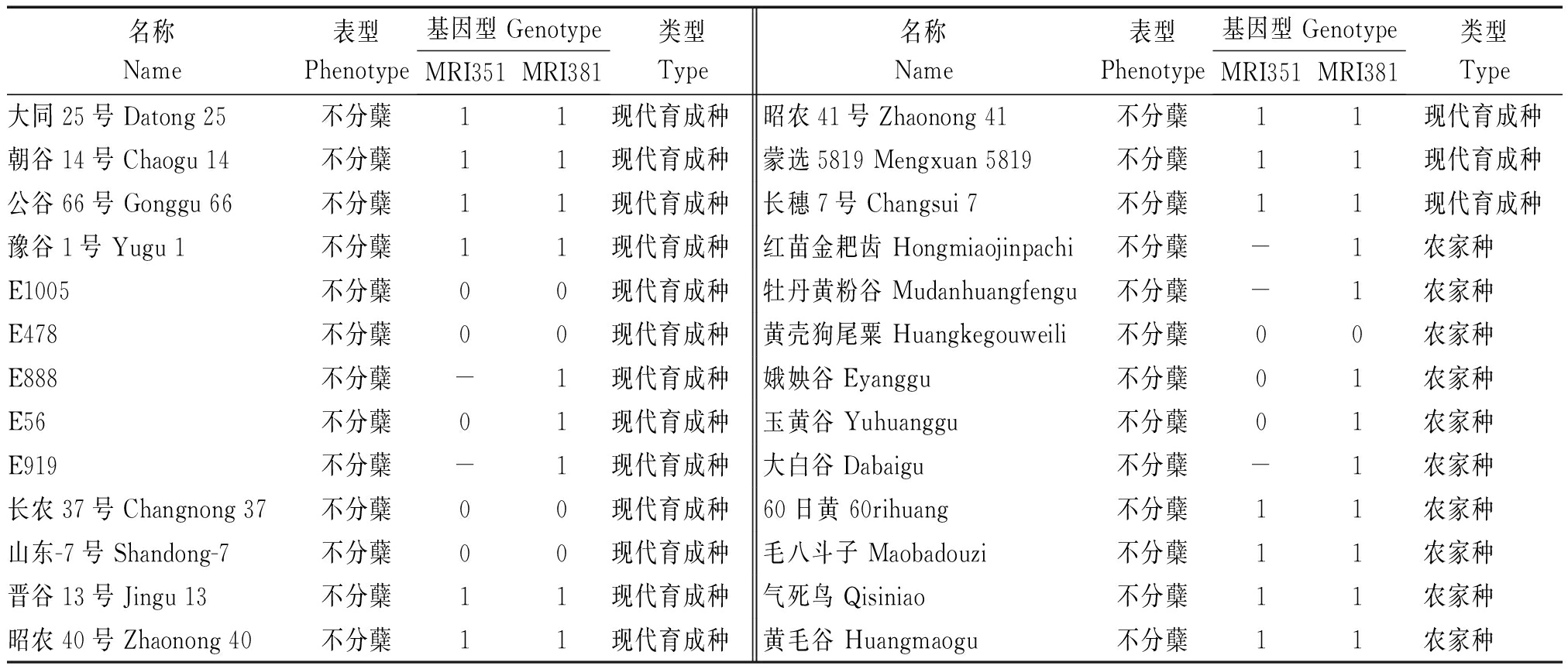
名称Name表型Phenotype基因型 GenotypeMRI351MRI381类型Type名称Name表型Phenotype基因型 GenotypeMRI351MRI381类型Type大同25号 Datong 25不分蘖11现代育成种昭农41号 Zhaonong 41不分蘖11现代育成种朝谷14号 Chaogu 14不分蘖11现代育成种蒙选5819 Mengxuan 5819不分蘖11现代育成种公谷66号 Gonggu 66不分蘖11现代育成种长穗7号 Changsui 7不分蘖11现代育成种豫谷1号 Yugu 1不分蘖11现代育成种红苗金耙齿 Hongmiaojinpachi不分蘖-1农家种E1005不分蘖00现代育成种牡丹黄粉谷 Mudanhuangfengu不分蘖-1农家种E478不分蘖00现代育成种黄壳狗尾粟 Huangkegouweili不分蘖00农家种E888不分蘖-1现代育成种娥姎谷 Eyanggu不分蘖01农家种E56不分蘖01现代育成种玉黄谷 Yuhuanggu不分蘖01农家种E919不分蘖-1现代育成种大白谷 Dabaigu不分蘖-1农家种长农37号 Changnong 37不分蘖00现代育成种60日黄 60rihuang不分蘖11农家种山东-7号 Shandong-7不分蘖00现代育成种毛八斗子 Maobadouzi不分蘖11农家种晋谷13号 Jingu 13不分蘖11现代育成种气死鸟 Qisiniao不分蘖11农家种昭农40号 Zhaonong 40不分蘖11现代育成种黄毛谷 Huangmaogu不分蘖11农家种
表4(续)
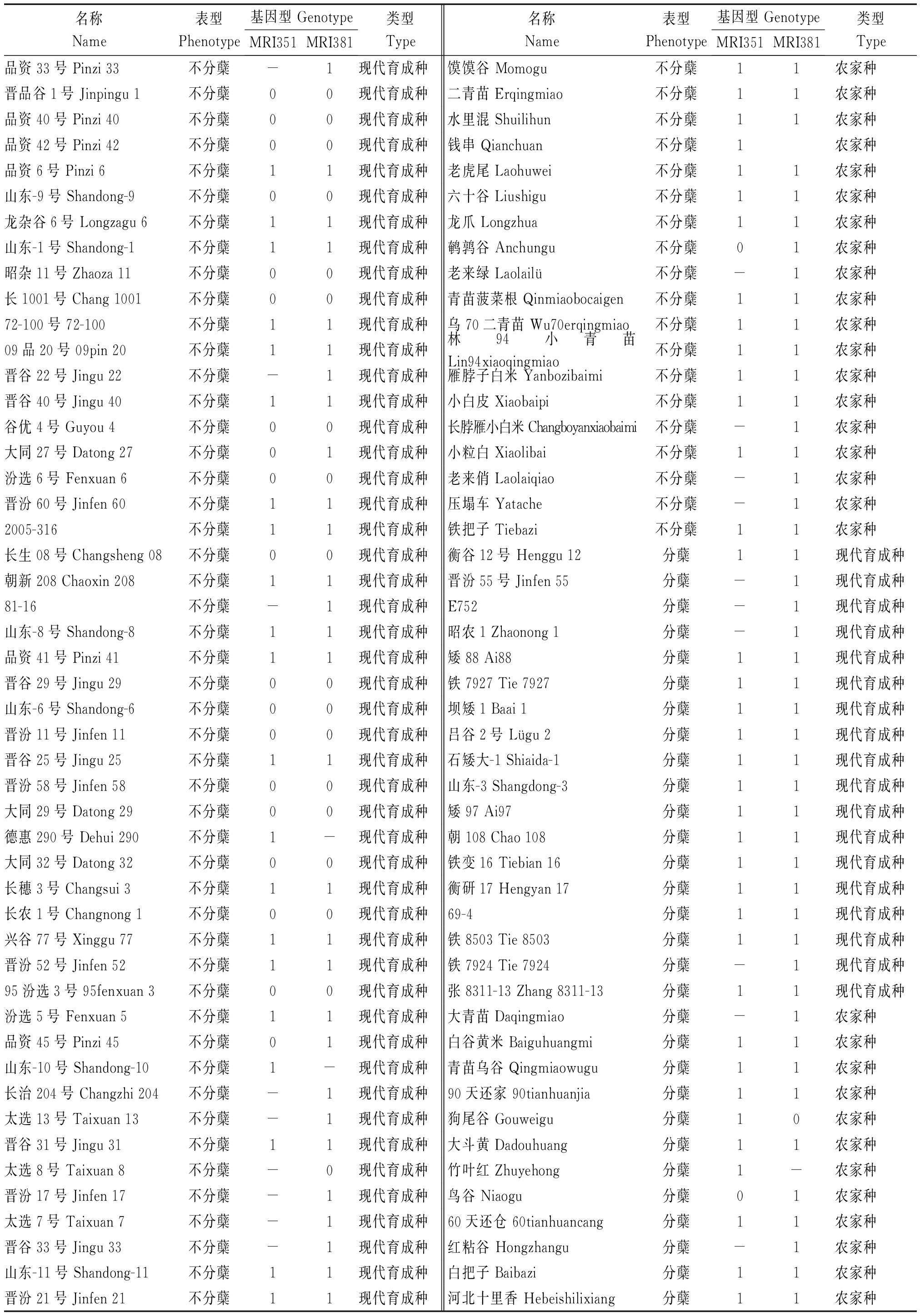
名称Name表型Phenotype基因型 GenotypeMRI351MRI381类型Type名称Name表型Phenotype基因型 GenotypeMRI351MRI381类型Type品资33号 Pinzi 33不分蘖-1现代育成种馍馍谷 Momogu不分蘖11农家种晋品谷1号 Jinpingu 1不分蘖00现代育成种二青苗 Erqingmiao不分蘖11农家种品资40号 Pinzi 40不分蘖00现代育成种水里混 Shuilihun不分蘖11农家种品资42号 Pinzi 42不分蘖00现代育成种钱串 Qianchuan不分蘖1农家种品资6号 Pinzi 6不分蘖11现代育成种老虎尾 Laohuwei不分蘖11农家种山东-9号 Shandong-9不分蘖00现代育成种六十谷 Liushigu不分蘖11农家种龙杂谷6号 Longzagu 6不分蘖11现代育成种龙爪 Longzhua不分蘖11农家种山东-1号 Shandong-1不分蘖11现代育成种鹌鹑谷 Anchungu不分蘖01农家种昭杂11号 Zhaoza 11不分蘖00现代育成种老来绿 Laolailü不分蘖-1农家种长1001号 Chang 1001不分蘖00现代育成种青苗菠菜根 Qinmiaobocaigen不分蘖11农家种72-100号 72-100不分蘖11现代育成种乌70二青苗 Wu70erqingmiao不分蘖11农家种09品20号 09pin 20不分蘖11现代育成种林94小青苗 Lin94xiaoqingmiao不分蘖11农家种晋谷22号 Jingu 22不分蘖-1现代育成种雁脖子白米 Yanbozibaimi不分蘖11农家种晋谷40号 Jingu 40不分蘖11现代育成种小白皮 Xiaobaipi不分蘖11农家种谷优4号 Guyou 4不分蘖00现代育成种长脖雁小白米 Changboyanxiaobaimi不分蘖-1农家种大同27号 Datong 27不分蘖01现代育成种小粒白 Xiaolibai不分蘖11农家种汾选6号 Fenxuan 6不分蘖00现代育成种老来俏 Laolaiqiao不分蘖-1农家种晋汾60号 Jinfen 60不分蘖11现代育成种压塌车 Yatache不分蘖-1农家种2005-316不分蘖11现代育成种铁把子 Tiebazi不分蘖11农家种长生08号 Changsheng 08不分蘖00现代育成种衡谷12号 Henggu 12分蘖11现代育成种朝新208 Chaoxin 208不分蘖11现代育成种晋汾55号 Jinfen 55分蘖-1现代育成种81-16不分蘖-1现代育成种E752分蘖-1现代育成种山东-8号 Shandong-8不分蘖11现代育成种昭农1 Zhaonong 1分蘖-1现代育成种品资41号 Pinzi 41不分蘖11现代育成种矮88 Ai88分蘖11现代育成种晋谷29号 Jingu 29不分蘖00现代育成种铁7927 Tie 7927分蘖11现代育成种山东-6号 Shandong-6不分蘖00现代育成种坝矮1 Baai 1分蘖11现代育成种晋汾11号 Jinfen 11不分蘖00现代育成种吕谷2号 Lügu 2分蘖11现代育成种晋谷25号 Jingu 25不分蘖11现代育成种石矮大-1 Shiaida-1分蘖11现代育成种晋汾58号 Jinfen 58不分蘖00现代育成种山东-3 Shangdong-3分蘖11现代育成种大同29号 Datong 29不分蘖00现代育成种矮97 Ai97分蘖11现代育成种德惠290号 Dehui 290不分蘖1-现代育成种朝108 Chao 108分蘖11现代育成种大同32号 Datong 32不分蘖00现代育成种铁变16 Tiebian 16分蘖11现代育成种长穗3号 Changsui 3不分蘖11现代育成种衡研17 Hengyan 17分蘖11现代育成种长农1号 Changnong 1不分蘖00现代育成种69-4分蘖11现代育成种兴谷77号 Xinggu 77不分蘖11现代育成种铁8503 Tie 8503分蘖11现代育成种晋汾52号 Jinfen 52不分蘖11现代育成种铁7924 Tie 7924分蘖-1现代育成种95汾选3号 95fenxuan 3不分蘖00现代育成种张8311-13 Zhang 8311-13分蘖11现代育成种汾选5号 Fenxuan 5不分蘖11现代育成种大青苗 Daqingmiao分蘖-1农家种品资45号 Pinzi 45不分蘖01现代育成种白谷黄米 Baiguhuangmi分蘖11农家种山东-10号 Shandong-10不分蘖1-现代育成种青苗乌谷 Qingmiaowugu分蘖11农家种长治204号 Changzhi 204不分蘖-1现代育成种90天还家 90tianhuanjia分蘖11农家种太选13号 Taixuan 13不分蘖-1现代育成种狗尾谷 Gouweigu分蘖10农家种晋谷31号 Jingu 31不分蘖11现代育成种大斗黄 Dadouhuang分蘖11农家种太选8号 Taixuan 8不分蘖-0现代育成种竹叶红 Zhuyehong分蘖1-农家种晋汾17号 Jinfen 17不分蘖-1现代育成种鸟谷 Niaogu分蘖01农家种太选7号 Taixuan 7不分蘖-1现代育成种60天还仓 60tianhuancang分蘖11农家种晋谷33号 Jingu 33不分蘖-1现代育成种红粘谷 Hongzhangu分蘖-1农家种山东-11号 Shandong-11不分蘖11现代育成种白把子 Baibazi分蘖11农家种晋汾21号 Jinfen 21不分蘖11现代育成种河北十里香 Hebeishilixiang分蘖11农家种
表4(续)
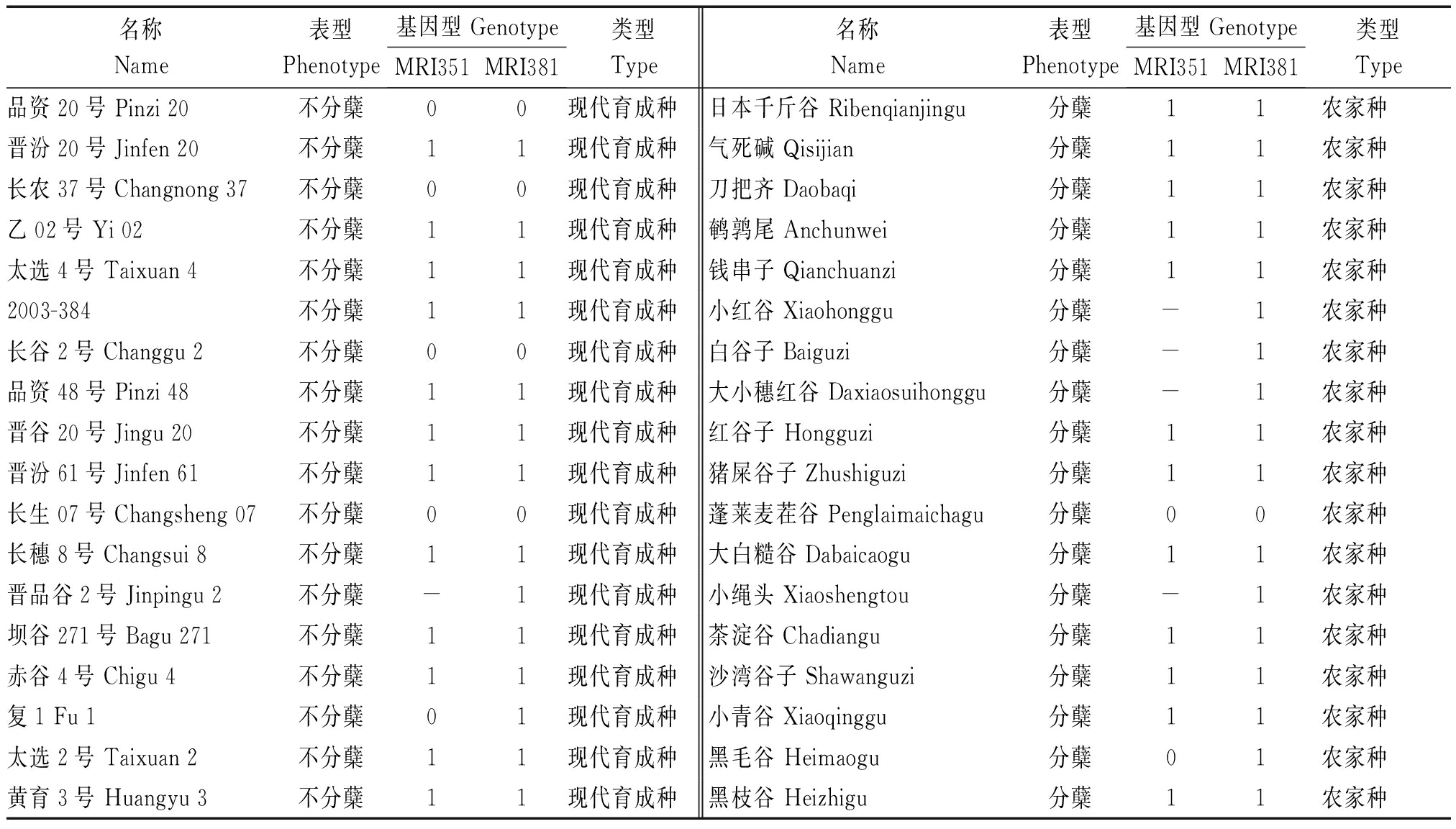
名称Name表型Phenotype基因型 GenotypeMRI351MRI381类型Type名称Name表型Phenotype基因型 GenotypeMRI351MRI381类型Type品资20号 Pinzi 20不分蘖00现代育成种日本千斤谷 Ribenqianjingu分蘖11农家种晋汾20号 Jinfen 20不分蘖11现代育成种气死碱 Qisijian分蘖11农家种长农37号 Changnong 37不分蘖00现代育成种刀把齐 Daobaqi分蘖11农家种乙02号 Yi 02不分蘖11现代育成种鹌鹑尾 Anchunwei分蘖11农家种太选4号 Taixuan 4不分蘖11现代育成种钱串子 Qianchuanzi分蘖11农家种2003-384不分蘖11现代育成种小红谷 Xiaohonggu分蘖-1农家种长谷2号 Changgu 2不分蘖00现代育成种白谷子 Baiguzi分蘖-1农家种品资48号 Pinzi 48不分蘖11现代育成种大小穗红谷 Daxiaosuihonggu分蘖-1农家种晋谷20号 Jingu 20不分蘖11现代育成种红谷子 Hongguzi分蘖11农家种晋汾61号 Jinfen 61不分蘖11现代育成种猪屎谷子 Zhushiguzi分蘖11农家种长生07号 Changsheng 07不分蘖00现代育成种蓬莱麦茬谷 Penglaimaichagu分蘖00农家种长穗8号 Changsui 8不分蘖11现代育成种大白糙谷 Dabaicaogu分蘖11农家种晋品谷2号 Jinpingu 2不分蘖-1现代育成种小绳头 Xiaoshengtou分蘖-1农家种坝谷271号 Bagu 271不分蘖11现代育成种茶淀谷 Chadiangu分蘖11农家种赤谷4号 Chigu 4不分蘖11现代育成种沙湾谷子 Shawanguzi分蘖11农家种复1 Fu 1不分蘖01现代育成种小青谷 Xiaoqinggu分蘖11农家种太选2号 Taixuan 2不分蘖11现代育成种黑毛谷 Heimaogu分蘖01农家种黄育3号 Huangyu 3不分蘖11现代育成种黑枝谷 Heizhigu分蘖11农家种
表5 分蘖数与标记间的相关性分析
Tab.5 Correlation analysis between tiller number and markers
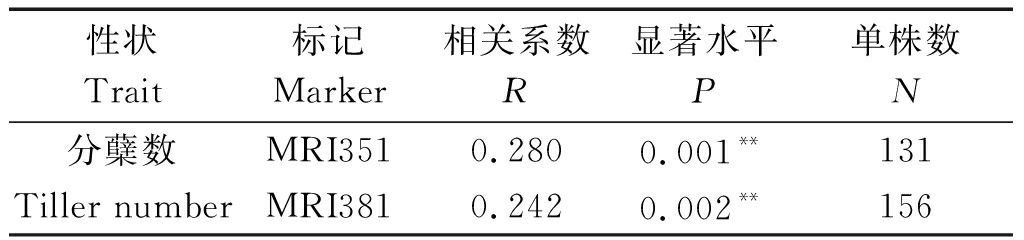
性状Trait标记Marker相关系数R显著水平P单株数N分蘖数MRI3510.280 0.001**131Tiller numberMRI3810.2420.002**156
注:**表示差异极显著(P<0.01)。
Note:** indicate extremely significant difference (P<0.01).
本研究基于简化基因组测序技术(RAD-seq),以2个F2群体(Cross AJ和Cross HC)为主要研究对象,共鉴定了8个控制分蘖数的QTL。其中,qAJTN1、qAJTN5、qAJTN7-2、qAJTN9和qHCTN5等5个QTL为本研究新鉴定的位点,其余3个QTL(qAJTN7-1、qAJTN7-3和qHCTN7)与Jia等[26]和Mauro-Herrera等[3]检测的QTL相一致。
在本研究中,鉴定的较大效应位点qAJTN5(9.8%)的临近区域,Doust等[24]检测到的分蘖相关QTL可解释表型变异率为28.1%,Mauro-Herrera等[3]检测到的分蘖相关QTL(Br5c)可解释表型变异率为23.6%,Zhang等[27]检测到的分蘖相关QTL(qtn5)可解释表型变异率为25.72%和11.31%,这些QTL效应值都比较大,这很可能是由于控制同一性状的QTL成簇分布或群体差异造成的。这些结果也暗示,在谷子第5染色体存在稳定的控制分蘖相关的QTL,相关QTL区间是发掘控制分蘖相关基因的关键区间。
同时,数量性状QTL定位准确性很容易受作图群体和环境因素影响。同一性状QTL在不同群体(F2、F2∶3、RIL和NIL)中效应大小可能不同,相对于F2和F2∶3群体来说,由于RIL和NIL群体能进行多年多点重复试验,其QTL定位结果更加可靠、稳定[3, 24-25, 49]。F2群体的表型数据不能重复,因此,利用F2群体鉴定的QTL效应需要用高级群体进一步验证。尽管如此,目前在农作物中也有一些利用F2群体定位QTL的研究报道[50-52],尤其是结合下一代测序技术能对目标性状进行快速定位[53-54]。本研究采用下一代测序技术,分别利用F2大群体(543个单株)和小群体(131个单株)对控制分蘖数的QTL进行定位研究,与前人研究相比,有一致位点QTL(qAJTN7-1、qAJTN7-3和qHCTN7),也有临近位点QTL(qAJTN5),比较而言,利用大群体检测到了更多与前人研究结果相同或相近的位点,而利用小群体定位的相同或相近位点较少,这表明基于F2大群体的简化基因组测序定位QTL可靠性较F2小群体更强,至于F2大群体与高级群体间的效率差异,还需要进一步利用该F2群体的重组近交系群体进行多年多点试验来验证。
InDel标记具有带型简单、密度高、变异稳定、多态性强、易检测等特点,被广泛应用于作物种质资源品种鉴定、遗传多样性分析和分子标记辅助育种中。赵庆英等[55]利用晋谷21重测序结果开发了一个晋谷21特异的InDel标记2G5501976,该标记可快速鉴定该品种是否为晋谷21或其衍生品种。姜童等[56]利用25对InDel标记对8个鲁西南地区簇生朝天椒品种构建InDel分子遗传图谱,从而对不同品种的相似度进行评价。田希辉等[57]开发了11个与白菜苗期TuMV-C4抗性连锁的InDel标记,这些标记对高抗单株选择的准确率达78%以上,这为白菜TuMV抗性分子育种奠定了良好基础。本研究开发的InDel标记MRI351和MRI381,统计分析表明,与分蘖性状紧密连锁,这为分蘖基因的克隆和分蘖型谷子分子标记辅助育种奠定了基础。
[1] Kebrom T H, Spielmeyer W, Finnegan E J. Grasses provide new insights into regulation of shoot branching[J]. Trends in Plant Science, 2013, 18(1): 41-48.
[2] Kuraparthy V, Sood S, Dhaliwal H S, et al. Identification and mapping of a tiller inhibition gene (tin3) in wheat[J]. Theoretical and Applied Genetics, 2007, 114(2): 285-294.
[3] Mauro-Herrera M, Doust A N. Development and genetic control of plant architecture and biomass in the panicoid grass, setaria[J]. PLoS One, 2016, 11(3): e0151346.
[4] McSteen P, Leyser O. Shoot branching[J]. Annual Review of Plant Biology, 2005, 56: 353-374.
[5] Doust A N. Grass architecture: genetic and environmental control of branching[J]. Current Opinion in Plant Biology, 2007, 10(1): 21-25.
[6] 刘瑞华, 崔贞玉, 冯 权. 水稻分蘖期的温度条件与适宜移栽密度的研究[J]. 吉林农业科学, 1993 (3): 40-43.
[7] 丁艳锋. 氮素营养调控水稻群体质量指标的研究[D]. 南京: 南京农业大学, 1997: 20-24.
[8] Dun E A, de Saint Germain A, Rameau C, et al. Antagonistic action of strigolactone and cytokinin in bud outgrowth control[J]. Plant Physiology, 2012, 158(1):487-498.
[9] Li X Y, Qian Q, Fu Z M, et al. Control of tillering in rice[J]. Nature, 2003, 422(6932): 618-621.
[10] Ishikawa S, Maekawa M, Arite T, et al. Suppression of tiller bud activity in tillering dwarf mutants of rice[J]. Plant Cell Physiology, 2005, 46(1): 79-86.
[11] Booker J, Auldridge M, Wills S, et al.MAX3/CCD7 is a carotenoid cleavage dioxygenase required for the synthesis of a novel plant signaling molecule[J]. Current Biology, 2004, 14(14): 1232-1238.
[12] Arite T, Iwata H, Ohshima K, et al.DWARF10, anRMS1/MAX4/DAD1 ortholog, controls lateral bud outgrowth in rice [J]. The Plant Journal, 2007, 51(6): 1019-1029.
[13] Arite T, Umehara M, Ishikawa S, et al.D14, a strigolactone-insensitive mutant of rice, shows an accelerated outgrowth of tillers[J]. Plant Cell Physiology, 2009, 50(8): 1416-1424.
[14] Lin H, Wang R X, Qian Q, et al.DWARF27, an iron-containing protein required for the biosynthesis of strigolactones, regulates rice tiller bud outgrowth[J]. The Plant Cell, 2009, 21(5): 1512-1525.
[15] Xu C, Wang Y H, Yu Y C, et al. Degradation ofMONOCULM 1 byAPC/C(TAD1) regulates rice tillering[J]. Nature Communications, 2012, 3: 750.
[16] Lin Q B, Wang D, Dong H, et al. RiceAPC/C(TE) controls tillering by mediating the degradation ofMONOCULM 1[J]. Nature Communications, 2012, 3: 752.
[17] Jiang L, Liu X, Xiong G S, et al.DWARF 53 acts as a repressor of strigolactone signalling in rice[J]. Nature, 2013, 504: 401-405.
[18] Tanaka W, Ohmori Y, Ushijima T, et al. Axillary meristem formation in rice requires the WUSCHEL ortholog TILLERS ABSENT1[J]. The Plant Cell, 2015, 27(4): 1173-1184.
[19] Xu J X, Zha M R, Li Y, et al. The interaction between nitrogen availability and auxin, cytokinin, and strigolactone in the control of shoot branching in rice (Oryza sativa L.)[J]. Plant Cell Reports, 2015, 34(9):1647-1662.
[20] Zuo J R, Li J Y. Molecular dissection of complex agronomic traits of rice: a team effort by Chinese scientists in recent years[J]. National Science Review, 2014, 1(2): 253-276.
[21] Li W L, Nelson J C, Chu C Y, et al. Chromosomal locations and genetic relationships of tiller and spike characters in wheat[J]. Euphytica, 2002, 125(3): 357-366.
[22] Li Z K, Peng T, Xie Q G, et al. Mapping of QTL for tiller number at different stages of growth in wheat using double haploid and immortalized F2 populations[J]. Indian Academy of Sciences, 2010, 89(4): 409-415.
[23] Naruoka Y, Talbert L E, Lanning S P, et al. Identification of quantitative trait loci for productive tiller number and its relationship to agronomic traits in spring wheat[J]. Theoretical and Applied Genetics, 2011, 123(6): 1043-1053.
[24] Doust A N, Devos K M, Gadberry M D, et al. Genetic control of branching in foxtail millet[J]. Proceeding of National Academy of Sciences of the United States of American, 2004, 101(24): 9045-9050.
[25] Doust A N, Kellogg E A. Effect of genotype and environment on branching in weedy green millet (Setaria viridis) and domesticated foxtail millet (Setaria italica) (Poaceae)[J]. Molecular Ecology, 2006, 15(5): 1335-1349.
[26] Jia G Q, Huang X H, Zhi H, et al. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (Setaria italica)[J]. Nature Genetics, 2013, 45(8): 957-961.
[27] Zhang K, Fan G, Zhang X, et al. Identification of QTLs for 14 agronomically important traits inSetaria italica based on SNPs generated from high-throughput sequencing[J]. G3 (Bethesda), 2017, 7(5): 1587-1594.
[28] Bennetzen J L, Schmutz J, Wang H, et al. Reference genome sequence of the model plantSetaria[J]. Nature Biotechnology, 2012, 30(6): 555-561.
[29] Zhang G Y, Liu X, Quan Z W, et al. Genome sequence of foxtail millet (Setaria italica) provides insights into grass evolution and biofuel potential[J]. Nature Biotechnology, 2012, 30: 549-554.
[30] Sato K, Mukainari Y, Naito K, et al. Construction of a foxtail millet linkage map and mapping of spikelet-tipped bristles 1(stb1) by using transposon display markers and simple sequence repeat markers with genome sequence information[J]. Molecular Breeding, 2013, 31(3): 675-684.
[31] Masumoto H, Takagi H, Mukainari Y, et al. Genetic analysis ofNEKODE1 gene involved in panicle branching of foxtail millet,Setaria italica (L.) P. Beauv., and mapping by using QTL-seq[J]. Molecular Breeding, 2016, 36: 59.
[32] Zhang S, Tang C J, Zhao Q, et al. Development of highly polymorphic simple sequence repeat markers using genome-wide microsatellite variant analysis in foxtail millet (Setaria italica (L.) P. Beauv.)[J]. BMC Genomics, 2014, 15: 78.
[33] Xue C X, Zhi H, Fang X J, et al. Characterization and fine mapping ofSiDWARF2 (D2) in foxtail millet[J]. Crop Science, 2016, 56(1): 95.
[34] Li W, Tang S, Zhang S, et al. Gene mapping and functional analysis of the novel leaf color geneSiYGL1 in foxtail millet (Setaria italica (L.) P. Beauv.)[J]. Physiologia Plantarum, 2016, 157(1): 24-37.
[35] Liu X T, Tang S, Jia G Q, et al. The C-terminal motif ofSiAGO1b is required for the regulation of growth, development and stress responses in foxtail millet (Setaria italica (L.) P. Beauv.)[J]. Joural of Experimental Botany, 2016, 67(11): 3237-3249.
[36] Xiang J S, Tang S, Zhi H, et al.Loose Panicle1 encoding a novel WRKY transcription factor, regulates panicle development, stem elongation, and seed size in foxtail millet (Setaria italica (L.) P. Beauv.)[J]. PLoS One, 2017, 12(6): e0178730.
[37] Fan X K, Tang S, Zhi H, et al. Identification and fine mapping ofSiDWARF3 (D3), a pleiotropic locus controlling environment-independent dwarfism in foxtail millet[J]. Crop Science, 2017, 57(5): 2431-2442.
[38] 王冠杰, 周 波, 李玉花. RAD标记测序及其在分子育种中的应用[J]. 中国生物化学与分子生物学报, 2012, 28(9): 797-803.
[39] Wang J, Wang Z L, Du X F, et al. A high-density genetic map and QTL analysis of agronomic traits in foxtail millet(Setaria italica (L.) P. Beauv.) using RAD-seq[J]. PLoS One, 2017, 12(6): e0179717.
[40] Wang J, Yang H Q, Du G H, et al. Mapping ofSihc1, which controls hull color, using a high-density genetic map based on restriction site-associated DNA sequencing in foxtail millet (Setaria italica (L.) P. Beauv. )[J]. Molecular Breeding, 2017, 37: 128.
[41] Chen D H, Ronald P C. A rapid DNA minipreparation method suitable for AFLP and other PCR applications[J]. Plant Molecular Biology Reporter, 1999, 17(1): 53-57.
[42] Baird N A, Etter P D, Atwood T S, et al. Rapid SNP discovery and genetic mapping using sequenced RAD markers[J]. PLoS One, 2008, 3(10): e3376.
[43] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754-1760.
[44] McKenna A, Hanna M, Banks E, et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research, 2010, 28(1): 1297-1303.
[45] Li H, Handsaker B, Wysoker A, et al. The Sequence Alignment/Map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079.
[46] Wu Y H, Bhat P R, Close T J, et al. Efficient and accurate construction of genetic linkage maps from the minimum spanning tree of a graph[J]. PLoS Genetics, 2008, 4(10): e1000212.
[47] Wang S, Basten C J, Zeng Z B. Windows QTL cartographer 2.5. Department of Statistics, North Carolina State University, Raleigh, NC[EB/OL]. [2012-02-05]. http://statgen.ncsu.edu/qtlcart/WQTLCart.htm.
[48] Marklund S, Chaudhary R, Marklund L, et al. Extensive mtDNA diversity in horses revealed by PCR-SSCP analysis[J]. Animal Genetics, 1995, 26(3): 193-196.
[49] Weng Y Q, Colle M, Wang Y H, et al. QTL mapping in multiple populations and development stages reveals dynamic quantitative trait loci for fruit size in cucumbers of different market classes[J]. Theoretical and Applied Genetics, 2015, 128(9): 1747-1763.
[50] Tomaszewski C, Byrne S L, Foito A, K et al. Genetic linkage mapping in an F2 perennial ryegrass population using DArT markers[J]. Plant Breeding, 2012, 131(2): 345-349.
[51] Barakat M N, Wahba L E, Milad S I. Molecular mapping of QTLs for wheat flag leaf senescence under water-stress[J]. Biologia Plantarum, 2013, 57(1): 79-84.
[52] Herlina L, Sobir S, Trijatmiko K R. Identification of quantitative trait loci (QTL) for awn, incomplete panicle exertion and total spikelet number in an F2 population derived from a backcross inbred line, Bio-148, and the recurrent parent, IR64[J]. Makara Journal of Science, 2016, 20(1): 17-27.
[53] Thyssen G N, Fang D D, Turley R B, et al. Next generation genetic mapping of the Ligon-lintless-2 (Li2) locus in upland cotton (Gossypium hirsutum L.)[J]. Theoretical and Applied Genetics, 2014, 127(10): 2183-2192.
[54] Li L B, Zhao S Q, Su J J, et al. High-density genetic linkage map construction by F2 populations and QTL analysis of early-maturity traits in upland cotton (Gossypium hirsutum L.)[J]. PLoS One, 2017, 12(8): e0182918.
[55] 赵庆英, 张瑞娟, 王瑞良, 等. 基于名优谷子品种晋谷21全基因组重测序的分子标记开发[J]. 作物学报, 2018, 4(5): 686-696.
[56] 姜 童, 王 辉, 陈 宁, 等. 利用InDel指纹图谱评价鲁西南地区簇生朝天椒品种的相似度[J]. 华北农学报, 2018, 33(2): 126-132.
[57] 田希辉, 于拴仓, 苏同兵, 等. 一个新的白菜苗期TuMV-C4抗性主效QTL定位及连锁分子标记开发[J]. 华北农学报, 2014, 29(6): 1-5.