雪菜BjSnRK2C基因的克隆及生物信息学分析
张春兰1,满丽莉1,向殿军2,李旭新2,包乌日娜2,刘 鹏2
(1.内蒙古民族大学 生命科学学院,内蒙古 通辽 028042;2.内蒙古民族大学 农学院, 内蒙古 通辽 028042)
摘要:SnRK2家族基因编码植物体内一类丝氨酸/苏氨酸蛋白激酶,是ABA通路中影响植物抗逆能力的关键调控基因。基于SnRK2成员cDNA的保守区序列信息,利用RT-PCR与SON-PCR相结合的方法从雪菜中克隆了一个SnRK2全长cDNA序列,命名为BjSnRK2C。测序结果显示,该cDNA序列全长1 380 bp,包含一个137 bp 的3′-非翻译区,一个214 bp 的5′-非翻译区,一个1 029 bp的完整开放阅读框,编码342个氨基酸,在GenBank数据库的登录号为MF983711。BjSnRK2C蛋白激酶理论分子量为38.2 ku,理论等电点为5.61,属于亲水性蛋白,存在2个显著跨膜结构域,含有30处磷酸化位点,没有信号肽,最可能定位的位置是在细胞质上。BjSnRK2C蛋白激酶的二级结构具有130个α-螺旋、126个无规则卷曲、59个延伸链和27个β-转角。BjSnRK2C蛋白包含1个丝氨酸/苏氨酸酶活性结构域和1个ATP结合位点,同拟南芥AtSnRK2.8蛋白激酶亲缘关系最近,处在同一进化分枝。雪菜BjSnRK2C蛋白激酶的三级结构与拟南芥AtSnRK2.8蛋白激酶极为相似,推测它们具有类似的功能。BjSnRK2C蛋白激酶的C末端仅含有结构域Ⅰ,缺少结构域Ⅱ,暗示雪菜BjSnRK2C基因在参与非生物胁迫过程中很可能也是不依赖ABA调控通路的。研究结果可为今后深入研究BjSnRK2C功能奠定理论基础。
关键词:雪菜;BjSnRK2C基因;克隆;生物信息学
高等植物在生存方式上有别于动物,其属于固着生物,在生长发育过程中容易遭受干旱、极端温度、高盐和重金属等多种非生物胁迫的影响,造成作物品质和产量的损失,严重时会造成绝收[1-4]。干旱胁迫是众多非生物胁迫中最为突出的一种,全世界每年大约有70%的粮食产量损失可以归因于干旱胁迫[5]。植物在长期进化过程中形成了一系列应答保护机制去忍受或适应各种极端环境条件[6-7]。通过特异的信号传导通路感知非生物胁迫,植物启动相关胁迫基因的表达,调节细胞内的各种新陈代谢反应去适应逆境胁迫。其中,蔗糖非发酵相关的蛋白激酶(Sucrose non-fermenting-1-related protein kinase, SnRK)调控靶蛋白的可逆磷酸化修饰在这一系列防御机制过程中发挥着重要的作用。
基于蛋白序列相似性和结构域特点,高等植物的SnRK家族成员可被分为SnRK1、SnRK2、SnRK3共3个亚家族。其中,SnRK2是植物所特有的一类丝氨酸/苏氨酸蛋白激酶,在植物生长发育、养分利用、ABA信号传导和响应渗透胁迫等过程中发挥着重要的作用。SnRK2蛋白激酶成员通过ABA非依赖和ABA依赖的2种调节途径参与渗透胁迫信号通路。目前,对于SnRK2非依赖ABA调控通路的研究还不太深入,而对ABA依赖信号通路的研究已经取得一定进展。SnRK2是ABA通路中至关重要的蛋白激酶,其作用的底物至少有58个,涉及表型遗传调控、RNA拼接、ROS稳定、叶绿体过程和离子通道等多种生物过程[8]。ABRE/AREB/bZIP转录因子是SnRK2蛋白激酶的重要底物,其活性严格受SnRK2蛋白激酶磷酸化的调节。ABA应答基因的启动子上含有多个保守的响应ABA的顺式作用元件ABRE,SnRK2蛋白激酶可以磷酸化转录因子如ABRE/AREB/bZIP,活性被磷酸化调节的反式因子随后与ABRE顺式作用元件结合,从而导致ABA响应基因mRNA过量累积[9-10]。与SnRK1和SnRK3成员相比,人们对SnRK2成员的研究较为深入,目前已经在拟南芥[11-12]、水稻[13]和小麦[14]等植物中分离了多个SnRK2家族成员,部分成员的抗逆作用已经得到了验证。Diédhiou等[15]将基因转入水稻,转基因水稻表现出对高盐胁迫的抗性。与对照相比,过量表达AtSnRK2.8基因的拟南芥植株的抗旱能力有显著的提高[16]。过表达小麦TaSnRK2.4基因的拟南芥植株也表现出对冻害、干旱和高盐胁迫的抗性[14]。
雪菜(Brassica juncea)为十字花科(Brassicaceae)芸薹属(Brassica)双子叶1年生草本植物,是我国长江流域普遍栽培的冬春2季重要蔬菜。雪菜含有大量活性很强的还原物质,参与机体中诸多氧化还原过程。此外,雪菜具有很强的抵御环境胁迫的能力,包含众多的优良性状及抗逆基因源。但是,相对于其他十字花科蔬菜,对雪菜抵抗逆境胁迫的研究较少,到目前为止,还未见有对SnRK2蛋白激酶成员的研究与报道。
本研究基于NCBI中的Nucleotide collection数据库信息,同源克隆了一个雪菜SnRK2基因,对其基因结构、理化性质、系统发生等进行了生物信息学分析。以期对雪菜SnRK2基因的抗逆功能研究提供理论依据,为揭示雪菜抗逆机制以及雪菜SnRK2基因在其他作物抗逆分子育种中的应用奠定理论基础。
1 材料和方法
1.1 试验材料
本研究所用的雪菜(Brassica juncea Coss. Var.Multiceps Tsen et Lee)种子为雪里蕻(九头鸟),为青县青风种业有限公司产品。将雪菜种子用70%乙醇进行表面消毒处理,用无菌水漂洗后播种到土壤中,培养温度为26 ℃,光照强度2 000 lx,光照16 h/d。用含有10% PEG6000的水溶液处理30 d苗龄的雪菜幼苗12 h,收集幼苗的全株,样品经液氮速冻后,置于-70 ℃冰箱保存,用于总RNA制备。
PrimeScriptTM 1st Strand cDNA,PrimeSTAR® HS DNA Polymerase,T-Vector pMD® 19(Simple)购于TaKaRa公司。DL2000 DNA Marker购于TaKaRa公司,Marker Ⅰ、Marker Ⅲ、RNAprep pure Plant Kit、琼脂糖凝胶回收试剂盒和DNA纯化试剂盒均购于天根生化科技(北京)有限公司。PCR引物合成及PCR产物测序由生工生物工程(上海)股份有限公司完成。
1.2 试验方法
1.2.1 总RNA的提取及第一链cDNA合成 采用RNAprep pure Plant Kit提取上述处理雪菜的总RNA,琼脂糖凝胶电泳检测其质量。第1链cDNA合成按照PrimeScriptTM 1st Strand cDNA Synthesis Kit说明书进行。
1.2.2BjSnRK2C基因克隆 以拟南芥AtSnRK2C(NP_974170.1)氨基酸序列作为检索序列,进行Protein Blast搜索,下载AlSnRK2C(XP_020874391.1)、CsSnRK2C(XP_019093971.1)、RsSnRK2C(XP_019577936.1)、BoSnRK2C(XP_013588502.1)、BrSnRK2C(XP_009104577.1)、BnSnRK2C(XP_013676330.1)6条氨基酸序列,登录Blockmaker(http://blocks.fhcrc.org/blocks/make_blocks.html),进行Block比对,在保守区域进行CodeHop引物搜索,设计1对简并引物(5′-GGAGRTT KTGTSGACGGCAAC-3′,5′-CCAWCSGCACRGGAGA GTCCAG-3′),利用PrimeSTAR® HS DNA Polymerase扩增BjSnRK2C基因保守区。扩增程序为:98 ℃ 10 s; 55 ℃ 15 s,72 ℃ 1 min,30个循环。PCR产物经回收、末端加A和纯化后克隆至T-Vector pMDTM 19(Simple)上进行测序。根据保守区的测序结果,以第1链cDNA为模板,分别设计2条同向嵌套引物,利用PrimeSTAR®HS DNA Polymerase对BjSnRK2C基因的3′端和5′进行SON-PCR(Single oligonucleotide nested PCR)扩增。扩增3′端的引物为5′-CAGC ACCACGTGTAAAGATATGTG-3′,5′-TTGGATACTCA AAGTCAGGAGTTC-3′,2条引物间距离为57 bp;扩增5′端的引物为5′-ATGACAGTAACTAACTCCT GAG-3′,5′-CCTCCGGCGGCATATTCCATTAC-3′,2条引物间距离为97 bp。SON-PCR扩增程序参照Antal等[17]方法进行。回收3′端和5′端扩增产物,克隆至T-Vector pMDTM 19(Simple)进行测序。利用Contig 1软件对上述3段序列进行拼接,根据拼接序列设计引物5′-AATCGCTTTGCATTATTATTACATC-3′,5′-TGAGAATCAAACACCTCAACAATAC-3′,对拼接序列进行RT-PCR验证。
1.2.3BjSnRK2C基因的生物信息学分析 基因编码区的预测、序列提交、蛋白质的理化性质分析,分别采用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)、Banki(https://www.ncbi.nlm.nih.gov/WebSub/?tool=genbank)和ProtParam tool(http://web.expasy.org/protparam/)在线程序完成。利用ScanProsite(http://prosite.expasy.org/scanprosite/)、TMpred Server(http://www.ch.embnet.org/software/TMPRED_form.html)、NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)、ProtScale(http://web.expasy.org/protscale/)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)、Signal P 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)在线程序分别对BjSnRK2C蛋白进行保守功能域、跨膜区、磷酸化位点、亲水性/疏水性、二级结构和信号肽分析预测。亚细胞定位和蛋白质三级结构预测分别采用PSORT Prediction(http://psort1.hgc.jp/form.html)和SWISS-MODEL(https://swissmodel.expasy.org/interactive)程序进行。BlastP搜索与BjSnRK2C同源的蛋白序列,基于MEGA 5.0软件以临近法完成蛋白系统发育树构建,利用GENEDOC软件生成蛋白多序列比对。
2 结果与分析
2.1 雪菜BjSnRK2C基因的克隆
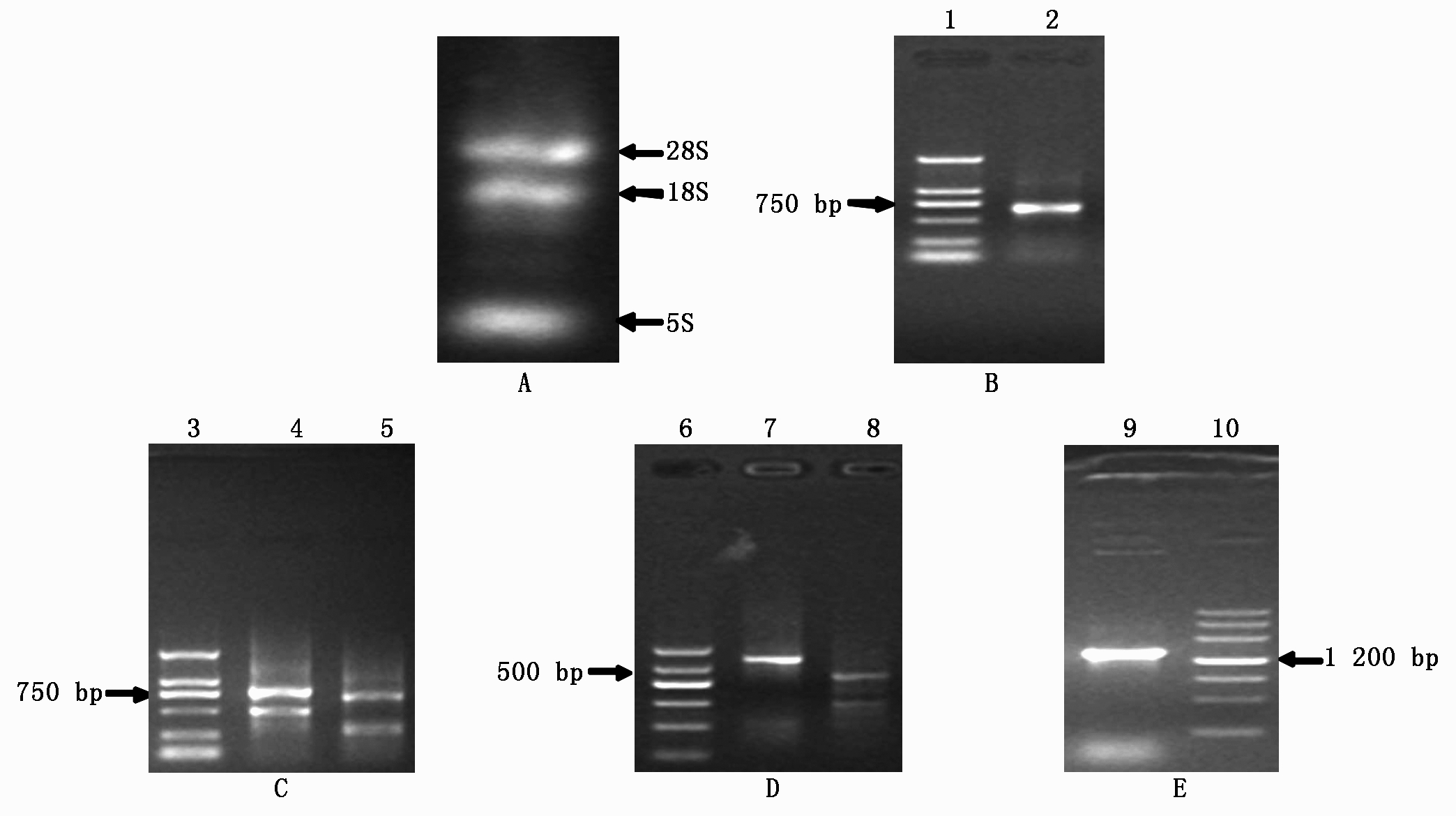
雪菜幼苗的总RNA提取结果显示(图1-A),所提取的总RNA 5S,18S,28S条带较完整和清晰,可以用于合成cDNA第1链的模板。以第1链cDNA为模板对目的基因保守区进行RT-PCR扩增,扩增产物经琼脂糖凝胶电泳显示,产生730 bp左右的特异性片段(图1-B)。扩增产物经测序得到729 bp的cDNA序列,与拟南芥AtSnRK2C(NM_202441.3)核苷酸序列相似性为91.7%。3′端扩增结果显示,第1轮和第2轮SON-PCR反应各产生2条目的条带(图1-C),产物大小符合引物设计的预期结果。回收第2轮反应产生的最大条带,经T-A克隆测序得到736 bp的cDNA序列。5′端扩增结果显示,第1轮SON-PCR反应产生大小约560 bp的特异条带(图1-D),第2轮SON-PCR反应产生2条目的条带(图1-D),最大条带符合引物设计的预期结果。回收第2轮反应产生的最大条带,经T-A克隆测序得到462 bp的cDNA序列。3段cDNA序列(729,736,462 bp)被Contig 1软件拼接成1条1 380 bp的cDNA序列。根据拼接序列两端设计1对引物进行RT-PCR验证,产生1条大小约1 400 bp的特异条带(图1-E),测序结果表明,扩增产物大小为1 380 bp,扩增产物序列与拼接序列完全一致。ORF Finder预测结果显示(图2),1 380 bp的cDNA具有1个137 bp的3′-非编码区(3′-UTR),1个1 029 bp的开放阅读框(ORF)和1个214 bp的5′-非编码区(5′-UTR),编码342个氨基酸。将该cDNA全长序列命名为BjSnRK2C,提交至GenBank,获得登录号为MF983711。
2.2 雪菜BjSnRK2C蛋白激酶的氨基酸组成和理化性质分析
BjSnRK2C全长cDNA编码342个氨基酸,理论等电点为5.61,理论分子量为38.2 ku;BjSnRK2C蛋白中Ser、Gly、Leu、Asp、Ile、Glu、Val、Lys含量较高,分别为9.4%,8.2%,7.9%,7.6%,7.6%,7.3%,6.7%,6.1%;BjSnRK2C蛋白中带负电荷的残基(Asp+Glu)总数为51个,带正荷的残基(Arg+Lys)总数为41个;BjSnRK2C蛋白分子式为C1688H2651N463O516S15,平均疏水性(GRAVY)为-0.278,脂肪氨基酸指数(AI)为85.5。BjSnRK2C蛋白的不稳定指数为42.63,属于不稳定蛋白,与拟南芥AtSnRK2C蛋白激酶的氨基酸组成极为相似。
2.3 BjSnRK2C蛋白激酶的序列分析
为了明确BjSnRK2C蛋白激酶的结构特征,对推导的BjSnRK2C蛋白激酶的氨基酸序列进行ScanProsite在线预测和多序列比对。ScanProsite预测结果表明,BjSnRK2C蛋白包含1个酸性氨基酸序列延伸的C-端激酶调控域及1个高度保守的N-端蛋白激酶催化域。高度保守的N-端催化域具有1个24个氨基酸的ATP结合位点(图3-黑框Ⅰ)和1个13个氨基酸的丝氨酸/苏氨酸蛋白激酶激活位点(图3-黑框Ⅱ)。C-端包含1个34个氨基酸的结构域Ⅰ(Domain Ⅰ)(图3-黑框Ⅲ),缺少1个44个氨基酸的结构域Ⅱ(Domain Ⅱ)(图3-黑框Ⅳ)。BjSnRK2C氨基酸序列与拟南芥AtSnRK2.8(NP_974170.1)、水稻OsSAPK9(BAD18005.1)、拟南芥AtSnRK2.3(NP_201489.1)、水稻OsSAPK6(BAD18002.1)和拟南芥AtSnRK2.10 (NP_176290.1)的氨基酸序列的相似性分别为84.21%,66.08%,65.20%,64.33%,64.33%(图3)。这与SnRK2家族成员的高度保守的N-端催化域和特异的C-端激酶调控域相关,SnRK2家族成员在功能上的相似性与差异性、响应胁迫的相似性与差异性等都可能归因于SnRK2s序列的一致性和多样性。
2.4 BjSnRK2C蛋白激酶的进化分析
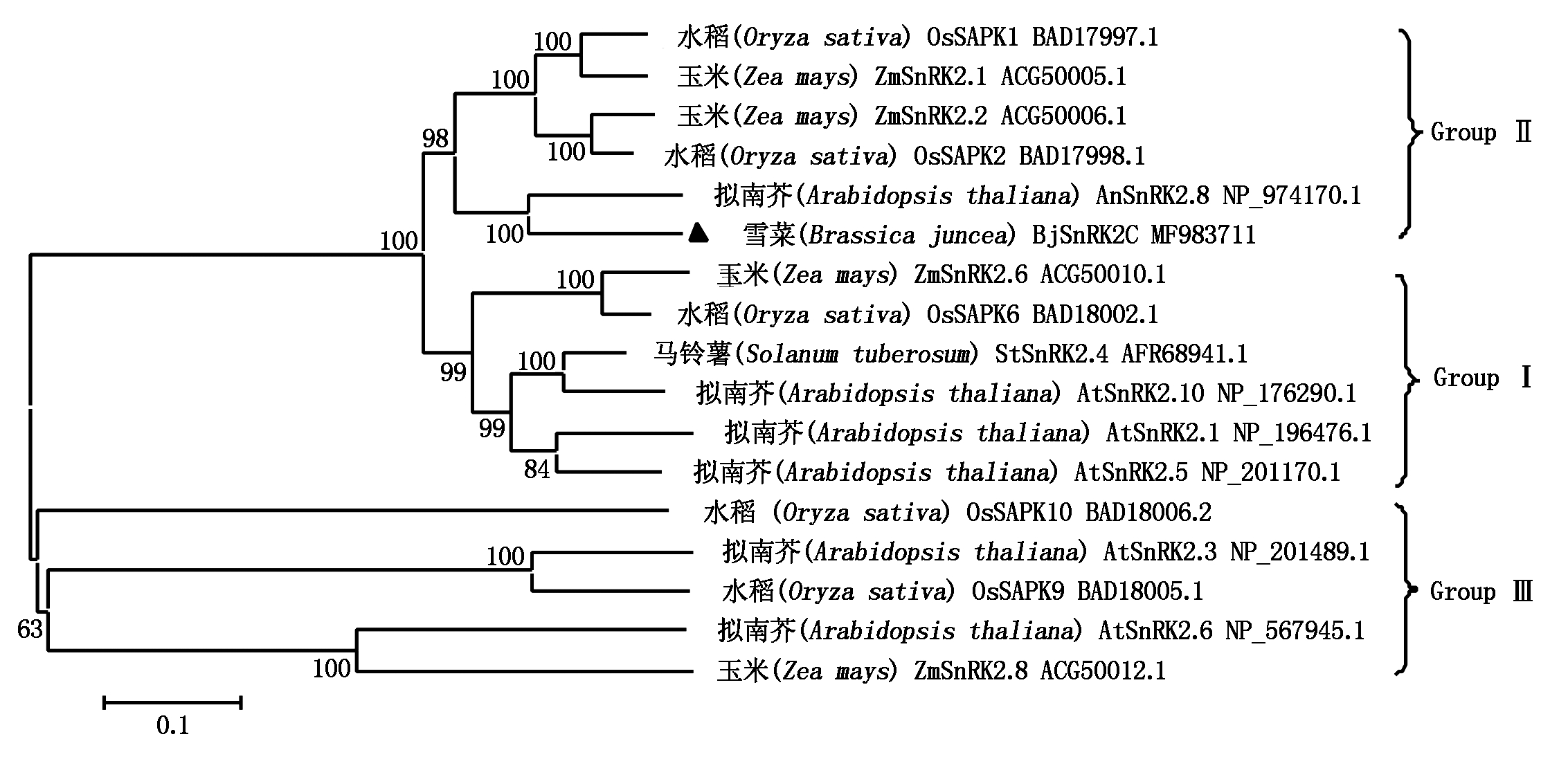
为了明确BjSnRK2C蛋白激酶同其他SnRK2s的进化关系,用来自雪菜、拟南芥、玉米、马铃薯的17个SnRK2成员的氨基酸序列构建进化树,结果如图4所示。17个SnRK2蛋白激酶可以分为3组,雪菜BjSnRK2C蛋白激酶同拟南芥AtSnRK2.8、水稻OsSAPK2、玉米ZmSnRK2.2、玉米ZmSnRK2.1和水稻OsSAPK1这6个成员聚为一组,其中,BjSnRK2C蛋白激酶与拟南芥AtSnRK2.8蛋白激酶关系最近,表明它们属于同源基因,可能具有类似的功能。
2.5 BjSnRK2C信号肽的预测和分析
信号肽是由长度5~30 个N-末端氨基酸组成的短肽链(有时不一定在N-端),其功能是引导新合成的蛋白质向分泌通路转移。利用SignalP4.1 Server对BjSnRK2C进行预测。结果显示,可能没有信号肽区域存在于BjSnRK2C蛋白激酶中(图5)。
2.6 BjSnRK2C蛋白激酶的的跨膜区分析
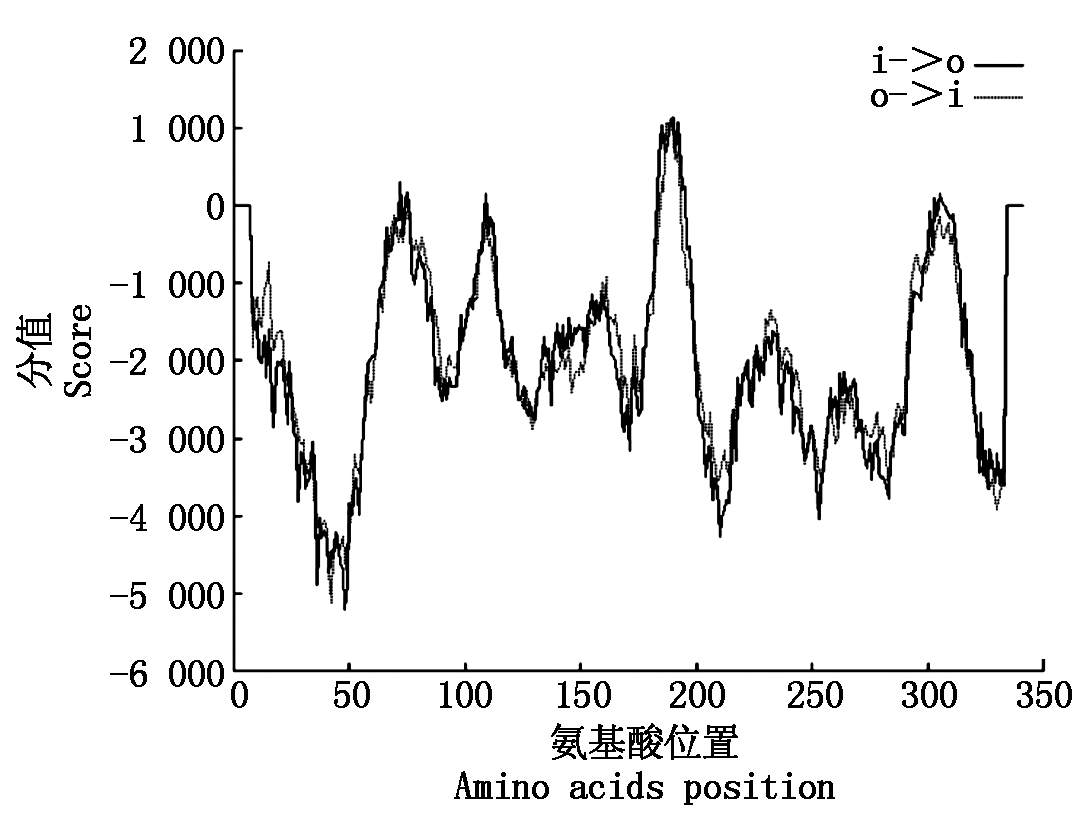
α-螺旋结构通常作为蛋白质序列中跨越细胞膜的区域,构成跨膜区的氨基酸大部分必须是疏水性氨基酸,这样才能使膜蛋白顺利通过细胞膜的磷脂双分子层。 预测结果显示(图6),可能有5个跨膜结构域存在于BjSnRK2C蛋白中。由外向内的跨膜结构域有1个,位于182-198位,长度为17个氨基酸,分数为1 109。由内向外的跨膜结构域有4个:第1个位于65-81位,长度为17个氨基酸,分数为304;第2个位于102-119位,长度为18个氨基酸,分数为153;第3个位于183-200位,长度为18个氨基酸,分数为1 139;第4个位于298-314位,长度为17个氨基酸,分数为148。所以,BjSnRK2C蛋白激酶可能存在2个显著跨膜结构域(分数大于500)。
2.7 BjSnRK2C蛋白激酶的亚细胞定位
为了掌握雪菜BjSnRK2C蛋白的表达和作用位置,利用在线预测软件PSORT Prediction对BjSnRK2C蛋白进行亚细胞定位。预测结果表明,BjSnRK2C蛋白的可能定位在细胞质(Cytoplasm)、微体(Microbody)、线粒体基质空间(Mitochondrial matrix space)和溶酶体(Lysosome)上。这4个位置的决定性系数(Certainty)分别为0.450,0.396,0.100,0.100,所以,推测BjSnRK2C蛋白最可能定位的位置是在细胞质上。
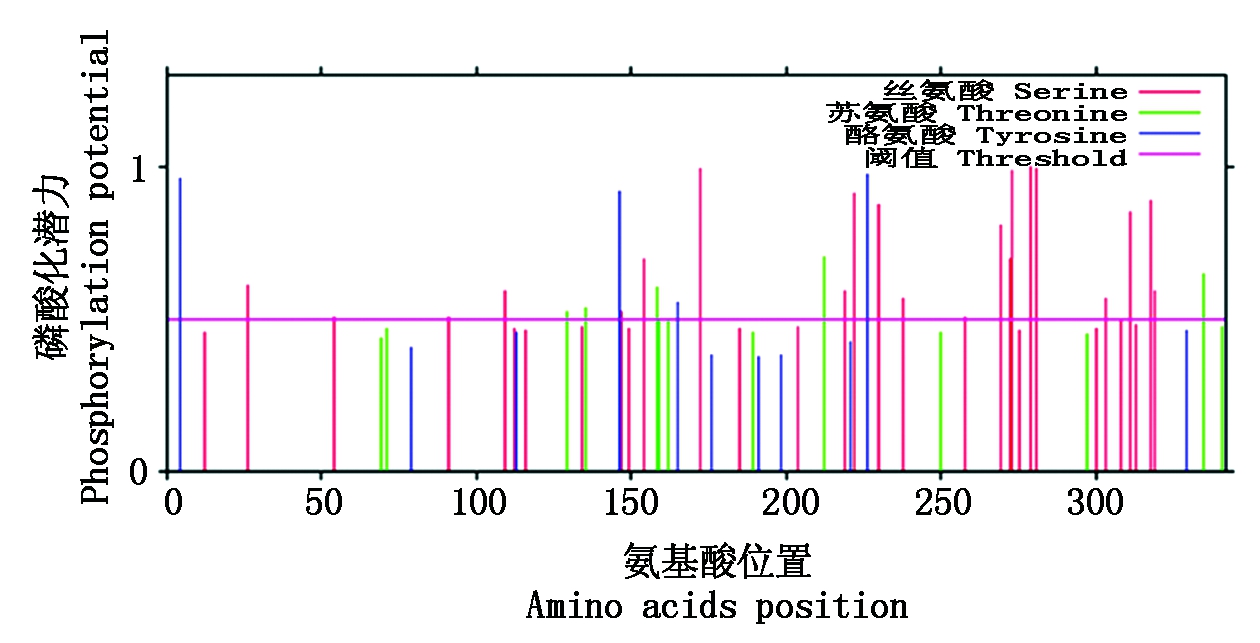
2.8 BjSnRK2C蛋白激酶磷酸化位点分析
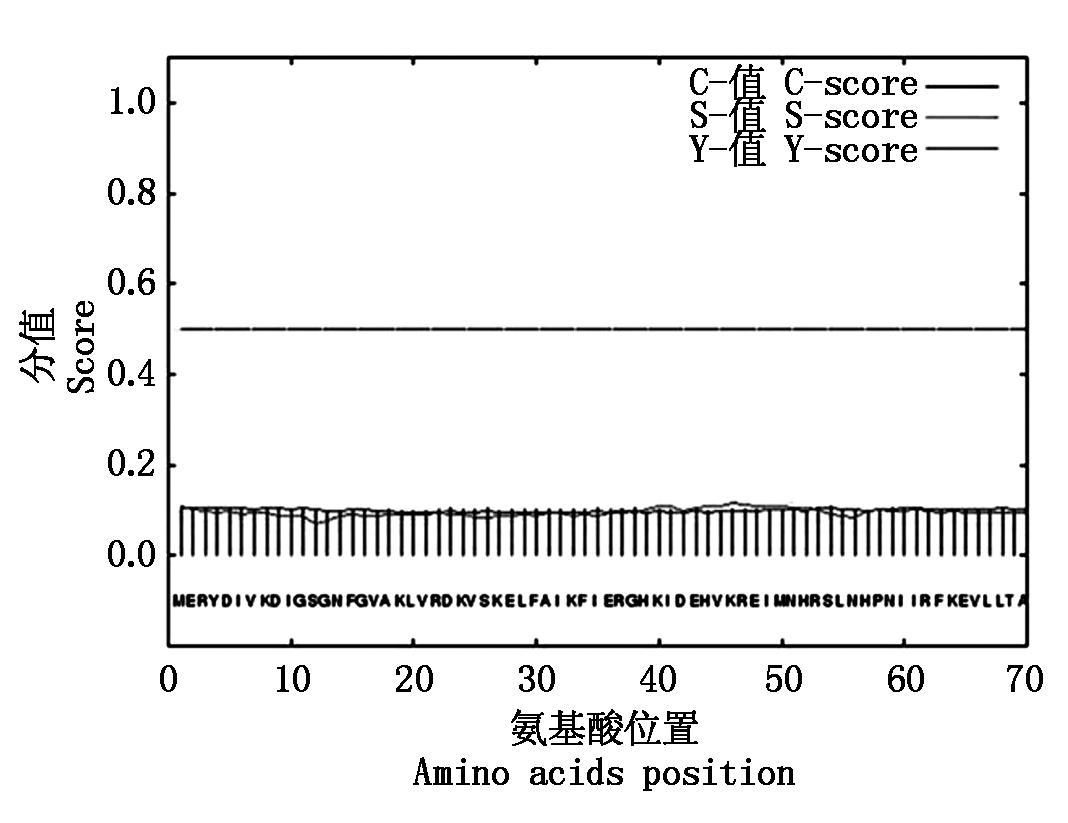
大部分前体蛋白需要经过翻译后修饰才能成为有功能的蛋白质,而蛋白质翻译后修饰中最重要、最常见的方式之一是蛋白质的磷酸化。磷酸化位点预测结果显示(图7),BjSnRK2C蛋白激酶可能有4个酪氨酸(Tyr)、5个苏氨酸(Thr)、21个丝氨酸(Ser)成为磷酸化位点。拟南芥AtSnRK2.8蛋白激酶可能有5个酪氨酸(Tyr)、8个苏氨酸(Thr)、16个丝氨酸(Ser)成为磷酸化位点。二者可能的磷酸化位点的数目和位置极为相似,它们可能具有类似的磷酸化过程。
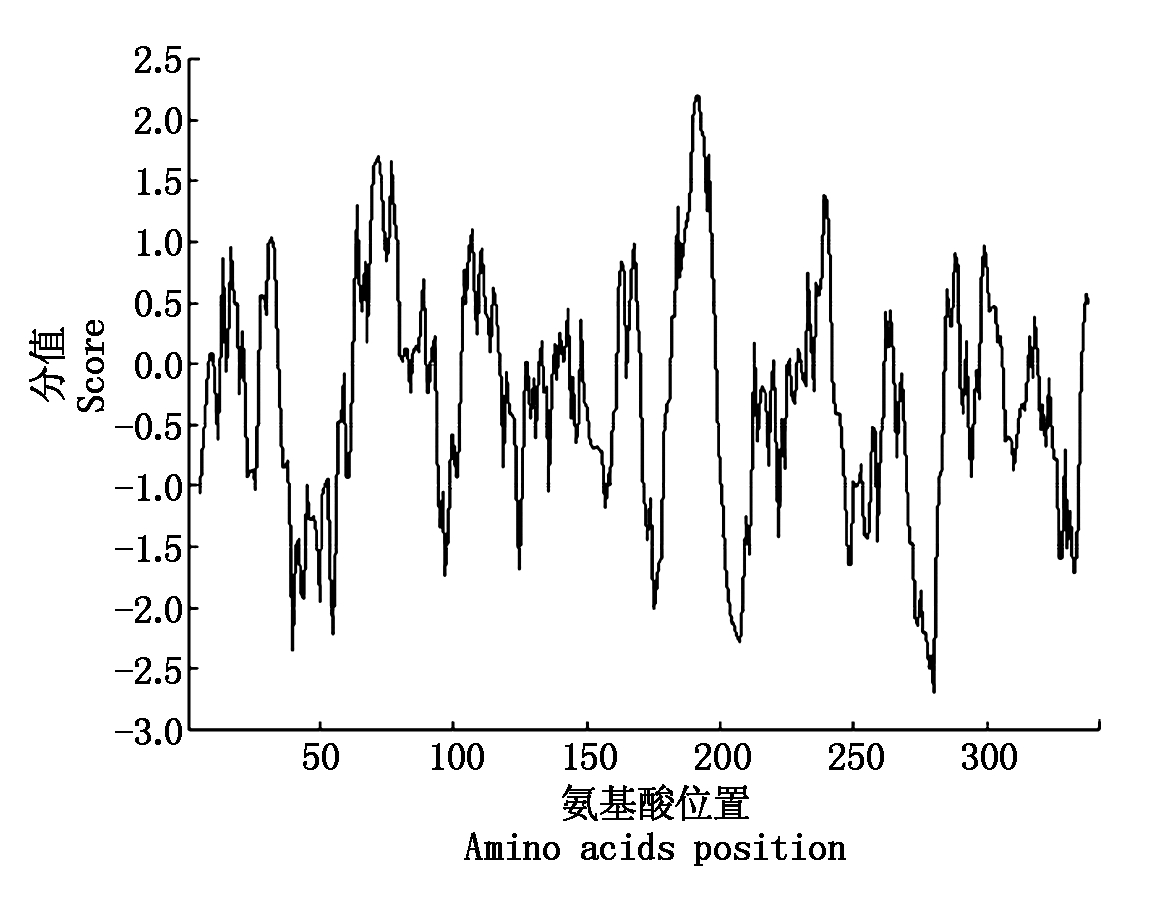
2.9 BjSnRK2C蛋白激酶的亲水性和疏水性分析
氨基酸的亲水性和疏水性是蛋白质固有的特征,氨基酸的亲水性和疏水性之间的平衡决定着蛋白质的结构特征,而氨基酸内的疏水作用在很大程度上决定着蛋白质结构的稳定性。所以,分析和预测蛋白质的亲水性和疏水性可以为蛋白质结构分析和功能预测提供重要的理论依据。氨基酸的亲水性和疏水性分析结果显示(图8),BjSnRK2C蛋白激酶最大疏水性氨基酸值为2.189,最小疏水性氨基酸值为-2.689,存在多个明显的亲水区域,而且组成BjSnRK2C蛋白激酶的大部分氨基酸属于亲水性氨基酸。据此推断,雪菜BjSnRK2C蛋白激酶属于亲水性蛋白。
2.10 BjSnRK2C蛋白激酶的二级结构预测
蛋白质二级结构预测结果显示(图9),雪菜BjSnRK2C蛋白激酶的二级结构具有130个α-螺旋(Alpha helix)、126个无规则卷曲(Random coil)、59个延伸链(Extended strand)和27个β-转角(Beta turn)。与拟南芥AtSnRK2.8蛋白激酶的139个α-螺旋、111个无规则卷曲、58个延伸链和35个β-转角极为相似,暗示雪菜BjSnRK2C蛋白激酶可能与拟南芥AtSnRK2.8蛋白激酶具有相似的功能。
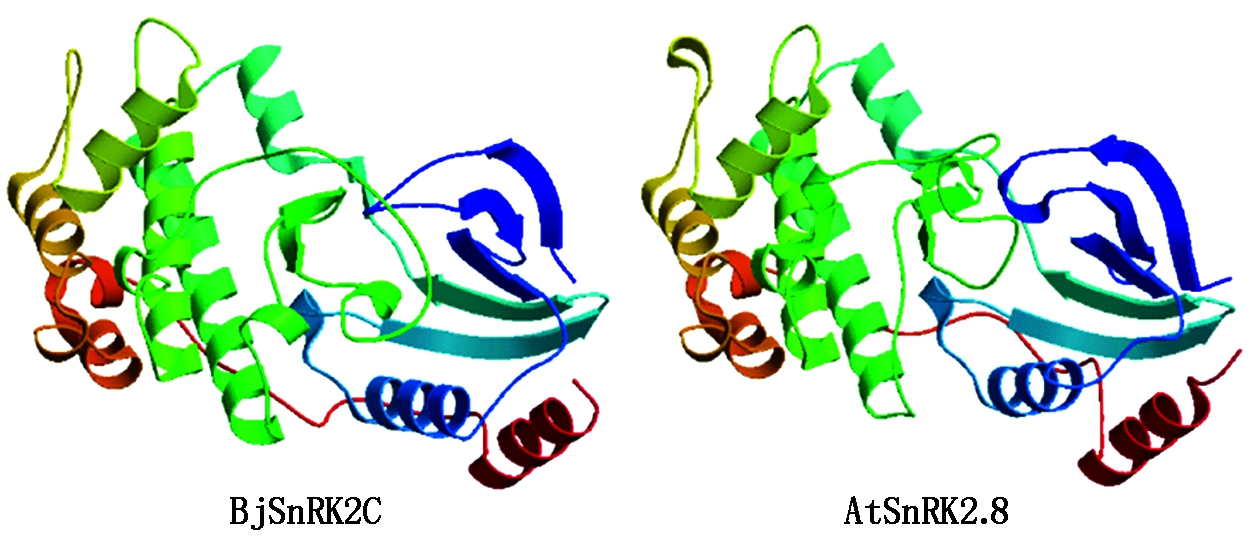
2.11 BjSnRK2C蛋白激酶的三级结构预测
三级结构预测结果如图10所示,雪菜BjSnRK2C蛋白激酶主要由α-螺旋、无规则卷曲、延伸链和β-转角构成,与二级结构预测结果基本一致。此外,雪菜BjSnRK2C蛋白激酶的三级结构与拟南芥AtSnRK2.8蛋白激酶极为相似,推测它们具有类似的功能。
3 结论与讨论
SnRK2蛋白激酶包含1个由27个氨基酸组成的丝氨酸/苏氨酸蛋白激酶激活环,其序列为DF GYSKSSVLHSQPKSTVGTPAYIAPE。对拟南芥AtSnRK2.6(NP_567945.1)和水稻SAPK2(BAD17998.1)蛋白激酶研究结果表明,丝氨酸/苏氨酸蛋白激酶激活环上的3个丝氨酸/苏氨酸残基(下划线处)对SnRK2蛋白激酶的酶活起到关键的作用[18-19]。用天冬氨酸替代这3个氨基酸中的前2个后,AtSnRK2.6蛋白激酶活性远远低于对照,只能使底物蛋白轻微磷酸化。同样,水稻SAPK2丝氨酸/苏氨酸蛋白激酶激活环上的3个丝氨酸/苏氨酸残基全部、两两或逐一被天冬氨酸取代后,无法检测到SAPK2蛋白激酶活性。多序列比对结果显示,雪菜BjSnRK2C蛋白激酶激活环序列与拟南芥AtSnRK2.8的该序列完全一致,推测雪菜BjSnRK2C蛋白激酶与拟南芥AtSnRK2.6蛋白激酶具有相似的磷酸化作用。
多序列比对结果显示,不同SnRK2蛋白激酶成员在N末端高度保守,而在C末端高度特异。SnRK2家族成员在C末端包含一个酸性补丁(D/E),一般富含天冬氨酸(D)或谷氨酸(E),依据这一特点将SnRK2成员分为SnRK2a(酸性补丁中富含D)和 SnRK2b(酸性补丁中富含E)2个组[20]。序列分析显示,雪菜BjSnRK2C蛋白激酶的C末端也包含1个谷氨酸(D)的酸性补丁,因此,BjSnRK2C蛋白激酶属于SnRK2a家族中的一员。基于C末端结构组成和系统发生,又可将SnRK2家族成员分为Group Ⅰ、GroupⅡ和Group Ⅲ共3个组[21]。进化树构建结果表明,雪菜BjSnRK2C蛋白激酶被归为Group Ⅱ成员,这与Group Ⅱ和Group Ⅲ属于SnRK2a,而Group Ⅰ属于SnRK2b的研究结果是一致的。SnRK2蛋白激酶家族成员的C末端主要包含2个结构域:一个是Domain Ⅰ,该区域是SnRK2蛋白激酶响应非生物胁迫的必要元件,Group Ⅰ、GroupⅡ和Group Ⅲ这3组成员都具备这一结构域;另一个是Domain Ⅱ,为Group Ⅲ成员所特有的一段序列,是响应ABA应答不可缺少的元件,在与PP2C、ABI1等磷酸酶的相互作用以及响应ABA应答等方面发挥着重要的作用[22-23]。除SnRK2.9外,拟南芥其余9个SnRK2成员都响应高渗胁迫,同时研究发现Group Ⅰ成员不响应ABA胁迫,Group Ⅱ成员对ABA胁迫不响应或具有微弱的响应,Group Ⅲ成员强烈响应ABA胁迫[24]。10个水稻SnRK2成员也都响应高渗胁迫,其中Group Ⅰ和Group Ⅱ中的3个成员(SAPK8、SAPK9和SAPK10)也响应ABA胁迫[19]。这些研究结果与SnRK2家族成员的高度保守的N端催化域和特异的C端激酶调控域是相关的,SnRK2家族成员在功能上的相似性与差异性、响应胁迫的相似性与差异性等都可能归因于SnRK2s序列的一致性和多样性。与拟南芥AtSnRK2.8蛋白激酶的研究结果极其相似,雪菜BjSnRK2C蛋白激酶的C末端仅含1个由34个氨基酸组成的Domain Ⅰ,缺少1个由44个氨基酸组成的Domain Ⅱ,暗示雪菜BjSnRK2C基因与拟南芥AtSnRK2.8有类似的功能,在参与非生物胁迫过程中很可能也是不依赖ABA调控通路的。
作为植物体内与胁迫相关的重要植物激素,ABA在植物许多生理生化的过程中起到尤为重要的作用。SnRK2蛋白激酶可通过SnRK2-AREB/ABF调控通路引发一系列ABA应答基因的转录。ABA应答基因的启动子上含有多个保守的响应ABA的顺式作用元件ABRE,SnRK2蛋白激酶可以磷酸化转录因子如ABFs/AREBs,活性被磷酸化调节的反式因子随后与ABRE顺式作用元件结合,启动ABA响应基因的表达[9-10]。小麦中的TaABF可以被PKABA1磷酸化[25],TRAB1可以被水稻的SAPK10、SAPK9及SAPK8蛋白激酶磷酸化[26]。拟南芥蛋白激酶SnRK1.1能够磷酸化开花调控途径中的转录因子FD[27],雪菜BjSnRK2C蛋白激酶能否磷酸化ABFs/AREBs还不清楚,需要进一步研究。
利用RT-PCR和SON-PCR技术从雪菜中克隆了1个SnRK2基因,命名为BjSnRK2C,经过生物信息学分析鉴定,它具有SnRK2蛋白激酶家族的特征,为雪菜SnRK2基因家族中的成员。
参考文献:
[1] Duan K, Sun G, Zhang Y, et al. Impact of air pollution induced climate change on water availability and ecosystem productivity in the conterminous United States[J]. Climatic Change, 2017, 140(2): 259-272.
[2] Man L L, Xiang D J, Wang L A, et al. Stress-responsive gene RsICE1 fromRaphanus sativus increases cold tolerance in rice[J]. Protoplasma, 2017, 254(2): 945-956.
[3] Ning W F, Zhai H, Yu J Q, et al. Overexpression ofGlycine soja WRKY20 enhances drought tolerance and improves plant yields under drought stress in transgenic soybean[J]. Molecular Breeding, 2017, 37(2): 19.
[4] Ye Y Y, Ding Y F, Jiang Q, et al. The role of receptor-like protein kinases (RLKs) in abiotic stress response in plants[J]. Plant Cell Reports, 2017, 36(2): 235-242.
[5] Karakas B, Ozias-Akins P, Stushnoff C, et al. Salinity and drought tolerance of mannitol-accumulating transgenic tobacco[J]. Plant Cell and Environment, 1997, 20(5): 609-616.
[6] Forni C, Duca D, Glick B R. Mechanisms of plant response to salt and drought stress and their alteration by rhizobacteria[J]. Plant and Soil, 2017, 410(1/2): 335-356.
[7] 张胜博, 刘景辉, 李立军, 等. 燕麦AsNAC1耐盐基因的克隆及初步验证[J]. 北方农业学报, 2016, 44(5): 1-7.
[8] Wang P C, Xue L, Batelli G, et al. Quantitative phosphoproteomics identifies SnRK2 protein kinase substrates and reveals the effectors of abscisic acid action[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(27): 11205-11210.
[9] Fujita Y, Yoshida T, Yamaguchi-Shinozaki K. Pivotal role of the AREB/ABF-SnRK2 pathway in ABRE-mediated transcription in response to osmotic stress in plants[J]. Physiologia Plantarum, 2013, 147(1, SI): 15-27.
[10] Yoshida T, Fujita Y, Maruyama K, et al. FourArabidopsis AREB/ABF transcription factors function predominantly in gene expression downstream of SnRK2 kinases in abscisic acid signalling in response to osmotic stress[J]. Plant Cell and Environment, 2015, 38(1): 35-49.
[11] Hrabak E M, Chan C W, Gribskov M, et al. TheArabidopsis CDPK-SnRK superfamily of protein kinases[J]. Plant Physiology, 2003, 132(2): 666-680.
[12] 高 天, 铁 英, 王 妍, 等. 拟南芥低钙诱导cDNA文库的构建[J]. 北方农业学报, 2016, 44(5): 8-14.
[13] Xu M R, Huang L Y, Zhang F, et al. Genome-wide phylogenetic analysis of Stress-activated protein kinase genes in rice (OsSAPKs) and expression profiling in response toXanthomonas oryzae pv.oryzicola infection[J]. Plant Molecular Biology Reporter, 2013, 31(4): 877-885.
[14] Mao X, Zhang H Y, Tian S J, et al. TaSnRK2.4, an SNF1-type serine/threonine protein kinase of wheat (Triticum aestivum L.), confers enhanced multistress tolerance inArabidopsis[J]. Journal of Experimental Botany, 2010, 61(3): 683-696.
[15] Diédhiou C J, Popova O V, Dietz K. The SNF1-type serine-threonine protein kinase SAPK4 regulates stress-responsive gene expression in rice[J]. BMC Plant Biology, 2008, 8(1): 49.
[16] Umezawa T, Yoshida R, Maruyama K, et al. SRK2C,a SNF1-related protein kinase 2,improves drought tolerance by controlling stress-responsive gene expression inArabidopsis thaliana[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(49): 17306-17311.
[17] Antal Z, Rascle C, Fevre M, et al. Single oligonucleotide nested PCR: a rapid method for the isolation of genes and their flanking regions from expressed sequence tags[J]. Current Genetics, 2004, 46(4): 240-246.
[18] Belin C, De Franco P O, Bourbousse C, et al. Identification of features regulating OST1 kinase activity and OST1 function in guard cells[J]. Plant Physiology, 2006, 141(4): 1316-1327.
[19] Kobayashi Y, Yamamoto S, Minami H, et al. Differential activation of the rice sucrose nonfermenting1-related protein kinase 2 family by hyperosmotic stress and abscisic acid[J]. The Plant Cell, 2004, 16(5): 1163-1177.
[20] Yoshida R, Hobo T, Ichimura K, et al. ABA-activated SnRK2 protein kinase is required for dehydration stress signaling inArabidopsis[J]. Plant and Cell Physiology, 2002, 43(12): 1473-1483.
[21] Kulik A, Wawer I, Krzywińska E, et al. SnRK2 protein kinases-key regulators of plant response to abiotic stresses[J]. OMICS-A Journal of Integrative Biology, 2011, 15(12): 859-872.
[22] Yoshida R, Umezawa T, Mizoguchi T, et al. The regulatory domain of SRK2E/OST1/SnRK2.6 interacts with ABI1 and integrates abscisic acid (ABA) and osmotic stress signals controlling stomatal closure inArabidopsis[J]. Journal of Biological Chemistry, 2006, 281(8): 5310-5318.
[23] 王永波, 高世庆, 唐益苗, 等. 植物蔗糖非发酵-1相关蛋白激酶家族研究进展[J]. 生物技术通报, 2010(11): 7-18.
[24] Boudsocq M, Barbier-Brygoo H, Lauriere C. Identification of nine sucrose nonfermenting 1-related protein kinases 2 activated by hyperosmotic and saline stresses inArabidopsis thaliana[J]. Journal of Biological Chemistry, 2004, 279(40): 41758-41766.
[25] Johnson R R, Wagner R L, Verhey S D, et al. The abscisic acid-responsive kinase PKABA1 interacts with a seed-specific abscisic acid response element-binding factor,TaABF,and phosphorylates TaABF peptide sequences[J]. Plant Physiology, 2002, 130(2): 837-846.
[26] Kobayashi Y, Murata M, Minami H, et al. Abscisic acid-activated SNRK2 protein kinases function in the gene-regulation pathway of ABA signal transduction by phosphorylating ABA response element-binding factors[J]. Plant Journal, 2005, 44(6): 939-949.
[27] 袁 敏, 葛伟娜, 王 莉, 等. 拟南芥蛋白激酶SnRK1.1对转录因子FD的磷酸化分析[J]. 华北农学报, 2017, 32(4): 37-41.
Cloning and Bioinformatics Analysis of BjSnRK2C Gene from Potherb Mustard
ZHANG Chunlan1, MAN Lili1, XIANG Dianjun2, LI Xuxin2, BAO Wurina2, LIU Peng2
(1.College of Life Science, Inner Mongolia University for Nationalities, Tongliao 028042, China; 2.College of Agriculture, Inner Mongolia University for Nationalities, Tongliao 028042, China)
Abstract: TheSnRK2 family genes, which are key regulators of plant resistance to abiotic stresses in the ABA pathway, encode a class of serine/threonine protein kinases in plants. Based on the conservative cDNA sequences of SnRK2 members, a full-length cDNA sequence of SnRK2, named asBjSnRK2C, was cloned fromBrassica juncea by RT-PCR and SON-PCR methods. The sequencing results showed that the length of full-length cDNA (GenBank accession No. MF983711)was 1 380 bp containing a 3′-UTR of 137 bp, a 5′-UTR of 214 bp, and a complete open reading frame of 1 029 bp with 342 amino acids. The putative molecular weight and theory isoelectric point of the BjSnRK2C protein were 38.2 ku and 5.61, respectively. The BjSnRK2C was a hydrophilic protein with 30 phosphorylation sites, contained two significant transmembrane domains, contained no signal peptide, and might played a physiological role in the cytoplasm. The secondary structure of the BjSnRK2C protein contained 130 α-helixes, 126 random coils, 59 extended strands, and 27 β-turns. The BjSnRK2C protein contained a serine/threonine enzyme activity domain and an ATP binding site, and was at the same evolutionary branch with AtSnRK2.8 fromArabidopsis. The three-dimensional structure of the BjSnRK2C protein kinase was very similar to that of AtSnRK2.8 protein kinase fromArabidopsis, suggesting that they had similar functions. The C terminal of the BjSnRK2C protein kinase contained only the domain Ⅰ, and lacked the domain Ⅱ, suggesting that theBjSnRK2C gene was probably independent of ABA regulatory pathway in abiotic stress process, which will lay a theoretical foundation for further studying theBjSnRK2C gene function.
Key words: Brassica juncea;BjSnRK2C gene;Cloning;Bioinformatics
中图分类号:S649
文献标识码:A
文章编号:1000-7091(2018)06-0024-09
doi:10.7668/hbnxb.2018.06.004
收稿日期:2018-08-08
基金项目:国家自然基金项目(31860389);内蒙古自治区自然科学基金项目(2018MS03057);内蒙古民族大学博士科研启动基金项目(BS404);内蒙古自治区饲用作物工程技术研究中心开放课题(MDK2017004;MDK2018033)
作者简介:张春兰(1972-),女,内蒙古赤峰人,实验师,硕士,主要从事作物生物技术研究。张春兰、满丽莉为同等贡献作者。
通讯作者:
向殿军(1978-),男,黑龙江望奎人,副教授,博士,主要从事植物非生物胁迫研究。
刘 鹏(1973-),男,内蒙古赤峰人,教授,博士,主要从事作物遗传育种研究。