小麦SBP基因家族生物信息学分析
吕亮杰,陈希勇,胡梦芸,刘玉平,孙丽静,马 乐,李 辉
(河北省农林科学院 粮油作物研究所,河北省作物遗传育种实验室,河北 石家庄 050035)
摘要:SBP基因家族是植物所特有的一类重要转录因子,含79个氨基酸残基保守结构域,主要参与植物生长发育、生理生化过程。为进一步探讨小麦SBP基因家族的基因功能,通过比对小麦最新基因组数据,结合公布的Chinese Spring的基因组数据,采用生物信息学方法对其基因结构、染色体分布、蛋白保守结构域、系统进化树及表达谱进行分析。结果获得了50个SBP基因,命名为TaSBPs,根据染色体编号排列为TaSBP1~TaSBP50。结果表明,50个小麦TaSBP基因分布于除4B、4D染色体外的其余19条染色体上,编码192~1 104个氨基酸,基因外显子数量2~11个变化不等;串联重复和片段复制是小麦SBP家族基因扩张的主要模式;7种作物SBP基因的系统进化树可分为4个类别,同一类之间的结构较为相似;小麦50个TaSBP基因家族含有10个motif,推测小麦TaSBP基因家族应都含有motif1、motif2、motif4。50个TaSBP基因都在13个组织器官中检测到转录本,不同组织器官中TaSBP基因的表达存在明显的差异。
关键词:小麦;SBP;生物信息学;进化树;表达谱
SBP(Squamosa promoter binding protein)基因家族是植物所特有的一类重要转录因子,含有一段保守的核苷酸序列(SBP-box),编码的蛋白质序列(SBP-domain)含79个氨基酸残基,并具有高度保守性,最初在金鱼草(Antirrhinum majus L.)植物中发现[1]。SBP基因家族的蛋白具有锌指结构,能识别并结合MAD-box基因SQUAM-OSA (SQUA)启动子,参与植物生长发育、抗逆以及多种生理生化过程。通过与启动子区域的顺式元件相互作用,转录因子能够激活或者抑制下游基因表达。1996年首次在金鱼草中发现SBP基因[2],后续在玉米[3-4]、拟南芥[5-6]、大豆[7]、番茄[8]、水稻[9-10]、土豆[11]和葡萄[12]等植物物种中也发现该基因家族成员[13-14]。SBP作为绿色植物特有的转录因子,在植物花和果实的发育[11]、赤霉素介导的发育[15]以及铜应答过程[16]的调控中发挥重要作用。Cardon等[17]在拟南芥的花和叶片中发现,SBP基因(SPL3)高度表达。Gandikota等[18]发现,SPL8基因参与拟南芥花粉发育的调控,SPL3、SPL4和SPL5中含有mi R156的调控位点。Moreno等[19]研究发现,SBP转录因子Liguleless1(LG1)对玉米舌叶和叶耳的发育有较大影响,LG1基因的缺失导致不能形成舌叶和叶耳。Lännenpää等[20]研究发现,BpSPL1基因特异结合BpMADS5启动子参与白桦树的花发育过程。曹雪等[21]发现葡萄的SBP基因(SPL9、SPL10)在不同组织的表达存在消长关系。Manning等[11]发现在番茄植株生发育长过程中SBP基因(LeSPL-CNR)是控制番茄果实成熟的关键基因。
随着模式植物拟南芥、水稻、小麦等重要农作物基因组数据的释放,通过生物信息学方法剖析基因家族序列特征和进化关系成为研究热门[22-23]。小麦是我国重要的粮食作物之一,小麦的产量和品质严重影响了我国小麦的发展。因此,研究小麦SBP家族对于研究小麦的生长发育具有重要意义。小麦基因组是由3个亚基因组整合在一起形成的异源六倍体(AABBDD),其基因组大小约为17 Gb,重复序列达85%。高质量的小麦基因组序列已释放(http://www.wheat genome.org/)[24],小麦A基因组供体乌拉尔图小麦(Triticum urartu)和D基因组供体粗山羊草(Aegliops tauschii)的基因组测序工作也已由中国完成[25-27],这为筛选小麦生长发育基因及其进化过程奠定了基础。目前,小麦中还未见关于SBP基因家族生物信息学和表达谱分析的报道,因此,有必要利用最新公布的小麦基因组数据对SBP基因家族进行系统研究分析。
本研究利用最新的小麦基因组数据通过生物信息学方法对小麦SBP基因进行筛选,并对其基因结构、蛋白结构、染色体分布、分子进化、串联重复片段及表达谱进行分析,旨在为进一步探讨小麦SBP基因的功能奠定基础,为利用分子生物学技术改良小麦性状提供理论依据。
1 材料和方法
1.1 小麦TaSBP基因家族的鉴定
从EnsemblPlants数据库(http://plants.ensembl.org/index.html/)下载小麦的基因组序列、基因注释和蛋白序列文件,利用NCBI(National center for biotechnology information) (https://www.ncbi.nlm.nih.gov/) Blast已报道的SBP蛋白序列并与Pfam数据库(http://pfam.xfam.org/)进行比对(e-value<1e-5),获得SBP基因家族的Pfam ID及其序列。搜索小麦SBP基因家族的同源蛋白,删除重复序列,利用在线软件Pfam(http://www.sanger.ac.uk/Software/Pfam/search.shtml)进行保守结构域分析验证[28],剔除冗余蛋白。将SBP基因家族成员按照染色体顺序编号命名并映射到不同染色体上,将散在的非染色体序列合并为U染色体。同时Blast各物种的蛋白序列与相应基因家族Pfam进行比对,得到每个物种SBP基因家族序列和蛋白序列。借助ProtParam(https://web.expasy.org/protparam/)对SBP基因家族蛋白进行分子量、等电点、氨基酸信息预测[29]。
1.2 小麦TaSBP基因家族结构分析
根据EnsemblPlants数据库中的DNA数据库检索小麦SBP基因的内含子、外显子、染色体位置等信息,利用GSDS 2. 0(Gene Structure Display Server)(http://gsds. cbi.pku.edu.cn /)在线绘制SBP基因的内含子和外显子组成及基因家族进化树[30]。利用Inparanoid分析小麦的同源蛋白(Orthologous groups,OG),使用circos基于基因注释信息对OG关系进行作图,删除OG聚类过程中没有同源关系的基因,使得同源基因在图片中显示。
1.3 小麦TaSBP基因家族的分子进化树构建
从EnsemblPlants数据库下载大麦(Hordeum vulgare)、拟南芥(Arabidopsis thaliana)、二穗短柄草(Brachypodium distachyum)、水稻(Oryza sativa)、玉米(Zea mays)、高粱(Sorghum bicolor)基因组和蛋白序列数据;利用 MUSCLE对小麦、大麦、短柄草、水稻、玉米、高粱、拟南芥SBP蛋白序列进行多重比对,将结果输入MEGA 7.0[31],采用邻接法(Neighbor-joining,NJ)分别构建小麦SBP基因家族进化树及小麦、大麦、短柄草、水稻、玉米、高粱、拟南芥的系统进化树,其中,校验参数(Bootstrap)设置为1 000,其余均为默认值[22]。借助FigTree绘制小麦SBP基因家族进化树及小麦、大麦、短柄草、水稻、玉米、高粱、拟南芥的系统进化树。
1.4 小麦TaSBP基因家族motif结构和三级结构预测分析
基于The MEME suite的在线工具MEME(http://meme-suite.org/tools/meme)对小麦SBP基因家族的 motif序列进行分析;小麦的SBP基因家族的蛋白质空间模型利用在线软件ExPaSy提供的SWISS-MODEL(https://swissmodel.expasy.org/interactive)进行三维结构同源建模。
1.5 小麦TaSBP基因的表达谱分析
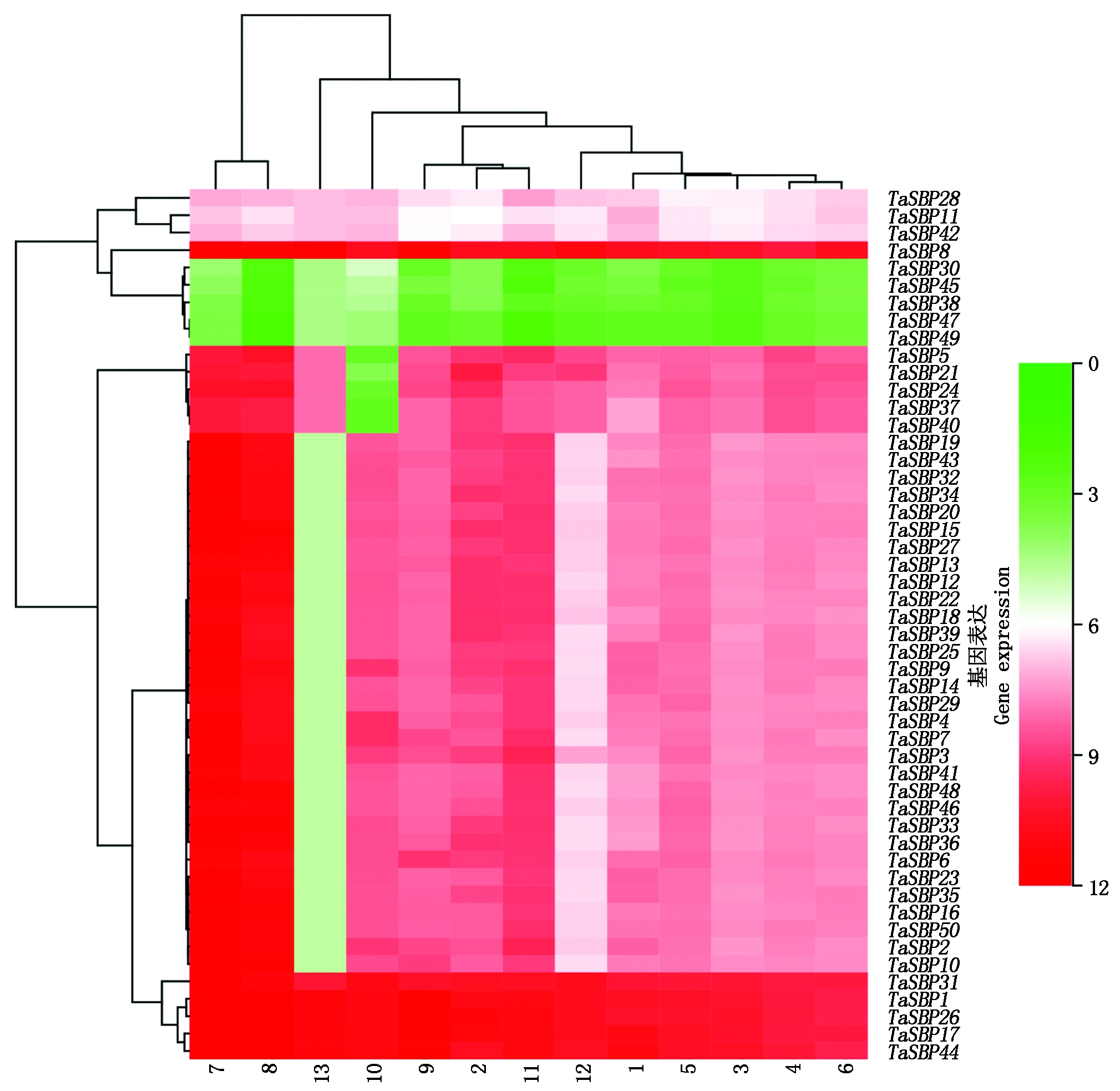
利用已公布的小麦RNA-seq数据,检索小麦SBP基因的表达谱(http://www.plexdb.org/modules/tools/plexdb_blast.php)。数据库中提供了Chinese Spring的13个不同组织器官的表达数据,包括胚芽鞘、胚芽根、胚乳、根、花冠、叶、幼穗、花苞、雌蕊、花药、3~5 DAP颖果、22 DAP胚、22 DAP胚乳,FPKM(Fragments Per Kilobase of transcript per Millionmapped reads)值作为SBP基因的表达谱数据,用Heatmapper构建基因表达热图(http://www.heatmapper.ca/)。
2 结果与分析
2.1 小麦TaSBP基因家族鉴定及蛋白特性分析
经EMBL-EBI确认,SBP基因家族的Pfam号为PF03110,利用HMM程序搜索得到小麦的SBP基因家族,结合已报道的水稻SBP基因、拟南芥SBP基因和葡萄SBP基因序列在Ensembl Plants数据库中进行BlastP比对,得到71个小麦候选SBP基因;利用Pfam(http: //www.ranger.ac.uk/Soft-ware/Pfam/search.shtml)分析比对的蛋白质序列保守结构域,剔除非典型SBP结构域的冗余蛋白后,获得50个家族成员,按照染色体顺序分别命名为TaSBP1~TaSBP50(表1)。对50个TaSBP基因综合分析发现,这50个小麦SBP基因分布于除4B、4D染色体外的其余19条染色体上,7D、7A、7B上含有的基因数目最多,分别为8,7,5个,其次为6A、6B、6D和2D,均为3个,1A、3A、4A、5A、1B、3B、1D和3D上含有的SBP基因数目最少,仅有1个。序列分析显示,50个TaSBP基因编码长度为192~1 104个氨基酸的蛋白质,其中TaSBP26所含氨基酸数目最多(1 104个),而TaSBP2和TaSBP18最少(192个);蛋白质的相对分子质量为20 117.43~120 626.35 ku,TaSBP26的相对分子质量最大(120 626.35 ku),而TaSBP32最小(20 117.43 ku);等电点为5.73~9.87,TaSBP5预测的等电点最低(5.73),而TaSBP2和TaSBP18的等电点最高(9.87)(表1)。
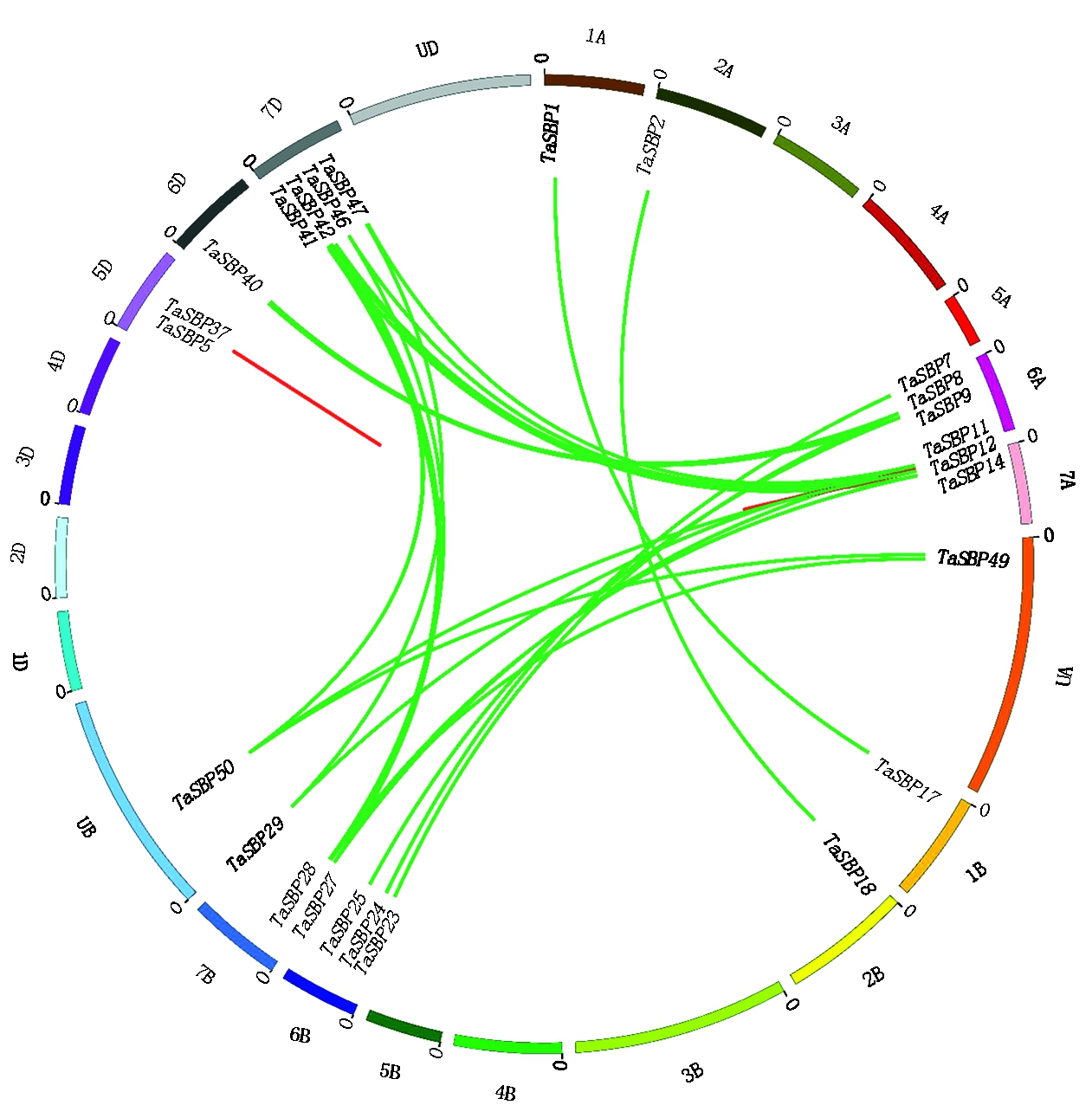
2.2 小麦TaSBP基因家族同源进化分析
片段复制和串联重复是家族基因扩张的主要模式,为了分析小麦SBP家族基因与祖先材料的同源进化关系,采用生物信息学方法将SBP基因定位到不同染色体,并对串联重复片段进行分析,具有同源关系的基因进行相关的连线说明(图1)。从同源关系来看,TaSBP1、TaSBP2、TaSBP7、TaSBP8等26个基因具有同源关系,而TaSBP3、TaSBP4、TaSBP16、TaSBP19等基因与其他染色体上的基因没有同源对应关系。7A染色体上的TaSBP12、TaSBP14基因与7B染色体上的TaSBP27、TaSBP28基因和7D染色体上的TaSBP41、TaSBP42基因串联重复,说明这6个基因具有一定的同源关系。1A染色体的TaSBP1、2A染色体的TaSBP2和6A染色体的TaSBP9仅有一个串联重复,分别对应1B染色体的TaSBP17、2B染色体的TaSBP18和6B的染色体的TaSBP23,说明这3类SBP基因分别在D基因组没有拷贝。
表1 50个小麦TaSBP基因的基本信息
Tab.1 The basic information of 50 wheat TaSBP genes
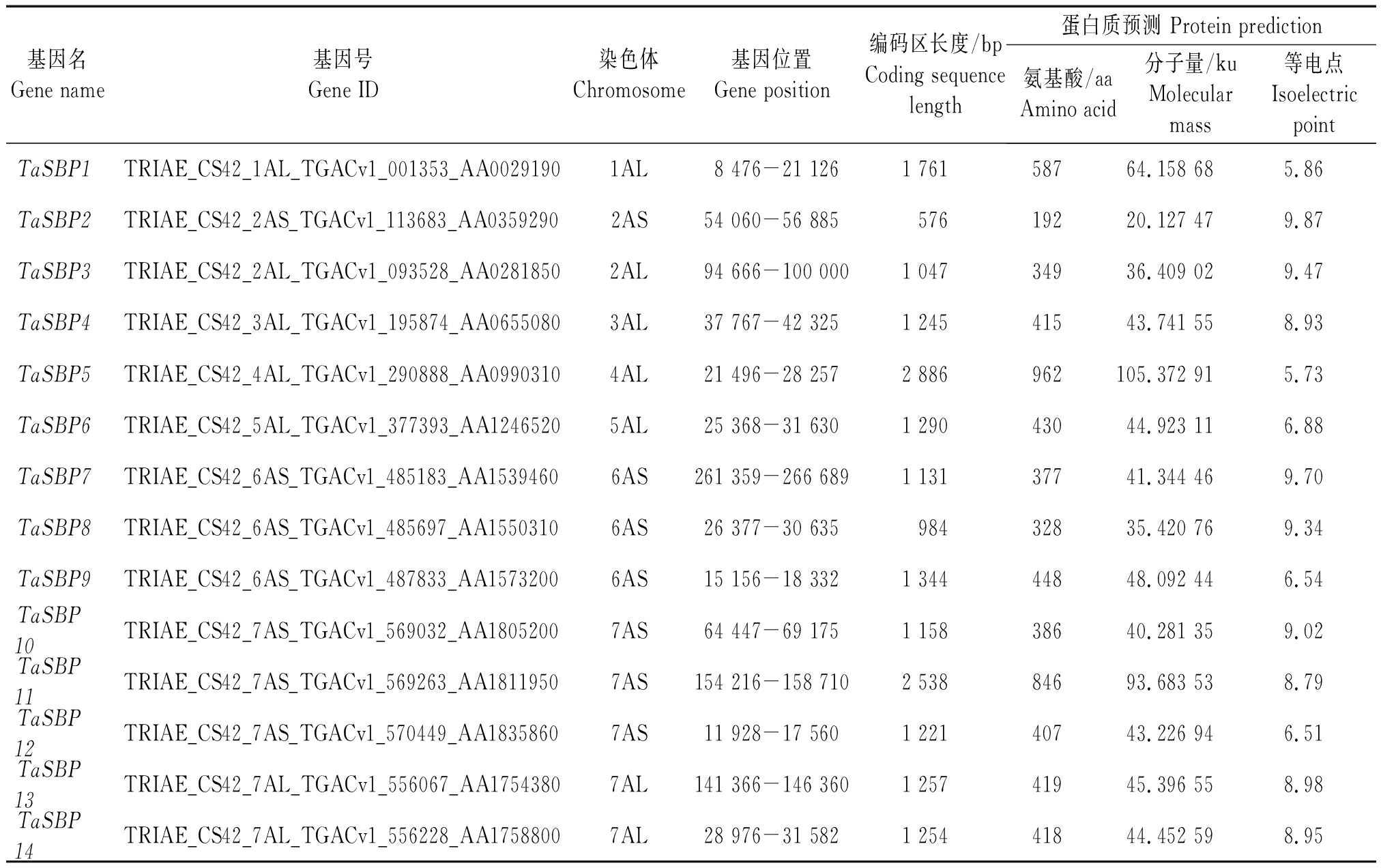
基因名Gene name基因号Gene ID染色体Chromosome基因位置Gene position编码区长度/bpCoding sequence length蛋白质预测 Protein prediction氨基酸/aaAmino acid分子量/kuMolecularmass等电点IsoelectricpointTaSBP1TRIAE_CS42_1AL_TGACv1_001353_AA00291901AL8 476-21 1261 76158764.158 685.86TaSBP2TRIAE_CS42_2AS_TGACv1_113683_AA03592902AS54 060-56 88557619220.127 479.87TaSBP3TRIAE_CS42_2AL_TGACv1_093528_AA02818502AL94 666-100 0001 04734936.409 029.47TaSBP4TRIAE_CS42_3AL_TGACv1_195874_AA06550803AL37 767-42 3251 24541543.741 558.93TaSBP5TRIAE_CS42_4AL_TGACv1_290888_AA09903104AL21 496-28 2572 886962105.372 915.73TaSBP6TRIAE_CS42_5AL_TGACv1_377393_AA12465205AL25 368-31 6301 29043044.923 116.88TaSBP7TRIAE_CS42_6AS_TGACv1_485183_AA15394606AS261 359-266 6891 13137741.344 469.70TaSBP8TRIAE_CS42_6AS_TGACv1_485697_AA15503106AS26 377-30 63598432835.420 769.34TaSBP9TRIAE_CS42_6AS_TGACv1_487833_AA15732006AS15 156-18 3321 34444848.092 446.54TaSBP10TRIAE_CS42_7AS_TGACv1_569032_AA18052007AS64 447-69 1751 15838640.281 359.02TaSBP11TRIAE_CS42_7AS_TGACv1_569263_AA18119507AS154 216-158 7102 53884693.683 538.79TaSBP12TRIAE_CS42_7AS_TGACv1_570449_AA18358607AS11 928-17 5601 22140743.226 946.51TaSBP13TRIAE_CS42_7AL_TGACv1_556067_AA17543807AL141 366-146 3601 25741945.396 558.98TaSBP14TRIAE_CS42_7AL_TGACv1_556228_AA17588007AL28 976-31 5821 25441844.452 598.95
表1(续)
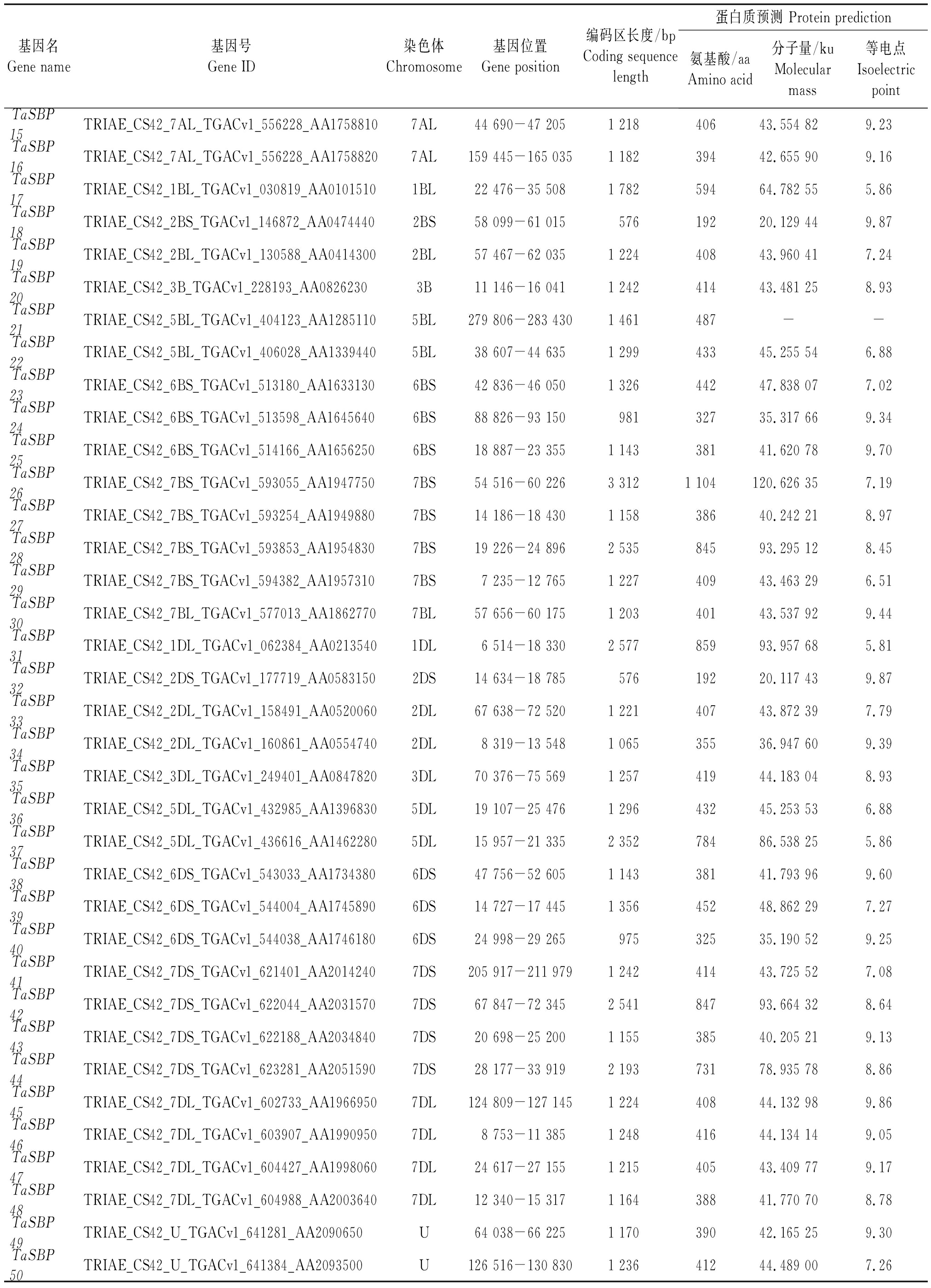
基因名Gene name基因号Gene ID染色体Chromosome基因位置Gene position编码区长度/bpCoding sequence length蛋白质预测 Protein prediction氨基酸/aaAmino acid分子量/kuMolecularmass等电点IsoelectricpointTaSBP15TRIAE_CS42_7AL_TGACv1_556228_AA17588107AL44 690-47 2051 21840643.554 829.23TaSBP16TRIAE_CS42_7AL_TGACv1_556228_AA17588207AL159 445-165 0351 18239442.655 909.16TaSBP17TRIAE_CS42_1BL_TGACv1_030819_AA01015101BL22 476-35 5081 78259464.782 555.86TaSBP18TRIAE_CS42_2BS_TGACv1_146872_AA04744402BS58 099-61 01557619220.129 449.87TaSBP19TRIAE_CS42_2BL_TGACv1_130588_AA04143002BL57 467-62 0351 22440843.960 417.24TaSBP20TRIAE_CS42_3B_TGACv1_228193_AA08262303B11 146-16 0411 24241443.481 258.93TaSBP21TRIAE_CS42_5BL_TGACv1_404123_AA12851105BL279 806-283 4301 461487--TaSBP22TRIAE_CS42_5BL_TGACv1_406028_AA13394405BL38 607-44 6351 29943345.255 546.88TaSBP23TRIAE_CS42_6BS_TGACv1_513180_AA16331306BS42 836-46 0501 32644247.838 077.02TaSBP24TRIAE_CS42_6BS_TGACv1_513598_AA16456406BS88 826-93 15098132735.317 669.34TaSBP25TRIAE_CS42_6BS_TGACv1_514166_AA16562506BS18 887-23 3551 14338141.620 789.70TaSBP26TRIAE_CS42_7BS_TGACv1_593055_AA19477507BS54 516-60 2263 3121 104120.626 357.19TaSBP27TRIAE_CS42_7BS_TGACv1_593254_AA19498807BS14 186-18 4301 15838640.242 218.97TaSBP28TRIAE_CS42_7BS_TGACv1_593853_AA19548307BS19 226-24 8962 53584593.295 128.45TaSBP29TRIAE_CS42_7BS_TGACv1_594382_AA19573107BS7 235-12 7651 22740943.463 296.51TaSBP30TRIAE_CS42_7BL_TGACv1_577013_AA18627707BL57 656-60 1751 20340143.537 929.44TaSBP31TRIAE_CS42_1DL_TGACv1_062384_AA02135401DL6 514-18 3302 57785993.957 685.81TaSBP32TRIAE_CS42_2DS_TGACv1_177719_AA05831502DS14 634-18 78557619220.117 439.87TaSBP33TRIAE_CS42_2DL_TGACv1_158491_AA05200602DL67 638-72 5201 22140743.872 397.79TaSBP34TRIAE_CS42_2DL_TGACv1_160861_AA05547402DL8 319-13 5481 06535536.947 609.39TaSBP35TRIAE_CS42_3DL_TGACv1_249401_AA08478203DL70 376-75 5691 25741944.183 048.93TaSBP36TRIAE_CS42_5DL_TGACv1_432985_AA13968305DL19 107-25 4761 29643245.253 536.88TaSBP37TRIAE_CS42_5DL_TGACv1_436616_AA14622805DL15 957-21 3352 35278486.538 255.86TaSBP38TRIAE_CS42_6DS_TGACv1_543033_AA17343806DS47 756-52 6051 14338141.793 969.60TaSBP39TRIAE_CS42_6DS_TGACv1_544004_AA17458906DS14 727-17 4451 35645248.862 297.27TaSBP40TRIAE_CS42_6DS_TGACv1_544038_AA17461806DS24 998-29 26597532535.190 529.25TaSBP41TRIAE_CS42_7DS_TGACv1_621401_AA20142407DS205 917-211 9791 24241443.725 527.08TaSBP42TRIAE_CS42_7DS_TGACv1_622044_AA20315707DS67 847-72 3452 54184793.664 328.64TaSBP43TRIAE_CS42_7DS_TGACv1_622188_AA20348407DS20 698-25 2001 15538540.205 219.13TaSBP44TRIAE_CS42_7DS_TGACv1_623281_AA20515907DS28 177-33 9192 19373178.935 788.86TaSBP45TRIAE_CS42_7DL_TGACv1_602733_AA19669507DL124 809-127 1451 22440844.132 989.86TaSBP46TRIAE_CS42_7DL_TGACv1_603907_AA19909507DL8 753-11 3851 24841644.134 149.05TaSBP47TRIAE_CS42_7DL_TGACv1_604427_AA19980607DL24 617-27 1551 21540543.409 779.17TaSBP48TRIAE_CS42_7DL_TGACv1_604988_AA20036407DL12 340-15 3171 16438841.770 708.78TaSBP49TRIAE_CS42_U_TGACv1_641281_AA2090650U64 038-66 2251 17039042.165 259.30TaSBP50TRIAE_CS42_U_TGACv1_641384_AA2093500U126 516-130 8301 23641244.489 007.26
2.3 小麦SBP基因家族蛋白进化树及基因结构分析
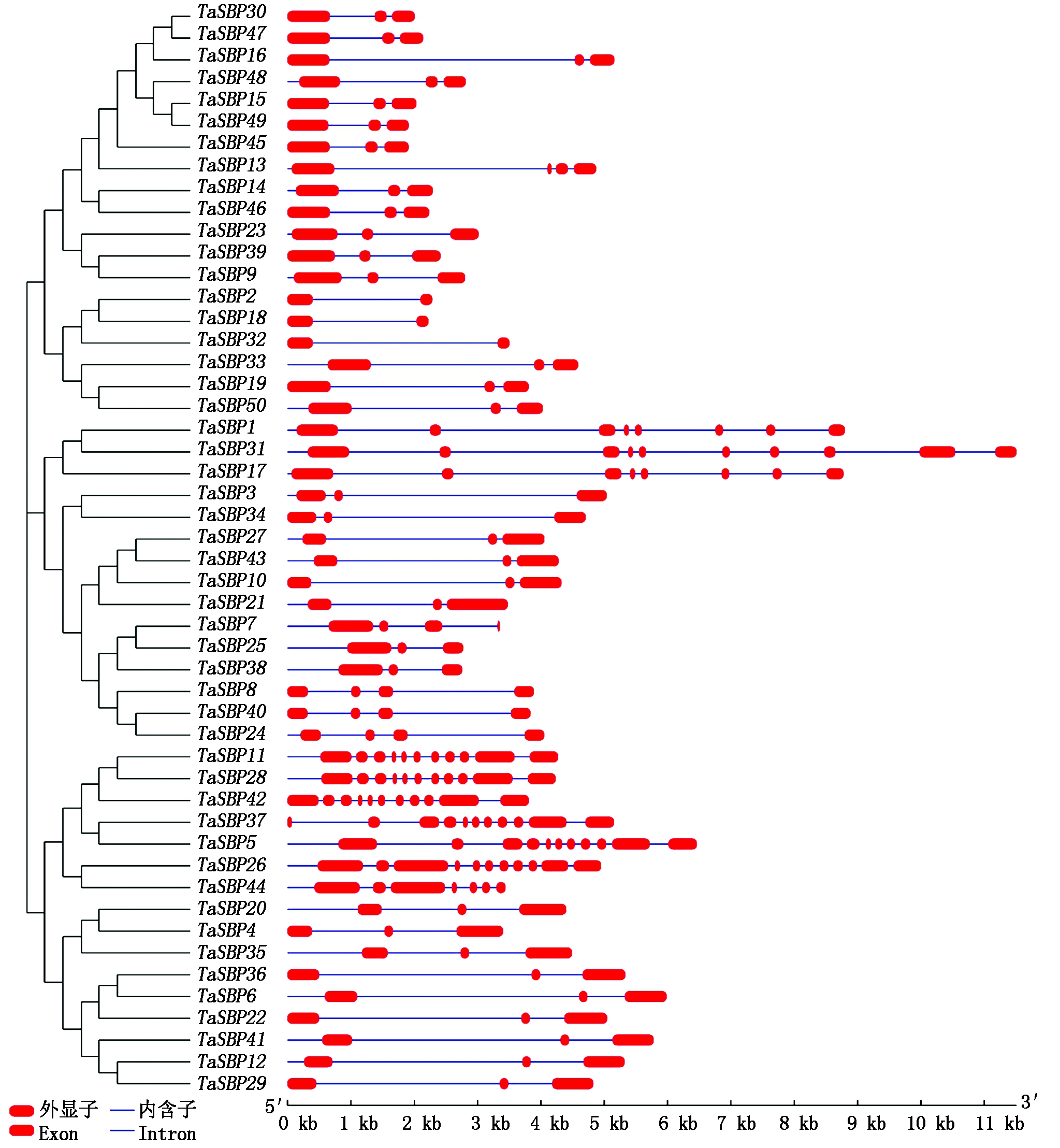
利用本研究中小麦TaSBP全长蛋白序列构建小麦系统进化树,结果显示(图2),在50个编码小麦TaSBP蛋白的基因中检测到了11个旁系同源基因对,即TaSBP30和TaSBP47、TaSBP15和TaSBP49、TaSBP27和TaSBP43、TaSBP7和TaSBP25、TaSBP40和TaSBP24、TaSBP11和TaSBP28、TaSBP37和TaSBP5、TaSBP26和TaSBP44、TaSBP20和TaSBP4、TaSBP36和TaSBP6、TaSBP12和TaSBP29。基因结构分析表明,小麦50个SBP基因外显子数量变化从2个(TaSBP2、TaSBP18、TaSBP32)到11个(TaSBP5、TaSBP11、TaSBP26、TaSBP28、TaSBP37、TaSBP42),小麦TaSBP基因家族蛋白进化树显示,同一类之间的结构较为相似。总体来看,SBP基因家族结构较为简单,多数还有2~3个外显子,这些基因可能产生或分化的时间较晚,推测其功能相对专一。具有相似外显子和内含子的结构,在蛋白进化树上也具有很高的同源性,表明亲缘关系近的基因在进化过程中其外显子、内含子具有一定的相似性。小麦(50个)、大麦(16个)、短柄草(18)、玉米(102个)、水稻(18个)、高粱(39个)及拟南芥(17个) SBP基因的系统进化树分析表明,来自7种作物的260个SBP基因分为4个类别(图3),而本研究中得到的50个小麦TaSBP基因可以归于3个类别(图2)。
2.4 小麦TaSBP基因家族的蛋白结构
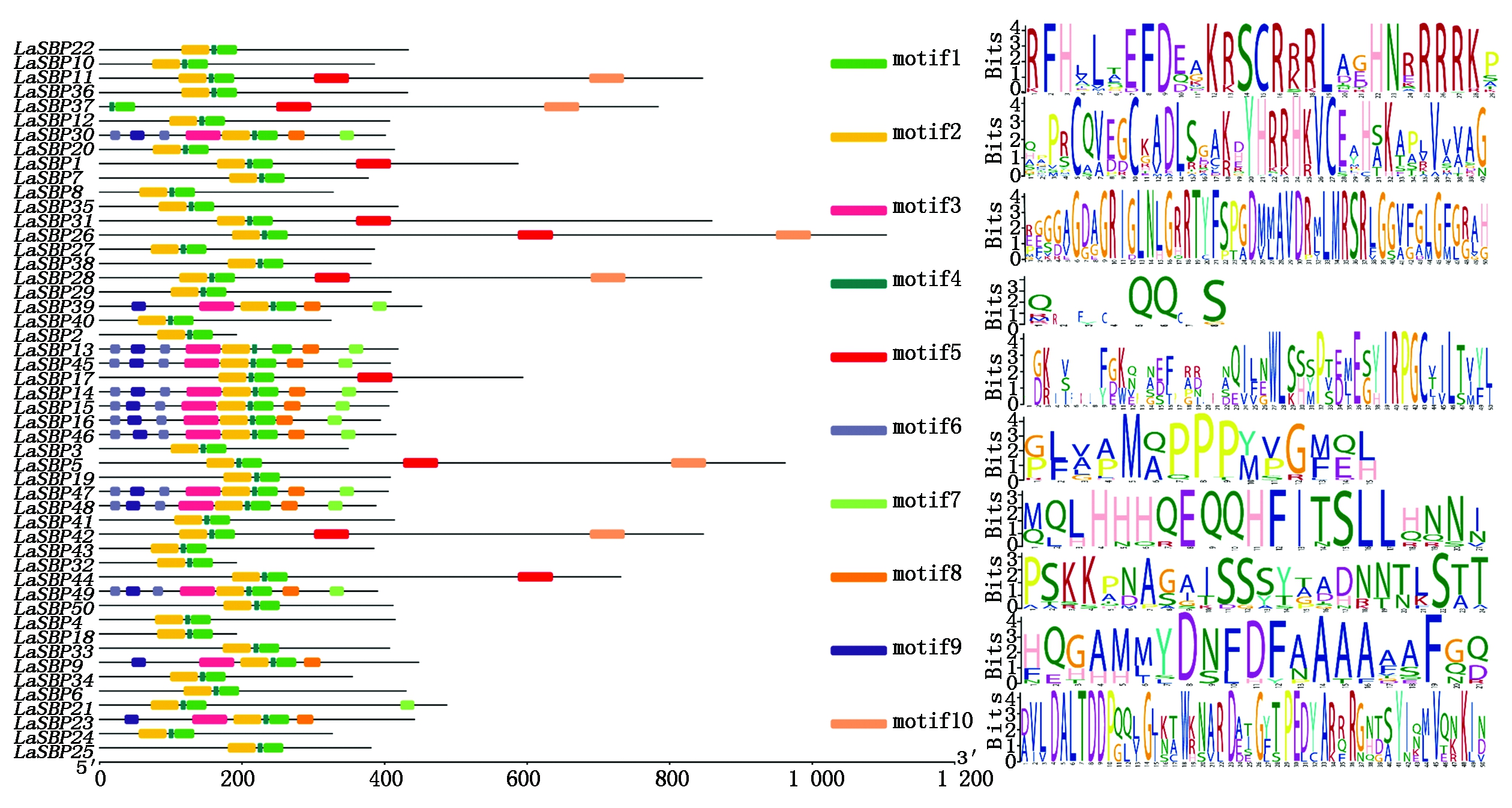
模体(motif)是蛋白质分子结构中介于二级结构与三级结构之间的结构层次,又称超二级结构,是蛋白质分子具有特定功能或作为独立结构域一部分的二级结构聚合体。基因家族所有的或者大多数成员共有的motif极可能是该家族执行重要功能或组成结构不可缺少的部分,如一些具有序列特异性的蛋白的结合位点(转录因子)或者是涉及重要生物过程的RNA起始、终止、剪切等。识别基因家族共同的motif就能刻画该基因家族的特征,从而可以利用这些特征来进行发掘基因家族新成员,所以,motif的识别对基因家族尤为重要。本研究中,小麦50个TaSBP基因家族含有10个motif,其中,TaSBP30、TaSBP13、TaSBP45等10个基因含有最多的motif结构(9个),其次,TaSBP39、TaSBP9、TaSBP23分别含有7,6,6个motif,最少的27个TaSBP基因都含有3个motif;50个TaSBP都含有motif1、motif2、motif4模型,仅TaSBP11、TaSBP37、TaSBP1等11个基因含有motif5,仅TaSBP30、TaSBP39、TaSBP13等11个基因含有motif7和motif9,且motif7和motif9大多数都是一起出现(图4)。分析结果说明,TaSBP基因家族应都含有motif1、motif2、motif4,TaSBP30、TaSBP13、TaSBP45、TaSBP39、TaSBP9、TaSBP23是TaSBP基因家族具有特异性功能的基因。这个预测有助于发现TaSBP基因家族的新成员。
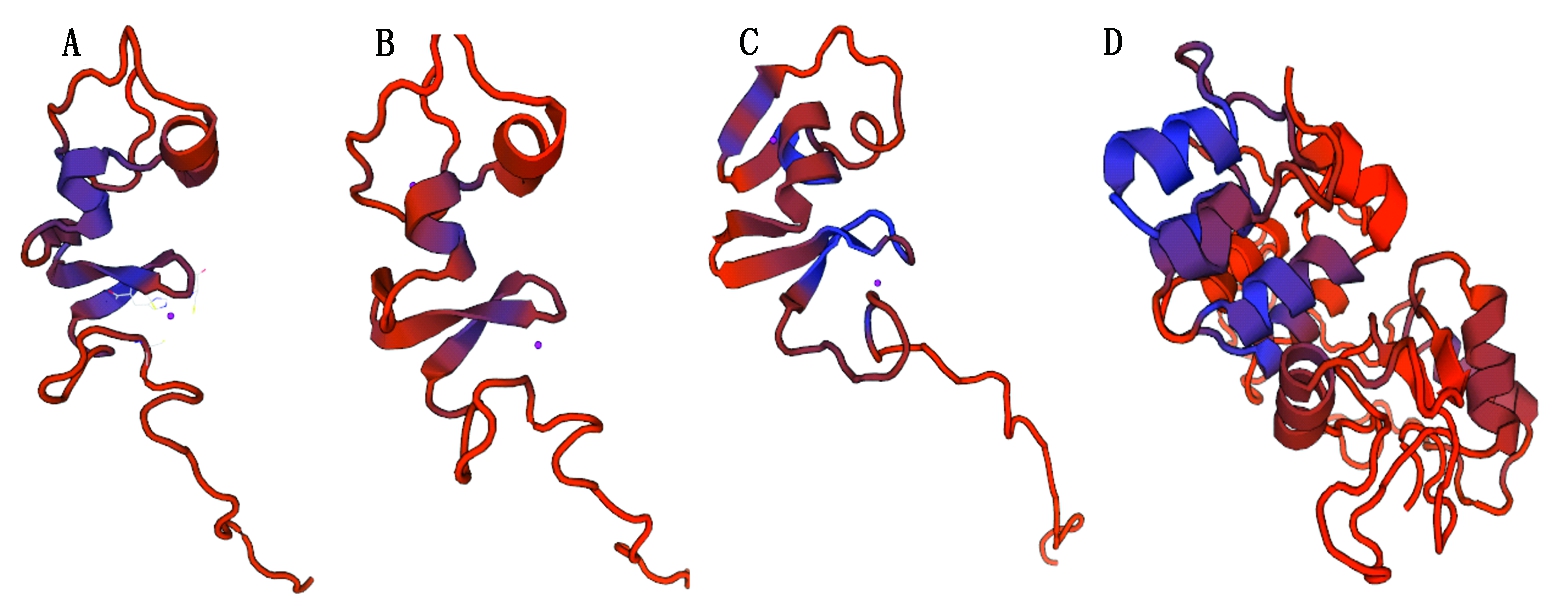
本研究通过对小麦50个TaSBP基因家族的氨基酸序列进行三维结构同源建模,利用Swiss-Model分析显示,50个TaSBP氨基酸序列的三级结构相似性较高。从中选取4条最具代表性的序列(TaSBP5、TaSBP12、TaSBP15、TaSBP31)进行同源三级结构建模(图5)。这4种序列都至少含有1个α-螺旋和2个β-折叠,但其三级结构不完全相同,TaSBP12、TaSBP15都含有2个α-螺旋和3个β-折叠,TaSBP31含有1个α-螺旋和3个β-折叠,而TaSBP5含有9个α-螺旋和2个β-折叠。这些α-螺旋、β-折叠的长度不同及无规则卷曲不同可能导致它们功能上相似或不同。
2.5 小麦TaSBP基因的表达谱分析
小麦品种Chinese Spring 13个组织器官的RNA-seq数据分析结果显示,50个TaSBP基因都在13个组织器官中检测到了转录本。如图6所示,TaSBP8、TaSBP31、TaSBP1、TaSBP26、TaSBP17、TaSBP44在13个组织器官中均具有较高的表达量,TaSBP30、TaSBP45、TaSBP38、TaSBP47、TaSBP49在13个组织器官中均具有较低的表达量;TaSBP19、TaSBP43、TaSBP32等42个基因在幼穗和花苞均具有最高的表达量,这与Manning等[11]研究的SBP基因(LeSPL-CNR)是控制番茄果实成熟的结果基本一致。除TaSBP5、TaSBP21、TaSBP24、TaSBP37、TaSBP40在花药中表达较低外,其他45个基因均具有较高的表达量,这与Schmid等[32]研究拟南芥SBP基因(SPL8)参与花粉发育的调控的结果基本一致,说明小麦的TaSBP基因可能与小麦的籽粒发育相关;TaSBP19、TaSBP43、TaSBP32等31个基因在22 DAP胚乳中均具有较低的表达量,说明小麦的TaSBP基因可能在小麦籽粒成熟后期表达量较低;TaSBP9、TaSBP14、TaSBP29等45个TaSBP基因在叶中的表达均不低,这与Moreno等[19]研究的SBP转录因子LG1能够影响玉米舌叶和叶耳的发育结果基本一致。
3 讨论
高等植物中存在数千种转录因子,其中部分已被证明与生长发育[33]、抗逆性相关,调控高盐、低温、干旱、激素及发育相关的转录因子相继被克隆[34],这些转录因子增加植物的逆境抵抗力和适应力,调控果实生长发育。小麦SBP基因家族的研究起步较晚,但对其功能的研究受到广泛重视。目前,SBP基因家族在模式植物拟南芥中的研究较为深入,同时构建了拟南芥转录因子数据库(DATF),为小麦转录因子的研究提供了很多经验和信息资源。从小麦最新数据库中筛选SBP转录因子相应的蛋白序列,结合生物信息学网站和分析软件,对小麦SBP家族基因结构、染色体定位和系统进化树等进行分析,得到小麦SBP基因家族不同成员之间的相互关系和演化历程。
利用NCBI Blast已报道的SBP蛋白序列并与Pfam数据库进行比对,获得SBP基因家族的Pfam ID及其序列。搜索小麦SBP基因家族的同源蛋白,删除重复序列,利用在线软件Pfam进行保守结构域分析验证,剔除冗余蛋白,最终在拟南芥、玉米和高粱中分别得到 17,102,39个SBP 基因。前人研究发现,拟南芥、高粱[35]和玉米[3]中分别有 16,18,42 个SBP基因。二者比较显示,本研究鉴定的 SBP 基因较多,原因是本研究采用最新的基因组数据库,进行全基因组多序列比对和隐马尔科夫模型查找。与拟南芥、大麦和水稻中的SBP基因相比,小麦中含有更多SBP基因,说明小麦SBP基因家族可能经历丢失、扩张以及进化导致基因家族成员增加。系统进化分析表明,小麦与拟南芥SBP基因间具有较高的保守性,同一类之间的结构较为相似,不同类间氨基酸组成和蛋白结构的区别可能导致SBP基因家族成员功能上的差异。在小麦基因组中存在 11对SBP旁系同源基因,并对具有代表性的SBP蛋白进行三维结构建模,这可为小麦全基因组加倍事件提供有利证据。本研究通过生物信息学的方法对小麦SBP基因家族进行分析,预测基因的结构与功能,并进行了相关的同源进化分析,这为SBP基因在小麦生长发育过程中基因的挖掘和利用提供了理论与试验依据,为下一步研究小麦SBP转录因子的生物学功能提供参考。
参考文献:
[1] Zhang Y, Schwarz S, Saedler H, et al. SPL8, a local regulator in a subset of gibberellin-mediated developmental processes inArabidopsis[J]. Plant Molecular Biology, 2007, 63(3): 429-439.
[2] Klein J, Saedler H, Huijser P. A new family of DNA binding proteins includes putative transcriptional regulators of theAntirrhinum majus floral meristem identity gene SQUAMOSA[J]. Molecular & General Genetics, 1996, 250(1): 7-16.
[3] 王维维, 单玲玲, 芮道晨. 玉米中SBP-box基因家族的生物信息学分析[J]. 基因组学与应用生物学, 2017, 36(1): 294-298.
[4] 彭 华, 何秀静, 高 健, 等. 玉米SBP转录因子全基因组鉴定与功能分析[J]. 作物学报, 2016, 42(2): 201-211.
[5] Unte U S, Sorensen A M, Pesaresi P, et al.SPL8, an SBP-box gene that affects pollen sac development inArabidopsis[J]. The Plant Cell, 2003, 15(4): 1009-1019.
[6] Yang Z, Wang X, Gu S, et al. Comparative study of SBP-box gene family inArabidopsis and rice[J]. Gene, 2008, 407(1/2): 1-11.
[7] 朱红霞, 胡利宗, 邓小莉, 等. 大豆SBP基因家族的序列特征,表达及进化分析[J]. 东北农业大学学报, 2012, 43(7): 26-33.
[8] 万红建,袁俞锞,刘云飞,等. 番茄SBP基因家族的全基因组鉴定、结构特征及表达分析[J]. 分子植物育种,2013,11(3):299-306.
[9] Wang Y, Hu Z L, Yang Y X, et al. Bioinformatics analysis of SBP-box gene family in rice[J]. China Journal of Bioinformatics, 2011, 9(1): 71-82.
[10] 丁 丹. 水稻 5 个粒型相关基因的分子标记开发与效应分析[D].南京:南京农业大学,2014.
[11] Manning K, Tör M, Poole M, et al. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening[J]. Nature Genetics, 2006, 38(8): 948-952.
[12] 曹 雪, 上官凌飞, 于华平, 等. 葡萄SBP基因家族生物信息学分析[J]. 基因组学与应用生物学, 2010, 29(4): 791-798.
[13] Guo A Y, Zhu Q H, Gu X, et al. Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family[J]. Gene, 2008, 418(1/2): 1-8.
[14] 葛安静, 张春华, 董清华, 等. 草莓SBP基因家族生物信息学初步分析[J]. 中国农学通报, 2012, 28(13): 215-220.
[15] 刘更森, 慕 茜, 戴洪义, 等. 苹果SBP基因家族生物信息学分析[J]. 江西农业学报, 2011, 23(12): 23-27.
[16] Kropat J, Tottey S, Birkenbihl R P, et al. A regulator of nutritional copper signaling in chlamydomonas is an SBP domain protein that recognizes the GTAC core of copper response element[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(51): 18730-18735.
[17] Cardon G H, Höhmann S, Nettesheim K, et al. Functional analysis of theArabidopsis thaliana SBP-box geneSPL3: a novel gene involved in the floral transition[J]. The Plant Journal: for Cell and Molecular Biology, 1997, 12(2): 367-377.
[18] Gandikota M, Birkenbihl R P, Höhmann S, et al. The miRNA156/157 recognition element in the 3′ UTR of the Arabidopsis SBP box geneSPL3 prevents early flowering by translational inhibition in seedlings[J]. The Plant Journal: for Cell and Molecular Biology, 2007, 49(4): 683-693.
[19] Moreno M A, Harper L C, Krueger R W, et al. Liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis[J]. Genes & Development, 1997, 11(5): 616.
[20] Lännenpää M, Jänönen I, Hölttä-Vuori M, et al. A new SBP-box geneBpSPL1 in silver birch (Betula pendula) [J]. Physiologia Plantarum, 2004, 120(3): 491-500.
[21] 曹 雪, 王 晨, 房经贵, 等. 葡萄SPL9和SPL10基因全长cDNA克隆、亚细胞定位和表达分析[J]. 园艺学报, 2011, 38(2): 240-250.
[22] 郭宝健, 李 赢, 袁泽宸, 等. 大麦ARF基因家族的全基因组分析[J]. 麦类作物学报, 2016, 36(11): 1426-1432.
[23] 李文芳, 陈佰鸿, 毛 娟, 等. 葡萄BRX基因家族生物信息学分析[J]. 生物信息学, 2015(3): 158-164.
[24] Avni R, Nave M, Barad O, et al. Wild emmer genome architecture and diversity elucidate wheat evolution and domestication[J]. Science, 2017, 357(6346): 93-97.
[25] Jia J Z, Zhao S C, Kong X Y, et al.Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation[J]. Nature, 2013, 496(7443): 91-95.
[26] Ling H Q, Zhao S, Liu D, et al. Draft genome of the wheat A-genome progenitorTriticum urartu[J]. Nature, 2013, 496(7443): 87-90.
[27] Choulet F, Alberti A, Theil S, et al. Structural and functional partitioning of bread wheat chromosome 3B[J]. Science, 2014, 345(6194): 1249721.
[28] Finn R D, Coggill P, Eberhardt R Y, et al. The Pfam protein families database: towards a more sustainable future[J]. Nucleic Acids Research, 2016, 44(D1): D279-D285.
[29] Artimo P, Jonnalagedda M, Arnold K A, et al. ExPASy: SIB bioinformatics resource portal[J]. Nucleic Acids Research, 2012, 40(W1): W597-W603.
[30] Hu B, Jin J P, Guo A Y, et al. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(8): 1296-1297.
[31] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[32] Schmid M, Uhlenhaut N H, Godard F, et al. Dissection of floral induction pathways using global expression analysis[J]. Development, 2003, 130(24): 6001-6012.
[33] 闫筱筱. 葡萄SBP11基因调控拟南芥早花及发育变化的机理[D].杨凌:西北农林科技大学,2014.
[34] 孙化雨, 杨意宏, 娄永峰, 等. 毛竹SBP转录因子基因的全基因组鉴定和表达分析[J]. 基因组学与应用生物学, 2017, 36(10): 4263-4274.
[35] 常建忠, 闫凤霞, 乔麟轶, 等. 高粱SBP-box基因家族全基因组鉴定及表达分析[J]. 遗传, 2016, 38(6): 569-580.
Bioinformatics Identification of SBP Genes and Expression Profiles in Wheat
LÜ Liangjie, CHEN Xiyong, HU Mengyun, LIU Yuping, SUN Lijing,MA Le, LI Hui
(Institute of Cereal and Oil Crops,Hebei Academy of Agriculture and Forestry Sciences, Crop Genetics and Breeding Laboratory of Hebei, Shijiazhuang 050035, China)
Abstract: The squamosa promoter binding protein (SBP) gene family is specific transcription factor in plant, containing 79 amino acid residue conserved domains, and is mainly involved in plant growth, development and physiological and biochemical processes.However,genome-wide analysis of the SBP gene family has not been reported in wheat.To further explore the function of the wheat SBP gene, 50 SBP genes, namedTaSBPs, were obtained by analyzing the latest genomic data of wheat, and were ranked according to the chromosome number asTaSBP1-TaSBP50. Combined with the published genome data of cultivar Chinese Spring, genes structure, chromosome distribution, conserved domain of proteins, phylogenetic trees and gene expression profiles of the wheat cultivars were analyzed using bioinformatics methods.The results showed that 50TaSBP genes were distributed on the remaining 19 chromosomes except 4B and 4D chromosomes. The genes encoded 192-1 104 amino acids in length and the number of gene exons was from 2 to 11.The results of tandem repeat analysis showed that fragment replication and tandem repeats were the main patterns of gene expansion in theTaSBP family. The phylogenetic tree of seven crops SBP proteins were divided into four categories, and the structure of the same class was similar; The 50TaSBP gene families in wheat contained 10 motifs, and it was presumed that theTaSBP gene family should contain motif1, motif2 and motif4.50TaSBP genes were all detected in 13 tissues and organs, and the expression ofTaSBP genes in different tissues were significantly different.
Key words: Wheat; SBP; Bioinformatics; Phylogenetic tree; Expression profiling
中图分类号:Q78;S512.03
文献标识码:A
文章编号:1000-7091(2018)06-0008-09
doi:10.7668/hbnxb.2018.06.002
收稿日期:2018-09-26
基金项目:河北省现代农业科技创新工程项目(F18R01;494-0402-YBN-RDC4);河北省农林科学院财政专政(F17R0013;2018060303);河北省农林科学院粮油作物研究所开放课题(LYS2016004)
作者简介:吕亮杰(1985-),男,河北石家庄人,助理研究员,博士,主要从事小麦遗传育种研究。
通讯作者:李 辉(1964-),男,河北石家庄人,研究员,博士,主要从事小麦分子技术与遗传育种研究。