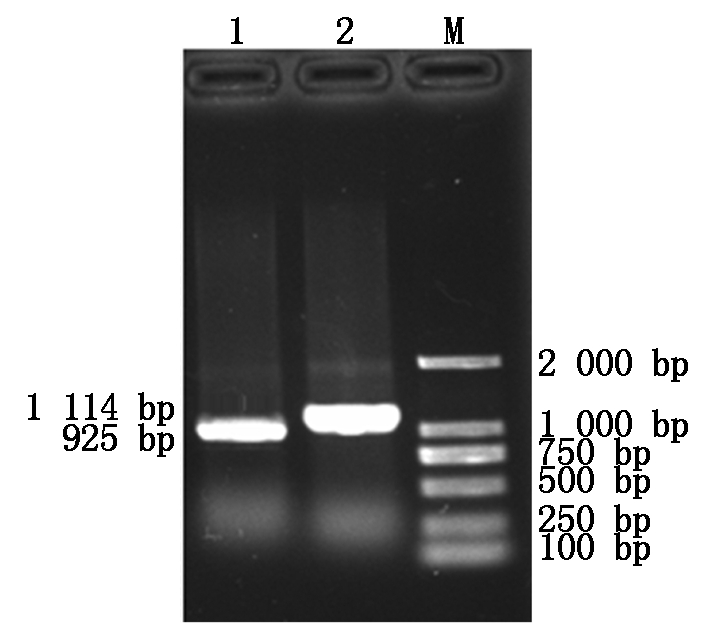
M.DL2000;1.cDNA;2.DNA。
图1 Tamyb59基因的克隆
Fig.1 Cloning of Tamyb59 gene
植物在生长发育进程中受到各种外界因素的影响,包括低温、干旱、高盐、高温等,这些非生物胁迫通过刺激特殊信号传导途径来激活胁迫应答基因的表达,进而通过调控相应生理进程应对外界刺激[1]。自1987年玉米转录因子(Transcription factor, TF)首次被报道以来,人们对转录因子的研究日趋增多,数百种转录因子陆续被发现。植物中常见的有NAC、MYB、WRKY、bZIP、AP/ERBP等转录因子家族[2-3],其中MYB是较大的转录因子家族,广泛存在于高等植物中[4-5]。玉米的C1基因是最早发现的植物MYB转录因子[6]。随着人们对转录调控的深入研究,已经从拟南芥、玉米、水稻等植物中鉴定出多个MYB转录因子[7-9]。大多数植物的MYB在其N端有一段51~52个氨基组成的MYB结构域[10-11]。诸多研究表明,MYB 转录因子参与植物的生长发育、激素信号转导、代谢产物的合成及逆境胁迫应答反应[12-15]。AtMYB21和AtMYB24通过参与植物激素茉莉酸的调控影响花青素的积累[16],AtMYB58和AtMYB63可以同时调控木质素、木聚糖和纤维素生物合成[17],AtMYB88和AtMYB124/FLP通过调控细胞周期相关的基因,诱导气孔的正常分化[18-19]。在干旱和盐胁迫下TaMYBsdul在耐盐小麦品种中的表达高于盐敏感品种[11],PEG、ABA和GA胁迫下棉花GbMYB5基因在茎尖、叶片和蕾铃中表达量增加,叶片中的表达水平显著高于茎尖和蕾铃[20],高盐和ABA胁迫处理下,拟南芥中的AtMybl02表达量明显高于对照。Seo等[21]研究表明,AtMYB96增强了拟南芥的抗旱和抗病性,干旱胁迫下促进表皮蜡质合成、水杨酸和脱落酸介导的抗病信号传递。
普通小麦(Triticum aestivum L.)MYB转录因子的研究起步较晚,目前发现的MYB转录因子数目远少于拟南芥、玉米和水稻等[22-24]。贾东升等[19]报道克隆的3种类型小麦转录因子TaMyb2均参与渗透胁迫的应答反应。Zhang等[25]克隆了60个小麦MYB基因并进行表达分析,发现过表达TaMYB32基因可提高拟南芥的耐盐性。过表达TaMYB73的拟南芥转基因植株同样可增加对盐胁迫的抵御[26]。李孟军等[27]报道,渗透胁迫下叶和根中TaMYBSM151的表达均受到诱导,叶中表现为上调,而根中表现为下调,呈现完全相反的表达模式,认为TaMYBSM151在叶和根中可能具有不同的调控机制。本研究从前期转录组测序结果中筛选到1个候选基因Unigene7811,经初步序列比对分析,该候选基因含有MYB 结构域,暂命名为Tamyb59。拟采用同源克隆方法克隆该基因,并对其在逆境胁迫中的表达和定位特性进行分析,旨在为深入研究Tamyb59基因的功能奠定基础。
本试验选用的小麦材料为冬性品种京841(J841)。选取饱满且大小均一的种子,先用75%酒精浸泡1 min,再经0.1%氯化汞消毒处理5 min,用无菌水冲洗5次,22 ℃黑暗条件下浸泡12 h,待种子萌动后将种子腹沟朝下,放置于铺有2层滤纸的培养皿,放入光照培养箱(25 ℃/18 ℃、16 h光照/8 h黑暗、光照强度350 μmol/(m2·s)中培养。待小麦长到两叶一心时进行PEG6000(20%)和NaCl(100 μmol/L)胁迫处理,剪取胁迫处理0,3,6,9,12,24,48,72 h的叶片和根尖组织,每次取3株样品,并设置3次重复,液氮速冻后保存于-80 ℃冰箱备用,未胁迫处理植株为对照(CK)。每天取样前保证光照1~2 h左右,避免基因节律表达对表达分析的影响。
取上述保存叶片和根组织在液氮中细研,采用TRIzol试剂(Invitrogen)提取总RNA,并通过1%琼脂糖凝胶电泳检测RNA质量。参照反转录试剂盒PrimerScriptRTreagent Kit with gDNA Eraser(TaKaRa)的操作说明进行cDNA的合成。
根据基因序列设计引物(Tamyb59-F1:5′-TC CAAGGTAGAAGCGAACAAC-3′;Tamyb59-R1:5′-GA TCACCCTCCATACTAATGC-3′),以J841的cDNA为模板进行PCR扩增。反应体系25 μL,包含LATaq(5 U/μL)0.2 μL、2×GC BufferⅡ 2 μL、dNTP mixture(10 mmol/L) 2 μL、上、下游引物(10 μmmol/L)各1 μL、cDNA模板1 μL,用ddH2O补齐至25 μL。PCR反应程序为95 ℃ 5 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃ 10 min,4 ℃保存。将PCR扩增产物回收纯化后,连接至pMD18-T载体后转化DH5α,经菌液PCR检测挑取阳性单克隆,送往北京华大基因有限公司测序,利用DNAMAN软件对测序结果进行比对分析。
根据引物设计原则设计Real-time PCR引物,前引物Tamyb59-F2:5′-GGCAGCACTCATAATGGCAC-3′,后引物Tamyb59-R2: 5′-CTCAGGAGGGCAGT AGCG-3′。反转录的cDNA为模板,进行实时荧光定量 PCR 分析,参照TaKaRa荧光定量PCR SYBR® Green Ⅰ试剂盒说明书进行操作。反应在 Bio-red CFX96 荧光定量 PCR 仪上运行,PCR反应体系为 20 μL,反应程序:95 ℃ 30 s;95 ℃ 15 s,60 ℃ 30 s,40个循环;以小麦的β-Actin基因为内参基因(β-actin-F:5′-GTTCCAATCTATGAGGGATACACGC-3′,β-actin-R:5′-GAACCTCCACTGAGAACAACATTACC-3′),每个样品设3次技术重复,采用2-ΔΔCt法进行定量分析。
根据Tamyb59的全长cDNA序列,利用Primer 5.0软件设计特异扩增引物Tamyb59-F3:5′-CGAC TAGTATGTCGCCACAAGAGGAA-3′和Tamyb59-R3:5′-TTGGCGCGCCAATAGCAGGAACGTTCTC-3′(下划
线为酶切位点SpeⅠ和Asc Ⅰ),以测序正确的质粒DNA为模板,用带有SpeⅠ和AscⅠ酶切位点的引物Tamyb59-F3/R3扩增出含有目标基因的cDNA 片段,连接至pMD18-T载体后转化至大肠杆菌DH5α,挑选阳性单菌落测序,测序正确后菌液进行扩大培养,提取质粒。用限制性内切酶SpeⅠ和AscⅠ进行双酶切,得到目的片段,同样双酶切表达载体pMDC83,将2个酶切后的片段用T4-DNA连接酶连接后,转化至大肠杆菌DH5α,涂布于卡那霉素抗性的LB固体平板,挑取单菌落,经PCR检测为阳性的菌液扩大培养后提取质粒,双酶切检测、测序鉴定。
将构建完成的pMDC83-Tamyb59重组载体转化农杆菌EHA105,并对幼嫩的本氏烟草进行侵染,2~5 d内在激光共聚焦显微镜下观察绿色荧光融合蛋白定位表达情况。

M.DL2000;1.cDNA;2.DNA。
图1 Tamyb59基因的克隆
Fig.1 Cloning of Tamyb59 gene

图2 Tamyb59的CDS序列及其氨基酸序列
Fig.2 Nucleotide and amino acid sequence of Tamyb59
分别以J841叶片提取的RNA反转录的cDNA和基因组DNA为模板,以Tamyb59-F1/R1为引物,PCR扩增得到产物大小分别为925 bp和1 114 bp,与预期产物大小一致(图1)。将扩增产物用琼脂糖凝胶回收试剂盒回收纯化,连接至pMD18-T载体后转化大肠杆菌DH5α,将鉴定正确的阳性单克隆测序。生物信息学分析显示,该基因的开放阅读框为522 bp,共编码173个氨基酸(图1,2),初步定位在4B染色体短臂上。
利用NCBI对Tamyb59基因的氨基酸序列保守结构域分析发现,其蛋白属于SANT superfamily超基因家族之一(图3)。对Tamyb59的DNA和cDNA序列比较分析表明,该基因有3个外显子和2个内含子(图4)。经过在线软件Expasy-ProtParam (http://www.expasy.org/)分析该基因所编码蛋白的理化性质,结果表明,预测的Tamyb59蛋白的分子质量为19.7 ku,理论等电点为7.61。在整个肽链中,亲水氨基酸分布均匀,且多于疏水氨基酸,因此整个肽链表现为亲水性,Tamyb59基因编码的MYB蛋白为亲水蛋白。
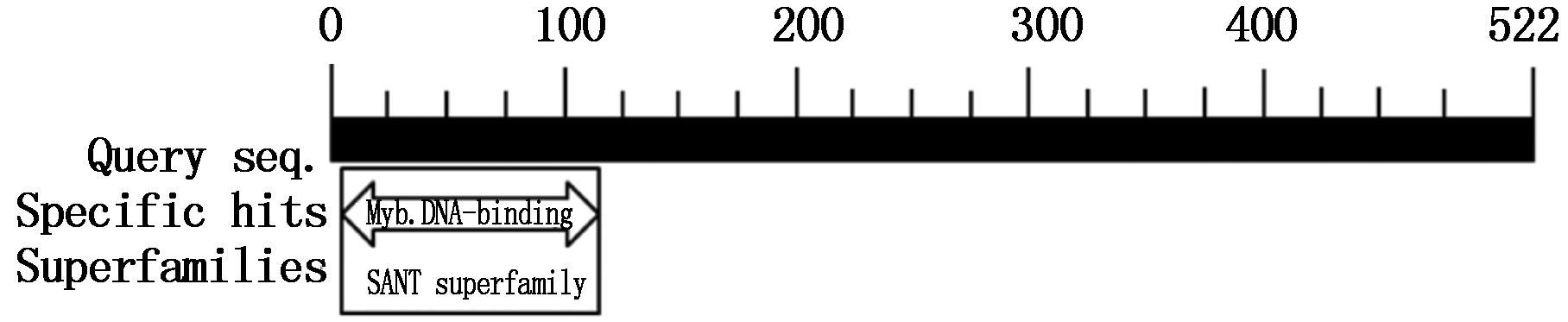
图3 Tamyb59蛋白的保守结构域分析
Fig.3 Conserved domain analysis of Tamyb59 protein
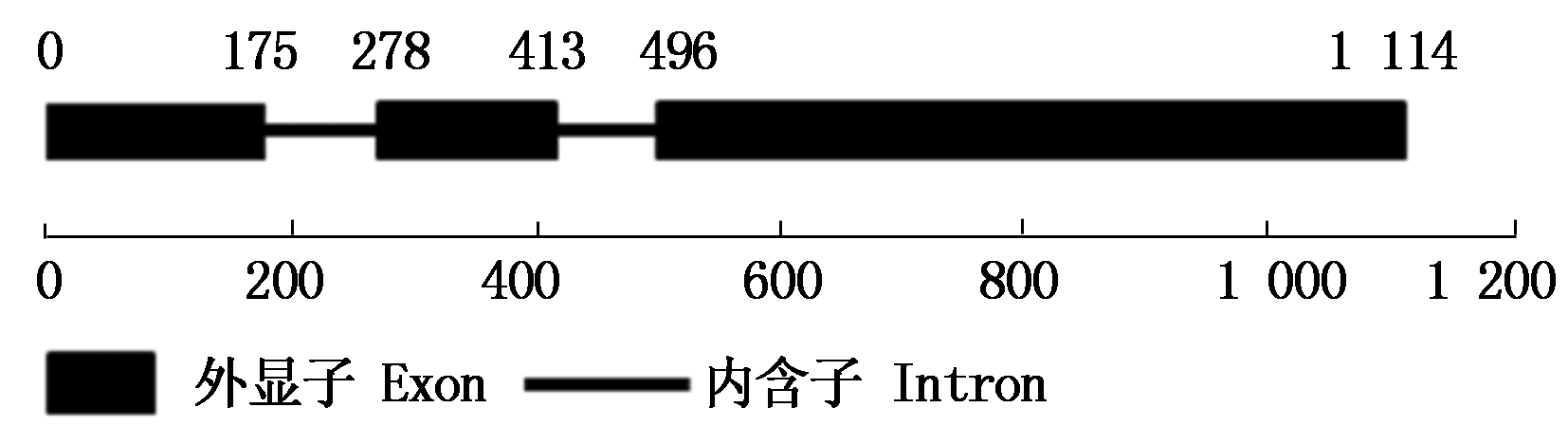
图4 Tamyb59的DNA结构分析
Fig.4 DNA structure analysis of Tamyb59
利用DNAMAN 8.0软件翻译获得Tamyb59的氨基酸序列,在NCBI (Chttp://www.ncbi.nlm.nih.gov/)蛋白数据库中比对发现,该蛋白与山羊草、粳稻、玉米等9种植物MYB转录因子有52.0%~85.6%的相似性,其中与谷子的MYB蛋白相似性最高,为85.6%。将Tamyb59与这9条转录因子序列进行多重比较并利用MEGA 6.0软件构建系统进化树,10条蛋白序列明显聚类为2组,Tamyb59与玉米、谷子、二穗短柄草、高粱为第1类,其中,与谷子的亲缘关系最近;而山羊草、籼稻、甘蔗等则为第2类(图5)。
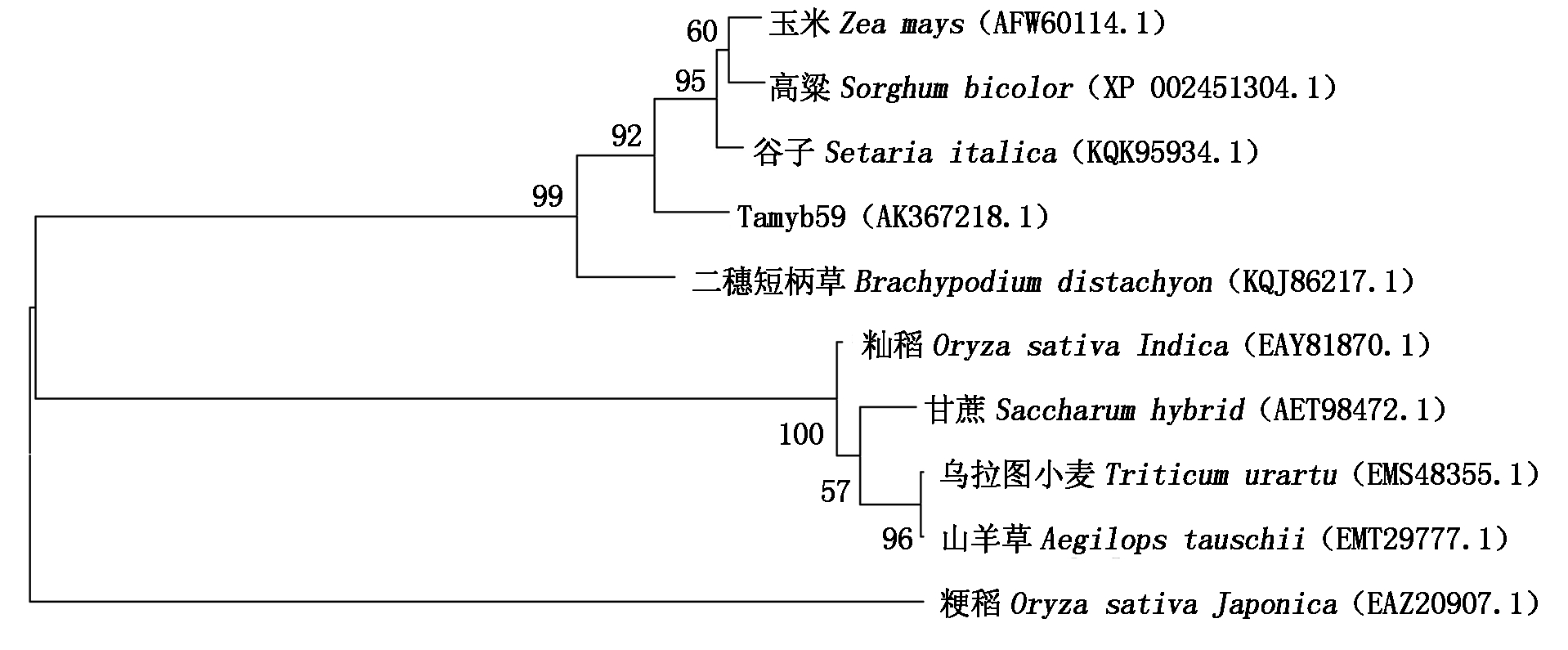
图5 基于NJ法构建Tamyb59蛋白与其他物种MYB蛋白的系统进化树
Fig.5 Phylogenetic tree analyses between Tamyb59 and other MYB proteins based on NJ method
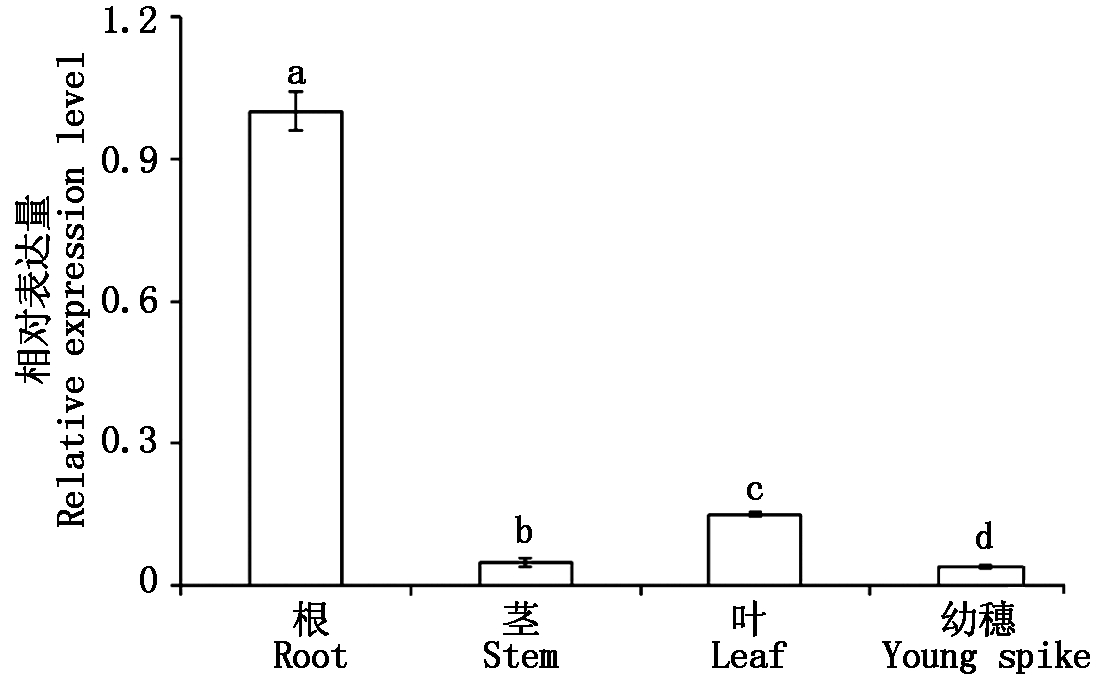
采用Real-time PCR 对Tamyb59基因在小麦不同组织器官中的表达进行分析,结果显示,Tamyb59在根、茎、叶、幼穗中均有表达,其中根中的表达量相对较高,其次是叶片,在茎和小麦幼穗中的表达量较低,差异达到显著水平(图6)。根中的表达量分别是茎的20.4倍、叶的6.7倍、幼穗的25.3倍。
将构建好的pMDC83-Tamyb59-GFP瞬时表达载体转化到烟草表皮细胞,注射烟草叶片,2~3 d后通过激光共聚焦荧光显微镜检测荧光信号。结果显示,不含外源基因的载体pMDC83-GFP分布在整个细胞核和细胞质膜;而转pMDC83-Tamyb59-GFP载体的烟草表皮细胞内绿色荧光仅分布在细胞核上(图7),说明该基因编码的蛋白定位于细胞核。
不同字母表示差异显著性(P<0.05)。图8-9同。
Different letters indicate significant differences (P<0.05).
The same as Fig.8-9.
图6 Tamyb59基因在小麦不同组织器官中的表达分析
Fig.6 Expression levels of Tamyb59 in different tissues and organs of wheat seedlings
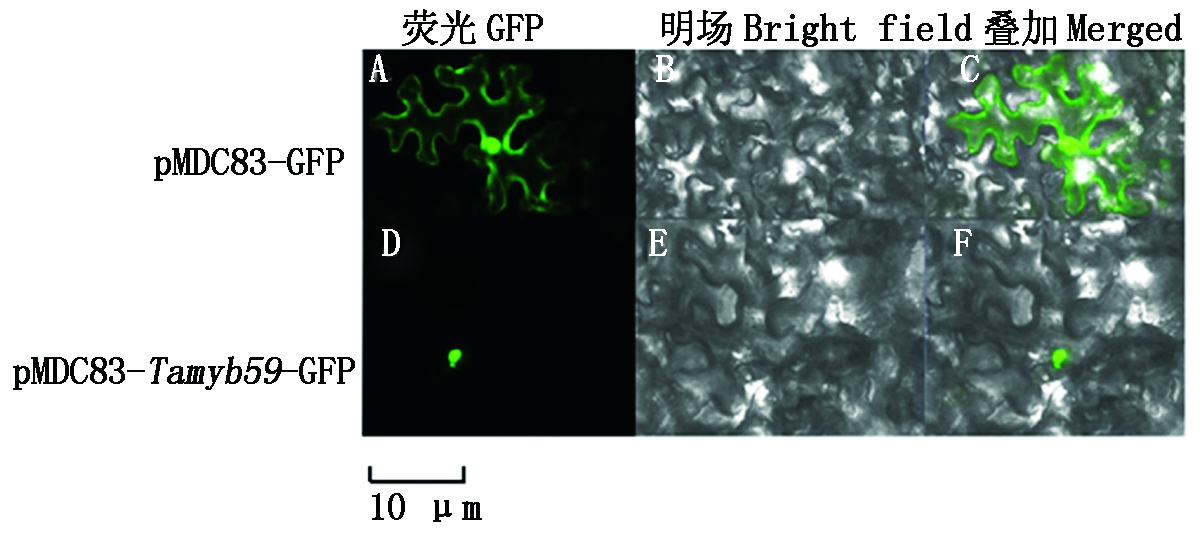
图7 Tamyb59在烟草表皮中的亚细胞定位
Fig.7 Subcellular localization of Tamyb59 in tobacco epidermis
当小麦植株长到两叶一心时,进行20%PEG6000胁迫。在根和叶片中的表达模式相同,整个胁迫处理过程中,Tamyb59基因的表达均呈现出先升高后降低的趋势,但Tamyb59基因在根中的表达量高于叶片。胁迫处理9 h时,Tamyb59基因在根尖中的表达量达到最高,随着胁迫处理时间的延长表达量逐渐降低(图8-A)。在叶片中,胁迫处理6 h时,Tamyb59基因在叶片的表达最高,与0 h相比,差异达到显著水平,胁迫处理9 h时表达量急剧下降,随着胁迫处理时间的延长,基因的表达量变化不明显,胁迫处理9,24,72 h,差异不显著(图8-B)。
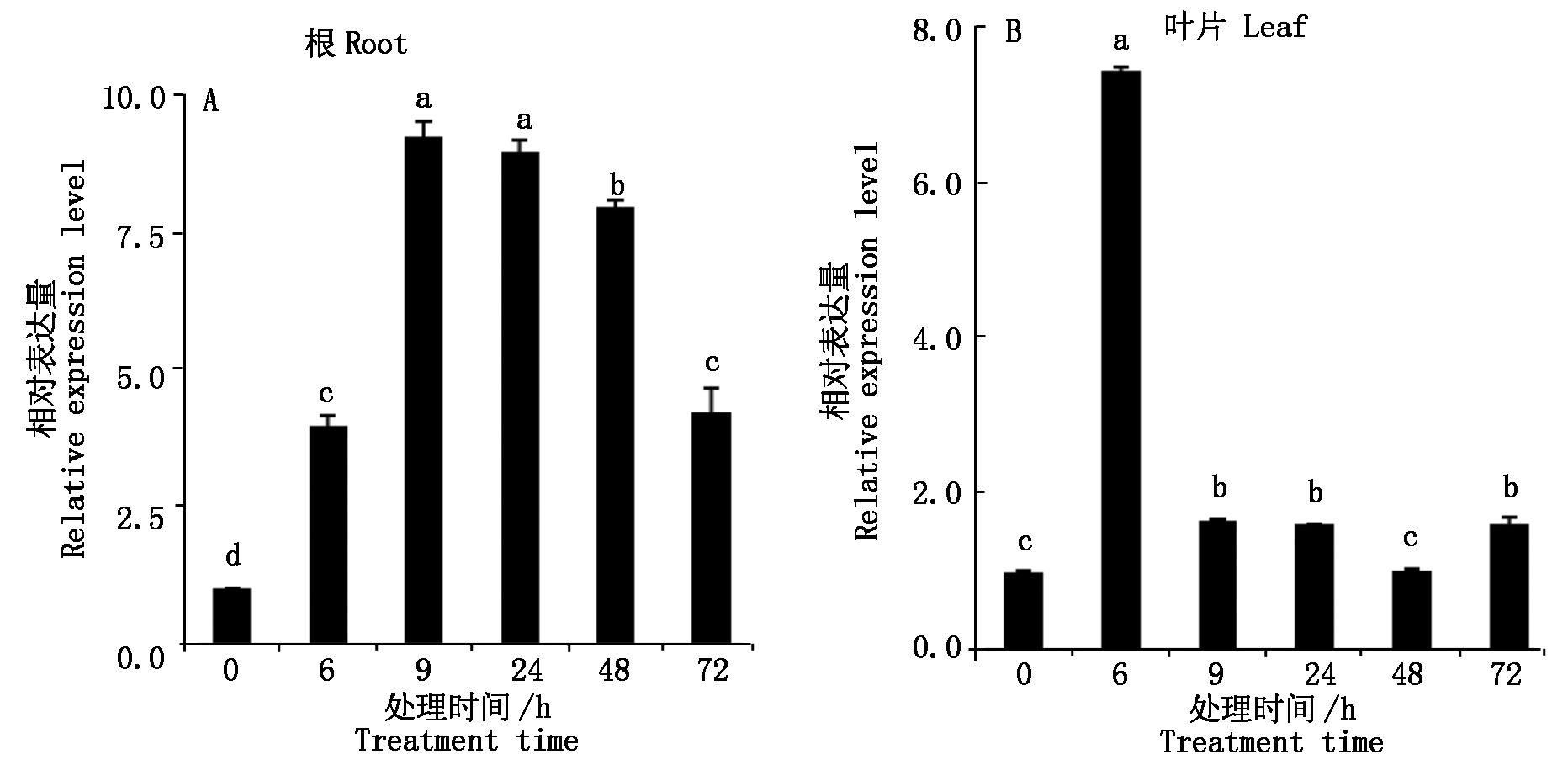
图8 PEG胁迫下小麦幼苗根和叶片中Tamyb59基因的表达变化
Fig.8 Expression patterns of Tamyb59 gene under PEG stress in root and leaf
盐胁迫处理下,Tamyb59基因的表达量呈先上升后下降的趋势。在根中,盐胁迫处理6 h时基因的表达量变化不明显,胁迫处理12 h时,Tamyb59基因表达量达到最大值,与0 h对照相比,差异达到显著水平。随着盐胁迫时间的延长,表达量逐渐降低,胁迫处理72 h时表达量最低,与0 h对照相比,差异水平不显著(图9-A)。在叶片中,Tamyb59基因在胁迫处理24 h时表达量最高,随着盐胁迫处理时间延长,基因的表达量逐渐降低,胁迫处理72 h,其基因表达量最低,差异达到显著水平(图9-B)。
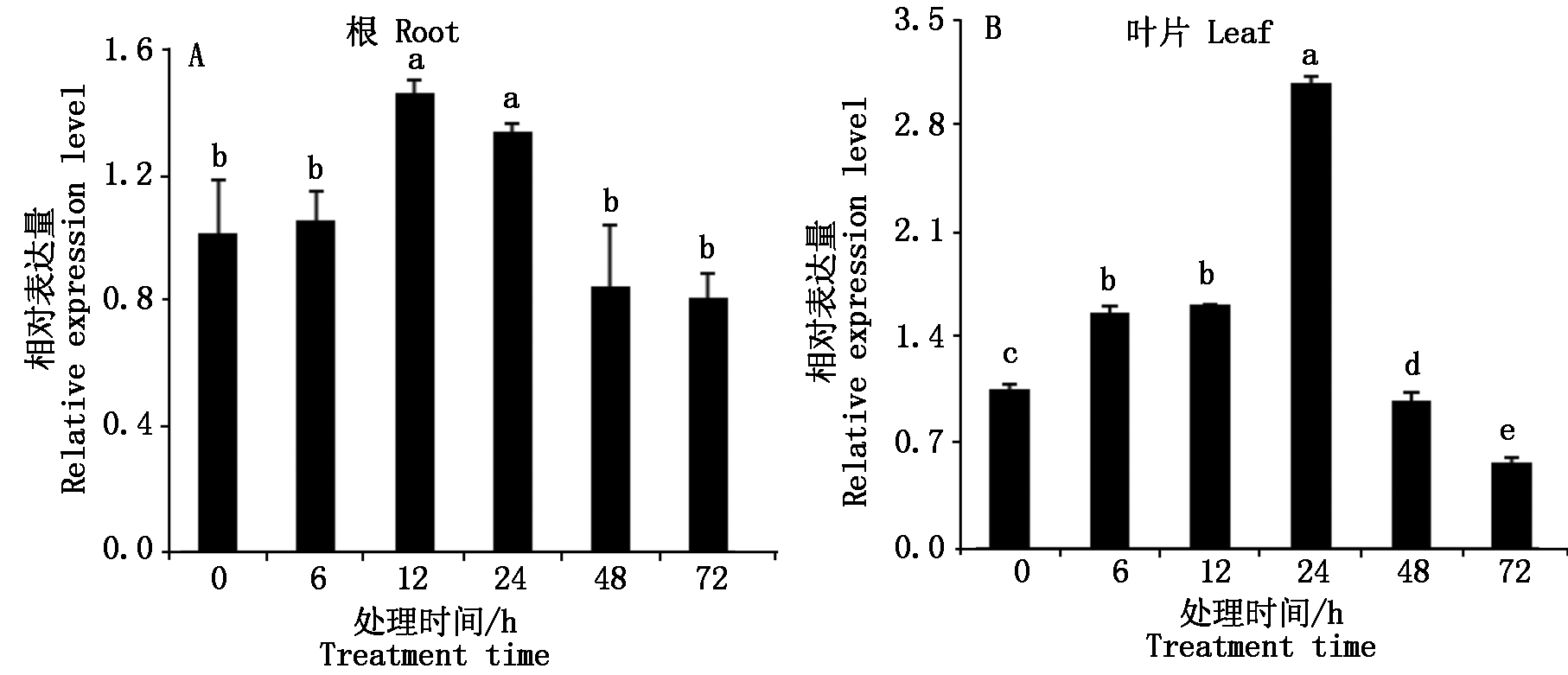
图9 盐胁迫下小麦Tamyb59基因在根和叶片的表达
Fig.9 Expression patterns of Tamyb59 gene under salt stress in root and leaf of wheat seedlings
干旱、高盐、低温等非生物胁迫都是影响植物生长发育和产量的重要因素。因此,挖掘响应非生物胁迫的基因、培育抗逆性新品种,对于提高作物产量具有重要的意义。转录因子在植物对非生物胁迫的应答中起着重要的作用。MYB转录因子为植物最大的转录因子家族之一,在植物的生长发育、代谢、生物和非生物胁迫应答中起着重要的调控作用[28-29]。MYB家族基因在许多植物中都有深入的研究,在普通小麦中报道较少。本研究中Tamyb59基因具有1个522 bp的ORF,编码173个氨基酸,含有3个外显子,2个内含子;Tamyb59与谷子的MYB蛋白亲缘关系最近,其次是高粱,和粳稻等的MYB蛋白亲缘关系相对较远。陈荣敏[30]在小麦根、茎、叶及授粉后3,6,12 d种子的cDNA中均克隆出MYB基因同源片段,说明小麦MYB基因广泛表达于不同组织器官,本研究发现Tamyb59基因在根、茎、叶及幼穗中均有表达,且在根中的表达量很高。
基因表达受多因素的影响,包括染色质修饰、基因转录和转录后调控、翻译修饰等,其中转录水平的调控是真核生物基因表达水平的主要调控方式,因此已成为分子生物学研究的热点。研究表明,不同MYB基因在同一胁迫处理下表达不同,同一个MYB基因在不同的非生物胁迫下的响应模式也不一致;并不是所有的MYB相关蛋白都是转录激活子,有的也起负调控作用,抑制目标基因的表达[31]。本研究中,PEG渗透胁迫下Tamyb59基因在小麦根和叶片的表达模式不同,根中呈上调表达趋势,胁迫处理下表达量均高于对照,而在叶片中大部分时间点表达量低于对照;盐胁迫下,Tamyb59基因在根和叶片中的表达均低于对照,其低水平的表达,可能促进该基因下游相关逆境基因的表达从而提高小麦植株对盐胁迫的耐受能力,研究结果与AtMYB44的表达模式类似[32]。
[1] Tan Y, Li M, Yang Y, et al. Overexpression ofMpCYS4, a phytocystatin gene fromMalus prunifolia (Willd.) Borkh.,enhances stomatal closure to confer drought tolerance in transgenicArabidopsis and apple[J].Frontiers in Plant Science, 2017, 8:33-48.
[2] Erpen L, Devi H S, Grosser J W, et al. Potential use of the DREB/ERF, MYB, NAC and WRKY transcription factors to improve abiotic and biotic stress in transgenic plants[J]. Plant Cell Tissue and Organ Culture, 2018, 132(1):1-25.
[3] Gao F, Zhao H X, Yao H P, et al. Identification, isolation and expression analysis of eight stress-relatedR2R3-MYB genes in tartary buckwheat (Fagopyrum tataricum)[J]. Plant Cell Reports, 2016, 35(6):1385-1396.
[4] Liu X L, Yu W W, Zhang X H, et al. Identification and expression analysis under abiotic stress of theR2R3-MYB genes inGinkgo biloba L.[J]. Physiology and Molecular Biology of Plants, 2017, 23(3):1-14.
[5] Mmadi M A,Dossa K, Wang L H,et al. Functional characterization of the versatile MYB gene family uncovered their important roles in plant development and responses to drought and waterlogging in sesame [J]. Genes, 2017, 8(12):362-380.
[6] Haga N, Kato K, Murase M, et al. R1R2R3-Myb proteins positively regulate cytokinesis through activation of KNOLLE transcription inArabidopsis thaliana[J]. Development, 2007, 134(6):1101-1110.
[7] Zhao Y, Tian X J, Wang F,et al.Characterization of wheat MYB genes responsive to high temperatures[J]. BMC Plant Biology, 2017, 17(1):208-222.
[8] Yin X, Cui Y, Wang M, et al. Overexpression of a novel MYB-related transcription factor, OsMYBR1, confers improved drought tolerance and decreased ABA sensitivity in rice [J]. Biochemical and Biophysical Research Communications, 2017, 490(4):1355-1361.
[9] Wei Q H, Zhang F, Sun F S, et al. A wheat MYB transcriptional repressor TaMyb1D regulates phenylpropanoid metabolism and enhances tolerance to drought and oxidative stresses in transgenic tobacco plants [J]. Plant Science, 2017, 265:112-123.
[10] Rahaie M, Xue G P, Naghavi M R, et al. A MYB gene from wheat (Triticum aestivum L.) is up-regulated during salt and drought stresses and differentially regulated between salt-tolerant and sensitive genotypes [J]. Plant Cell Reports, 2010, 29(8):835-844.
[11] D′Amelia V,Aversano R, Ruggiero A, et al. Subfunctionalization of duplicate MYB genes inSolanum commersonii generated the cold-induced ScAN2 and the anthocyanin regulator ScAN1[J].Plant Cell and Environment, 2018, 41(5):1038-1051.
[12] Guo H Y, Wang L Q, Yang C P, et al.Identification of novel cis-elements bound by BplMYB46 involved in abiotic stress responsesand secondary wall deposition [J]. Journal of Integrative Plant Biology, 2018, 60(10):1000-1014.
[13] Li W B,Ding Z H,Ruan M B, et al.Kiwifruit R2R3-MYB transcription factors and contribution of the novel AcMYB75 to red kiwifruit anthocyanin biosynthesis [J]. Scientific Reporters,2017, 7(1):16861-16875.
[14] Sun M, Feng X X,Gao J J, et al. VvMYBA6 in the promotion of anthocyanin biosynthesis and salt tolerance in transgenicArabidopsis[J]. Plant Biotechnology Reports, 2017,11(5):299-314.
[15] Huang W J, Lü H Y, Wang Y.Functional characterization of a novel R2R3-MYB transcription factor modulating the flavonoid biosynthetic pathway from epimediumsagittatum[J].Frontiers in Plant Science, 2017, 8, 1274-1285.
[16] Golldack D, Luking I,Yang O. Plant tolerance to drought and salinity: stress regulating transcription factors and their functional significance in the cellular transcriptional network [J]. Plant Cell Reports, 2011, 30(8):1383-1391.
[17] Seo P J, Park C M. MYB96-mediated abscisic acid signals induce pathogen resistance response by promoting salicylic acid biosynthesis inArabidopsis [J]. New Phycologists, 2010, 186(2):471 483.
[18] Denekamp M, Smeekens S C. Integration of wounding and osmotic stress signals determines the expression of the AtMYB102 transcription factor gene [J]. Physiologia Plantarum, 2003, 132(3):1415-1423.
[19] 贾东升, 毛新国, 景蕊莲, 等. 小麦转录因子TaMyb2s的克隆及表达[J]. 作物学报, 2008,34(8):1323-1329.
[20] Song S S, Qi T C,Huang H, et al. The Jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to effect jasmonate-regulated stamen development inArabidopsis [J]. Plant Cell, 2011, 23(3):1000-1013.
[21] Seo P J, Lee S B, Suh M C, et al. The MYB96 transcription factor regulates cuticular wax biosynthesis under drought conditions inArabidopsis [J]. Plant Cell, 2011, 23(3):1138-1152.
[22] Xu F C, Liu H L, Xu Y Y, etal. Heterogeneous expression of the cotton R2R3-MYB transcription factor GbMYB60 increases salt sensitivity in transgenicArabidopsis [J].Plant Cell Tissueand Organ Culture, 2018, 133(1):15-25.
[23] Wei Q H, Zhang F, Sun F S, et al. A wheat MYB transcriptional repressor TaMyb1D regulates phenylpropanoid metabolism and enhances tolerance to drought and oxidative stresses in transgenic tobacco plants [J]. Plant Science, 2017, 265:112-123.
[24] Zhang L C, Liu G X, Zhao G Y, et al. Characterization of a wheat R2R3-MYB transcription factor gene,TaMYB19, involved in enhanced abiotic stresses inArabidopsis [J].Plant and Cell Physiology,2014,55(10):1802-1812.
[25] Zhang L C, Zhao G Y, Jia J Z, et al. Molecular characterization of 60 isolated wheat MYB genes and analysis of their expression during abiotic stress [J]. Journal of Experimental Botany, 2012, 63(1):203-214.
[26] He Y N, Li W, Lv J, et al. Ectopic expression of a wheat MYB transcription factor gene,TaMYB73, improves salinity stress tolerance inArabidopsis thaliana [J].Journal of Experimental Botany, 2011, 63(3):1511-1522.
[27] 李孟军, 史占良, 郭进考. 小麦渗透胁迫诱导基因TaMYBSM151的克隆与分析[J]. 华北农学报, 2014,29(4):19-24.
[28] Saha G,Park J I, Ahmed N U, et al. Characterization and expression profiling of MYB transcription factors against stresses and duringmale organ development in Chinese cabbage (Brassica rapassppekinensis)[J]. Plant Physiology and Biochemistry, 2016, 104:200-215.
[29] Chen N,Yang Q L,Pan L J, et al.Identification of 30 MYB transcription factor genes and analysis of their expression during abioticstress in peanut (Arachis hypogaea L.)[J].Gene, 2014, 533(1):332-345.
[30] 陈荣敏. 普通小麦Myb和Dof转录因子家族基因的克隆和表达研究[D]. 北京:中国农业大学,2004.
[31] Jin H,Martin C.Multifunctionality and diversity within the plant MYB-gene family [J].Plant molecular biology, 1999, 41(5):577-585.
[32] Jung C, Seo J S, Han S W.Overexpression of AtMYB44 enhances stomatal closure to confer abiotic stress tolerance in transgenicArabidopsis [J]. Plant Physiol, 2008, 146(2): 623-635.
卫 丽(1966-),女,河南郑州人,研究员,博士,主要从事小麦发育研究。
刘毓侠(1965-),女,河南商丘人,研究员,主要从事作物遗传育种及期刊编辑工作。