小反刍兽疫(Peste des petits ruminants,PPR)主要感染小反刍动物,山羊和绵羊易感,也称羊瘟。野生的小反刍兽也具有易感性。其潜伏期是2~7 d。 动物感染后的主要症状是发热、流涕、腹泻、白细胞减少、呼吸困难、鼻及口腔黏膜溃烂,进而化脓并伴有恶臭气味,怀孕动物会出现流产现象[1-3]。其感染率、死亡率高,被世界动物卫生组织(World Organisation for Animal Health,OIE)定为必须上报A类疫病。该病造成了巨大的经济损失,尤其对发展中国家的养羊业[4]。引起PPR的病原为小反刍兽疫病毒(Peste des petits ruminants virus,PPRV)。其属副粘病毒科(Paramyxoviridae)麻疹病毒属(Morbillivirus),为单股负链RNA病毒,病毒粒子呈多形性。基因组全长15 948 bp,共有N-P(C、V)-M-F-H-L 6个基因,可编码8种蛋白,6种结构蛋白及非结构蛋白C、V。病毒复制时利用宿主内膜形成囊膜蛋白H、F,并由基质蛋白M将H、F与核蛋白相连。病毒入侵宿主细胞时,H与宿主表面受体结合后F改变构型,发生膜融合,核酸注入细胞质中。装配完全的病毒颗粒通过出芽生殖释放出来。另外,F和H蛋白,可诱导宿主的免疫保护,形成针对H蛋白的中和抗体,参与体液免疫[5-6]。高度的免疫压力使 F蛋白易变异、保守性差[7]。
本研究结合同源模建及多种预测蛋白抗原表位的算法,预测PPRV H 蛋白3D结构及表面特性,分析了PPRV H蛋白结构和功能的关系,旨在为研究和开发PPRV H基因工程疫苗提供参考。
1 材料和方法
1.1 试验材料
1.1.1 质粒、菌种和毒株 试验所用E.coli 为JM109和Rosetta。所用原核表达载体为pET32a。 所用毒株为PPRV Nigeria75/1 株,由甘肃省动物细胞工程中心保存。
1.1.2 主要试剂、工具酶及引物 试验所用T4 DNA 连接酶、Ex Taq DNA 聚合酶、限制性内切酶为BamH Ⅰ、Hind Ⅲ,购自大连宝生物公司。RNA提取试剂盒购自北京博凌科为生物科技有限公司。质粒提取试剂盒购自兰州美博生物有限公司。一抗为鼠抗His, 二抗为山羊抗鼠,均购自Sigma公司。His 标签镍离子蛋白纯化柱购自GE 公司。参考PPRV Nigeria75/1毒株H基因序列设计一对PCR引物。上游引物F1:5′-TCCGCACAAAGGGAAAG-3′;下游引物F2:5′-TCAGACTGGATTACATGTTACC-3′。
1.2 试验方法
1.2.1 目的基因扩增 提取病毒基因总RNA,反转录,PCR 扩增。反应体系:Ex Taq DNA 聚合酶0.25 μL,dNTP Mixture 4 μL,10 ×Ex Taq Buffer 5 μL,上、下游引物各 1 μL,反转录产物 cDNA 500 ng,补加 ddH2O 至 50 μL。反应条件:95 ℃ 5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min, 35个循环;72 ℃延伸 10 min。琼脂糖凝胶电泳鉴定所得产物后回收目的片段。
1.2.2 克隆目的基因、测序 回收后的H基因与 pMD18-T 载体连接,转化感受态 JM109细胞,涂布Amp 抗性的平板上(所用抗生素浓度为 1‰),37 ℃培养12 h;挑取单克隆菌落,1∶100比例接种Amp 抗性的 LB 液体培养基中(所用抗生素浓度为 1‰),37 ℃摇菌12 h,提取重组质粒,BamHⅠ、 Hind Ⅲ双酶切鉴定,命名阳性质粒pMD18-T-H,测序。
1.2.3 构建重组表达质粒 原核表达载体 pET-32a、阳性重组质粒 pMD18-T-H 分别用BamHⅠ和 Hind Ⅲ双酶切,回收片断,16 ℃过夜连接,总体系(10 μL):10×缓冲液 1 μL,T4 DNA 连接酶0.5 μL,达到H2O 2.5 μL目的片段5 μL,载体1 μL,H2O 2.5 μL;转化 JM109 ,双酶切鉴定,阳性重组质粒 pET-32a-H。
1.2.4 诱导表达目的蛋白、可溶性分析 转化重组质粒pET-32a-H ,所用大肠杆菌为Rosetta,取转化菌接种至 LB 液体培养基中,37 ℃,220 r/min 过夜;1∶100 比例接种至3 mL新鲜 LB 液体培养基中,37 ℃,220 r/min 振荡培养 1.5 h 至 A 600约为0.6时,取1 mL菌液作为对照,加入 IPTG 至终浓度为1 mmol/L,于 37 ℃诱导培养 6 h,取1 mL 菌液,离心收集菌体进行Lysis Buffer冰上裂解30 min,12 000 r/min离心 10 min,分离上清及沉淀。分别加入 2× SDS 上样缓冲液沸水浴10 min,进行 10% SDS-PAGE 分析。对照组为重组菌诱导前的诱导表达产物及诱导前后pEG32a空质粒。
1.2.5 鉴定表达产物 诱导表达的重组 H 蛋白进行 Western Blot 鉴定。一抗为鼠抗His 标签(1∶3 000 稀释),二抗为山羊抗小鼠 IgG(1∶10 000 稀释)。
1.2.6 纯化目的蛋白 将诱导培养的 2 L 重组菌以8 000 r/min 离心 30 min,加入Lysis Buffer重悬菌体,超声裂解至澄清后,收集上清,将其加入Ni柱中4 ℃孵育2 h。分别用含50,300,500 mmol/L咪唑浓度的洗脱缓冲液洗脱,收集洗脱液 ,加入 2 × SDS 上样缓冲液沸水浴 10 min 后,进行Western Blot 分析。
1.3 搜索PDB数据库及PPRV Nigeria75/1 H蛋白3D模型的模拟
利用NCBI数据库Blast功能分析PPRV Nigeria75/1 H蛋白。并于ExPAEy 网站SWIAA-MODLE模块寻找同源蛋白,在线建立蛋白3D模型。于NCBI数据库Structure功能查找蛋白表面模型。
1.4 PPRV Nigeria75/1 H蛋白二级结构预测
利用DNAStar软件提供的Protean模块,采用chou-Fasman算法和Garnier-Robson算法综合分析。
1.5 PPRV Nigeria75/1 H蛋白亲水性预测
通过DNAStar软件Protean模块,采用Kyte-Poolittle预测。
1.6 PPRV Nigeria75/1 H蛋白可塑性预测[8]
通过DNAStar软件Protean模块,采用Karplus-Schultz预测。
1.7 PPRV Nigeria75/1H蛋白表面可及性预测
通过DNAStar软件Protean模块进行Emini预测。
1.8 PPRV Nigeria75/1 H蛋白抗原性指数预测
通过DNAStar软件Protean模块进行Jameson-Wolf预测。
2 结果与分析
2.1 PPRV Nigeria75/1 H 基因PCR产物鉴定
PCR产物通过1%琼脂糖凝胶电泳鉴定,电压150 V经15 min,得到图1所示结果,目的条带大小1 830 bp,与预期相符。
M.DNA Marker DL2000;1.Nigeria75/1 株病毒H基因扩增产物。 M.DNA Marker DL2000;1.H gene product from Nigeria75/1 PPRV.
图1 Nigeria75/1 株病毒H基因PCR电泳
Fig.1 The electrophoresis of H gene from Nigeria75/1 PPRV
2.2 PPRV Nigeria75/1 H 基因克隆质粒的鉴定及测序
质粒 pMD18-T-H经BamHⅠ、Hind Ⅲ双酶切,产物通过1%琼脂糖凝胶电泳分析,得到图2所示结果,载体2 692 bp、目的基因1 830 bp ,大小符合预期;PCR所得H基因测序结果与 GenBank 上参考序列相似度为100%。
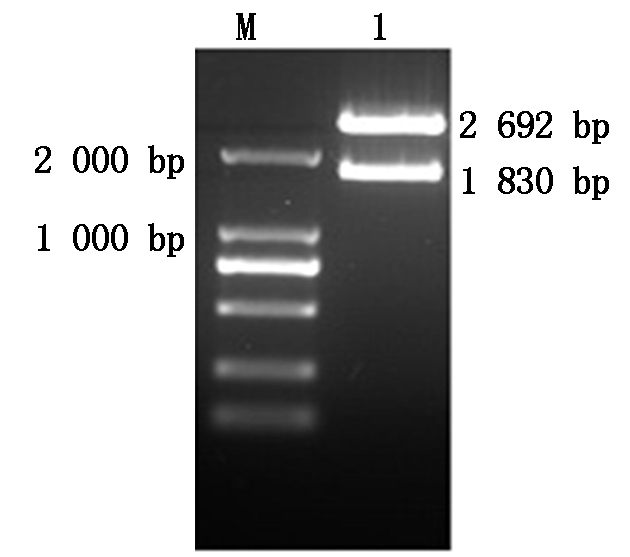
M.DNA Marker DL2000;1.质粒pMD18-T-H双酶切产物 (BamH Ⅰand Hind Ⅲ)。 M.DNA Marker DL2000;1.Products of plasmid pMD18-T-H digested by BamH Ⅰ and Hind Ⅲ.
图2 质粒 pMD18-T-H BamH Ⅰ/Hind Ⅲ双酶切鉴定
Fig.2 The electrophoresis of plasmid pMD18-T-H digested by BamH Ⅰ and Hind Ⅲ
2.3 重组表达质粒的鉴定
采用BamHⅠ、Hind Ⅲ对重组质粒pET-32a-H 双酶切,通过1%琼脂糖凝胶电泳分析,可得图3所示结果1 830 bp 的目的基因片段和5 900 bp 的载体片段,表明质粒 pET-32a-H 构建正确。
M.DNA Maker DL10000;1.质粒 pET-32a-H的双酶切产物。 M.DNA Marker DL10000;1.The electrophoresis of plasmid pET-32a-H.
图3 重组表达质粒 pET-32a-H 的BamH Ⅰ/Hind Ⅲ双酶切鉴定
Fig.3 The electrophoresis of plasmid pET-32a-H digested by BamH Ⅰ and Hind Ⅲ
2.4 目的蛋白表达的鉴定
2.4.1 SDS-PAGE 分析 诱导表达重组菌,SDS-PAGE鉴定可得图4结果:蛋白相对分子质量约67 ku,对照均无目的蛋白带。H蛋白以包涵体形式表达。
2.4.2 Western Blot 分析 通过抗 His 标签抗体的识别,在67 ku处有目的蛋白条带,结果与预测一致,且在上清中发现有少量可溶性表达(图5)。表明 H 蛋白获得正确表达,且具有良好的反应原性。
M.蛋白质 Marker;1.未诱导的空载体宿主菌;2.空载体宿主菌诱导 6 h;3.IPTG 诱导前重组表达菌;4.pET32a-H IPTG 诱导6 h后的重组表达菌。
M.Standard protein Marker;1.No induced vector host bacteria;2.Vector induced 6 h;3.pET32a-H host bacteria before IPTG induction; 4.pET32a-H host bacteria induced 6 h by IPTG.
图4 重组表达H蛋白的 SDS-PAGE分析
Fig.4 The electrophoresis of recombinant H protein analyzed by SDS-PAGE
M.蛋白质Marker;1.重组表达菌诱导6 h:2.重组表达菌诱导6 h上清;3.重组表达菌未诱导。
M.Standard protein Marker;1.pET32a-H host bacteria induced 6 h by IPTG; 2.Supernatant of pET32a-H host bacteria induced 6 h by IPTG;3.No induced pET32a-H host bacteria.
图5 重组表达H蛋白的 Western Blot 分析
Fig.5 Western Blot of expressed recombinant H protein
2.5 纯化产物的鉴定
经镍柱纯化后,Western Blot 分析,得到图6结果:
在约67 ku处有单一蛋白条带,表明纯化效果良好。
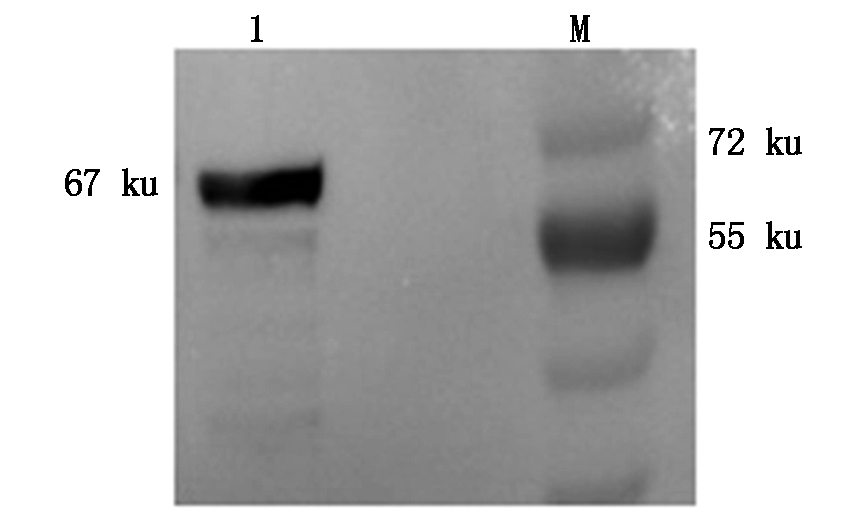
M.蛋白质 Marker;1.纯化的重组 PPRV H 蛋白。 M.Standard protein Marker;1.Purified recombinant H protein of PPRV.
图6 纯化的重组表达H蛋白的 Western Blot 分析
Fig.6 Western Blot profiles of purified PPRV H protein
2.6 PPRV Nigeria75/1 H 蛋白3D模型和蛋白表面
通过ExPAEy 网站SWIAA-MODLE功能在线建立同源蛋白3D模型(图7-A)。所得模型序列一致性为39.26%,H蛋白的三级结构主要由无规则卷曲、β折叠片及α螺旋组成。 β-折叠桶由β折叠片构成,其外部有大量亲水性侧链集团,丰富的非极性氨基酸残基侧链聚集其内部,疏水侧链构成一个封闭的疏水核心区域,使H 蛋白高级结构更加稳定。β-转角和无规卷曲组成松散的结构,更好接触周围极性环境,这些都使其更有可能成为抗原表位。并以同源蛋白为模型于NCBI数据库Structure查询相关蛋白表面模型(图7-B)。
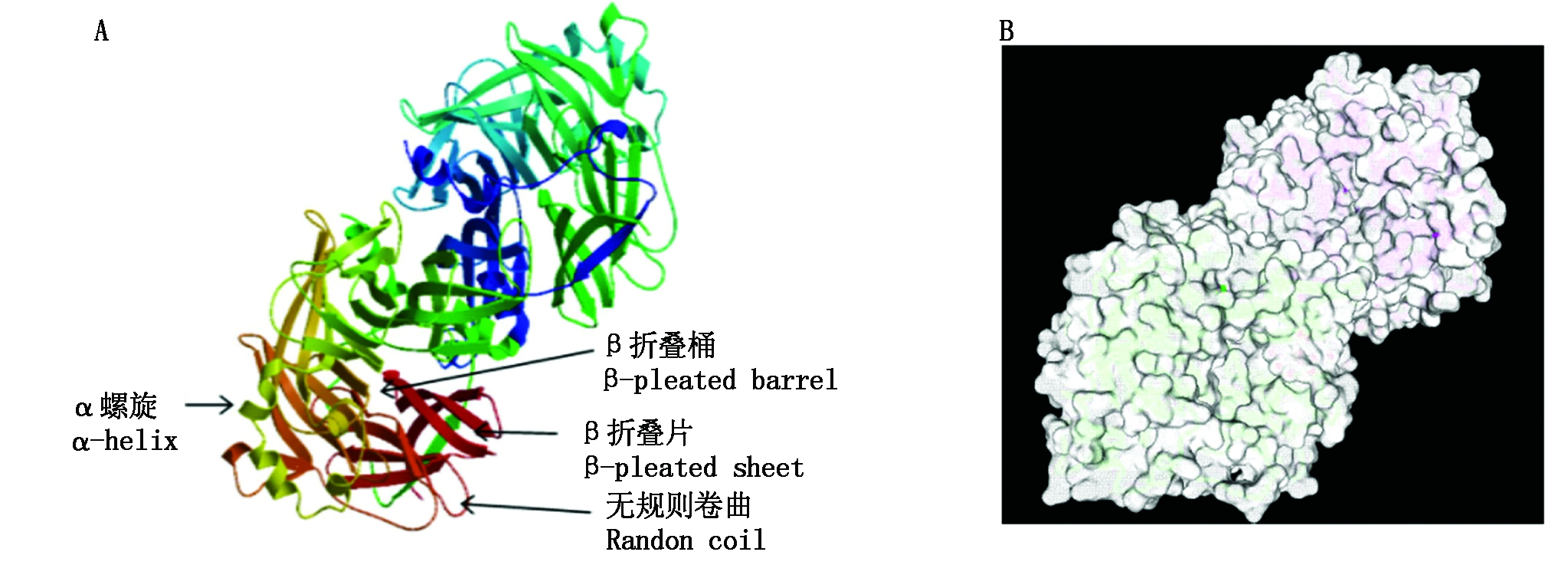
图7 PPRV Nigeria75/1 H蛋白3D模型(A)和表面模型(B)
Fig.7 The 3D model(A) and surface representation(B) of H protein from PPRV Nigeria 75/1
2.7 PPRV Nigeria75/1 H蛋白理化性质
经DNAStar软件Protean模块采用Chou-Fasman和Garnier-Robson算法综合分析可知,PPRV Nigeria75/1 H蛋白等电点为4.95,α螺旋占29.6%,β折叠片占47.9%,转角占12.5%,无规则卷曲占10.3%。
2.8 PPRV Nigeria75/1 H蛋白亲水性
如图8所示,PPRV Nigeria75/1 H蛋白亲水性区域分散分布。亲水性较高区域:1-12,13-27,29-34,69-80,81-92,123-150,151-156,168-178,193-197,210-216,232-247,278-287,311-317,356-366,370-379,386-395,399-408,440-450,487-496,501-511,520-527,530-535,543-553,556-566,571-580,589-598。
2.9 PPRV Nigeria75/1 H蛋白可塑性
PPRV Nigeria75/1 H可塑性较高区域(图9):5-8,14-16,18-21,26-31,32-35,66-78,80-85,86-95,107-114,119-124,126-131,142-148,168-177,183-188,190-196,224-228,236-248,281-286,301-307,308-319,328-335,340-348,353-356,359-364,368-381,387-393,395-408,421-424,445-450,457-463,473-476,484-494,500-513,518-522,532-536,544-551,560-565,588-602这些区段的柔韧性较高,发生扭曲、折叠的可能性较大。能形成丰富的二级结构。
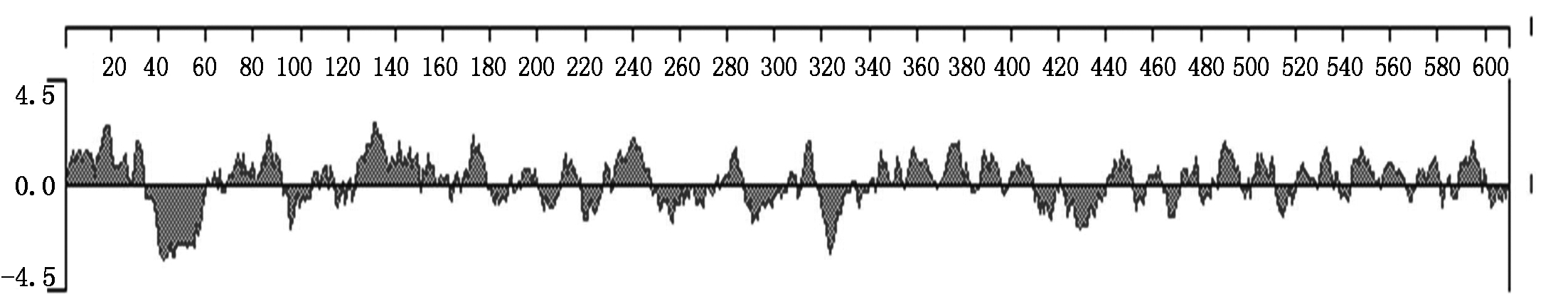
图8 Nigeria75/1 H蛋白亲水性分析
Fig.8 Analysis of hydrophilicity of Nigeria75/1 H
图9 Nigeria75/1 H蛋白可塑性分析
Fig.9 Analysis of flexible regions of Nigeria75/1 H
2.10 PPRV Nigeria75/1 H蛋白表面可及性分析
应用DNAStar软件,采用Emini方法分析H蛋白表面可及性结果如图10所示。其表面可及性较高的区段为:2-8,11-21,26-32,73-75,83-90,125-135,142-147,170-177,192-195,211-214,235-245,281-285,303-305,312-318,341-346,360-363,370-379,388-391,443-445,446-449,457-461,474-476,486-489,503-505,519-523,530-535,544-552,588-591,592-595。
图10 Nigeria75/1 H蛋白表面可及性分析
Fig.10 Analysis of surface accessibility of Nigeria75/1 H
2.11 PPRV Nigeria75/1 H蛋白抗原表位预测
DNAStar软件中Jameson-Wolf方案预测PPRV Nigeria75/1 H蛋白的抗原表位(图11)。可预测PPRV Nigeria75/1 H蛋白抗原表位可能位于2-9,12-33,69-93,99-106,107-114,118-151,157-164,169-179,183-187,208-217,223-231,235-248,261-266,279-287,292-296,301-305,307-320,328-332,340-351,352-365,370-393,395-399,400-410,419-426,443-450,457-461,471-479,486-495,500-514,518-524,530-537,543-551,557-567,589-601。
图11 Nigeria75/1 H蛋白抗原指数分析
Fig.11 Analysis of the antigenic index of Nigeria75/1 H
2.12 PPRV Nigeria75/1 H蛋白B细胞表位综合分析
综合PPRV Nigeria75/1 H蛋白亲水性、可塑性、表面可及性、抗原表位预测,以及二级结构分析可知,在5-8,14-16,73-75,83-90,125-131,142-147,170-177,236-245,281-285,312-317,360-363,370-379,388-391,445-449,487-489,503-505,520-522,532-535,544-551,592-595这些区段中亲水性、可塑性、表面可及性、抗原表位可能性均较高。且在这些区段均位于α折叠、β螺旋等二级结构中。这使得所预测区段有更大几率成为抗原表位。
3 讨论
虽然用灭活苗免疫动物已可有效防治PPR,但是出于安全考虑,多年来人们致力于安全高效的小反刍兽疫亚单位疫苗[9]。PPRV H 蛋白诱发宿主体液免疫产生中和抗体,体外表达H蛋白及其抗原表位预测是为建立 PPRV 血清学检测方法和制备亚单位疫苗及建立提供基础[10]。
基于原核表达系统表达量高、简便、方便纯化等特点[11-15]。本试验将PPRV H基因克隆于原核表达载体pEG-32a,在大肠杆菌中得到有效表达,主要以包涵体的形式存在,有部分可溶性表达。本研究中表达载体 pET-32a带有His 标签,对目的蛋白免疫原性及功能无影响。
目前,笔者主要通过多维核磁共振技术与X射线晶体学技术研究蛋白高级结构。但现有技术远不能追上探索蛋白质结构功能的步伐。前者不能检测分子量大于30 ku的蛋白质,后者要求得到高标准的蛋白晶体。因此,理论模拟和结构预测十分重要。
随着生物信息学的发展,分析蛋白质的软件多种多样。Hopp等[16]在20世纪80年代首次提出亲水性参数对抗原表位预测的方法。现被广泛认可的方法主要是:二级结构分析、亲水性方案、可塑性方案、可及性方案、抗原性方案[17-18]。B细胞抗原表位的预测需要综合上述5种方案,这是因为蛋白抗原氨基酸残基可分为亲水残基和疏水残基。亲水残基位于蛋白表面,与抗原位点紧密相关[19]。但疏水残基与抗原表位的形成也有联系。蛋白抗原构象不是刚性不变的,其多肽骨架有一定的活动性[20],活动性强的氨基酸残基形成抗原表位的可能性较大。另外,在蛋白质二级结构中α-螺旋、β-折叠片位于蛋白质的疏水区域或蛋白质内部,化学键能较高、结构规则;而β转角和无规卷曲,结构突出,亲水性强,处于蛋白质的表面与抗原表位联系紧密[21]。每种方法具有其优势的同时也存在缺陷。总之,B细胞表位需与B细胞抗原受体(bcr)和抗体结合,故要位于蛋白表面。约由4~6个氨基酸残基或者糖基组成。具有柔韧性,构象可发生变化。
综上所述,本试验成功表达了PPRV H蛋白,得到可溶性表达并纯化。采用同源模建的方法来分析PPRV Nigeria75/1 H的高级结构,提供PPRV Nigeria75/1 H蛋白的直观分子模型,使H蛋白抗原表位预测更准确。结果可为建立针对PPRV的检测方法和疫苗的研制提供依据。
[1] Ismail I M,House J. Evidence of identification of peste des petits ruminants from goats in Egypt[J]. Archiv Fur Experimentelle Velerinarmedizin,1990,44(3):471-474.
[2] 李林杰,谢 军,徐文凯,等. 小反刍兽疫病毒的研究进展[J]. 黑龙江畜牧兽医,2018(1):66-70.
[3] 邢泽黎,朱利塞,郭昌明. 吉林省某羊群暴发小反刍兽疫[J]. 中国兽医科学,2014,5(15):1005-4545.
[4] Diallo A,Minet C,Goff C L,et al. The threat of peste des petits ruminants:progress in vaccine development for disease control[J]. Vaccine,2007,25(30):5591-5597.
[5] 王秋霞,杨香芳,窦永喜. 小反刍兽疫病毒样颗粒装配的研究[J]. 中国兽医科学,2014,44(12):1297-1303.
[6] Sinnathamby G,Gj R,Rajasekhar M,et al. Immune reponses in goats to recombinant hemagglutinin-neuraminidase glycoprotein of Peste des petits ruminants virus identification of a T cell determinant[J]. Vaccine,2001,19(32):4816-4823.
[7] 信爱国,杨仕标,向文彬. 小反刍兽疫研究进展[J].中国预防兽医报,2003,25(2):152-155.
[8] Karplus P A,Schulz G E. Prediction of chain flexibility in proteins[J]. Naturwissenschaften,1985,72(4):212-213.
[9] 李海侠,毛旭虎. 蛋白质抗原表位研究进展[J]. 微生物学免疫学进展,2007,35(1):54-58.
[10] Wang Y,Liu G Q,Shi L J,et al. Immune responses in mice vaccinated with a suicidal DNA vaccine expressing the hemagglutinin glycoprotein from the Peste des petits ruminants virus[J]. Journal of Virological Methods,2013,193(2):525-530.
[11] 闫飞虎,王化磊,邱泊宁,等. 小反刍兽疫病毒H蛋白的原核表达及其多克隆抗体的制备[J]. 中国生物制品学杂志,2016,29(6):576-581.
[12] Zhang X,Zhao W,Li C,et al.
Differential expression of miR-34c and its predicted target genes in testicular tissue at Different development stages of swine[J]. Asian-Australas J Anim Sci,2015,28(11):1532-1536.
[13] Tanaka T,Kanatsu-Sshnohara M,Shinohara T. The CDKN1B-RB1-E2F1 pathway protects mouse spermatogonial stem cells from genomic damage [J]. J Reproud Dev,2015,61(4):305-316.
[14] Kwom J T,Jin S,Choi H,et al. Identification and characterization of germ cell genes expressed in the F9 testicular teratoma stem cell line[J]. PLoS One,2014,9(8):e103837.
[15] Kumar N,Maherchandani S,Kashyap S K,et al. Peste des petits ruminants virus infection of small ruminants:a comprehensive review[J]. Viruses,2014,6(6):2287-2327.
[16] Hopp T P,Woods K R. Prediction of protein antigenic determinants from amino acid sequences[J]. Proc Natl Acad Sci USA,1981,78(6):3824-3828.
[17] 王艳华,张德林,殷 宏,等. 抗原表位预测方法的研究进展[J]. 中国兽医科学,2009,39(10):938-940.
[18] 高位相. 小反当兽疫病毒H蛋白的表达及其B细胞线性表位筛选[D]. 合肥: 安徽农业大学,2015.
[19] 郑 伟. 蛋白质结构研究及B细胞表位预测[D]. 天津: 南开大学,2014.
[20] 周建华,丛国正,高闪电,等. FMDV OA/58病毒株VP1蛋白结构构建与B细胞抗原表位的预测[J].华北农学报,2007,22(4):176-179
[21] ApostolopoulosV,Yu M,Corper A L,et al. Crystal structure of a noncanonical low affinity peptide complexed with MHC class Ⅰ:a new approach for vaccine design[J]. Journal of Molecular Biology,2002,318(5):1293-1305.