水稻是世界上重要的粮食作物之一[1],是我国主要的粮食作物[2]。水稻栽培区的土壤盐碱化日趋严重。在盐碱稻作区的水稻在苗期时对盐碱危害更为敏感,故而,提高水稻在苗期时的耐盐性及耐碱性对盐碱稻作区的作用重大。近年来,水稻耐盐QTL研究取得了较大进展,大量与耐盐性状有关的QTL相继被报道,但对于根系相关性状的研究相对较少,而水稻根系对于水稻整个生育期的生长发育有着十分重要的作用[3]。大多数研究利用群体作图进行遗传连锁图谱的绘制,与群体耐盐碱性鉴定的结果相结合,对耐盐碱相关基因进行初步定位。以便有效地利用种质资源,挖掘与水稻耐盐碱相关的优质基因,为水稻耐盐相关基因及耐碱相关基因的精确定位以及分子标记辅助育种提供理论依据。根系既能固定植株还能吸收水分和养分,并且还是水稻合成内源激素的主要器官,是水稻重要的组织器官[4-5]。并且,根系还是水稻地上部分与地下部分的物质交换与信息交流的系统之一[6],在盐碱胁迫下水稻苗期根数和根长能较直观地反映出水稻耐盐碱的强度[7]。顾兴友等[8]利用RLFP标记以Pokkali及Peta杂交构建的回交群体鉴定了与水稻苗期鲜干质量比、Na+含量以及盐害级别相关的5个QTL。Lee等[9]利用群体定位到与水稻苗期耐盐相关的2个QTL。林鸿宣等[10]利用RIL群体定位到4个与秧苗存活天数相关的QTL。Kim等[11]以回交导入系群体为材料定位到8个与盐胁迫条件下苗高、叶片面积及鲜质量相关的QTL。杨静等[12]以Lemont和特清的双向导入系为作图群体定位到了18个与苗期耐盐相关性状有关的QTL。许多研究已经证明,水稻的耐盐性基因和耐碱性基因具有遗传多样性,并利用不同的群体或不同的品种对水稻耐盐性及水稻的耐碱性相关的性状进行QTL定位,然而目前研究盐、碱条件下产量性状的QTL较多,在苗期根系相关性状研究却鲜有报道,水稻的根系在水稻的生育过程中起重要作用。因此,在盐和碱胁迫条件下研究水稻苗期根系表型性状有重要意义。
本试验以东农425和长白10号衍生的重组自交系群体为遗传研究材料,对水稻苗期时的根数和根长在盐胁迫和碱胁迫条件下进行QTL分析,深入挖掘水稻耐盐碱性的QTL,为水稻耐盐碱性相关基因的QTL精细定位和水稻耐碱品种的选育提供理论和科学支持。
1 材料和方法
1.1 试验材料
本试验以优质粳稻品种东农425(DN425)为母本,以耐盐、耐碱性强的粳稻品种长白10号(CB10)为父本配置杂交组合,经过多次自交,通过单粒传获得的F7 群体作为试验材料。
1.2 水稻苗期耐盐碱性鉴定
本试验在东北农业大学国家水稻区域技术创新中心的温室进行,共设3种处理:自然条件、盐胁迫条件和碱胁迫条件。首先将处理材料进行预处理,打破休眠:选取50粒籽粒饱满的种子在50 ℃的条件下烘干3 d;表面消毒:使用0.5%的NaClO处理10 min进行表面消毒;浸种:用清水冲洗干净后,在28 ℃的条件下浸种2 d。然后在塑料秧盘内选取每个家系和亲本种子的20粒露白发芽一致的种子进行播种,放置在20 L盛有水的塑料盆内,使用pH值在5.5左右的清水培养至三叶一心期。盐胁迫时将清水换成含50 mmol/L NaCl的Yoshida 营养液[13]预处理3 d后,再将NaCl溶液的浓度增至140 mmol/L,营养液的pH值维持在5.5左右。碱胁迫时将清水换成0.05% Na2CO3 的Yoshida营养液预处理3 d后,再将Na2CO3溶液的浓度增至0.15%,使溶液的pH值维持在8.5左右,每5 d换一次营养液[14]。在处理12 d后,收获根部,测出群体及亲本的根数及根长。
在自然条件、盐胁迫和碱胁迫3个环境:E1(2015年对照)、E2(2015 年盐处理)、E3(2015年碱处理),每株系随机选取非边株的5个单株。测得DN425和CB10及F7 群体的2项根系性状:根数(Root number,RN)、根长(Root length,RL),以3次重复的平均值作为统计单元。
1.3 根系相关性状数据统计分析
使用Excel 2016对根系的根数和根长数据进行处理,再用SPSS(Version 17.0 for Windows)对根数和根长进行统计及相关性检验分析。
1.4 构建遗传连锁图谱并定位QTL
本试验叶片的DNA使用由天根生化科技(北京)有限公司提供的DNA提取试剂盒提取。基于本试验已检测到的多态性SSR标记102对,具有多态性的Indel引物113对,用于构建连锁图谱;由华大基因合成SSR引物,由新海基因合成Indel引物。利用两亲本间具有多态性的215对引物对F7的180个单株DNA进行PCR扩增,PCR反应和电泳方法参考邢军等[14]方案,利用Mapchart 2.2软件绘制遗传连锁图谱。取2015年耐盐碱性根长和根数数据的平均值进行分析。利用QTL IciMapping v3.3软件的完备区间作图法(ICIM)进行加性QTL定位,设定LOD值为2.0[15]。QTL命名参照McCouch等[16]的方法。
2 结果与分析
2.1 盐、碱胁迫条件下水稻苗期根系性状的表型值分析
2.1.1 水稻耐盐根数和根长的表型分析 在盐胁迫、碱胁迫和自然条件下亲本及RIL群体单株根数和根长的表型分析(表1)。在自然条件下母本DN425和父本CB10的根数和根长存在极显著差异(P<0.01),且根数DN425高于CB10,根长CB10高于DN425。盐胁迫条件下根数、根长在两亲本之间表现出极显著差异,且根数DN425高于CB10,根长CB10高于 DN425。自然条件与盐胁迫的差值下,根数、根长在两亲本之间表现出极显著差异,且根数、根长DN425 高于CB10。DN425与CB10间根数和根长的遗传差异大,在耐盐性方面有明显差别,为QTL分析提供了良好的遗传背景。
表1 水稻苗期亲本及重组RIL群体在不同环境下根系的表型分析
Tab.1 Phenotypic analysis of different environments root for RIL population and parents at the seedling stage in rice
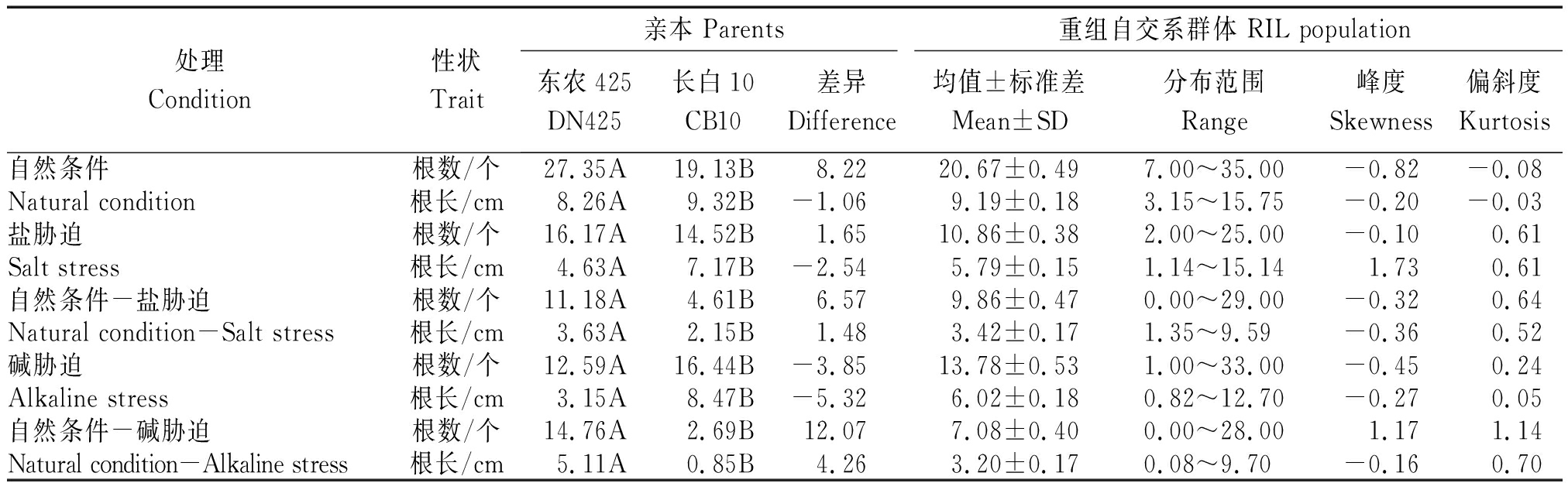
处理Condition性状 Trait亲本 Parents重组自交系群体 RIL population东农425DN425长白10CB10差异Difference均值±标准差Mean±SD分布范围Range峰度Skewness偏斜度Kurtosis自然条件根数/个27.35A19.13B8.2220.67±0.497.00~35.00-0.82-0.08Natural condition 根长/cm8.26A9.32B-1.069.19±0.183.15~15.75-0.20-0.03盐胁迫根数/个16.17A14.52B1.6510.86±0.382.00~25.00-0.100.61Salt stress根长/cm4.63A7.17B-2.545.79±0.151.14~15.141.730.61自然条件-盐胁迫根数/个11.18A4.61B6.579.86±0.470.00~29.00-0.320.64Natural condition-Salt stress根长/cm3.63A2.15B1.483.42±0.171.35~9.59-0.360.52碱胁迫根数/个12.59A16.44B-3.8513.78±0.531.00~33.00-0.450.24Alkaline stress根长/cm3.15A8.47B-5.326.02±0.180.82~12.70-0.270.05自然条件-碱胁迫根数/个14.76A2.69B12.077.08±0.400.00~28.00 1.171.14Natural condition-Alkaline stress根长/cm5.11A0.85B4.263.20±0.170.08~9.70-0.160.70
注:不同大写字母表示在0.01 水平上显著相关。
Note:Different capital letter indicate significance at the level of 1%.
2.1.2 水稻耐碱根数和根长的表型分析 碱胁迫条件下根数、根长在两亲本之间表现出极显著差异,且根数、根长CB10高于DN425。自然条件与碱胁迫的差值下,根数、根长在两亲本之间表现出极显著差异,且根数、根长DN425高于CB10。DN425与CB10在根数和根长间的遗传差异大,在耐碱性方面有明显差别,为之后的QTL分析提供良好的遗传背景。
在盐胁迫条件和碱胁迫条件下,DN425、CB10以及RIL群体的的根数和根长都受到明显影响,其中在碱胁迫条件下水稻更为敏感,苗期根系的根数和根长降低更明显。其中CB10显示出了较强的耐盐性以及耐碱性,其根数和根长的稳定性明显优于DN425。在盐胁迫和碱胁迫条件下,水稻DN425的根数和根长在自然条件与碱胁迫条件下差值的平均值高于在自然条件与盐胁迫条件下差值的平均值,由此可知,DN425对碱胁迫条件比CB10更为敏感,即在碱胁迫的条件下DN425的根数和根长受到了更大程度的限制。除了盐胁迫下的根数以外,RIL群体根数和根长在其他条件下都处在DN425与CB10间,并且根数和根长的变异范围广,具有很明显的超亲分离现象,峰度、偏斜度的绝对值基本上都小于或接近1.0。对数据进行正态分布的适合性检验,表明根数和根长基本都符合正态分布,与数量性状的遗传特性相符合(图 1)。
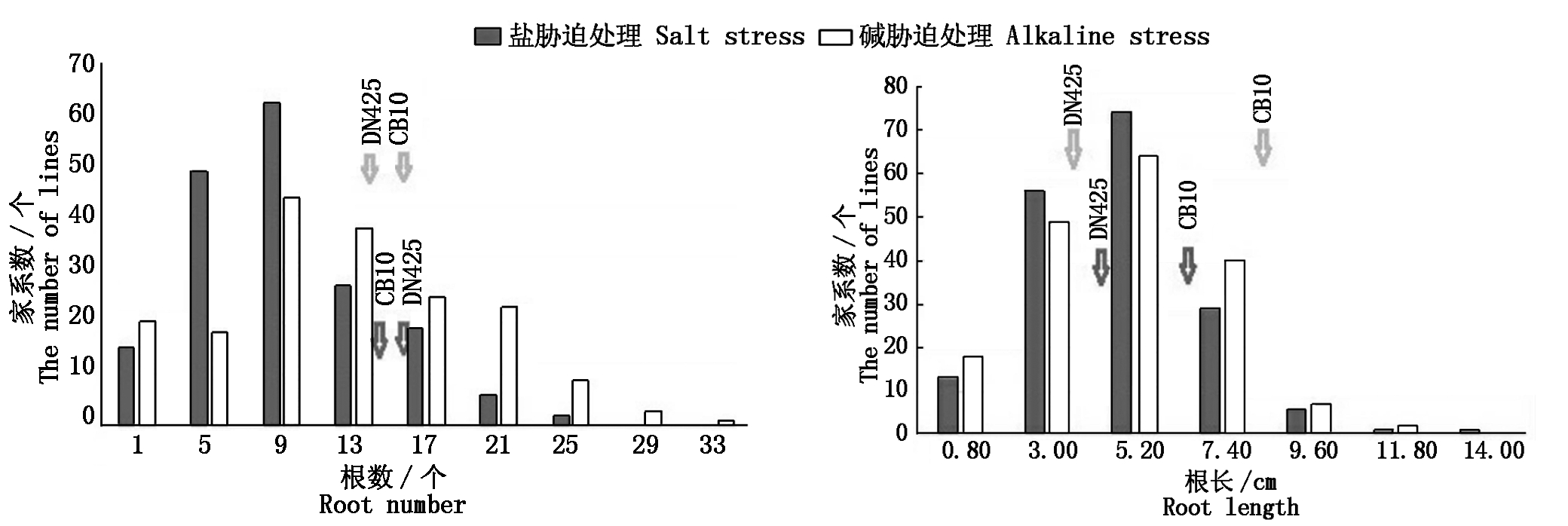
图1 盐胁迫和碱胁迫下水稻根数根长的分布
Fig.1 Distribution of root number and root length under salt stress and alkaline stress in rice
2.2 水稻盐胁迫和碱胁迫条件下根数、根长的相关分析
通过对水稻盐、碱胁迫和自然条件下根数和根长的相关性分析可知(表 2),在自然条件下,根数与根长呈极显著正相关;在盐胁迫条件下,根数与根长呈极显著正相关;在碱胁迫条件下,根数与根长呈极显著正相关。
表 2 性状间的相关系数
Tab.2 Correlation coefficients among traits
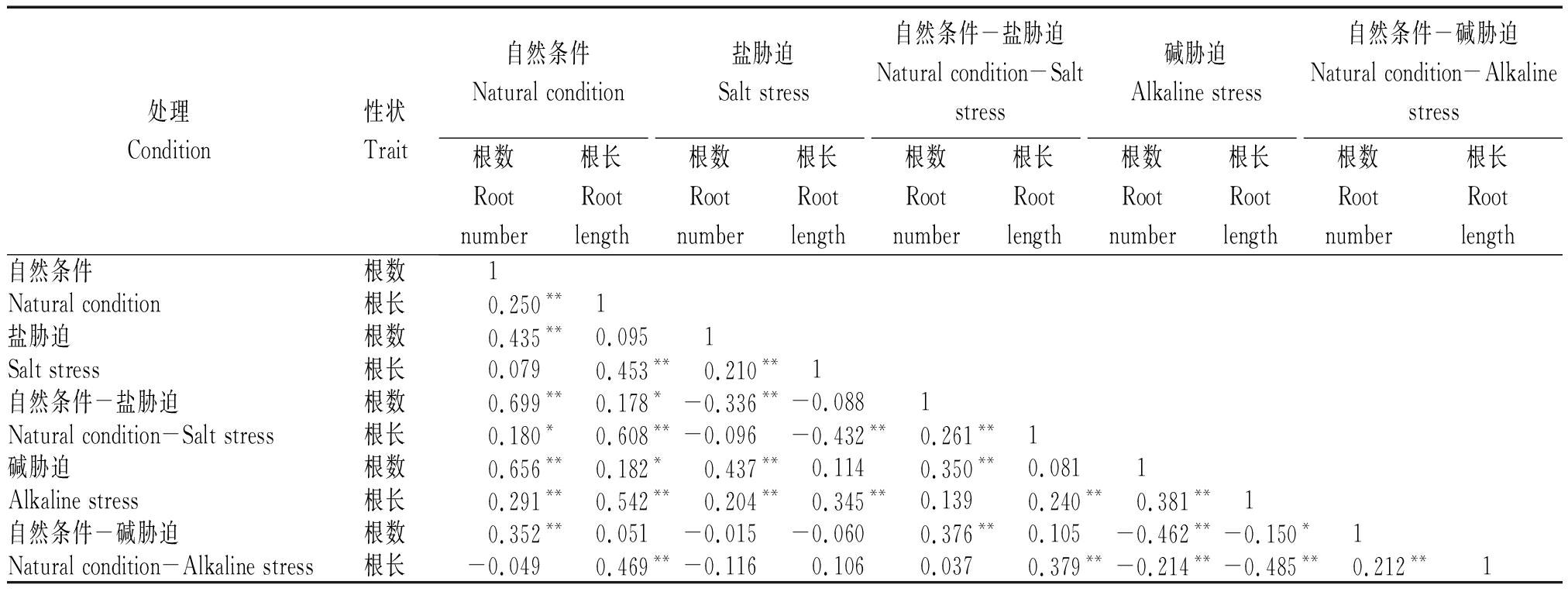
处理Condition 性状Trait自然条件 Natural condition盐胁迫 Salt stress自然条件-盐胁迫Natural condition-Salt stress碱胁迫 Alkaline stress自然条件-碱胁迫Natural condition-Alkaline stress根数Rootnumber根长Rootlength根数Rootnumber根长Rootlength根数Rootnumber根长Rootlength根数Rootnumber根长Rootlength根数Rootnumber根长Rootlength自然条件根数1Natural condition 根长0.250**1盐胁迫根数0.435**0.095 1Salt stress根长0.0790.453**0.210**1自然条件-盐胁迫根数0.699**0.178*-0.336**-0.088 1Natural condition-Salt stress根长0.180*0.608**-0.096-0.432**0.261**1碱胁迫根数0.656**0.182*0.437**0.1140.350**0.081 1Alkaline stress根长0.291**0.542**0.204**0.345**0.1390.240**0.381**1自然条件-碱胁迫根数0.352**0.051-0.015-0.0600.376**0.105-0.462**-0.150* 1Natural condition-Alkaline stress根长-0.0490.469**-0.1160.1060.0370.379**-0.214**-0.485**0.212**1
注:*和**分别表示在 0.05 和 0.01 水平上显著相关。
Note:*and**indicate significance at the level of 5% and 1% .
2.3 水稻盐、碱胁迫条件下根数和根长相关性状的QTL分析
使用QTL IciMapping v3.3完备区间作图法(ICIM)对水稻的耐盐性和耐碱性的根数和根长进行加性QTL的定位分析,结果见表3。在自然条件、盐胁迫条件、碱胁迫条件下检测到了18个具有加性效应的QTL,分别分布在第1,2,3,4,5,7,8,9,10号染色体上,LOD值为2.01~3.35,表型变异的贡献率为6.02%~20.06%。
在自然条件下,检测到4个与根数相关的QTL,分布在第4,7,10染色体上,分别位于第4号染色体的Indel31~Indel38区间内,第7号染色体的RM182~RM346、Indel61~Indel65区间内,第10号染色体的RM474~Indel89区间内,其中,qNRN7-2贡献率最大,为12.46%。qNRN10的增效基因来自CB10,其余的qNRN4、qNRN7-1、qNRN7-2的增效基因皆来自于DN425,且未检测到与根长相关的QTL。
在盐胁迫条件下,检测到5个与根数、根长相关QTL,分别分布在第1,3,5,7和9号染色体上。其中,共检测到4个与根数相关的QTL,分别位于1号染色体的RM529~Indel1区间内、3号染色体的RM1324~RM517区间内、7号染色体的RM560~RM182区间内和9号染色体的Indel77~RM215区间内,其中,qSRN3贡献率最大,为20.06%, qSRN3和qSRL5的增效等位基因皆来自于CB10;qSRN1、qSRN7、qSRN9 的增效等位基因均来自于DN425。
在碱胁迫条件下,共检测到3个与根数和根长相关的QTL,分别分布在第2,3,5 染色体上,其中,位于第2号染色体的Indel20~RM207区间内qARN2为与根数相关的QTL,贡献率为12.99%,其增效等位基因来自 CB10,与根长相关的QTL分别位于第3号染色体的Indel29~RM232区间内和第5号染色体的Indel45~Indel43 区间内,qARL3和qARL5贡献分别为7.04%,8.88%,增效等位基因分别来自DN425和CB10。
在自然条件与盐胁迫的差值条件下,共检测到4个与根根数、根长相关的QTL,分布在第1,7,8号染色体上。检测到了2个与根数相关的QTL,都位于8号染色体上,分别位于RM1384~Indel69和Indel74~Indel71区间内,贡献率为6.99%和14.01%,其中,qN-SRN8-1的增效等位基因来自于DN425,qN-SRN8-2的则来自于CB10;并且检测到了2个与根长相关的QTL,位于1号染色体的Indel8~Indel4区间内和7号染色体的Indel62~RM418 区间内,贡献率分别为14.12%和9.40%,其中qN-SRL1的增效等位基因来自CB10,qN-SRL7的增效等位基因来自DN425。
在自然条件与碱胁迫的差值条件下,共检测到2个与根数、根长相关的QTL,分布在第3,10号染色体上。第3号染色体上的QTL在区间RM1230~RM1352内与根数相关,贡献率为6.02%,qN-ARN3的增效等位基因来自DN425;检测到1个与根长相关的QTL,位于第10号染色体的Indel94~Indel88区间内,贡献率为7.45%,其中,qN-ARL10的增效等位基因来自CB10。
表3 水稻苗期耐盐碱性的QTL及遗传效应
Tab.3 QTL and gentic effects of salt and alkali tolerance
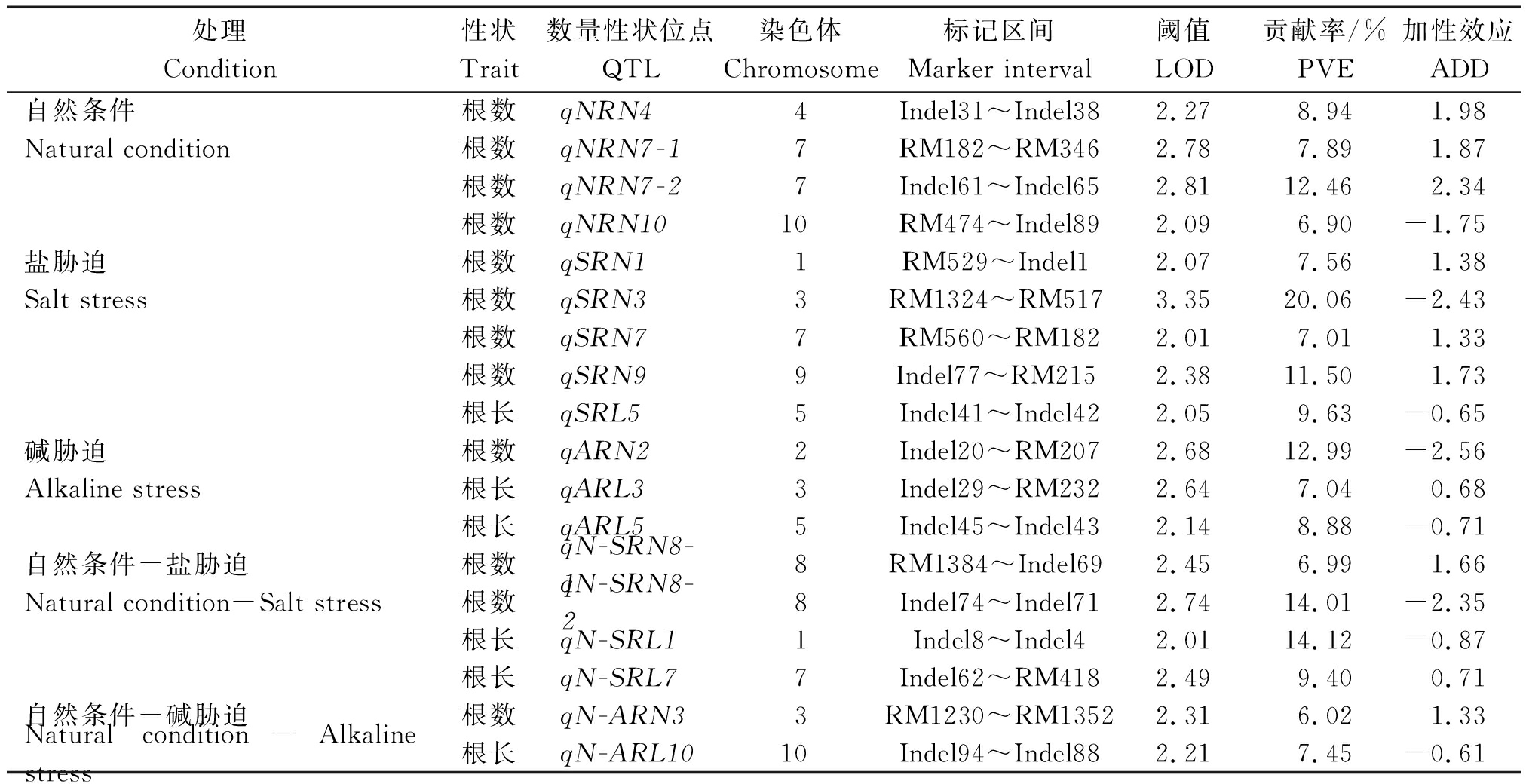
处理Condition 性状Trait数量性状位点QTL染色体Chromosome标记区间Marker interval 阈值LOD 贡献率/%PVE 加性效应ADD自然条件根数qNRN44Indel31~Indel382.278.941.98Natural condition根数qNRN7-17RM182~RM3462.787.891.87根数qNRN7-27Indel61~Indel652.8112.462.34根数qNRN1010RM474~Indel892.096.90-1.75盐胁迫根数qSRN11RM529~Indel12.077.561.38Salt stress根数qSRN33RM1324~RM5173.3520.06-2.43根数qSRN77RM560~RM1822.017.011.33根数qSRN99Indel77~RM2152.3811.501.73根长qSRL55Indel41~Indel422.059.63-0.65碱胁迫根数qARN22Indel20~RM2072.6812.99-2.56Alkaline stress根长qARL33Indel29~RM2322.647.040.68根长qARL55Indel45~Indel432.148.88-0.71自然条件-盐胁迫根数qN-SRN8-18RM1384~Indel692.456.991.66Natural condition-Salt stress根数qN-SRN8-28Indel74~Indel712.7414.01-2.35根长qN-SRL11Indel8~Indel42.0114.12-0.87根长qN-SRL77Indel62~RM4182.499.400.71自然条件-碱胁迫根数qN-ARN33RM1230~RM13522.316.021.33Natural condition-Alkaline stress根长qN-ARL1010Indel94~Indel882.217.45-0.61
注:QTL命名中的第一个大写字母N、S、A代表自然条件、盐胁迫条件、碱胁迫条件;N-S、N-A则代表自然条件与盐胁迫、碱胁迫的差值。
Note:The first capital letter N,S and A represent natural conditions,salt stress and alkaline stress;And N-S and N-A represent the difference between natural conditions and salt stress and alkali stress.
2.4 构建遗传连锁图谱
利用125对SSR引物对RIL群体进行基因型鉴定,构建遗传连锁图谱,其总长为2 387.33 cM,覆盖水稻12条染色体,标记间平均距离为11.10 cM(图2)。
3 讨论与结论
水稻的耐盐性以及耐碱性是复杂的数量性状,在试验中测得的根数与根长也易受到外界环境条件的影响。所以,在对比研究自然条件与盐胁迫条件和碱胁迫条件下其QTL的表现规律时,必须将与本试验无关的环境因子对结果的影响消除。所以,本试验是在温室内进行的,并且在试验过程中保证它们具有相同的土肥条件。
在之前的苗期耐盐性及耐碱性的研究当中,CB10比DN425有较强的耐盐、碱性[17] ,而由这2个品种建立起来的RIL群体也被证明是一个有效的作图群体[14]。因此,在本试验中,大多数QTL提供加性效应的等位基因来源于亲本CB10。
通过对自然条件与盐、碱胁迫条件下总共检测到18个QTL,自然条件下检测到4个(36. 19 %),盐胁迫环境中检测到5个(55.85%),碱胁迫条件中检测到3个(28.91%),在自然条件与盐胁迫差值下检测到4个(44.52%),在自然条件与碱胁迫差值下检测到2个(13.47%),说明不同的QTL在各自的环境中更为有效。检测出在7号染色体上包含多个QTL的位点,在该染色体上定位到的QTL中,qNRN7-1和qSRN7 2个QTL的标记区间相邻,分别位于第7号染色体的RM182~RM346区间和RM560~RM182区间,贡献率为7.89% 和 7.01%。在自然条件下检测到qNRN7-2位于7号染色体上,贡献率为12.46%;在盐胁迫下检测到qSRN3位于3号染色体上,贡献率为20.06%;在碱胁迫下检测到qARN2位于 2 号染色体上,贡献率为12.99%;在自然条件与盐胁迫的差值下检测到位于第 8 号染色体的qN-SRN8-2和位于1号染色体的qN-SRL1,贡献率分别为14.01%和14.12%;在自然条件与碱胁迫的差值下检测到位于第10号染色体的qN-ARL10,贡献率为7.45%。在所统计的10个性状中,盐胁迫下检测到的QTL最多,且总贡献率达到了55.85%,其中位于3号染色体上的qSRN3,贡献率达到20.06%,且被证实来源于CB10。

▲.自然条件下的根数的QTL;![]() .盐胁迫下根数和根长的QTL;
.盐胁迫下根数和根长的QTL;![]() .碱胁迫下的根数和根长的QTL;
.碱胁迫下的根数和根长的QTL;![]() .自然条件与盐胁迫的差值下的根数和根长的QTL;
.自然条件与盐胁迫的差值下的根数和根长的QTL;![]() .自然条件与碱胁迫的差值下的根数和根长的QTL.
.自然条件与碱胁迫的差值下的根数和根长的QTL.
▲.QTL for root number under natural conditions;![]() .QTL for root number and root lenght under salt stress;
.QTL for root number and root lenght under salt stress;![]() .QTL for root number and root length under alkaline;
.QTL for root number and root length under alkaline;![]() .QTL for root number and root length under difference between natural and salt;
.QTL for root number and root length under difference between natural and salt;![]() .QTL for root number and root length under difference between natural and alkaline.
.QTL for root number and root length under difference between natural and alkaline.
图2 盐、碱胁迫条件下水稻苗期根数、根长QTL在染色体上的分布
Fig.2 Root number and root length QTL under salt and alkaline stress at the seedling stage in rice on chromosomes
本研究在相同的SSR标记或比较图谱的基础上,将与根数和根长相关的QTL与耐盐耐碱QTL结果进行比较。在自然条件下,第7号染色体一起定位到的与根数相关的qNRN7-1与影响相对分蘖数的qRTN7-4和影响结实率的qRSS7-2 定位在相邻区间[18-19];另外,在第10号染色体上定位到的qNRN10与影响叶绿素含量相对碱害率的qRCC10定位在相邻区间[20]。在盐胁迫下,本研究定位到的qSRN7与影响千粒质量的qRGW7定位在相邻区间[19],与影响株高的qPH7-1定位在相同区间[18];另外,本研究定位到的qSRN1与影响相对分蘖数的qRTN1在相邻区间[18];在第3号染色体定位到的qSRN3与影响单株有效穗数的qPN3-2定位到相邻区间[19]。在碱胁迫下,qARL3与影响碱胁迫下根部Na+浓度的qARNC3以及影响碱胁迫下根部Na+ /K+ 的qARNK3定位在相邻区间[14]。在自然条件与盐胁迫的差值条件下,定位在第8号染色体的qN-SRN8-1与影响叶相对Na+含量的qRLNC8、与影响叶Na+/K+ 的qLNa/K8 定位在相邻区间[18]。在自然条件与碱胁迫的差值条件下,定位在第3号染色体的qN-ARN3与影响单株有效穗数的qPN3-3定位在相邻区间[19]。结果表明,QTL可以在不同的环境、不同的遗传背景、不同的指标下进行检测,这些QTL稳定可靠,对水稻的耐盐性和耐碱性研究具有重要的意义。
在盐胁迫和碱胁迫条件下,水稻的根数和根长都受到明显影响,其中在碱胁迫条件下水稻更为敏感,苗期根系的根数和根长降低更明显。从QTL定位的结果也能反映出在盐胁迫和碱胁迫条件下根长和根数遗传机理存在联系但也存在差异,还需进一步结合其他根系性状进行研究。
[1] 公婷婷. 中国水稻起源,驯化及传播研究[D]. 北京:中央民族大学,2017.
[2] 朱德峰,张玉屏,陈惠哲,等. 中国水稻高产栽培技术创新与实践[J]. 中国农业科学,2015,48(17):3404-3414.
[3] 孙 凯,胡丽燕,张 伟,等. 水稻根系泌氧对土壤微生物区系及氮素矿化影响的研究进展[J]. 生态学杂志,2016,35(12):3413-3420.
[4] 徐国伟,王贺正,翟志华,等. 不同水氮耦合对水稻根系形态生理、产量与氮素利用的影响[J]. 农业工程学报,2015,31(10):132-141.
[5] 梁永书,周军杰,南文斌,等. 水稻根系研究进展[J]. 植物学报,2016,51(1):98-106.
[6] 陈 晨,龚海青,张敬智,等. 水稻根系形态与氮素吸收累积的相关性分析[J]. 植物营养与肥料学报,2017,23(2):333-341.
[7] 王小虎,方云霞,张 栋,等. 水稻根基粗适宜取样量及其与抗倒伏性状的相关分析[J]. 江苏农业科学,2016,44(5):85-89.
[8] 顾兴友,梅曼彤,严小龙,等. 水稻耐盐性数量性状位点的初步检测[J]. 中国水稻科学,2000,14(2):2-7.
[9] Lee S Y,Ahn J H,Cha Y S,et al. Mapping of quantitative trait loci for salt tolerance at the seedling stage in rice[J]. Molecules and Cells,2006,21(2):192-196.
[10] 林鸿宣,柳原城司,庄杰云,等.应用分子标记检测水稻耐盐性的QTL[J].中国水稻科学,1998,12(2):72-78.
[11] Kim D M,Ju H G,Kwon T R,et al. Mapping QTLs for salt tolerance in an introgression line population between Japonica cultivars in rice[J]. Journal of Crop Science and Biotechnology,2009,12(3):121-128.
[12] 杨 静,孙 勇,程立锐,等. 利用双向导入系群体检测遗传背景对耐盐QTL定位的影响[J]. 作物学报,2009,35(6):974-982.
[13] Yoshida S,Forno D A,Cock J H,et al. Laboratory manual for physiological studies of rice[M].2 nd edition.Philippines:The International Rice Research Institute Philippines,1972:57-63.
[14] 邢 军,常汇琳,王敬国,等. 盐,碱胁迫条件下粳稻Na+,K+浓度的QTL分析[J]. 中国农业科学,2015,48(3):604-612.
[15] 龚继明,郑先武,杜保兴,等. 控制水稻重要农艺性状的QTL在盐胁迫与非胁迫条件下的对比研究[J]. 中国科学:C辑,2000,30(6):561-569.
[16] McCouch S R,Cho Y G,Yano M,et al.Report on QTL nomenclature[J].Rice Genet Newslett,1997,14:11-13
[17] 张俊国,张三元,赵劲松,等. 耐盐碱、高产、优质水稻新品种长白10的选育报告[J]. 吉林农业科学,2003,28(1):20-22.
[18] 孙 健. 水稻耐盐相关性状的发育动态QTL分析[D]. 哈尔滨:东北农业大学,2013.
[19] 梁银培,孙 健,索艺宁,等. 水稻耐盐性和耐碱性相关性状的QTL定位及环境互作分析[J]. 中国农业科学,2017,50(10):1747-1762.
[20] 邹德堂,马 婧,王敬国,等. 粳稻幼苗前期耐碱性的QTL检测[J]. 东北农业大学学报,2013,44(1):12-18.