低温是影响农作物生产的重要危害因素之一。在前期育苗过程中,水稻经常会受到低温影响,导致水稻生长迟缓或停止生长[1-3]。早稻直播面积的逐渐扩大,水稻苗期受低温胁迫的可能性日益增加[4]。因此,提高水稻抗低温能力、深入研究水稻耐低温机制成为许多研究人员关注的热门话题[5]。水稻是世界上最重要的粮食作物之一,全世界超过1/3的人口以水稻为主食[6]。然而,水稻产量受到包括低温等不同非生物胁迫的影响很大[7]。水稻苗期在遭受低温胁迫后会打破细胞渗透压稳态、调节渗透压的物质积累及代谢异常等,导致叶片失绿卷曲、生长缓慢、育性下降和产量降低等[8]。在农业生产中,低温胁迫会致使幼苗枯黄、秧苗腐烂等现象,影响其穗发育和产量等[9]。低温冷害是水稻种植所面临的主要困难之一[10]。目前,关于作物耐冷性研究主要停留在生理阶段[11]。水稻抗逆相关功能基因组学的研究一直是水稻研究核心内容。分析低温胁迫下幼苗体内代谢物变化特征和研究水稻苗期耐低温机理有助于解析水稻应对低温胁迫的生理机制与分子机制,为以后水稻分子育种奠定基础,这对农业经济和社会都有着深远意义。水稻可以通过抵御代谢反应、改变体内激素的含量及活性来调节生理过程,使其在低温条件下能维持正常的生理活动[12-13]。转录水平的调控是生物体最重要的调控方式,与基因组学相比具有针对性强、研究范围小的优点[14]。因此,被认为是一种在转录水平上更精准的测定分析方法[15]。目前,利用高通量转录组测序技术已发现了细枝木麻黄[16]、玉米[17]、西葫芦[18]、油菜[19]等植物与耐低温相关的转录、病害防御、信号转导、转运、次级代谢等大量相关基因。
本研究采用高通量测序方法-转录组测序(RNA-Seq)技术,在转录组水平上研究水稻苗期应答低温差异基因的表达特性,为增强对水稻耐低温机理的认识,明确水稻应答低温的分子机制,以及培育水稻耐低温品种提供参考。
1 材料和方法
1.1 试验材料
供试材料为粳稻品种中花-11,由浙江大学生命科学学院植物生理与生化国家重点实验室提供。
1.2 试验方法
1.2.1 培养条件 种子催芽方法和水培方法参考国际水稻所的水稻水培方法[20]。幼苗长至一叶一心时,移栽到规格为6 cm×6 cm×6 cm的培养钵中,三叶一心时小心将生育进程一致的幼苗取出避免伤到根系。将其中1/2的幼苗放置在光温培养箱(HPG-280,哈尔滨市东联电子技术开发有限公司)作为对照组(WTCK-2),培养条件为:昼夜温度为28 ℃/22 ℃,12 h/12 h,相对湿度为80%;另1/2的幼苗作为低温处理组(WTCOLD-2),移入另一光照培养箱(HPG-280,哈尔滨市东联电子技术开发有限公司),昼夜温度保持在17 ℃,12 h/12 h,相对湿度80%,保持10 d。
1.2.2 RNA提取、文库构建 水稻幼苗在低温处理后,分别取对照和处理后的苗期叶片放入液氮速冻,存至-80 ℃冰箱保存备用。委托北京诺禾致源生物信息科技有限公司完成RNA的提取、质控、建库及Illumina HiSeqTM 4000测序工作。
1.2.3 原始数据的组装 由高通量测序(Illumina HiSeqTM)得到的原始数据文件,利用CASAVA碱基识别(Base calling)分析转化成原始测序序列(Sequenced reads),即Raw reads。将低质量和带接头的reads进行过滤得到Clean reads,从而提高信息分析质量,Clean reads将作为后续分析的研究基础。本研究使用Q30(碱基准确率达到99.9%)的质量控制标准。
1.2.4 差异表达基因的筛选 通过使用DESeq自带的标准化方法对原始数据进行标准化处理。在差异分析过程中,使用负二项分布法对Read count的分布进行估计,评估且计算完P value后,对P value进行多重假设检验校正,来减少假阳性。差异基因筛选标准为:|log2(Fold Change)|>1并且校正的P值(Padj)<0.05。
1.2.5 GO基因功能注释分析 使用GOseq[21]将组装后的Unigene序列按照Corrected_P value<0.05筛选差异基因,为寻找出基因显著富集的GO Term,对筛选的差异基因进行GO基因功能注释分析,获得所有差异表达基因的功能注释、生物学功能以及相关的代谢途径,从而推测这些基因对应的特定过程。
1.2.6 KEGG pathway分析 使用软件KOBAS(2.0)进行特定基因的通路定位,找出显著富集的通路,最后对KEGG注释的基因功能信息进行生物学通路的注释与预测,解释低温胁迫下植物对应的调控机制。
1.2.7 聚类分析 使用聚类软件Pheatmap对差异基因的相对表达水平值log2(ratios) 进行聚类。以FPKM值的读数来计算差异表达水平,颜色从红到蓝,表示log10(FPKM+1)从大到小。使用与之匹配的距离算法,计算出基因间的距离,反复迭代,算出基因之间的相对距离,最后依据基因的相对距离远近来分成不同的Subcluster,从而实现聚类。
1.2.8 qRT-PCR验证 利用TRIzol Reagent提取对照组和处理组叶片的总 RNA,cDNA合成利用PrimeScriptTMRT reagent Kit with gDNAEraser(Perfect Real Time),荧光定量检测使用abm® EvaGreen qPCR MasterMix-no dye试剂盒。利用PrimerQuest Tool(http://sg.idtdna.com/Primerquest/Home/Index)设计引物,优先选择靠近基因3′端的引物并进行 Real-time PCR 分析。反应程序为95 ℃,10 min;反应循环数为45次,循环过程是94 ℃,15 s,60 ℃,1 min;72 ℃,20 s。用ABI StepOne Plus(ABI 公司,美国)进行荧光定量检测。利用2-ΔΔCt法,以对照组中各基因表达量为1,Log2FC表示log2倍,误差线表示标准偏差,以OsActin 为内参基因对目的基因进行转录水平的相对定量分析。
1.2.9 新基因功能预测 将测序所得的全部reads数据和已知的基因模型进行比较,对比原有的基因注释文件,可以发现新基因。根据转录组数据所提供的新基因碱基序列信息,在MSU(http://rice.plantbiology.msu.edu/analyses_search_locus.shtml)网站上查找相关需要信息。
1.2.10 蛋白互作预测 应用STRING蛋白质互作数据库(http://string-db.org/)中的互作关系分析差异基因蛋白互作网络。利用数据库中提取目标基因集互作关系和目标基因集中的序列,应用BlastX比对到String数据库中包含的参考物种的蛋白质序列上来构建蛋白质互作关系。
2 结果与分析
2.1 低温影响水稻幼苗期的生长发育
17 ℃低温处理10 d后,水稻在正常条件和低温处理下,幼苗长势差异明显(图1)。低温胁迫后的幼苗叶片偏黄、叶尖变黄枯且株高较正常条件下偏低。表明低温胁迫严重抑制了水稻幼苗的生长发育。

图1 正常条件(左)与低温处理(右)下的中花-11表型
Fig.1 Zhonghua-11 phenotype under normal condition (left) and low temperature (right) treatment
2.2 测序结果的质量分析
分别收集处理组和对照组水稻苗期叶片并制备其RNA样品。这些样品通过illumina HiSeqTM测序平台进行测序,一共生成约1.01亿条长度约100 bp的原始数据,2种条件下的原始片段分别是47 576 488,53 790 724条。根据生物信息学分析,舍弃低质量的读数,经过序列拼接,最后获得约0.99亿干净的高质量序列,分别得到46 384 074,52 483 052条干净的高质量序列,占原始数据的97.53%。其中,平均Q20、Q30都分别可达到96%,91%以上,GC含量分别为54.91%和55.65%,不确定碱基比例仅为0.02%。使用TopHat2将过滤后的数据定位到中花-11(对照组和处理组)的基因组。在2个样本组的高质量序列读数中,在参考序列上有唯一比对位置的分别占86.29%和84.95%、有多个比对位置的占1.57%和1.75%、总测序序列定位数占87.86%,86.70%。结合以上数据可以说明转录组测序质量较高,符合进一步的生物信息学分析(表1)。
表1 测序数据统计
Tab.1 Sequencing date statistics
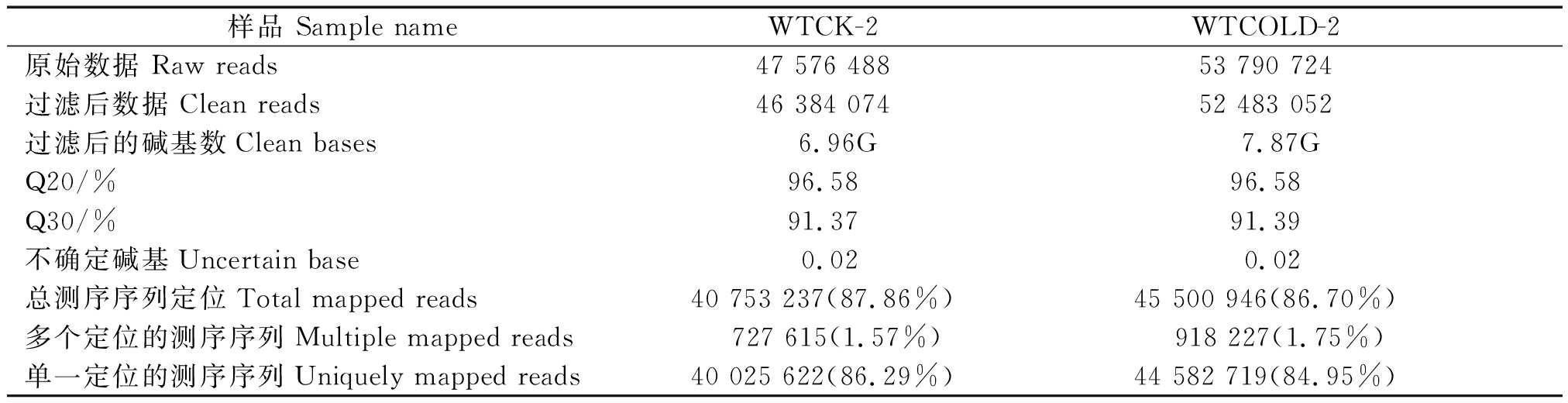
样品 Sample nameWTCK-2WTCOLD-2原始数据 Raw reads47 576 48853 790 724过滤后数据 Clean reads46 384 07452 483 052过滤后的碱基数Clean bases6.96G7.87GQ20/%96.5896.58Q30/%91.3791.39不确定碱基Uncertain base0.020.02总测序序列定位Total mapped reads40 753 237(87.86%)45 500 946(86.70%)多个定位的测序序列Multiple mapped reads 727 615(1.57%) 918 227(1.75%)单一定位的测序序列Uniquely mapped reads40 025 622(86.29%)44 582 719(84.95%)
注:Q20.质量值≥20的碱基所占比例;Q30.质量值≥30的碱基所占比例;GC含量.计算碱基G和C的数量总和占总的碱基数量的百分比。
Note:Q20.The proportion of the bases whose quality value≥20; Q30.The proportion of the bases whose quality value≥30; GC content.Calculate the percentage of the total number of bases G and C as a percentage of the total number of bases.
2.3 差异表达基因的GO功能注释分析
通过GO功能富集分析,对照组与处理组存在2 044个差异表达基因(Differentially expressed genes,DEGs),其中,1 252个基因表达上调,792个基因表达下调。被富集到3 405个GO terms中。其中生物学过程(Biological process,BP)占63.29%、细胞组分(Cellular component,CC)占8.81%、分子功能(Molecular function,MF)占27.90%。通过数据所占比例可以初步判断大多数差异表达基因与一些生物学功能显著相关。单一生物体代谢过程(9.55E-16)、质体(3.94E-21)和阳离子结合(4 .02×10-3)分别是BP、CC、和MF中最显著富集的功能类别。低温胁迫下DEGs涉及的20个最显著富集的功能如图2所示。
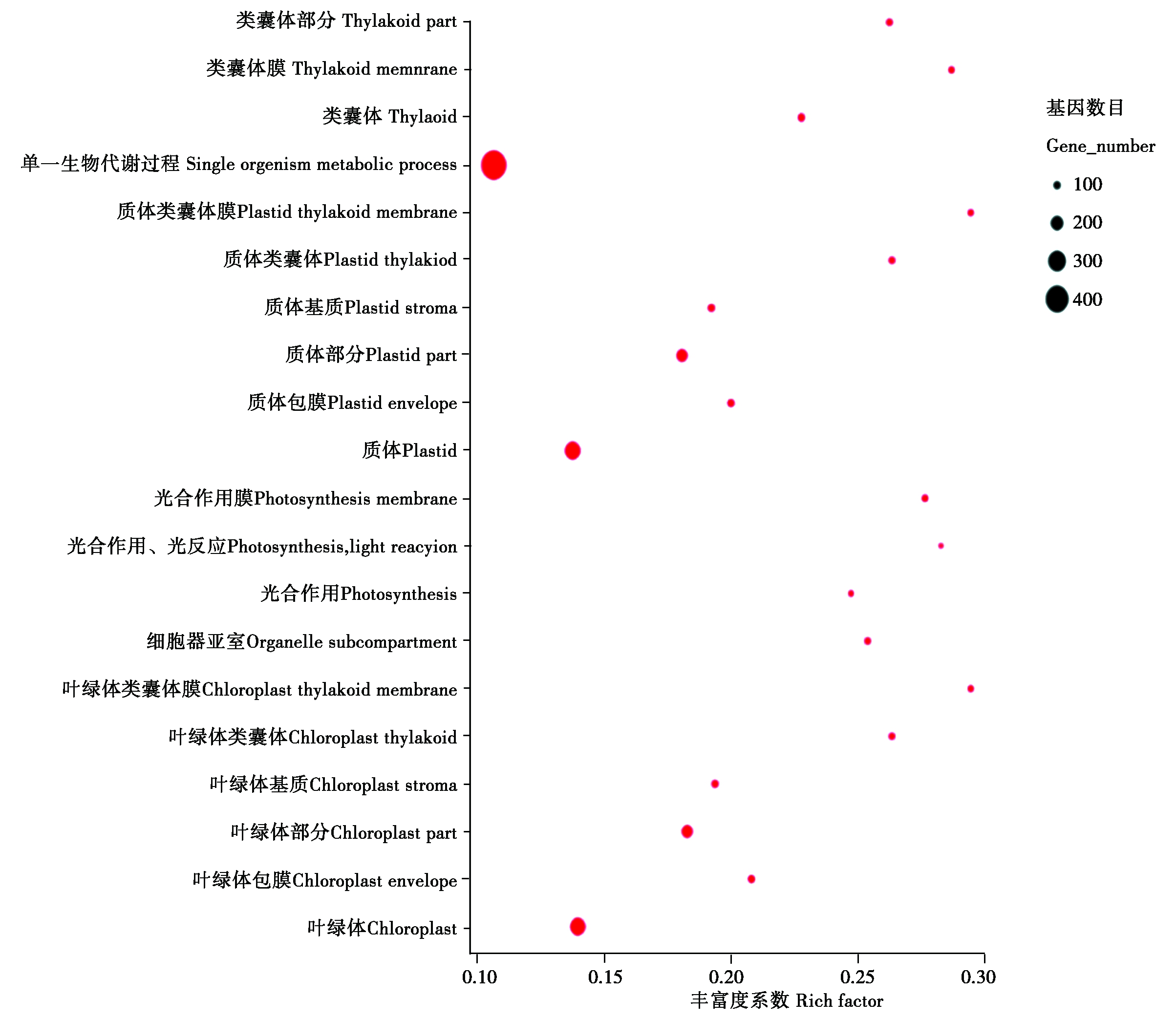
圆点所在的位置代表着富集的条目,圆点的大小表示差异基因的数量。 The position of the dot represents the enriched item,the size of the dot indicates the number of
Differential genes.
图2 GO term富集统计
Fig.2 GO term enrichment
2.4 低温胁迫下差异表达基因的鉴定与筛选
通过将表达模式相同或相近的基因聚集成类,用以推测已知基因的新功能或未知基因的功能[22]。为了鉴定具有功能富集的聚类,利用差异基因的相对表达水平值log2(ratios)进行层次聚类(图3),从图3可以看出,该材料在低温胁迫下基因表达量有变化。
基于以上发现,在对照组和处理组中鉴定了2 044个差异表达基因,其中有关于光合作用、苯丙氨酸代谢、WRKY转录因子等多种功能途径。其中右侧虚线的右侧圆点代表差异表达上调的基因有1 252个,左侧虚线的左侧圆点代表差异表达下调的基因有792个,两侧虚线中间表示无显著性差异表达的基因(图4)。
2.5 差异表达基因的KEGG通路分析
为了精准分析低温胁迫对水稻发育过程中参与的代谢途径发生的变化,对得到的Unigene进行KEGG代谢通路分析(表2)。结果表明,在代谢通路中,主要是光合作用天线蛋白(ko00196)、光合作用(ko00195)、类胡萝卜素生物合成(ko00906)、次生代谢产物的生物合成(ko01110)。利用KEGG数据库对差异基因进行数据分析,得到差异基因KEGG注释通路图,其中水稻苗期对受低温胁迫影响较大的代谢通路分析如下:
通路一:光合作用
低温胁迫下,此通路中的光合系统Ⅰ存在11个差异表达基因,且全部表现为下调;光系统Ⅱ中有24个差异表达基因,其中3个基因表现为上调,21个基因表现为下调;细胞色素b6/f复合体中含有1个差异表达基因且表现为下调;光合电子运输中有20个差异表达基因,有8个基因表现为上调,其余基因均表现为下调;F型ATP酶中有6个表现为下调基因(图5)。上述结果得出,在光合作用通路里这些差异表达基因可能是受低温处理影响较大的基因。
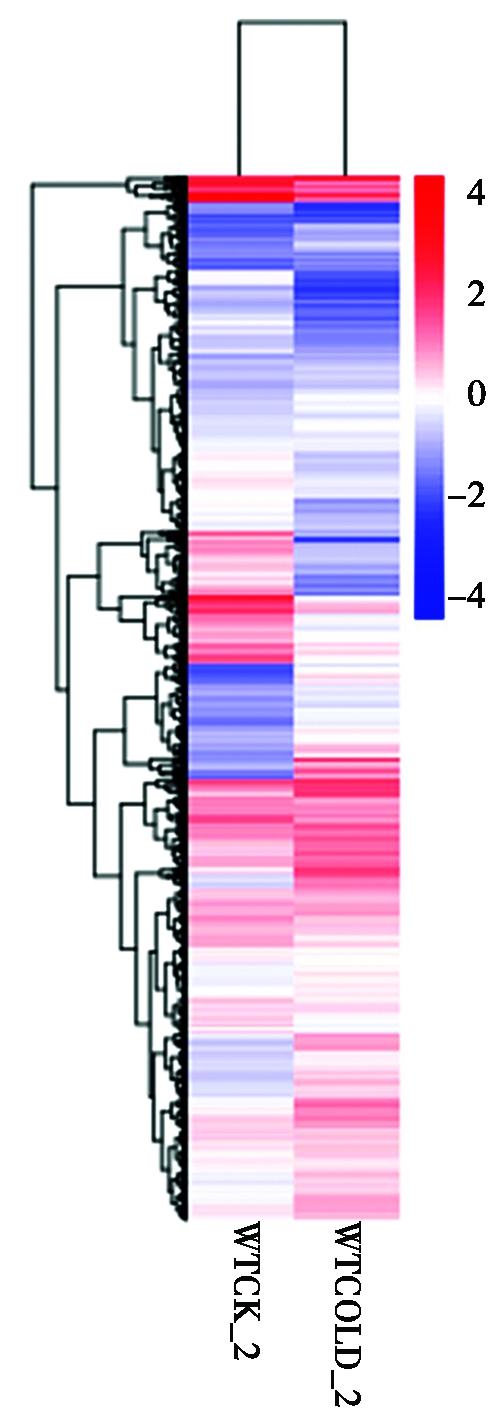
WTCK_2.对照条件下的中花-11;WTCOLD_2.低温胁迫下的中花-11。 WTCK_2.Zhonghua-11 under control conditions; WTCOLD_2.Zhonghua-11 under low temperature stress.
图3 差异基因的层次聚类
Fig.3 Hierarchical clustering of differential genes
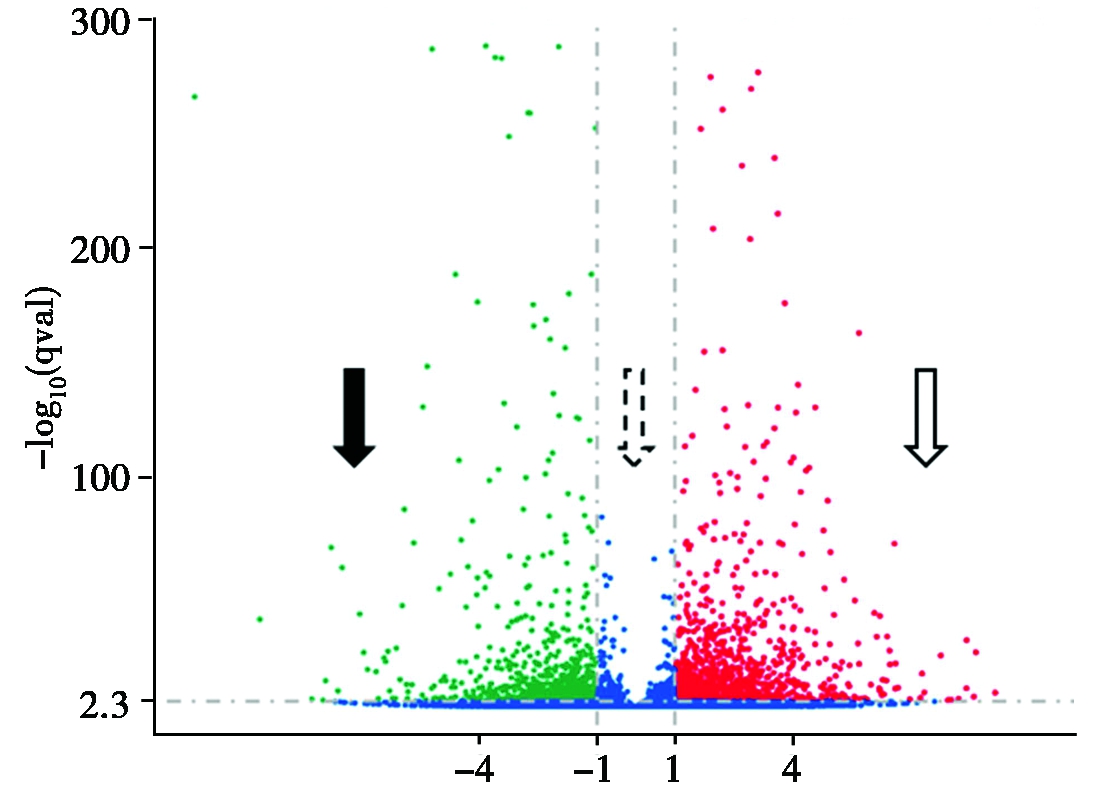
空心箭头.上调的显著差异表达基因;实心箭头.下调的显著差异表达基因;虚线空心箭头.无显著性差异表达的基因;横坐标.基因在不同样本中表达倍数变化;纵坐标.基因表达量变化差异的统计学显著性。
Hollow arrow.Significant Different expressed genes up-regulated; Solid arrow. Significantly Different expressed genes down-regulated; Dotted hollow arrow. Genes with no significant
Differential expression Abscissa. Gene expression fold change in Different samples; Ordinate.Statistically significant difference in gene expression changes.
图4 差异基因的火山
Fig.4 Volcanic map of differential genes
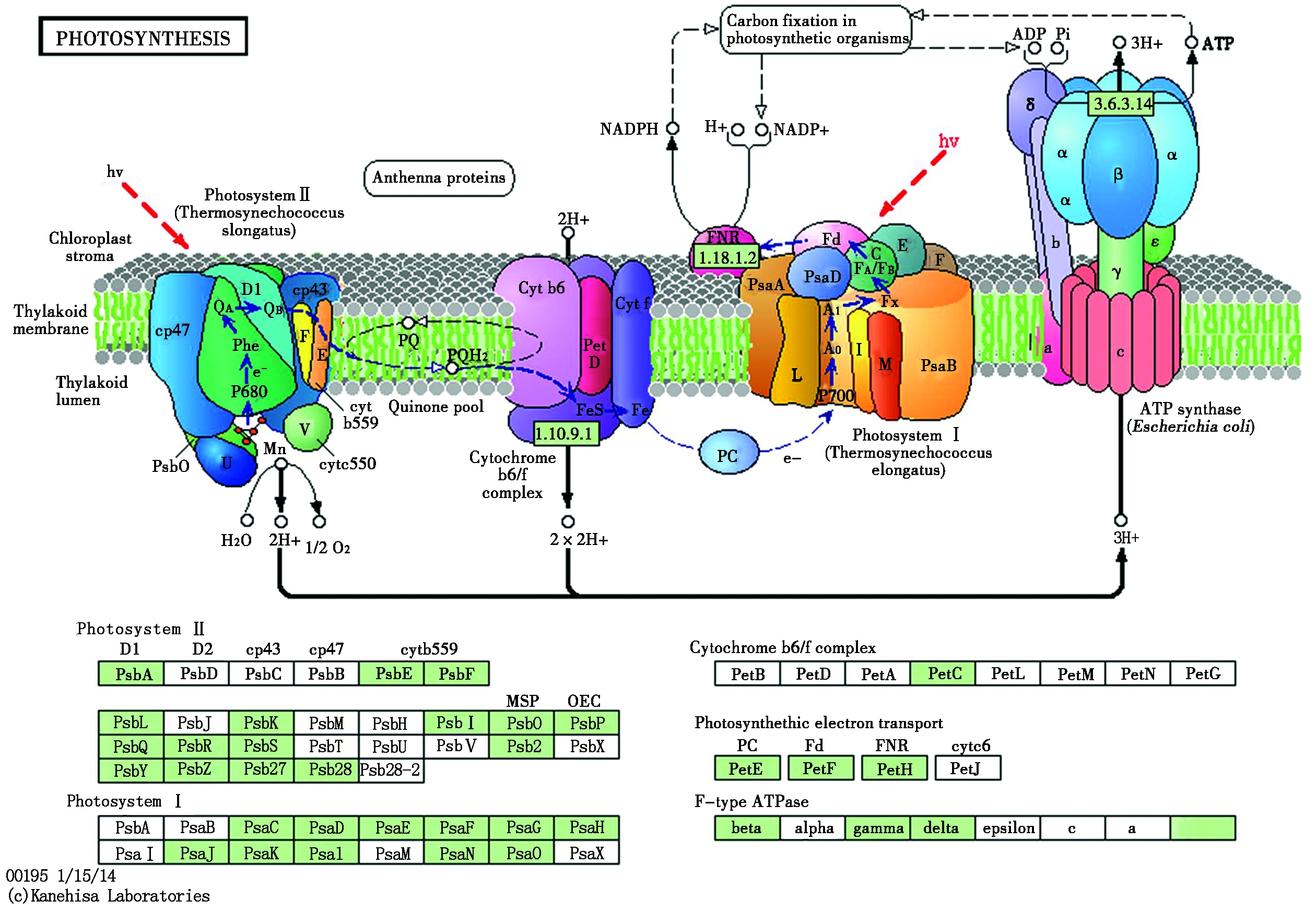
PhotosystemⅡ.光系统Ⅱ;PhotosystemⅠ.光系统Ⅰ;Cytochrome b6/f complex.细胞色素b6/f复合体;Photosynthethic electron transport.光合电子运输;F-type ATPase. F型ATP酶;Carbon fixation in photosynthetic organisms.光合生物中的碳固定;Chloroplast stroma.叶绿体基质;Thylakoid membrane.类囊体膜;Thylakoid lumen.类囊体腔。
图5 差异表达基因参与的光合作用代谢途径
Fig.5 Photosynthesis metabolic pathways involved in differentially expressed genes
通路二:苯丙氨酸代谢
苯丙氨酸代谢途径是植物最重要的次生代谢途径之一,苯丙氨酸解氨酶(Phenylalanine ammonia-lyase,PAL)基因表达量高,与之对应的次生代谢产物产量就高,水稻幼苗的整体抗逆能力就强[23-24]。低温胁迫下,在此通路中发现17个差异表达基因,8个DEGs表达上调,其余9个DEGs表达下调。分别在4.3.1.24(K01110)和4.3.1.25(K01110)处PAL相关基因OS02G0626400(1.654 5)、OS02G0626100(1.229 9)、OS04G0518100(1.544 4)上调表达,进而调节水稻次生代谢反应(图6)。从这个通路图可以看出,在低温胁迫下PAL基因表达上调,说明低温与苯丙氨酸代谢通路具有相关性,差异性表达的基因可能是低温应答的关键基因。
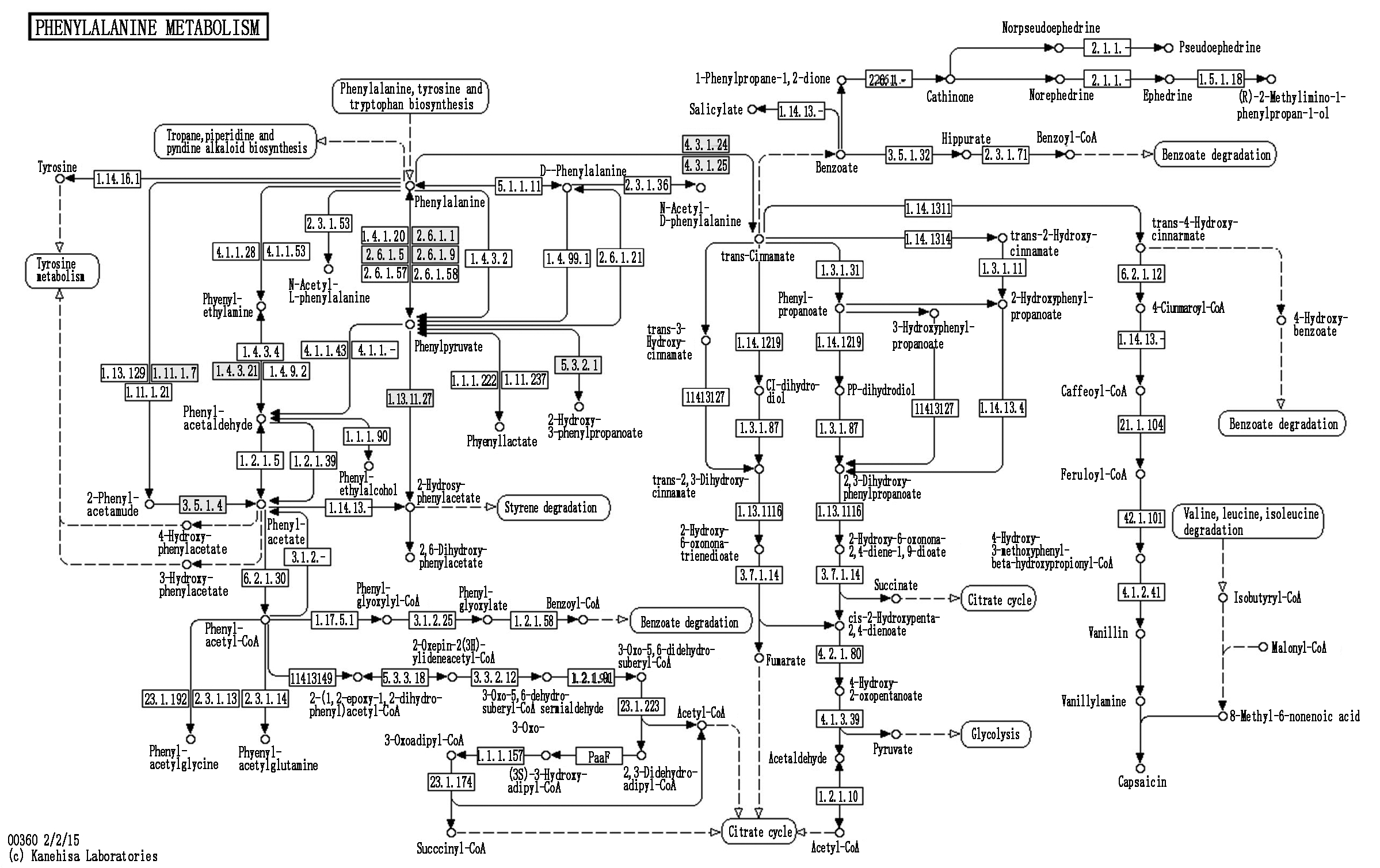
图6 差异表达基因参与苯丙氨酸代谢通路
Fig.6 Differentially expressed genes involved in phenylalanine metabolic pathway
表2 代谢通路及其差异性表达基因数量
Tab.2 Metabolic pathways and the number of differentially expressed genes
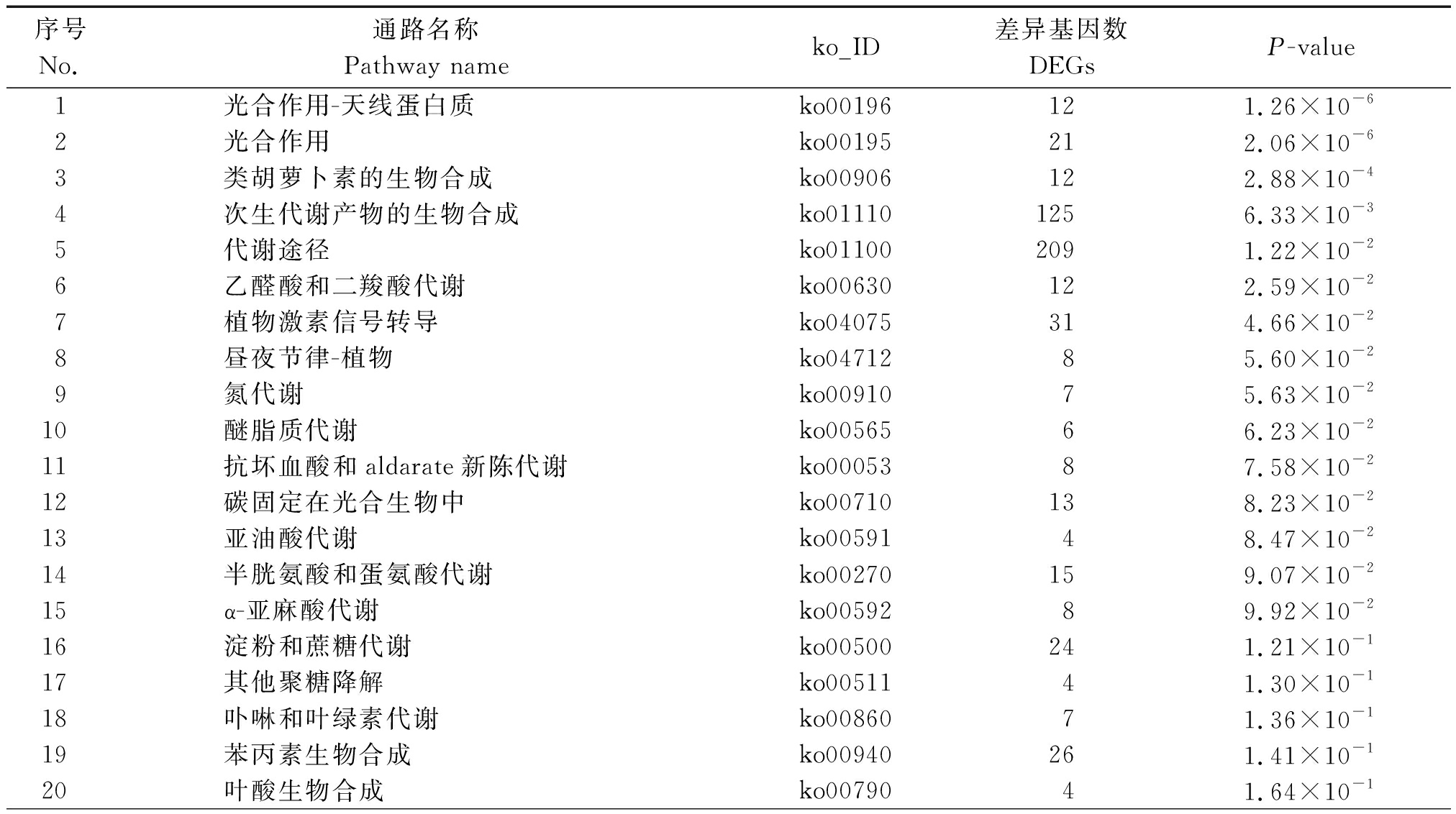
序号No.通路名称Pathway nameko_ID差异基因数DEGsP-value1 光合作用-天线蛋白质ko00196121.26×10-62 光合作用ko00195212.06×10-63类胡萝卜素的生物合成ko00906122.88×10-44次生代谢产物的生物合成ko011101256.33×10-35代谢途径ko011002091.22×10-26乙醛酸和二羧酸代谢ko00630122.59×10-27植物激素信号转导ko04075314.66×10-28昼夜节律-植物ko0471285.60×10-29氮代谢ko0091075.63×10-210醚脂质代谢ko0056566.23×10-211抗坏血酸和aldarate新陈代谢ko0005387.58×10-212碳固定在光合生物中ko00710138.23×10-213亚油酸代谢ko0059148.47×10-214半胱氨酸和蛋氨酸代谢ko00270159.07×10-215α-亚麻酸代谢ko0059289.92×10-216淀粉和蔗糖代谢ko00500241.21×10-117其他聚糖降解ko0051141.30×10-118卟啉和叶绿素代谢ko0086071.36×10-119苯丙素生物合成ko00940261.41×10-120叶酸生物合成ko0079041.64×10-1
表2(续)
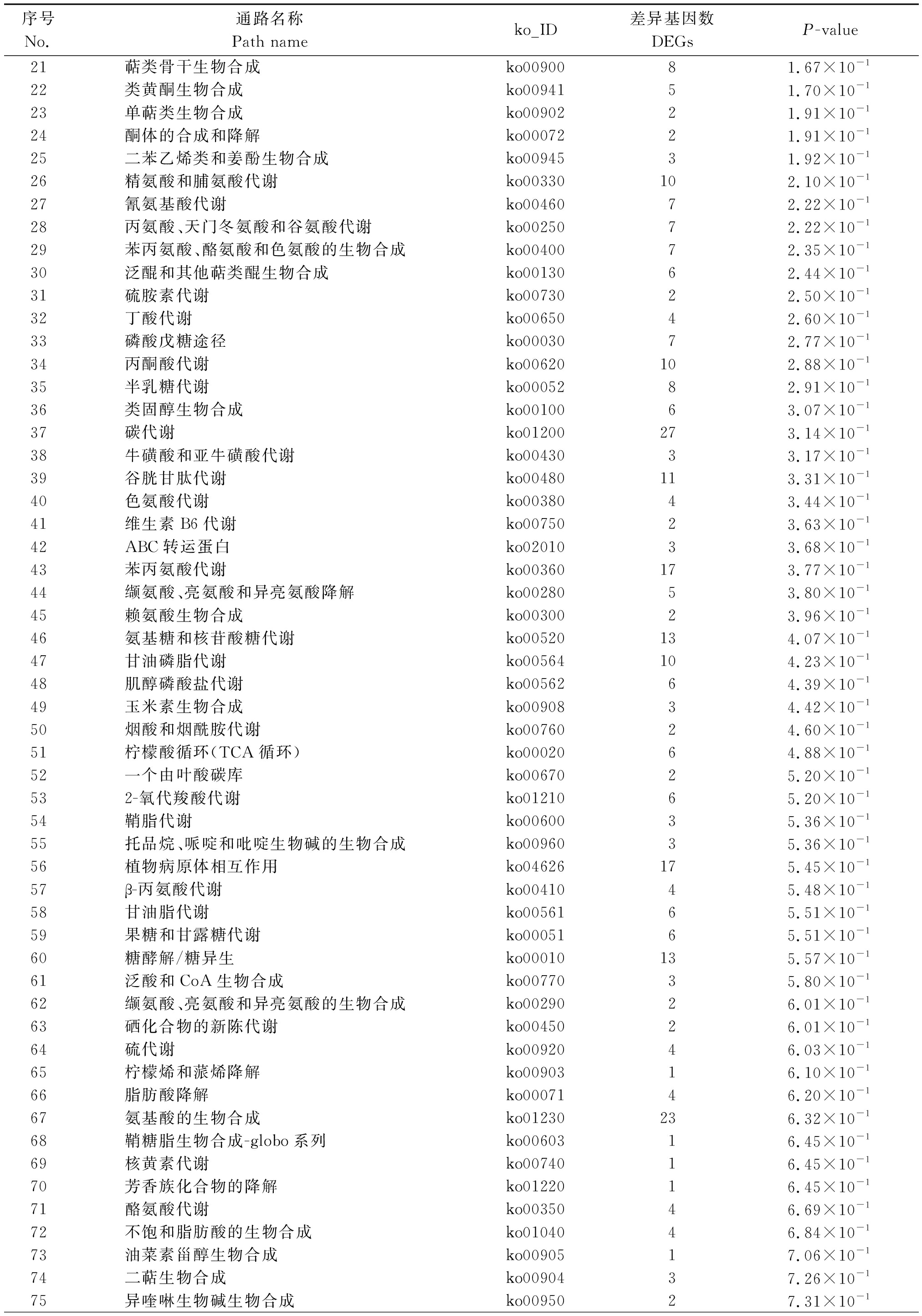
序号No.通路名称Path nameko_ID差异基因数DEGsP-value21萜类骨干生物合成ko0090081.67×10-122类黄酮生物合成ko0094151.70×10-123单萜类生物合成ko0090221.91×10-124酮体的合成和降解ko0007221.91×10-125二苯乙烯类和姜酚生物合成ko0094531.92×10-126精氨酸和脯氨酸代谢ko00330102.10×10-127氰氨基酸代谢ko0046072.22×10-128丙氨酸、天门冬氨酸和谷氨酸代谢ko0025072.22×10-129苯丙氨酸、酪氨酸和色氨酸的生物合成ko0040072.35×10-130泛醌和其他萜类醌生物合成ko0013062.44×10-131硫胺素代谢ko0073022.50×10-132丁酸代谢ko0065042.60×10-133磷酸戊糖途径ko0003072.77×10-134丙酮酸代谢ko00620102.88×10-135半乳糖代谢ko0005282.91×10-136类固醇生物合成ko0010063.07×10-137碳代谢ko01200273.14×10-138牛磺酸和亚牛磺酸代谢ko0043033.17×10-139谷胱甘肽代谢ko00480113.31×10-140色氨酸代谢ko0038043.44×10-141维生素B6代谢ko0075023.63×10-142ABC转运蛋白ko0201033.68×10-143苯丙氨酸代谢ko00360173.77×10-144缬氨酸、亮氨酸和异亮氨酸降解ko0028053.80×10-145赖氨酸生物合成ko0030023.96×10-146氨基糖和核苷酸糖代谢ko00520134.07×10-147甘油磷脂代谢ko00564104.23×10-148肌醇磷酸盐代谢ko0056264.39×10-149玉米素生物合成ko0090834.42×10-150烟酸和烟酰胺代谢ko0076024.60×10-151柠檬酸循环(TCA循环)ko0002064.88×10-152一个由叶酸碳库ko0067025.20×10-153 2-氧代羧酸代谢ko0121065.20×10-154鞘脂代谢ko0060035.36×10-155托品烷、哌啶和吡啶生物碱的生物合成ko0096035.36×10-156植物病原体相互作用ko04626175.45×10-157β-丙氨酸代谢ko0041045.48×10-158甘油脂代谢ko0056165.51×10-159果糖和甘露糖代谢ko0005165.51×10-160糖酵解/糖异生ko00010135.57×10-161泛酸和CoA生物合成ko0077035.80×10-162缬氨酸、亮氨酸和异亮氨酸的生物合成ko0029026.01×10-163硒化合物的新陈代谢ko0045026.01×10-164硫代谢ko0092046.03×10-165柠檬烯和蒎烯降解ko0090316.10×10-166脂肪酸降解ko0007146.20×10-167氨基酸的生物合成ko01230236.32×10-168鞘糖脂生物合成-globo系列ko0060316.45×10-169核黄素代谢ko0074016.45×10-170芳香族化合物的降解ko0122016.45×10-171酪氨酸代谢ko0035046.69×10-172不饱和脂肪酸的生物合成ko0104046.84×10-173油菜素甾醇生物合成ko0090517.06×10-174二萜生物合成ko0090437.26×10-175异喹啉生物碱生物合成ko0095027.31×10-1
表2(续)
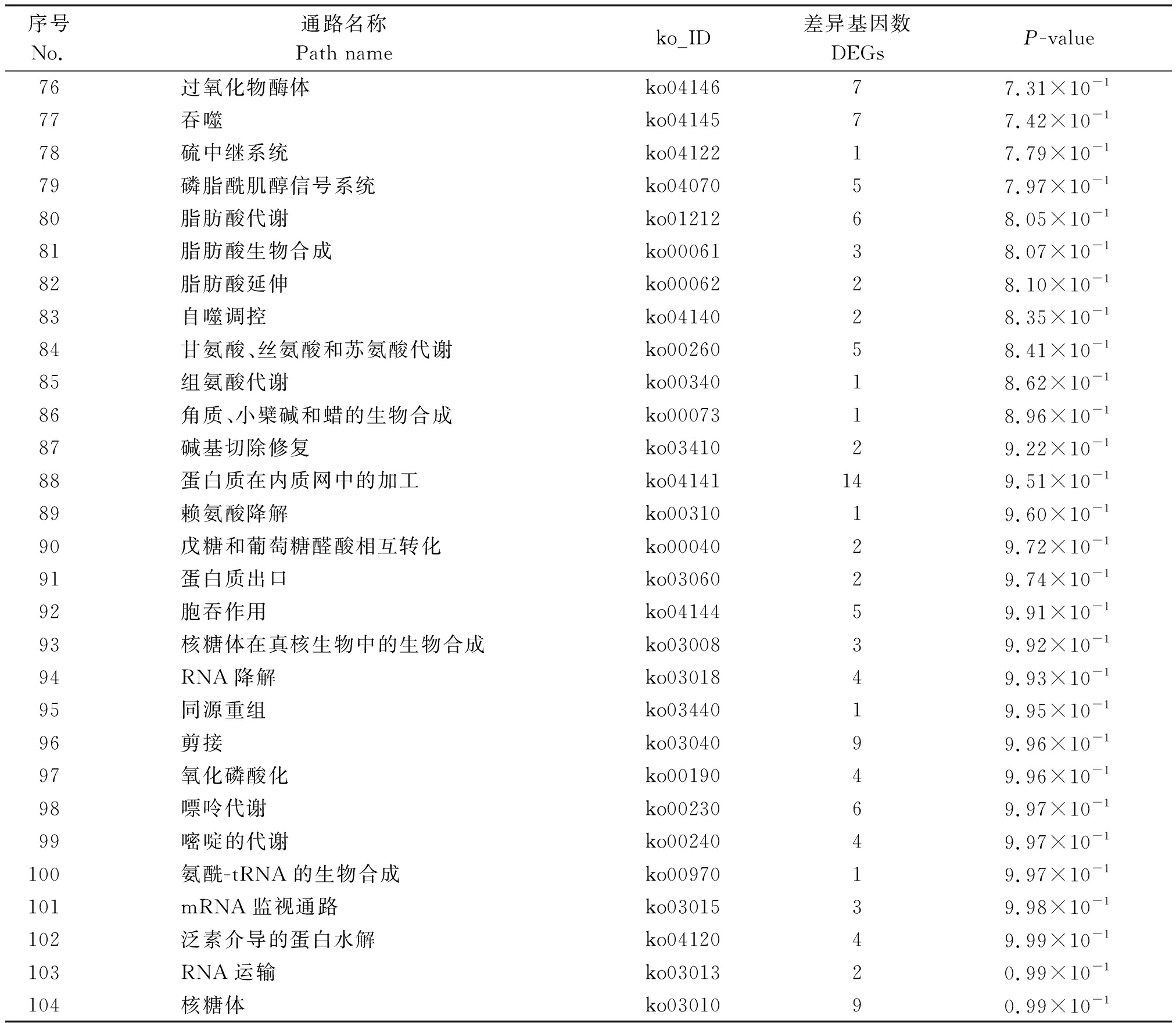
序号No.通路名称Path nameko_ID差异基因数DEGsP-value76过氧化物酶体ko0414677.31×10-177吞噬ko0414577.42×10-178硫中继系统ko0412217.79×10-179磷脂酰肌醇信号系统ko0407057.97×10-180脂肪酸代谢ko0121268.05×10-181脂肪酸生物合成ko0006138.07×10-182脂肪酸延伸ko0006228.10×10-183自噬调控ko0414028.35×10-184甘氨酸、丝氨酸和苏氨酸代谢ko0026058.41×10-185组氨酸代谢ko0034018.62×10-186角质、小檗碱和蜡的生物合成ko0007318.96×10-187碱基切除修复ko0341029.22×10-188蛋白质在内质网中的加工ko04141149.51×10-189赖氨酸降解ko0031019.60×10-190戊糖和葡萄糖醛酸相互转化ko0004029.72×10-191蛋白质出口ko0306029.74×10-192胞吞作用ko0414459.91×10-193核糖体在真核生物中的生物合成ko0300839.92×10-194RNA降解ko0301849.93×10-195同源重组ko0344019.95×10-196剪接ko0304099.96×10-197氧化磷酸化ko0019049.96×10-198嘌呤代谢ko0023069.97×10-199嘧啶的代谢ko0024049.97×10-1100氨酰-tRNA的生物合成ko0097019.97×10-1101mRNA监视通路ko0301539.98×10-1102泛素介导的蛋白水解ko0412049.99×10-1103 RNA运输ko0301320.99×10-1104 核糖体ko0301090.99×10-1
2.6 qRT-PCR验证基因表达
为了验证转录组分析结果中差异基因表达的可靠性,上调基因中选取了一个与植物病原体相互作用的基因OS05G0502000、一个与氮代谢相关的基因OS02G0770800、2个参与苯丙氨酸代谢的基因OS01G0869800、OS07G0677200;下调基因中挑选了4个与光合作用有关的基因OS07G0558400、OS01G0501800、OS03G0592500、OS02G0581100,利用qRT-PCR方法进行验证(表3)。使用RNA-Seq和qRT-PCR比较基因表达水平,以对照组中各基因表达量为1,统计低温处理下各基因的相对表达量。变化结果表明,qRT-PCR验证的基因表达量变化趋势和RNA-Seq差异基因表达趋势保持一致,如图7所示。表明转录组分析所得的数据具有较高的可靠性。
表3 实时定量PCR引物
Tab.3 Primers used for qRT-PCR analysis

序号No.基因号Gene ID正向引物(5'-3')Forward primer反向引物(5'-3')Reverse primer片段长度/bpFragment length1OS05G0502000GGGAGAACCATGCTCACTAAAGCTTCCTCATCCCATCTTTCA1252OS02G0770800AGTCCTGCCTCGTCTTCGAACTCGAGAGAGCTCCAAAC1173OS01G0869800CCTCTTCAACGTCGTCTTCTTCGATCGATCCTCGTAATTCTACTC1004OS07G0677200ACACCTTCTCCTCATCCTCT-TAATGACCTCTCCTCTCTCCATAC1405OS07G0558400GACAAGAGGTGGTTCAAGGGGGGATTAAAGAGAAGCTGAGAT-AGAG1056OS01G0501800GATACATGGAGAGAGGGAAG-GACACGAGAACAGAGGAAGAAACA1127OS03G0592500CCGCCATGATGATGCAGTAGCTGGTCGAAGTCCATGTT908OS02G0581100AGCTGTGGTTAGAGGGAAGACATCCATGGCGACAGTCTAATC1099OsActinTCCATCTTGGCATCTCTCAGCAGCCTTGGCAATCCACATCT60
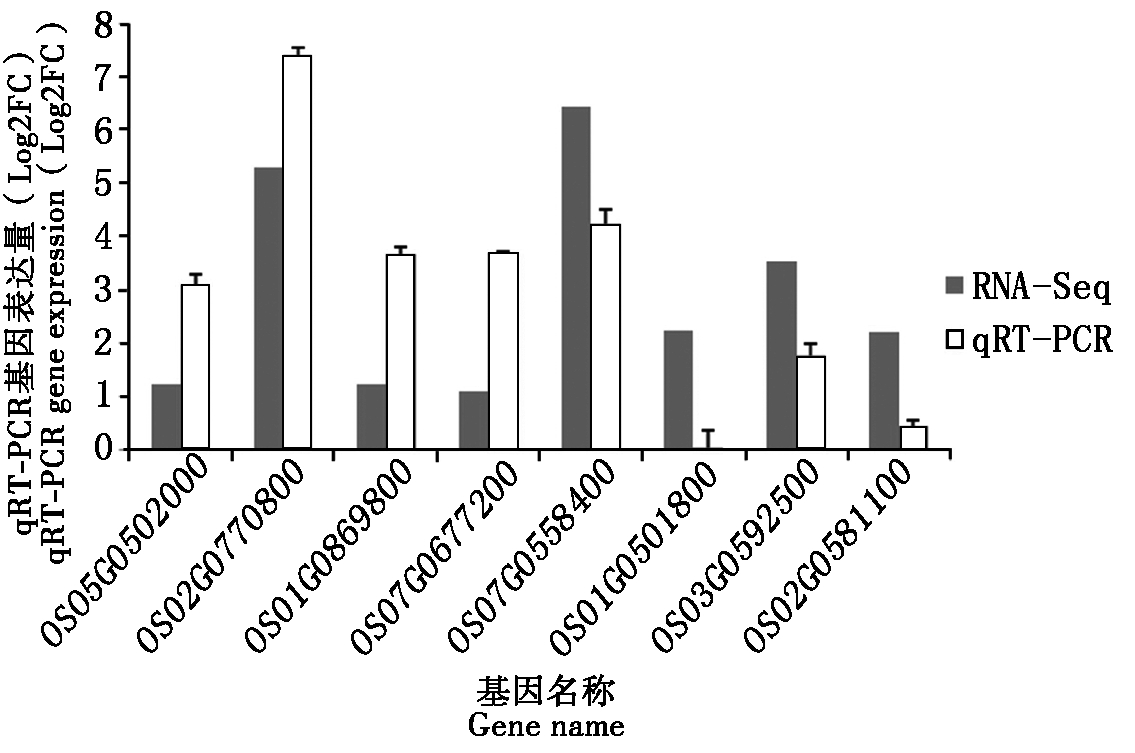
图7 RNA-Seq和qRT-PCR基因表达水平比较
Fig.7 Comparison of RNA-Seq and qRT-PCR gene expression levels
2.7 新基因功能预测
综合各种外显子预测的算法和人们对基因结
构信息的认识,可预测出可能的完整基因[25]。新基因的预测有助于分析的完整性。转录组数据分析发现了25个新基因,它们的长度在779~6 551 bp不等,主要出现在1,2,3,4,5,6,7,8,10,12号染色体上,有6个基因没有具体的信息描述,其他的主要表现在线粒体前体、钙转运ATP酶、氯通道蛋白和转绿因子WRKY等方面(表4)。
水稻的WRKY、MYB、bHLH、bZIP、C2H2等家族调控水稻的生长发育和耐逆抗病过程 [26-28]。本试验发现,2个WRKY转录因子,即 Novel00699和Novel00786。这2个WRKY转录因子可能参与了应答低温环境的信号刺激,被诱导表达,提高作物抗逆能力。
表4 新基因功能预测
Tab.4 Novel gene function prediction
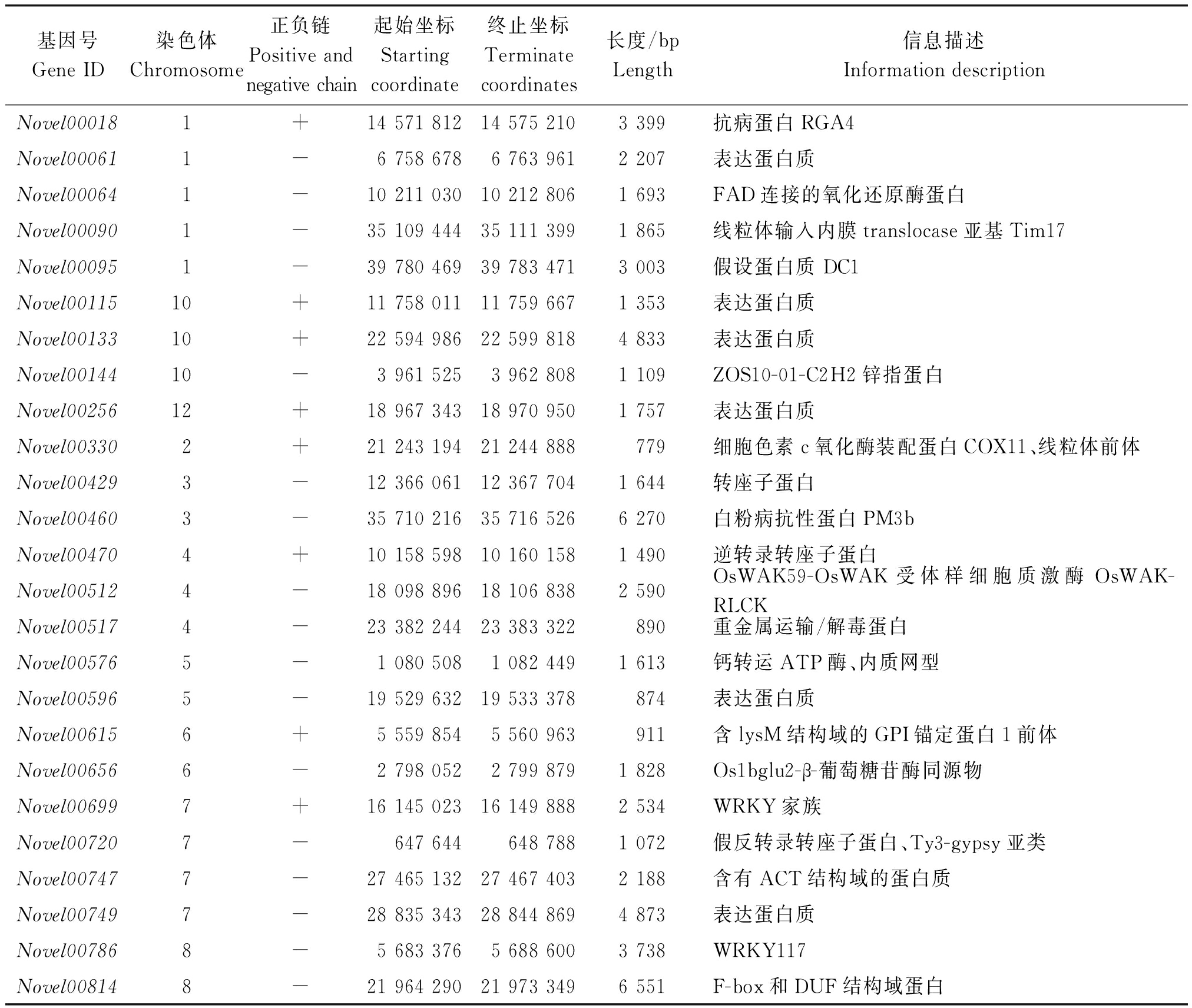
基因号Gene ID染色体Chromosome正负链Positive andnegative chain起始坐标Startingcoordinate终止坐标Terminatecoordinates长度/bpLength信息描述Information descriptionNovel000181+14 571 81214 575 2103 399抗病蛋白RGA4 Novel000611-6 758 6786 763 9612 207表达蛋白质Novel000641-10 211 03010 212 8061 693FAD连接的氧化还原酶蛋白Novel000901-35 109 44435 111 3991 865线粒体输入内膜translocase亚基Tim17 Novel000951-39 780 46939 783 4713 003假设蛋白质 DC1 Novel0011510+11 758 01111 759 6671 353表达蛋白质Novel0013310+22 594 98622 599 8184 833表达蛋白质Novel0014410-3 961 5253 962 8081 109ZOS10-01-C2H2锌指蛋白Novel0025612+18 967 34318 970 9501 757表达蛋白质Novel003302+21 243 19421 244 888779细胞色素c氧化酶装配蛋白COX11、线粒体前体Novel004293-12 366 06112 367 7041 644转座子蛋白Novel004603-35 710 21635 716 5266 270白粉病抗性蛋白PM3b Novel004704+10 158 59810 160 1581 490逆转录转座子蛋白Novel005124-18 098 89618 106 8382 590OsWAK59-OsWAK受体样细胞质激酶OsWAK-RLCKNovel005174-23 382 24423 383 322890重金属运输/解毒蛋白Novel005765-1 080 5081 082 4491 613钙转运ATP酶、内质网型Novel005965-19 529 63219 533 378874表达蛋白质Novel006156+5 559 8545 560 963911含lysM结构域的GPI锚定蛋白1前体Novel006566-2 798 0522 799 8791 828Os1bglu2-β-葡萄糖苷酶同源物Novel006997+16 145 02316 149 8882 534WRKY家族Novel007207-647 644648 7881 072假反转录转座子蛋白、Ty3-gypsy亚类Novel007477-27 465 13227 467 4032 188含有ACT结构域的蛋白质Novel007497-28 835 34328 844 8694 873表达蛋白质Novel007868-5 683 3765 688 6003 738WRKY117 Novel008148-21 964 29021 973 3496 551F-box和DUF结构域蛋白
2.8 预测的互作蛋白
本研究分析得到5个与光合作用有关的基因,分别为LOC_Os01g31690、LOC_Os03g56670、LOC_Os04g33830、LOC_Os07g04840、LOC_Os08g44680,预测有16个基因与其互作;一个与PAL有关的基因LOC_Os02g41630,且预测到有3个基因与其互作(表5)。这些结果有助于对水稻幼苗响应低温胁迫的调节机制的进一步研究。
表5 预测的互作蛋白
Tab.5 Predicted interaction proteins
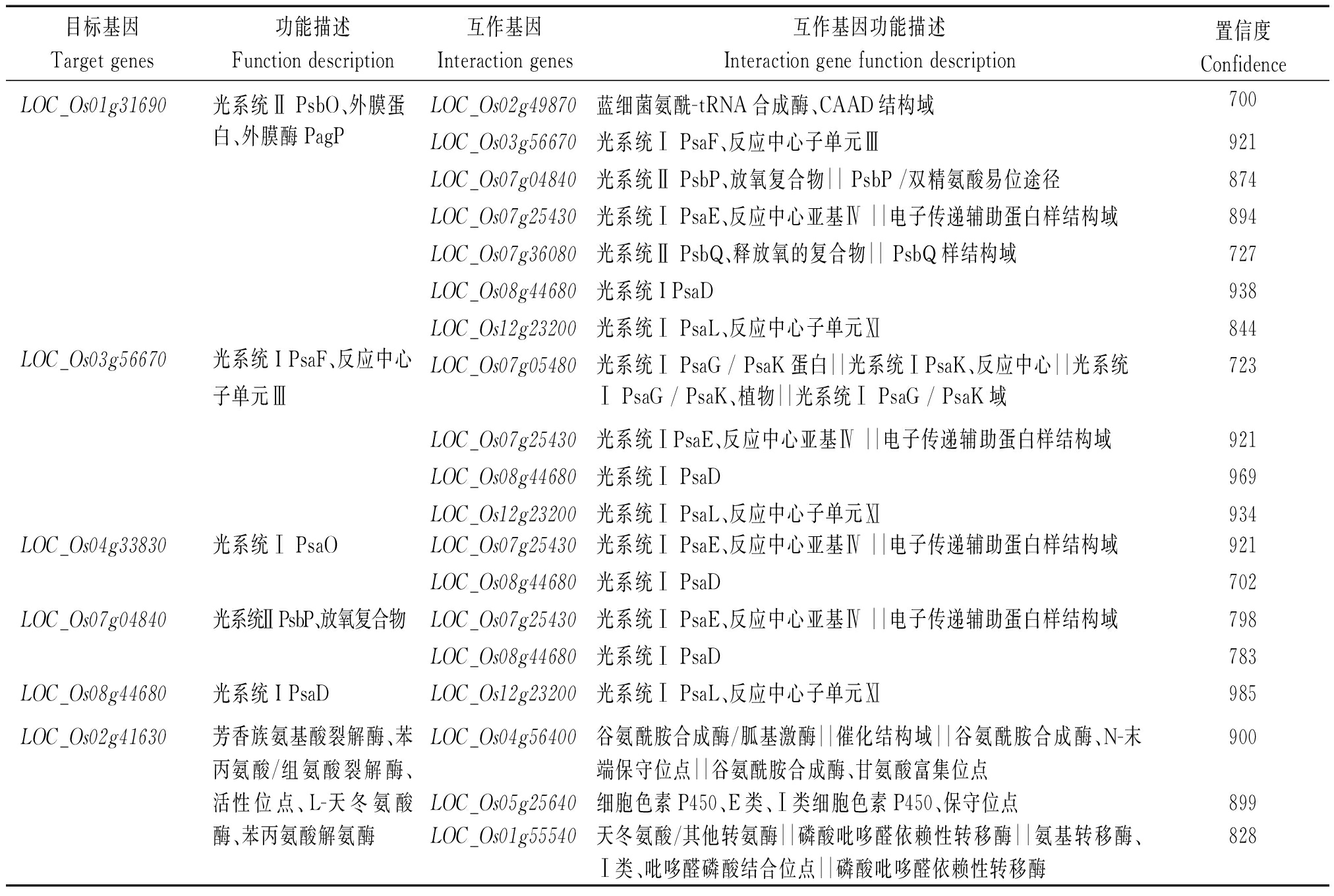
目标基因Target genes功能描述Function description互作基因Interaction genes互作基因功能描述Interaction gene function description置信度ConfidenceLOC_Os01g31690光系统Ⅱ PsbO、外膜蛋LOC_Os02g49870蓝细菌氨酰-tRNA合成酶、CAAD结构域700白、外膜酶PagPLOC_Os03g56670光系统Ⅰ PsaF、反应中心子单元Ⅲ921LOC_Os07g04840光系统Ⅱ PsbP、放氧复合物|| PsbP /双精氨酸易位途径874LOC_Os07g25430光系统Ⅰ PsaE、反应中心亚基Ⅳ ||电子传递辅助蛋白样结构域894LOC_Os07g36080光系统Ⅱ PsbQ、释放氧的复合物|| PsbQ样结构域727LOC_Os08g44680光系统I PsaD938LOC_Os12g23200光系统Ⅰ PsaL、反应中心子单元Ⅺ844LOC_Os03g56670光系统I PsaF、反应中心LOC_Os07g05480光系统Ⅰ PsaG / PsaK蛋白||光系统ⅠPsaK、反应中心||光系统723子单元ⅢⅠ PsaG / PsaK、植物||光系统Ⅰ PsaG / PsaK域LOC_Os07g25430光系统ⅠPsaE、反应中心亚基Ⅳ ||电子传递辅助蛋白样结构域921LOC_Os08g44680光系统Ⅰ PsaD969LOC_Os12g23200光系统Ⅰ PsaL、反应中心子单元Ⅺ934LOC_Os04g33830光系统Ⅰ PsaOLOC_Os07g25430光系统Ⅰ PsaE、反应中心亚基Ⅳ ||电子传递辅助蛋白样结构域921LOC_Os08g44680光系统Ⅰ PsaD702LOC_Os07g04840光系统Ⅱ PsbP、放氧复合物LOC_Os07g25430光系统Ⅰ PsaE、反应中心亚基Ⅳ ||电子传递辅助蛋白样结构域798LOC_Os08g44680光系统Ⅰ PsaD783LOC_Os08g44680光系统I PsaDLOC_Os12g23200光系统Ⅰ PsaL、反应中心子单元Ⅺ985LOC_Os02g41630芳香族氨基酸裂解酶、苯丙氨酸/组氨酸裂解酶、活性位点、L-天冬氨酸酶、苯丙氨酸解氨酶LOC_Os04g56400LOC_Os05g25640LOC_Os01g55540谷氨酰胺合成酶/胍基激酶||催化结构域||谷氨酰胺合成酶、N-末端保守位点||谷氨酰胺合成酶、甘氨酸富集位点 细胞色素P450、E类、Ⅰ类细胞色素P450、保守位点天冬氨酸/其他转氨酶||磷酸吡哆醛依赖性转移酶||氨基转移酶、Ⅰ类、吡哆醛磷酸结合位点||磷酸吡哆醛依赖性转移酶900899828
3 结论与讨论
3.1 结论
本研究对正常条件和低温胁迫下水稻苗期叶片进行转录组测序。通过GO功能注释、KEGG Pathway、聚类分析等方法在正常条件和低温胁迫下进行差异基因表达水平上的比较,筛选出2 044个DEGs,其中,1 252个基因表达上调,792个基因表达下调;发现低温胁迫后差异表达基因主要参与光合作用、苯丙氨酸代谢等途径;在低温胁迫下,水稻幼苗的光合作用受到影响,其中该作用中光系统Ⅱ与光合运输电子受到的影响较大,基因变化趋势主要体现为下调;并且低温诱导了PAL活性升高,促进苯丙烷类次生代谢产物的合成,增强水稻幼苗在低温胁迫下的耐受力。通过新基因预测发现,差异表达基因可能参与转录因子的调控。本研究提供了水稻苗期低温研究方面一定的表达数据,同时也有助于对水稻苗期低温响应的分子调控机理的进一步研究。
3.2 讨论
国内外学者针对低温胁迫对植物光合作用的影响有着大量研究。王春萍等[29]研究了低温胁迫对水稻幼苗不同叶龄叶片叶绿素荧光特性,结果发现,低温胁迫对水稻幼苗三叶期的光合利用能力造成了一定影响。PSⅡ是植物光合作用过程中进行光反应的重要结构,低温胁迫会导致光能和光合系统Ⅱ(PSⅡ)电子传递转化效率降低,造成水稻的净光合速率、气孔导度与蒸腾速率均明显下降[30-32]。Kaniuga等[33-34]研究证明,低温条件下降低了叶绿体的希尔反应活性和类PSⅡ的电子传递活性。本研究发现,低温处理水稻幼苗后在光合作用通路上的差异表达基因,分别体现在光系统Ⅰ、光系统Ⅱ、细胞色素b6/f复合体、光合运输电子和F型ATP酶5个方面,受温度影响DEGs数量较多的为光系统Ⅱ和光合电子运输2个部分。光系统Ⅱ中的DEGs有21个表现下调,3个表现上调;光合电子运输中有20个差异表达基因,有8个表现上调,其余均为下调。通过这些数据可以补充说明:低温胁迫下,水稻幼苗的光合作用受到影响,且该作用中光系统Ⅱ与光合运输电子受到的影响较大,基因变化趋势主要体现下调。
PAL在植物生长发育和抵御环境胁迫等方面具有重要的生理意义[35-36]。黄小贞等[24]发现在不同的组织和时间段PAL基因的表达具有特异性,并且受内部发育信号等的调节。董春娟等[37]研究得出,黄瓜幼苗中的PAL基因表达和酶活性可在10 ℃低温条件下被诱导升高,证明了在低温胁迫下,黄瓜中的PAL基因参与黄瓜幼苗对低温胁迫的响应且存在基因功能的重叠。油菜在2 ℃处理后,叶片经PAL抑制剂氨基茚磷酸处理,叶片物质积累会发生迟缓,且Fv/Fm的值下降。证实PAL活性受到抑制会导致幼苗对低温胁迫的抗性降低[38]。本试验发现,水稻幼苗在低温胁迫下,有17个DEGs参与苯丙氨酸代谢通路,其中有3个注释到PAL功能,表达量均表现为上调。结合本试验结果说明,水稻幼苗为在低温条件下提高自身的抗逆性,保证其正常生长发育。所以,低温诱导了PAL的活性升高,促进苯丙烷类次生代谢产物的合成,缓解低温引起的不利因素。
众多研究表明,拟南芥与水稻 WRKY 基因家族的生物学功能和复杂相关特征已被广泛的挖掘并进行了功能鉴定[39-42]。目前,粳稻和籼稻分别鉴定出98,102个WRKY转录因子[41],生物和非生物逆境因子可大量诱导WRKY基因表达[43-45]。鄂志国等[46]将水稻WRKY家族按WRKY结构域基本结构特征的差异为3类,且得出它们通过调控生长调节物质所介导的信号途径,广泛的影响水稻的生长发育、生物与非生物胁迫应答。彭喜旭等[47]研究表明, WRKY80可能通过茉莉酸/乙烯介导的信号途径参与或调控防御反应。Shimono等[48-54]在水稻中找到WRKY45、WRKY13、WRKY03等多个WRKY家族基因被证明参与抗病防御调节。仇玉萍等[55]证实水稻中的7个WRKY因子(OsWRKY8、OsWRKY 12、OsWRKY 13、OsWRKY 14、OsWRKY 17、OsWRKY 23 和OsWRKY 45)能被4 ℃低温诱导表达;Zhou等[56]研究发现GmWRKY21过量表达可提高拟南芥抵抗低温的能力。本试验研究发现,对照组和处理组的水稻幼苗共有10个WRKY转录因子发生差异性表达,有8个DEGs表现上调,分别为WRKY21(OS01G0821600)、WRKY24(OS01G0826400)、WRKY121(OS03G0741400)、WRKY68(OS04G0605100)、WRKY7(OS05G0537100)、WRKY8(OS05G0583000)、WRKY69(OS08G0386200)、WRKY97(OS12G0116400);2个DEGs表现下调,分别是WRKY30(OS08G0499300)、 WRKY90(OS09G0481700)。新基因预测中也发现了2个WRKY转录因子WRKY115(Novel00699)和WRKY117(Novel00786),经过低温处理后,水稻幼苗中部分WRKY转录因子的表达量发生变化,说明一些WRKY转录因子参与低温胁迫。
[1] 王 品,魏 星,张 朝,等. 气候变暖背景下水稻低温冷害和高温热害的研究进展[J]. 资源科学,2014,36(11):2316-2326.
[2] 李健陵,霍治国,吴丽姬,等. 孕穗期低温对水稻产量的影响及其生理机制[J]. 中国水稻科学,2014,28(3):277-288.
[3] 聂元元,蔡耀辉,颜满莲,等. 水稻低温冷害分析研究进展[J]. 江西农业学报,2011,23(3):63-66.
[4] 余肇福. 作物冷害[M]. 北京:农业出版社,1994.
[5] 向 丹. 水稻苗期低温耐性差异及其调控研究[D]. 北京:中国农业科学院,2013.
[6] 林 剑. 水稻空育131芽期耐冷候选基因OsGST的克隆和功能分析[D]. 哈尔滨:黑龙江大学,2016.
[7] Jangam A P,Pathak R R,Raghuram N. Microarray analysis of rice d1 (RGA1) mutant reveals the potential role of G-Protein alpha subunit in regulating multiple abiotic stresses such as drought,salinity,heat,and cold[J]. Frontiers in Plant Science,2016,7(1000):11.
[8] Wenzel A, Frank T, Reichenberger G, et al. Impact of induced drought stress on the metabolite profiles of barley grain[J]. Metabolomics, 2015, 11(2):454-467.
[9] Song S,Qi T,Huang H,et al. The Jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to affect jasmonate-regulated stamen development in Arabidopsis[J]. The Plant Cell,2011,23(3):1000-1013.
[10] 赵秀琴,张 婷,王文生,等. 水稻低温胁迫不同时间的代谢物谱图分析[J]. 作物学报,2013,39(4):720-726.
[11] Rokebul Anower M, Anne Fennell, Arvid Boe, et al. Physiological and molecular characterisation of lucerne (Medicago sativa L.) germplasm with improved seedling freezing tolerance[J]. Crop & Pasture Science, 2016, 67(6):655-665.
[12] 刘雄锋. CYPPDK在水稻种子萌发中对温度的响应及磷酸化调节研究[D]. 武汉:华中农业大学, 2016.
[13] 李合生. 现代植物生理学[M]. 北京:高等教育出版社,2002.
[14] 赵 航,贾富强,张富春,等. 盐胁迫下盐穗木差异表达基因的转录组信息分析[J]. 生物信息学,2014,12(2):90-98.
[15] 周 华,张 新,刘腾云,等. 高通量转录组测序的数据分析与基因发掘[J]. 江西科学,2012,30(5):607-611.
[16] 刘 芬. 低温胁迫对细枝木麻黄无性系生理指标和转录组的影响[D]. 长沙:中南林业科技大学,2015.
[17] 李 钊. 玉米苗期抗冻生理响应及其转录组调控分析[D]. 哈尔滨:东北农业大学,2017.
[18] 张宏亮. 低温胁迫下西葫芦转录组分析与SSR分子标记开发[D]. 太谷:山西农业大学,2015.
[19] 杜春芳. 甘蓝型油菜低温诱导的转录组和蛋白组分析[D]. 武汉:华中农业大学,2016.
[20] 张忠臣. 水稻侧根发育基因OsCYP2(cyclophilin2)的克隆和功能分析[D]. 杭州:浙江大学,2007.
[21] Young M D,Wakefield M J,Smyth G K. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biology,2010,11(2):R14.
[22] 吕 顺. 蛋白激发子MoHrip1诱导水稻抗病机理的研究[D]. 北京:中国农业科学院,2016.
[23] 朱海生, 温文旭, 刘建汀,等. 丝瓜苯丙氨酸解氨酶基因PAL克隆及表达分析[J]. 植物遗传资源学报, 2018,19(2):305-313.
[24] 黄小贞,赵德刚. 植物苯丙氨酸解氨酶表达调控机理的研究进展[J]. 贵州农业科学,2017,45(4):16-20.
[25] 张丽娟,成 军,罗 军. 新基因功能预测的理论及方法[J]. 医学分子生物学杂志,2006,3(4):279-282.
[26] 刘梦佳, 李海峰. 水稻WRKY转录因子家族研究进展[J].河南农业科学, 2016, 45(3):1-8.
[27] Moon S J, Han S Y, Kim D Y, et al. Ectopic expression of CaWRKY1, a pepper transcription factor, enhances drought tolerance in transgenic potato plants[J].Journal of Plant Biology, 2014, 57(3):198-207.
[28] 冯盼盼, 陈 鹏, 洪文杰, 等. 拟南芥转录因子家族 MYB 研究进展[J].生命科学研究, 2016,20(6):555-560.
[29] 王春萍,雷开荣,李正国,等. 低温胁迫对水稻幼苗不同叶龄叶片叶绿素荧光特性的影响[J]. 植物资源与环境学报,2012,21(3):38-43.
[30] Sui N. Photoinhibition of suaeda salsa,to chilling stress is related to energy dissipation and water-water cycle[J]. Photosynthetica,2015,53(2):207-212.
[31] Woo N S,Badger M R,Pogson B J. A rapid,non-invasive procedure for quantitative assessment of drought survival using chlorophyll fluorescence[J]. Plant Methods,2008,4(1):27.
[32] 王亚男,范思静. 低温胁迫对水稻幼苗叶片生理生化特性的影响[J]. 安徽农业科学,2017,45(5):8-9.
[33] Kaniuga Z,Sochanowicz B,Zabek J,et al. Photosynthetic apparatus in chilling-sensitive plants :I. Reactivation of hill reaction activity inhibited on the cold and dark storage of detached leaves and intact plants[J]. Planta,1978,140(2):121-128.
[34] 苗妍秀. 黄瓜幼苗对红蓝光质响应的生理机制[D]. 北京:中国农业大学,2015.
[35] Dixon R A,Achnine L,Kota P,et al. The phenylpropanoid pathway and plant defence a genomics perspective-Dixon-2002-Molecular Plant Pathology-Wiley Online Library[J]. Molecular Plant Pathology,2010,3(5):371-390.
[36] Dong C J, Li L, Cao N, et al. Roles of phenylalanine ammonia-lyase in low temperature tolerance in cucumber seedlings[J]. Chinese Journal of Applied Ecology, 2015, 26(7):2041-2049.
[37] 董春娟,李 亮,曹 宁,等. 苯丙氨酸解氨酶在诱导黄瓜幼苗抗寒性中的作用[J]. 应用生态学报,2015,26(7):2041-2049.
[38] Solecka D,Kacperska A. Phenylpropanoid deficiency affects the course of plant acclimation to cold[J]. Physiologia Plantarum,2003,119(2):253-262.
[39] Jang J Y, Choi C H, Hwang D J. The WRKY superfamily of rice transcription factors[J]. Plant Pathology Journal, 2010, 26(2):110-114.
[40] Rushton P J,Somssich I E,Ringler P,et al. WRKY transcription factors[J]. Trends in Plant Science,2010,15(5):247-258.
[41] Ding W W,Fang W B,Shi S Y,et al. Wheat WRKY type transcription factor gene TaWRKY1 is essential in mediating drought tolerance associated with an ABA-Dependent pathway[J]. Plant Molecular Biology Reporter,2016,34(6):1111-1126.
[42] 孟 姣,王海华,向建华,等. 水稻WRKY转录因子基因家族响应外源一氧化氮的表达谱分析[J]. 中国水稻科学,2016,30(2):111-120.
[43] Ross C A,Liu Y E,Shen Q J. The WRKY gene family in rice (Oryza sativa) [J]. Journal of Integrative Plant Biology,2007,49(6):827-842.
[44] Dou L, Zhang X, Pang C, et al. Genome-wide analysis of the WRKY gene family in cotton[J]. Molecular Genetics & Genomics Mgg, 2014, 289(6):1103.
[45] Kim C Y, Vo K T X, Cong D N, et al. Functional analysis of a cold-responsive rice WRKY gene, OsWRKY71[J].Plant Biotechnology Reports, 2016, 10(1):13-23.
[46] 鄂志国,王 磊. 水稻WRKY基因家族功能研究进展[J]. 核农学报,2012,26(5):750-755.
[47] 彭喜旭,唐新科,周平兰,等. 水稻WRKY80转录调节蛋白基因的分离与表达模式[J]. 中国农业科学,2013,46(19):4035-4043.
[48] Shimono M,Sugano S,Nakayama A,et al. Rice WRKY45 plays a crucial role in benzothiadiazole-inducible blast resistance[J]. The Plant Cell,2007,19(6):2064-2076.
[49] Qiu D Y,Xiao J,Ding X H,et al. OsWRKY13 mediates rice disease resistance by regulating defense-related genes in salicylate-and jasmonate-dependent signaling[J]. Molecular Plant-Microbe Interactions,2007,20(5):492-499.
[50] Liu X Q,Bai X Q,Qian Q,et al. OsWRKY03,a rice transcriptional activator that functions in defense signaling pathway upstream of OsNPR1[J]. Cell Research,2005,15(8):593-603.
[51] Liu X,Bai X,Wang X,et al. OsWRKY71,a rice transcription factor,is involved in rice defense response[J]. Journal of Plant Physiology,2007,164(8):969-979.
[52] Sahu G K. Salicylic Acid: Role in Plant Physiology and Stress Tolerance[M]// Molecular Stress Physiology of Plants. India: Springer, 2013:217-239.
[53] Zhang J,Peng Y,Guo Z. Constitutive expression of pathogen-inducible OsWRKY31 enhances disease resistance and affects root growth and auxin response in transgenic rice plants[J]. Cell Research,2008,18(4):508-521.
[54] Wang H,Hao J,Chen X,et al. Overexpression of rice WRKY89 enhances ultraviolet B tolerance and disease resistance in rice plants[J]. Plant Molecular Biology,2007,65(6):799-815.
[55] 仇玉萍,荆邵娟,付 坚,等. 13个水稻WRKY基因的克隆及其表达谱分析[J]. 科学通报,2004,49(18):1860-1869.
[56] Zhou Q Y,Tian A G,Zou H F,et al. Soybean WRKY-type transcription factor genes,GmWRKY13,GmWRKY21,and GmWRKY54,confer
Differential tolerance to abiotic stresses in transgenic Arabidopsis plants[J]. Plant Biotechnology Journal,2008,6(5):486-503.