内蒙古绒山羊是我国优秀绒山羊品种,所产羊绒洁白、柔软、纤细、光泽好,是一类高档的纺织原料,具有很高的经济价值[1]。内蒙古绒山羊包括3个不同类群:阿拉善型、阿尔巴斯型及二狼山型,其中,以阿拉善型所产羊绒品质最佳,其细度和色泽上乘,享誉国内外[2]。线粒体是真核细胞的一种细胞器,拥有其自己的基因组,也能编码细胞器的一些蛋白质[3]。通常,除少数低等真核生物的线粒体基因组是线状DNA分子外,其他生物的线粒体基因组DNA均为闭合环状分子。由于一个细胞内拥有许多线粒体,而且一个线粒体里也有多份基因组拷贝,所以,一个细胞里也就包含多个线粒体基因组。线粒体 DNA (mtDNA)是一种核外遗传物质,与核 DNA 相比,具有分子质量小、结构简单、进化速度快、母性遗传、无组织特异性及提取方便等特点。由于这些特点,使得线粒体基因组成为研究动物起源进化及群体遗传分化的理想对象及研究真核生物分子遗传学、发育生物学和分子系统进化的重要模式体系[4]。就目前来说,线粒体基因组的测定方法主要包括:基于Sanger 测序法的经典线粒体基因组测序;基于PCR方法的线粒体基因组测序;基于靶序列杂交和体外扩增的滚环扩增测序以及基于二代高通量测序技术(NGS)的线粒体基因组测序方法[5-7]。近年来,关于内蒙古绒山羊的研究大多集中于核基因组及其核基因组中功能基因表达调控[8],而有关线粒体基因组的研究鲜有报道。
本研究基于NGS方法分析内蒙古绒山羊线粒体基因组结构和序列,同时对不同绒山羊品种个体之间线粒体DNA序列进行比较分析,旨在通过对内蒙古绒山羊线粒体基因组序列的测定和分析,丰富和扩展数据库,为不同物种间线粒体基因组的结构、特点、进化等分析提供依据。
1 材料和方法
1.1 试验动物
选取内蒙古绒山羊(阿拉善型)成年母羊,使用经灭菌处理过的耳缺钳采集黄豆粒大小耳组织样品,置于2 mL含有75%乙醇的无菌Eppendorf管中带回实验室,组织样品可短期保存于-20 ℃冰箱,用于基因组DNA的提取。
1.2 基因组DNA的提取
按照常规“酚-氯仿”法进行操作和提取[9],通过NanoDrop2000和琼脂糖凝胶电泳并对所提取的绒山羊基因组DNA质量进行检测。
1.3 小插入片段文库制备
基因组DNA经纯化后,利用Covaris进行基因组DNA片段化,构建基因组测序文库,将打断后的小片段基因组分别与接头A、B引物连接,通过琼脂糖凝胶电泳检测,仅筛选DNA片段一侧为接头A、另一侧为接头B的基因组片段,双链变性,得到分离的DNA片段,用于下一步桥式PCR。
1.4 桥式PCR
引物特异性识别互补配对结合于DNA一端,然后结合于芯片上;没有被引物结合的另一端,与旁边的其他引物序列互补识别,同样结合上来,形成一个“桥”,通过PCR扩增得到DNA簇,DNA扩增得到的产物经过线性化形成单链DNA分子,用于下一步高通量测序。
1.5 Illumina PE测序
将dNTP用4种不同的荧光进行标记,加入修饰后的聚合酶,每个循环的反应,只在体系中添加一种含有荧光标记的碱基;通过激光扫描芯片,分别读取每一轮反应过去互补结合上去的核苷酸序列;通过化学法,对“荧光基团”和“终止基团”进行切割,使得3′末端黏性得以恢复,开启下一轮的核苷酸聚合过程;对每一轮反应得到的荧光信号进行收集转化,可转化为模板DNA的序列。
1.6 基因组序列组装
原始数据比对参考基因组,筛选满足条件的reads,进行基因组组装和预测。
1.7 系统发育树分析
登陆NCBI官网,进入BLAST网页(http://blast.ncbi.nlm.nih.gov/Blast.cgi),依次选择Web BLAST→Nucleatide BLAST→blastn的分析模块。下载用于进行系统发育树分析的其他品种的山羊线粒体基因组序列信息,存储为文本文件。在Enter Query Sequence下的选择上传ARS1的参考线粒体序列,同时勾选:“Align two or more sequences”选项。在Enter Subject Sequence步骤中上传本研究的山羊线粒体基因组,在“Algorithm parameters”选项中的“Filters and Masking”,选择“Specifics-specific repeats for Capra hircus (Goat)”,然后进行Blast分析。在生成的结果中查看相关分析内容,选择“Distance tree of results”点击,查看构建的系统发育树的结果。根据分析方法的不同,本研究选择Neighbor Joining方法构建绒山羊的系统发育树,并把结果以Newich file格式保存在本地。随后利用MEGA软件包对此系统发育树进行修剪调整。
1.8 线粒体基因组碱基组成成分分析
利用MEGA软件中的序列分析软件包对不同线粒体的碱基组成进行分析。
2 结果与分析
2.1 基因组DNA提取结果
采用浓度为1%的琼脂糖凝胶,对基因组DNA提取的结果进行了电泳检测,结果显示,所提取基因组DNA为一条较致密、清晰、整齐的条带,并且没有拖尾,说明DNA完整性较好,可以用于后续试验。Nano drop检测结果如表1所示。
表1 DNA检测结果
Tab.1 The quality of total DNA

样品SampleDNA浓度/(ng/μL)Concentration of DNAOD260/280Results of OD260/280内蒙古绒山羊(阿拉善型)981.9Inner Mongolia Cashmere goat(Alashan type)
2.2 高通量测序结果
2.2.1 原始测序数据质控结果 高通量测序得到的原始图像数据经过Base Calling软件,转化为序列数据,结果以FASTQ格式的文件进行储存。对测序reads的每个circle进行碱基分布和质量波动的统计,可以直观地反映出测序样本的测序质量和文库构建质量(图1)。在基因组测序的文库构建中,当建库质量较为理想时,代表不同碱基的不同灰度的界线通常波动极小,基本在一条水平线上。从结果可知,本试验所构建的基因组测序文库碱基分布均一性良好,可用于后续分析。
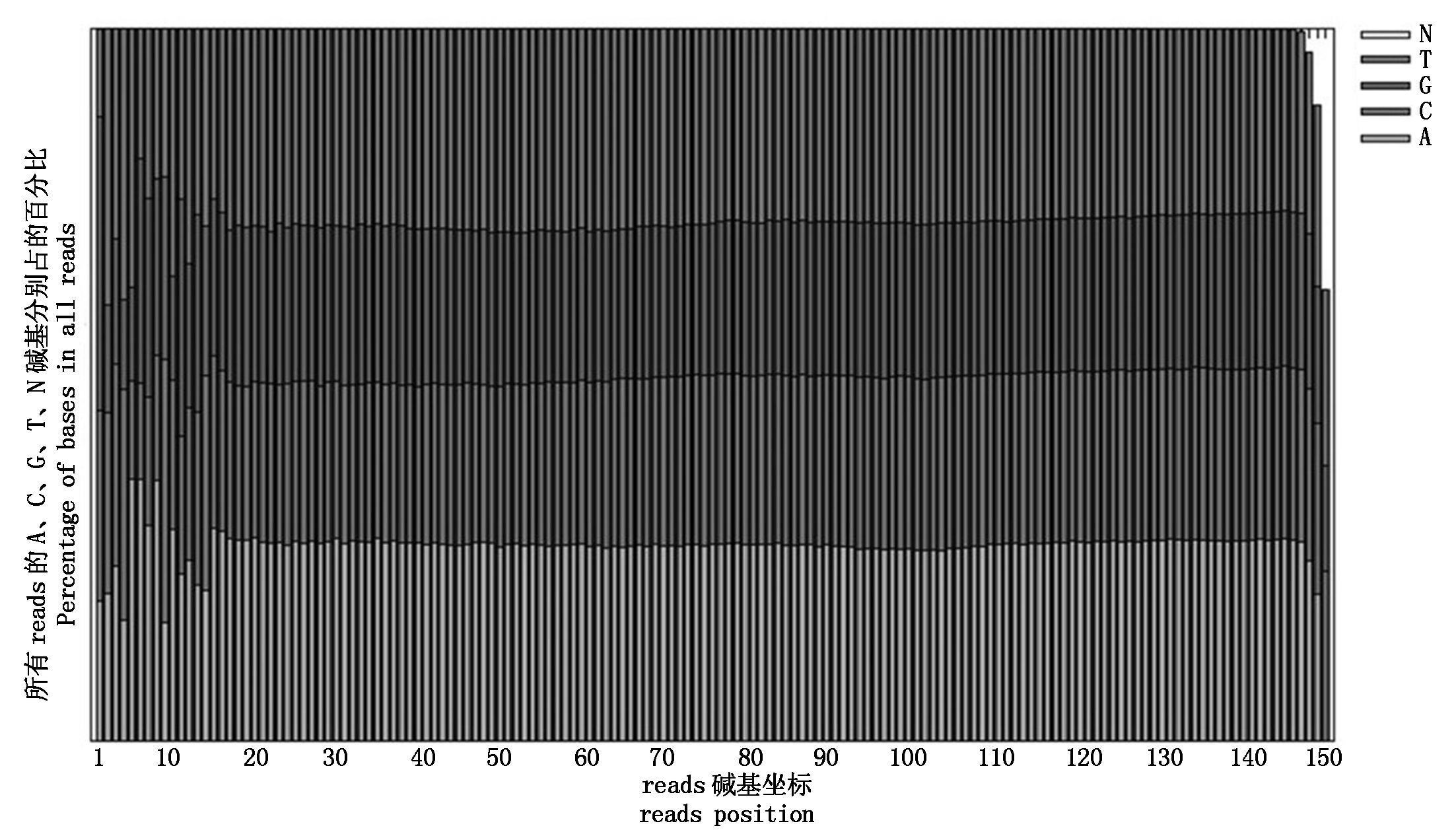
图1 碱基分布
Fig.1 The distribution of bases
2.2.2 测序数据统计结果 比对参考基因组前后的数据分别对测序reads数、总碱基数、Q20进行统计,结果表明(表2),测序共得到2 551 175 100 bp冗余数据,过滤后得到Clean data为1 553 400 bp,质量百分数为95.40%。由于线粒体DNA测序为双末端测序,所以,实际片段数分别为原始测序片段数与过滤后片段数的2倍。
表2 测序数据统计
Tab.2 Sequencing data
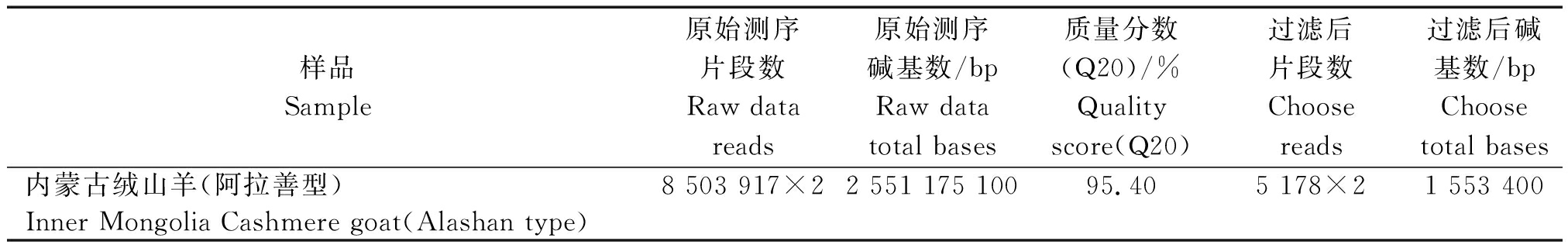
样品Sample原始测序片段数Raw data reads原始测序碱基数/bpRaw data total bases质量分数(Q20)/%Quality score(Q20)过滤后片段数Choose reads过滤后碱基数/bpChoose total bases内蒙古绒山羊(阿拉善型)8 503 917×22 551 175 10095.405 178×21 553 400Inner Mongolia Cashmere goat(Alashan type)
2.2.3 基因组组装结果 采用线粒体专业组装软件mitoMaker http://sourceforge.net/projects/mitomaker/对过滤后的数据进行组装,组装采用默认参数,组装统计结果如表3所示,组装后的线粒体基因组长度为16 642 bp的闭合环状DNA(图2),其基因组GC含量为39.17%,K-mer大小为53 bp。
表3 线粒体基因组组装统计结果
Tab.3 The statistical results of mitochondrial genome assembly
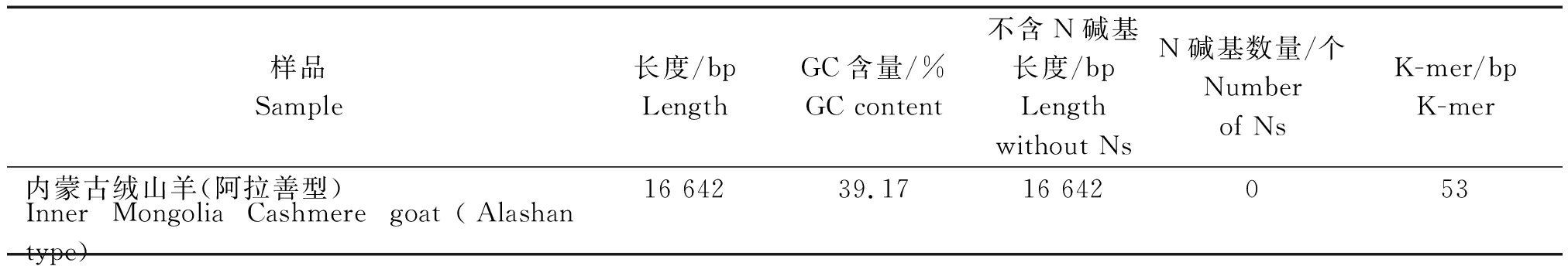
样品Sample长度/bpLengthGC含量/%GC content不含N碱基长度/bpLengthwithout NsN碱基数量/个Number of NsK-mer/bpK-mer内蒙古绒山羊(阿拉善型)16 64239.1716 642053Inner Mongolia Cashmere goat(Alashan type)
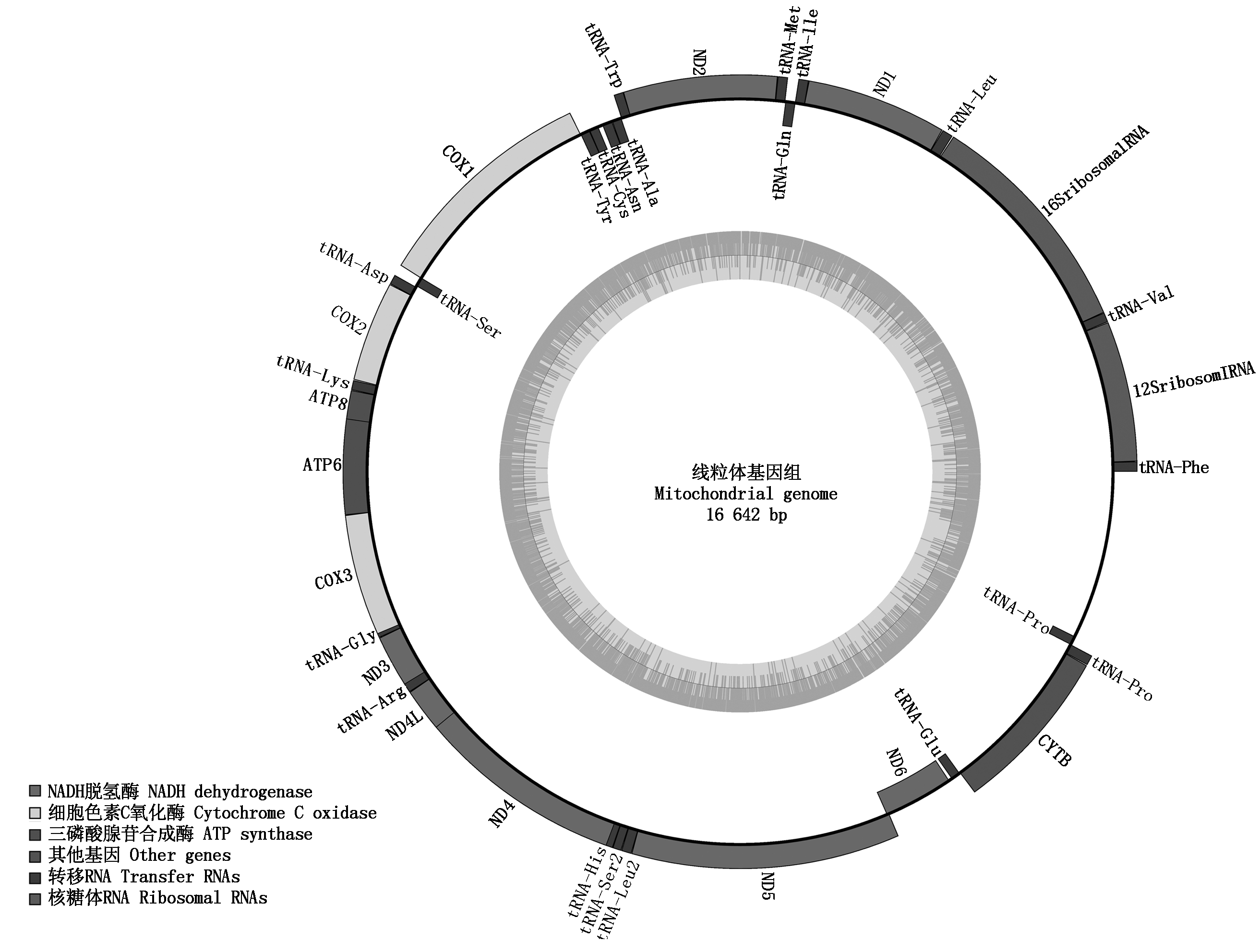
图2 内蒙古绒山羊(阿拉善型)线粒体基因组组装
Fig.2 mtDNA of Inner Mongolia Cashmere goat(Alashan type)
2.2.4 系统发育树构建结果 由图3可知,内蒙古绒山羊的3个类群距离相对比较近,其中,阿拉善型和二狼山型的关系最近,阿尔巴斯型次之;内蒙古绒山羊和辽宁绒山羊可以分为单独的2个类群;与国外品种San Clemente的距离最远。该结果与我国绒山羊选育的历程基本一致。
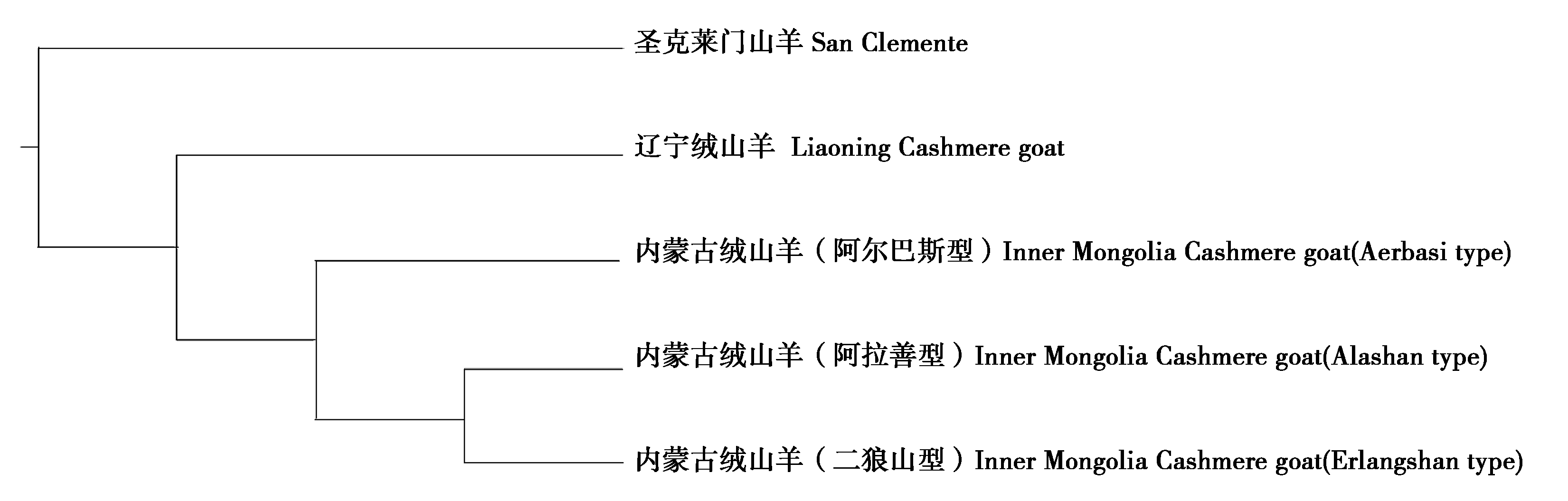
图3 不同品种山羊线粒体基因组系统发育树结果
Fig.3 The results of phylogenetic of different goat mtDNA genome
2.2.5 碱基成分分析结果 利用MEGA软件中的序列分析软件包对不同线粒体的碱基组成进行分析,结果如表4所示,不同品种绒山羊,其线粒体基因组的碱基成分含量基本一致,这表明线粒体基因组在绒山羊的进化过程中比较保守,具有较强的种属特异性。
表4 线粒体基因碱基组成
Tab.4 The base composition of different mitochondrial genome %

样品SampleT(U)CAG圣克莱门山羊San Clemente27.326.033.613.1辽宁绒山羊Liaoning Cashmere goat27.326.133.513.1内蒙古绒山羊(二狼山型)Inner Mongolia Cashmere goat(Erlangshan type)27.326.033.513.1内蒙古绒山羊(阿拉善型)Inner Mongolia Cashmere goat(Alashan type)27.326.033.513.1内蒙古绒山羊(阿尔巴斯型)Inner Mongolia Cashmere goat(Aerbasi type)27.326.033.513.1
3 讨论与结论
本研究中高通量测序采用的是Illumina PE测序平台,可以实现覆盖基因组100×深度的高通量测序,根据序列组装结果,可以获取较为准确的基因组序列信息。该测序方法测序深度高,数据量大,可靠性强[10-11]。由于mi-seq采用双末端测序,需要对数据进行合并,使用FASTX软件去掉序列末尾质量低于Q15的碱基并使用FLASH软件对数据进行合并[12-13];在测序试验的过程中得到的产物,有时候会包含有一些非特异性的扩增片段,通过特异性引物信息可以将其去掉[14-15]。本研究中,在分析的时候,将标签序列去除,并对处理后的有效序列进行数据及长度分布统计。
同其他家畜品种不同,关于羊的线粒体基因组及线粒体DNA的研究较为有限,不如牛、猪等其他家畜研究深入[16-17],然而也取得了一些研究成果,但大多集中在线粒体DNA及其多态性方面。例如,对来自我国18个不同地方山羊品种的线粒体DNA进行了多态性研究,结果表明,我国地方山羊品种可能起源于2个不同的母系祖先[18]。叶绍辉等[19]通过限制性酶切方法对云南当地山羊和朝鲜的土种山羊进行了检测和分析,结果表明,亚洲山羊很可能是源于相同的祖先。贾永红等[20]也通过对线粒体DNA多态性的检测,对贵州不同山羊品种进行了检测和分析,结果表明,贵州山羊可能存在2个不同的母系祖先,分化的年代大约在19 万年前。在绵羊相关的研究中,霍俊宏[21]对我国6个不同的地方绵羊品种的线粒体 DNA 的遗传多样性进行了研究,结果表明,在这些绵羊群体中确实存在 “C”单倍型,表明它们可能共有3个母系起源。通过对线粒体基因组及线粒体DNA进行研究,将对研究羊的起源和进化有帮助[22]。
内蒙古绒山羊的3个不同类型是在内蒙古自治区独特的自然选择和人工选择的条件下形成的独特品种,其中内蒙古绒山羊(阿拉善型)所产的山羊绒是我国绒山羊品种中质量最好的绒毛之一[23-24]。开展内蒙古绒山羊线粒体基因组结构和功能的研究,对于深入了解内蒙古绒山羊生长发育过程中重要经济性状相关的基因表达与调控的遗传机制、核外基因与核内遗传信息的互作、绒山羊重要生理功能及其有关母性遗传的遗传关系具有重要的作用。系统进化树分析,为绒山羊的起源、进化、不同类群的分化以及特定遗传特性的形成机制奠定重要基础。另外,内蒙古绒山羊线粒体基因组序列的测定和分析可以丰富和扩展数据库,为不同物种间线粒体基因组的结构、特点、进化等分析提供依据。通过系统进化树分析亲缘关系的远近,为研究绒山羊起源进化和特定遗传特性的形成机制奠定了重要基础。
[1] 苏 蕊. 内蒙古绒山羊绒毛生长,脱落期皮肤差异表达基因的研究[D]. 呼和浩特:内蒙古农业大学,2009.
[2] 张燕军. 内蒙古绒山羊胎儿期皮肤毛囊形态发生和发育规律及部分Hox基因家族成员研究[D]. 呼和浩特:内蒙古农业大学,2007.
[3] 汪泰初,刘朝良,肖林珍. 线粒体基因组(mtDNA)的研究进展[J]. 安徽农业科学,2006,34(10):2068-2071.
[4] 廖顺尧,鲁 成. 动物线粒体基因组研究进展[J]. 生物化学与生物物理进展,2000,27(5):508-512.
[5] Besnard G,Juhling F,Chapuis E,et al. Fast assembly of the mitochondrial genome of a plant parasitic nematode(Meloidogyne graminicola)using next Generation sequencing[J]. Comptes Rendus Biologies,2014,337(5):295-301.
[6] Williams S T,Foster P G,Littlewood D. The complete mitochondrial genome of a turbinid vetigastropod from MiSeq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny[J]. Gene,2014,533(1):38-47.
[7] 滕婕华,倪 敏,李超群,等. 基于cpDNA和nrITS对濒危植物掌叶木的基因检测[J]. 北方农业学报,2017,45(3):23-28.
[8] 江 玮. 绒山羊皮肤毛囊发育不同时期初级,次级毛囊比较转录组学研究[D]. 呼和浩特:内蒙古农业大学,2015.
[9] J萨姆布鲁克,DW拉塞尔,萨姆布鲁克,等. 分子克隆实验指南[M]. 3版. 北京:科学出版社,2002.
[10] 王兴春,杨致荣,王 敏,等. 高通量测序技术及其应用[J]. 中国生物工程杂志,2012,32(1):109-114.
[11] Chen K,Wang Y,Li X Y,et al. Sequencing and analysis of the complete mitochondrial genome in Anopheles sinensis(Diptera:Culicidae) [J]. Infectious Diseases of Poverty,2017,6(1):149.
[12] Hua Y Q,Ding Y R,Yan Z T,et al. The complete mitochondrial genome of Anopheles minimus (Diptera:Culicidae) and the phylogenetics of known Anopheles mitogenomes[J]. Insect Science,2016,23(3,SI):353-365.
[13] Escalona T,Weadick C J,Antunes A. Adaptive patterns of mitogenome evolution are associated with the loss of shell scutes in turtles[J]. Molecular Biology and Evolution,2017,34(10):2522-2536.
[14] Schiavo G,Hoffmann O I,Ribani A A,et al. A genomic landscape of mitochondrial DNA insertions in the pig nuclear genome provides evolutionary signatures of interspecies admixture[J]. DNA Research,2017,24(5):487-498.
[15] Nejsuma P,Hawash M B,Betson M A,et al. Ascaris phylogeny based on multiple whole mtDNA genomes[J]. Infection Genetics and Evolution,2017,48:4-9.
[16] Horsburgh K A,Prost S,Gosling A,et al. The genetic diversity of the nguni breed of African cattle (Bos spp.):complete mitochondrial genomes of haplogroup T1[J]. PLoS One,2013,8(8):e71956.
[17] Adeola A C,Oluwole O O,Oladele B M,et al. Analysis of the genetic variation in mitochondrial DNA,Y-chromosome sequences,and MC1R sheds light on the ancestry of nigerian indigenous pigs[J]. Genetics Selection Evolution,2017,49(1):52.
[18] 曹丽荣,王小明,饶 刚,等. 从细胞色素b基因全序列分析岩羊和山羊,绵羊的系统发生关系[J]. 兽类学报,2004,24(2):109-114.
[19] 叶绍辉,王 文. 云南保种山羊线粒体DNA限制性酶切研究[J]. 中国畜牧杂志,1998,34(3):11-12.
[20] 贾永红,简承松. 贵州四个山羊品种mtDNA多态性及起源分化[J]. 动物学研究,1999,20(2):88-92.
[21] 霍俊宏. 中国六个地方绵羊品种mtDNA的遗传多样性研究[D]. 晋中:山西农业大学,2005.
[22] 涂正超,张亚平. 藏绵羊线粒体DNA遗传多样性研究[J]. 畜牧兽医学报,1998,29(2):132-135.
[23] Qiao X,Su R,Wang Y,et al. Genome-wide target enrichment-aided chip design:a 66 K SNP chip for cashmere goat[J]. Scientific Reports,2017,7(1):8621.
[24] Li X K,Su R,Wan W T,et al. Identification of selection signals by large-scale whole-genome resequencing of cashmere goats[J]. Scientific Reports,2017,7(1):15142.