由谷瘟病菌引起的谷瘟病是谷子生产中的重要病害之一[1]。近几年,由于谷子种植品种中抗病品种缺乏及谷瘟病菌生理小种的改变,谷瘟病发病有逐渐加重的趋势,在谷子生产上造成严重减产[2]。谷瘟病菌有性态为Magnaporthe oryzae,属于子囊菌门大角间座壳属;无性态为Pyricularia grisea,属有丝分裂孢子真菌中的梨孢属。该梨孢菌除侵染谷子并引致谷瘟病外,还可侵染水稻、大麦、马唐等多种禾本科作物和杂草[3-4],寄主为水稻的灰梨孢菌(Magnaporthe oryzae)称为稻瘟病菌。迄今为止,田间仍未发现谷瘟病菌自然交配形成的有性态。但是,前人研究表明可以在特定的条件下通过人工配对使稻瘟病菌在培养基上产生子囊孢子[5]。子囊真菌交配型控制菌株的交配亲和性及有性生殖,其有性生殖分为同宗配合和异宗配合2种[6-7]。同宗配合的子囊菌的同一株菌株具有2种交配型基因[8],而异宗配合的大多数子囊真菌含有一对高度异源的交配型基因座[7],分别命名为MAT1-1和MAT1-2,其中MAT1-1-1基因中包含一个编码保守alpha盒子结构域(α-box domain),而MAT1-2-1编码高流动蛋白家族(Highly mobility group protein-box,HMG-box)结构域[9-10]。交配型基因编码蛋白主要作为调控因子,参与调控不同交配型菌株在异宗配合时细胞之间相互识别与融合等过程[11]。通过检测菌株是否含有MAT1-1-1和MAT1-2-1交配型基因可确定异宗配合的灰梨孢菌交配型[12]。
传统方法采用标准交配型菌株测定稻瘟病菌交配型,但这种方法具有耗时长,易受温度、光照及菌株自生育性等因素的影响。近年来,利用PCR分子检测的方法研究子囊菌的交配型已十分普遍[12-15]。Brewer等[12]利用多重PCR的方法对葡萄白粉病菌的交配型基因MAT1-1-1和MAT1-2-1进行检测,明确了不同葡萄白粉病菌菌株的交配型。乔广行等[13]应用PCR技术检测灰葡萄孢交配型基因MAT1-1-1和MAT1-2-1,结果发现各种植区交配型菌株所占比例有较大的差异,多数种植区灰葡萄孢同时存在这2种交配类型。Sirisathaworn等[14]利用Samanta等[15]设计的交配型引物对泰国的稻瘟病菌进行检测,结果表明,所有供试稻瘟病菌株属于交配型MAT1-2,表明群体内缺乏有性重组。史文琦等[16]根据白粉病菌的MAT1-1-1与MAT1-2-1部分基因序列设计并筛选特异性引物,利用PCR扩增检测了516株小麦白粉病菌菌株的交配型,结果显示在不同地区均能检测出2种交配型基因,且28个地区的2种交配型菌株约为1∶1。
谷瘟病菌与稻瘟病菌存在寄主专化性,我国不同谷子产区谷瘟病菌交配型基因类型的分布尚未见报道,为明确我国谷子产区谷瘟病菌包含的交配型基因及其分布情况,本研究根据已知的稻瘟病菌交配型基因MAT1-1-1和MAT1-2-1的部分序列设计特异性引物,利用PCR技术对我国不同采集地的谷瘟病菌的交配型基因进行检测,通过分析不同谷子产区谷瘟病菌的不同交配型菌株的分布频率,可用于推测田间谷瘟病菌菌株有性生殖发生的可能性,为谷瘟病的有效防治提供重要指导作用,同时为进一步阐明谷瘟病菌的遗传变异机理提供理论依据。
1 材料和方法
1.1 供试材料
2010-2016年从河北、山东、山西、陕西、吉林、黑龙江、辽宁、内蒙古、新疆和海南等地采集,经过室内分离纯化后得到的谷瘟病菌单孢菌株186株。
1.2 谷瘟病菌基因组DNA的提取
将谷瘟病菌在PDA固体培养基活化,转接到PDA液体培养基中,室温静止培养8 d,收集菌丝,采用常规CTAB法提取谷瘟病菌的基因组DNA。用1.1%的琼脂糖凝胶电泳检测DNA质量及完整性,-20 ℃保存备用。
1.3 谷瘟病菌菌株交配型基因特异性引物的设计及筛选
根据已知稻瘟病菌的交配型基因MAT1-1-1(GenBank登录号AB080672.2)和MAT1-2-1(GenBank登录号AB080673.2)的部分基因序列分别设计3对特异性引物,引物序列参见表1。随机挑选16株谷瘟病菌菌株的DNA进行特异性引物的筛选,PCR反应体系包括2 × Ex Taq Master Mix 12.5 μL,引物各0.5 μL,模板DNA 2 μL(≤200 ng),ddH2O 9.5 μL,总体积25 μL。扩增程序为95 ℃预变性5 min;94 ℃ 30 s,54 ℃ 30 s,72 ℃ 1 min共25次循环;72 ℃ 6 min。1.2%琼脂糖凝胶,恒压110 V电泳对PCR扩增产物进行检测。通过对部分目的条带切胶回收,克隆测序确定为非特异性引物,最终筛选得到特异性较好的2对引物,并对提取的186个不同区域谷瘟病菌基因组DNA进行扩增检测,扩增及电泳条件同上。挑选不同交配型谷瘟病菌菌株各4株进行克隆测序及比对分析,用于比对分析的稻瘟病菌为菌株Y93-164g-1(GenBank登录号AB080672.2)。
表1 交配型基因的引物序列
Tab.1 Primers sequence of mating type genes

交配型基因Mating type genes引物Primer序列(5′-3′)Sequence碱基数Base number预期片段大小/bpExpected fragment sizeMAT1-1-1JPX1-1-S1CATCACCAAACCCTGCGACT20907JPX1-1-A1GTTGGGGATTGTGACGGGTA21JPX1-1-S2CTGGAGGATTTAGAGGTAGCC 20681JPX1-1-A2GAAGGCTTACAAAGACGGCTC 20JPX1-1-S3GAGAGACGCAAGGGATGAAGT21713JPX1-1-A3GGTAGTCGCAGGGTTTGGTGA21MAT1-2-1JPX1-2-S1TCAGCTCGCCCAAATCAACAAT22809JPX1-2-A1ACTCAAGACCCGGCACGAACAT22JPX1-2-S2GCTTTGGGATCTCCAGGTTG201 078JPX1-2-A2TTCTTTGCTTTGCTCGGCTT20JPX1-2-S3GGCTTTGGGATCTCCAGGTT201 053JPX1-2-A3CCCGTTCGTTGGGGTCTTTC20
2 结果与分析
2.1 谷瘟病菌交配型基因MAT1-1-1和MAT1-2-1特异性引物的筛选
随机挑选16株谷瘟病菌菌株的DNA,进行谷瘟病菌交配型基因特异性引物的筛选。检测结果如图1所示,因此,试验确定引物对JPX1-1-S3/JPX1-1-A3和JPX1-2-S1/JPX1-2-A1为检测谷瘟病菌交配型基因MAT1-1-1和与MAT1-2-1的特异性引物并利用这对特异性引物对提取的186株不同区域谷瘟病菌基因组DNA进行扩增检测。
2.2 谷瘟病菌与稻瘟病菌交配型基因编码蛋白的比对分析
利用2对特异性引物(JPX1-1-S1/JPX1-1-A1和JPX1-1-S3/JPX1-1-A3)克隆了4株谷瘟病菌交配型基因MAT1-1-1的alpha盒子结构域(图1)并对谷瘟病菌与稻瘟病菌交配型基因MAT1-1-1的alpha盒子结构域的氨基酸序列进行比对分析,结果发现谷瘟病菌菌株交配型基因MAT1-1-1的alpha盒子结构域不存在变异。以HBhd1菌株为代表(GenBank登录号MF993977),与已知稻瘟病菌参考菌株Y93-164g-1(GenBank登录号AB080672.2)的比对发现,仅在第172位氨基酸存在差异,前者为赖氨酸,后者为谷氨酸(图2)。利用特异性引物(JPX1-2-S1/JPX1-2-A1)克隆了4株谷瘟病菌交配型基因MAT1-2-1的高迁移率蛋白盒子部分序列,比对表明谷瘟病菌之间并不存在变异,以谷瘟病菌菌株SD1(GenBank登录号MF993976)的MAT1-2-1的高迁移率蛋白盒子结构域与稻瘟病菌参考菌株Y93-164a-1(GenBank登录号AB080673.2)之间比对发现,两者存在2个位点的差异,分别在77位与438位的氨基酸处,+77位为酪氨酸/组氨酸、+438位为脯氨酸/精氨酸(图3),表明谷瘟病菌与稻瘟病菌之间存在差异,及进一步研究谷瘟病菌交配型基因的必要性。
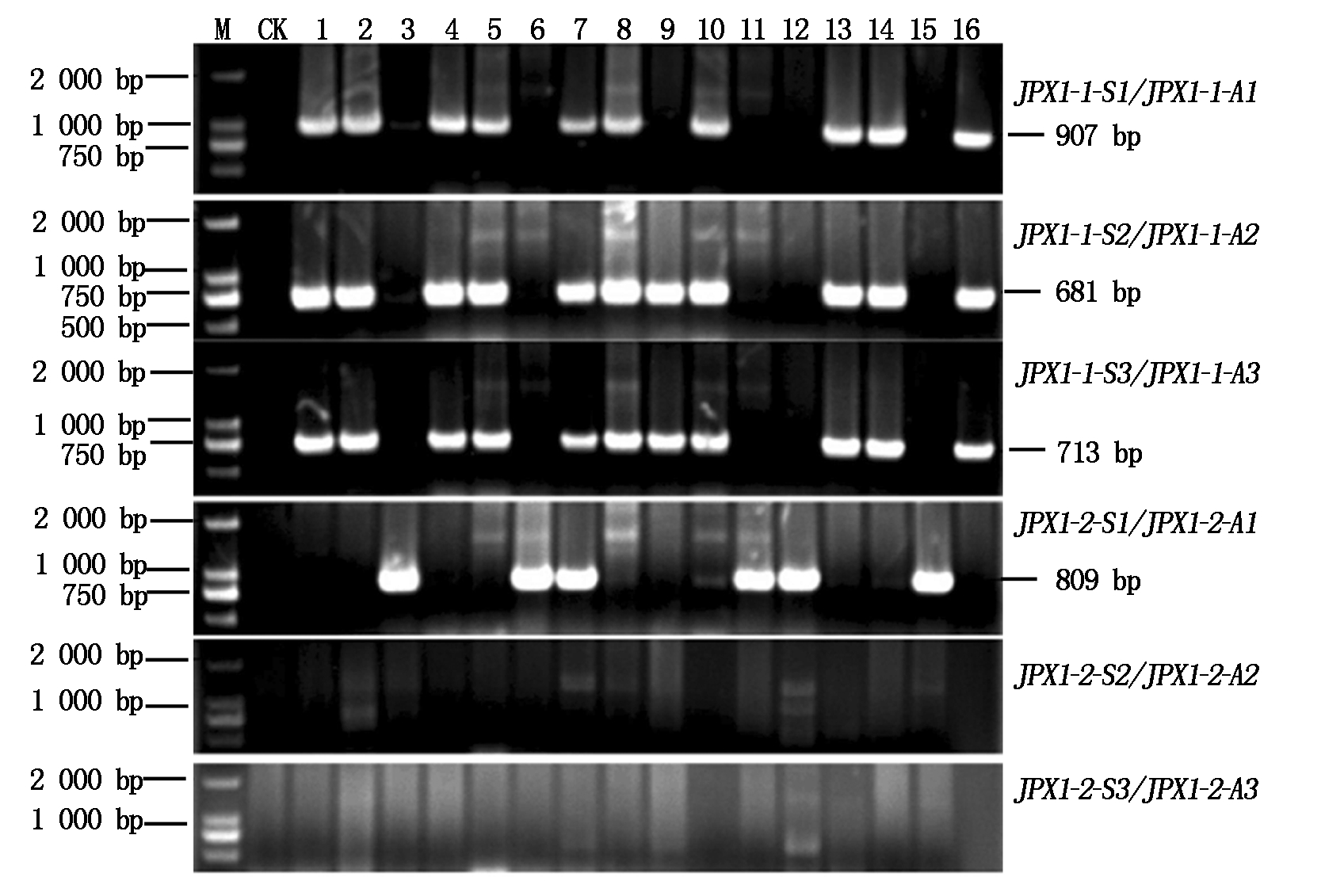
M.DL2000 Marker;CK.阴性对照(ddH2O);1~16.谷瘟病菌菌株样本。 M.Marker;CK.Negative control(ddH2O);1-16.DNA samples of Magnaporthe oryzae isolates.
图1 PCR检测谷瘟病菌交配型基因电泳
Fig.1 Electrophoretogram of PCR-based detection of Magnaporthe oryzae mating type genes

图2 谷瘟病菌与稻瘟病菌的MAT1-1-1 alpha盒子部分氨基酸序列比对
Fig.2 Alignment of MAT1-1-1 alpha-box partial amino acid sequences of Magnaporthe oryzae of Foxtail millet and Magnaporthe oryzae of rice

图3 谷瘟病菌与稻瘟病菌的MAT1-2-1高迁移率蛋白盒子部分氨基酸序列比对
Fig.3 Alignment of MAT1-2-1 HMG-box partial amino acid sequences of Magnaporthe oryzae of Foxtail millet and Magnaporthe oryzae of rice
2.3 不同地区谷瘟病菌交配型类型及分布频率
利用谷瘟病菌交配型基因MAT1-1-1和MAT1-2-1的特异性引物对谷瘟病菌交配型基因 MAT1-1-1和MAT1-2-1的部分基因片段进行扩增,检测结果分别用于鉴定交配型基因座 MAT1-1与MAT1-2不同类型的菌株。PCR 产物的电泳结果表明:谷瘟病菌交配型基因MAT1-1-1引物扩增得到大小为713 bp单一条带(图4),MAT1-2-1引物扩增得到 809 bp单一条带(图5)。通过比对图4与图5可知,谷瘟病菌菌株2号、5号和15号(菌株分别为HBsjz1、HN2和HBxt1)为双交配型菌株。
通过PCR 技术对谷瘟病菌交配型进行地理分布类型检测,结果发现,186个谷瘟病菌菌株中,交配型为MAT1-1的谷瘟病菌共101株,占比54.30%;交配型为MAT1-2的谷瘟病菌共75株,占比40.32%,总体上接近1∶1;同时具有MAT1-1和MAT1-2的双交配型菌株共10株,占5.38%。除黑龙江与内蒙古地区只含有单一的交配型菌株外,其余各地区均含有2种交配型基因。各种植区间两交配型菌株所占的比例有较大差异,其中夏谷区不同交配型菌株所占比例存在明显差异,其交配型为MAT1-1的谷瘟病菌占69.15%,而交配型为MAT1-2的菌株仅占26.60%,其余4.25%菌株为双交配型菌株,出现在河北和河南地区;春谷区谷瘟病菌交配型基因MAT1-1占38.55%,而交配型MAT1-2占比为56.63%,其余4.82%菌株为双交配型菌株,出现在山西、陕西及辽宁地区,但总体水平上谷瘟病菌的2种交配型保持平衡状态(表 2)。对表2数据分析可知,不同地区交配型菌株分布也不同,如辽宁MAT1-2交配型菌株占比为50%,吉林省MAT1-2交配型菌株占比75%,黑龙江MAT1-2交配型菌株占比100%。
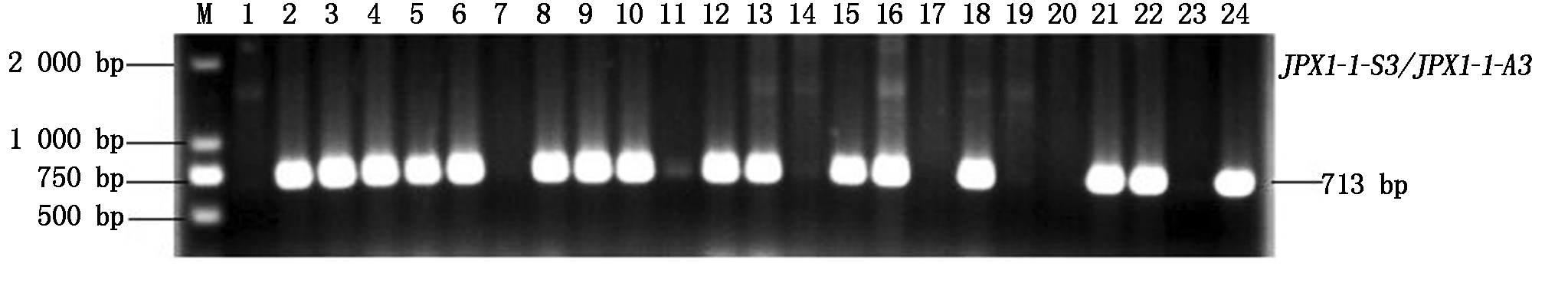
M.DL2000 Marker;713 bp.MAT1-1-1基因条带;1~24菌株编号:HBcd1、HBsjz1、SD2、JL1、HN2、Sx4、Sx3、SD1、Hn2、Hn1、LN1、HBhd1、HBxt2、HBsjz2、HBxt1、Sx2、HBxt3、SX1、SX2、HBsjz4、XJ1、XJ2、HLJ1、HN6。图5同。
M.DL2000 Marker;713 bp.MAT1-1-1;1-24.The following isolates in order:HBcd1,HBsjz1,SD2,JL1,HN2,Sx4,Sx3,SD1,Hn2,Hn1,LN1,HBhd1,HBxt2,HBsjz2,HBxt1,Sx2,HBxt3,SX1,SX2,HBsjz4,XJ1,XJ2,HLJ1,HN6.The same as Fig.5.
图4 PCR检测谷瘟病菌交配型基因MAT1-1-1电泳
Fig.4 Electrophoretogram of PCR-based detection of Manaporthe oryzae MAT1-1-1

M.DL2000 Marker;809 bp.MAT1-2-1基因条带。
M.DL2000 Marker;809 bp.MAT1-2-1.
图5 PCR检测谷瘟病菌交配型基因MAT1-2-1电泳
Fig.5 Electrophoretogram of PCR-based detection of Manaporthe oryzae MAT1-2-1
表2 谷瘟病菌交配型分布分子测定结果
Tab.2 The result of molecular detection mating types of Manaporthe oryzae

采集地点Location菌株数No.MAT1-1MAT1-2MAT1-1和MAT1-2MAT1-1 and MAT1-2菌株数No.频率/%Frequency菌株数No.频率/%Frequency 菌株数No.频率/%Frequency 夏谷区 河北保定844.2644.2600.00Summer sowing region河北张家口222.1300.0000.00河北邢台211414.8966.3811.06河北石家庄1366.3866.3811.06河北邯郸131212.7711.0600.00河北沧州222.1300.0000.00河南211718.0922.1322.13山东1488.5166.3800.00合计946569.152526.6044.25春谷区山西2589.641619.2811.20Spring sowing region陕西1267.2344.8222.41吉林411.2033.6100.00黑龙江500.0056.0200.00辽宁1467.2378.4311.20内蒙古333.6100.0000.00新疆422.4122.4100.00河北承德1256.0278.4300.00北京411.2033.6100.00合计833238.554756.6344.82海南Hainan9444.44333.33222.22合计Total18610154.307540.32105.38
3 讨论
Hebert[17]从马唐(Digitaria sanguinalis L.)上分离到灰梨孢菌(P. grsiea),并在人工培养基上成功的诱发形成子囊壳。随后Barr[18]根据稻瘟病菌的形态学特征、细胞学特征及寄主范围等多方面来分析,将稻瘟病菌的有性世代修正为Magnaporthe grisea。Couch等[19]根据稻瘟病菌(M. grisea)的肌动蛋白、β微管蛋白和钙调素等基因基于系统发育分析,将从马唐中分离株(马唐瘟菌)有性态称为Magnaporthe grisea。其他来自禾本科植物包括水稻和谷子的分离株(灰梨孢菌)有性态统一命名为新物种Magnaporthe oryzae。李成云等[20]对稻瘟病菌有性世代形成能进行研究,通过单胞菌株与标准菌株杂交证明MAT1-1型有较强的有性世代形成能力,单株培养能产生子囊壳证明稻瘟病菌是从同宗配合进化到异宗配合的。交配型基因(Mating type gene,MAT)作为调控真菌有性生殖的一类重要转录因子,在子囊菌中广泛存在[21-22]。近年来,关于交配型基因结构和功能及基于分子进化的亲缘关系研究有诸多报道[23-25]。随着分子生物学的发展,近年来更多的植物病原菌的交配型通过PCR技术进行了分子测定[13-14,16]。与传统的方法相比,PCR技术具有更快速、准确、可靠的特点。
我国不同谷子产区谷瘟病菌交配型基因类型的分布尚未见报道,关于稻瘟病菌交配型基因的相关研究也都集中在南方地区。本研究首次利用PCR技术对来自我国河北、山东、山西、陕西、吉林、黑龙江、辽宁、内蒙古、新疆和海南等不同谷子种植区186个谷瘟病菌菌株的交配型进行分析,结果发现MAT1-1、MAT1-2以及双交配型分别占54.30%,40.32%,5.38%,交配型MAT1-1、MAT1-2总体接近1∶1的比例,推测其原因可能是谷瘟病菌在自然界中存在有性世代。不同省份谷子种植区中除黑龙江与内蒙古外均含有2种交配型基因,黑龙江与内蒙古只含有单一的交配型菌株,原因可能为无性分生孢子繁殖占主导地位,也可能是该地区地理环境特异性导致。如黑龙江省属于春谷区的最北端,是我国谷子栽培区域的北限,该区具有气候寒冷、无霜期短等环境特征,可能会对谷瘟病菌交配型基因的类型产生影响。以东北三省为例,从南向北辽宁MAT1-2交配型菌株所占比例50%<吉林省MAT1-2交配型菌株所占比例75%<黑龙江MAT1-2交配型菌株所占比例100%。不同谷子种植生态区中,谷瘟病菌不同交配型菌株所占比例有较大差异,夏谷区尤为明显,其中谷瘟病菌交配型基因MAT1-1占69.15%,而交配型MAT1-2仅占26.60%。春谷区谷瘟病菌交配型基因MAT1-1占38.55%,而交配型MAT1-2占比为56.63%。春谷区与夏谷区中双交配型菌株占比差别不大分别为4.25%与4.82%。这种不同生态种植区存在的交配型菌株的偏好性还需要更多谷瘟病菌菌株进行验证。在河北、河南、山西和陕西等谷子种植区发现了双交配型的谷瘟病菌菌株,谷瘟病菌中双交配型菌株的发现证实谷瘟病菌是由同宗配合进化到异宗配合的,谷瘟病菌在自然界中可能存在有性世代。通过对谷瘟病菌与稻瘟病菌的交配型基因MAT1-1的alpha盒子部分氨基酸序列和MAT1-2高迁移率蛋白盒子氨基酸序列比对分析发现,两者亲缘关系极为接近,但序列并不完全相同,任世龙等[26]对谷瘟病菌和稻瘟病菌IGS序列研究同样表明,两者亲缘关系很近,但存在超越种内之间的差异。2对引物可完成对186株谷瘟病菌交配型基因的检测,其余2对MAT1-1-1引物的互补验证结果表明,这2对引物特异性较好。史文琦等[16]对利用PCR方法鉴定的不同交配型小麦白粉菌株随机选取16个进行配对接种,16个杂交组合都能产生闭囊壳,证实了PCR检测交配型基因的可靠性。
本研究建立了快速PCR检测谷瘟病菌交配型的方法,通过分子生物学方法快速测定谷瘟病菌菌株交配型的类型,可对不同交配型遗传背景的谷瘟病菌进行室内有目的的有性杂交,对基于不同交配型遗传背景的谷瘟病菌群体结构与遗传分化分析具有重要意义。但是对谷瘟菌株交配型鉴定需要通过2对引物扩增,分别电泳检测,步骤比较繁琐,今后可以摸索建立多重PCR检测体系,可以更加快速检测谷瘟病菌交配型基因。另外不同谷瘟病菌即使有不同交配型,也不一定形成有性世代,因此对本研究确定的不同交配型还需要进一步对峙培养,以确定其育性。通过有性生殖产生新的遗传重组后代,是植物病原真菌变异进化的重要来源之一,所以了解谷瘟病菌的不同交配型菌株的分布频率,可用于推测田间谷瘟病菌菌株有性生殖发生的可能性,这对于该病害的有效防治具有非常重要的指导作用。
参考文献:
[1] Chiapello H,Mallet L,Guerin C,et al. Deciphering genome content and evolutionary relationships of isolates from the fungus Magnaporthe oryzae attacking different host plants[J]. Genome Biology and Evolution,2015,7(10):2896-2912.
[2] 李志江,贾冠清,李祥羽,等. 谷瘟病菌生理小种鉴别及谷子标准品种体系的构建[J]. 中国农业科学,2016,49(17):3308-3318.
[3] Choi J,Park S Y,Kim B R,et al. Comparative analysis of pathogenicity and phylogenetic relationship in Magnaporthe grisea species complex[J]. PLoS One,2013,8(2):e57196.
[4] Gallet R,Fontaine C,Bonnot F,et al. Evolution of compatibility range in the Rice-Magnaporthe oryzae system:an uneven distribution of R genes between rice subspecies[J]. Phytopathology,2016,106(4):348-354.
[5] Notteghem J L,Silué D. Distribution of the mating type alleles in Magnaporthe grisea populations pathogenic on rice[J]. Phytopathology,1992,82(4):423-426.
[6] Angelini R M D M,Rotolo C,Pollastro S,et al. Molecular analysis of the mating type ( MAT1 ) locus in strains of the heterothallic ascomycete Botrytis cinerea[J]. Plant Pathology,2016,65(8):1321-1332.
[7] Debuchy R,Turgeon B G. Mating-type structure,evolution,and function in euascomycetes[J]. Growth Differentiation & Sexuality,2006,1(1):293-323.
[8] Desjardins A E,Brown D W,Yun S H,et al. Deletion and complementation of the mating type (MAT) locus of the wheat head blight pathogen Gibberella zeae[J]. Applied and Environmental Microbiology,2004,70(4):2437-2444.
[9] Amorim R,Savi D C,Ferreira-Maba L A,et al. MAT gene idiomorphs suggest a heterothallic sexual cycle in the citrus pathogen Phyllosticta citricarpa[J]. European Journal of Plant Pathology,2017,147(2):325-337.
[10] Pöggeler S. Mating-type genes for classical strain improvements of ascomycetes[J]. Applied Microbiology and Biotechnology,2001,56(5/6):589-601.
[11] Kanamori M,Kato H,Yasuda N,et al. Novel mating type-dependent transcripts at the mating type locus in Magnaporthe oryzae[J]. Gene,2007,403(1/2):6-17.
[12] Brewer M T,Cadle-Davidson L,Cortesi P,et al. Identification and structure of the mating-type locus and development of PCR-based markers for mating type in powdery mildew fungi[J]. Fungal Genetics & Biology,2011,48(7):704-713.
[13] 乔广行,李兴红,黄金宝,等. 灰葡萄孢交配型基因的分析与检测[J]. 菌物学报,2015,34(1):108-116.
[14] Sirisathaworn T,Srirat T,Longya A,et al. Evaluation of mating type distribution and genetic diversity of three Magnaporthe oryzae,avirulence genes,PWL-2,AVR-Pii,and Avr-Piz-t,in Thailand rice blast isolates[J]. Agriculture & Natural Resources,2017,51(1):7-14.
[15] Samanta S,Dhua U,Nayak S,et al. Mating types analysis of Magnaporthe oryzae populations by molecular methods[J]. Open Biotechnology Journal,2014,8(1):6-12.
[16] 史文琦,龚双军,曾凡松,等. 小麦白粉病菌有性生殖与自然群体交配型检测[J]. 植物病理学报,2016,46(5):645-652.
[17] Hebert T T. The perfect stage of Pyricularia grísea[J]. Phytopathology,1971,61(1):83-87.
[18] Barr M E. Magnaporthe,telimenella,and hyponectria(Physosporellaceae)[J]. Mycologia,1977,69(5):952-966.
[19] Couch B C,Kohn L M. A multilocus gene genealogy concordant with host preference indicates segregation of a new species, Magnaporthe oryzae,from M. grisea[J]. Mycologia,2010,94(4):683-693.
[20] 李成云,李家瑞,沈 锐,等. 稻瘟病菌的有性世代形成能——南省稻瘟病菌的有性世代研究之三[J]. 西南农业学报,1992,5(1):53-58.
[21] Bihon W,Wingfield M J,Slippers B,et al. MAT gene idiomorphs suggest a heterothallic sexual cycle in a predominantly asexual and important pine pathogen[J]. Fungal Genetics and Biology,2014,62:55-61.
[22] Kano R,Yoshida E,Yaguchi T,et al. Mating type gene (MAT1-2) of Trichophyton verrucosum[J]. Mycopathologia,2014,177(1/2):87-90.
[23] Sun Y,Corcoran P,Menkis A,et al. Large-scale introgression shapes the evolution of the Mating-type chromosomes of the Filamentous ascomycete neurospora tetrasperma[J]. PLoS Genetics,2012,8(7):e1002820.
[24] Bolton M D,De Jonge R,Inderbitzin P,et al. The heterothallic sugar beet pathogen Cercospora beticola contains exon fragments of both MAT genes that are homogenized by concerted evolution[J]. Fungal Genetics and Biology,2014,62:43-54.
[25] Kano R,Anzawa K,Mochizuki T,et al. Sporothrix schenckii(sensu strict S. globosa)mating type 1-2(MAT1-2)gene[J]. The Journal of Dermatology,2013,40(9):726-730.
[26] 任世龙,白 辉,董 立,等. 中国不同地理来源谷瘟病菌rDNA-IGS序列分析[J]. 植物病理学报,2017,47(3):305-312.