干旱胁迫正在严重影响着全球的水稻产量和种植面积[1],因此,揭示水稻抗逆的分子机制、克隆出与干旱胁迫相关的重要功能基因以及相应的基因工程改良对于提高水稻抗逆性和环境适应性、扩大水稻生产面积具有重要的意义[2]。转氨酶(EC 2.6.1.x)是催化氨基酸与酮酸之间氨基转移的一类酶,参与氨基酸的分解和合成,普遍存在于动物、植物组织和微生物中。通过生物信息学及酶工程学方法对转氨酶基因编码的相应产物及其特异底物、非自然底物进行了分析,初步验证了其在生物代谢中的各种功能[3]。由转氨酶催化的反应在各种代谢途径中起着不可或缺的作用,包括氨基酸的生物合成和分解代谢、维生素代谢、碳和氮的同化和穿梭、次生代谢、光呼吸、乙醛酸解毒和糖异生作用[4]。通常大多数氨基转移酶反应是自由可逆的;然而,存在由产物不稳定性,产物通道化或氨基转移酶对反应产物的低亲和力产生的单向转氨酸的情况[5-7]。在植物体内,转氨酶参与植物的生长、代谢和抵抗非生物胁迫。转氨酶Ⅲ家族是转氨酶家族中重要的一个亚族,通过对拟南芥中的转氨酶进行基因组分析和与其他物种的比对以及对转氨酶已知与未知功能的基因序列进行MPSS分析,预测了未知基因的功能,并将其分为4个家族[4]。现已在拟南芥体内确定了6种转氨酶Ⅲ家族基因AcOAT、δ-OAT、GSA1、GSA2、GABA-T、DAPA-AT,其功能也已确定,其中,δ-OAT(δ-鸟氨酸转氨酶)基因的表达与提高植物渗透胁迫耐性和脯氨酸的含量有密切联系,而将拟南芥δ-OAT基因转入水稻体内后对高盐和干旱胁迫都表现出较强的忍耐力。GABA-T在拟南芥生长过程中不仅参与叶的生长、乙醛酸的合成,同时也响应盐胁迫。在水稻中,通过将水稻与拟南芥中、粳稻与籼稻中的转氨酶Ⅲ家族基因进行了系统发育分析并在低温、干旱、高盐条件下对其进行了实时定量 PCR分析,结果发现,12个转氨酶基因在非生物胁迫下对至少一种逆境有响应[8]。
本研究拟通过RT-PCR法克隆粳稻转氨酶Ⅲ家族基因中的OsAMTR310基因并分析其在水稻苗期干旱胁迫下的表达模式,将克隆出的目的基因片段与超表达载体连接,并通过农杆菌介导法将其转入水稻体内,培育出转基因水稻植株,进一步分析转基因植株在干旱条件下的表型差异和生理变化,为揭示OsAMTR310基因的功能和利用其培育抗逆水稻新品种奠定基础。
1 材料和方法
1.1 试验材料
水稻品种野生型日本晴(Oryza sativa L. subsp. japonica cv. Nipponbare)与转基因受体水稻东农427种子由东北农业大学寒地水稻研究中心保存。TRIzol RNA 提取试剂盒(RNAisoTM Plus)、M-MLV反转录酶、DNase Ⅰ、质粒小提试剂盒和荧光定量PCR试剂盒、大肠杆菌DH5α、Taq DNA 聚合酶、DL2000 DNA Marker、克隆载体pMD18-T、限制性内切酶购自宝生物(大连)有限公司(TaKaRa Co.Ltd. Dalian,China);超表达载体pCAMBIA1302、根癌土壤农杆菌株EHA105由东北农业大学生命科学与生物技术研究中心保存。
1.2 试验方法
1.2.1 植物材料培养 水稻种子经10%次氯酸钠溶液表面消毒后,播于1/2 MS固体培养基上,置于光照培养箱(22 ℃)中培养。14 d后,当幼苗长出2片真叶时,将幼苗移栽于土壤中,然后转入人工气候室中培养,光照周期为光/暗16 h/8 h,温度为28 ℃/22 ℃,相对湿度70%[9]。
1.2.2 水稻总RNA 的提取和cDNA 第一链的合成 用TRIzol法提取上述水稻叶片的RNA[10],具体方法参照产品说明书,并用DNase Ⅰ去除RNA中可能存在的基因组DNA。再以RNA为模板,参照反转录试剂盒的说明反转录合成cDNA,再以其为模板进行RT-PCR反应。
1.2.3 OsAMTR310基因的克隆与序列生物信息学分析 用Primer premier 5.0软件在基因的编码序列两端设计引物OsAMTR310F(5′-CATGCCATGGATGATTTGCCGCAGTCTCCT-3′)和OsAMTR310R(5′-GACTAGTACCCCTCTTGGCTTTGAGCTC-3′),在引物的5′端分别加入一个Nco Ⅰ酶切位点(上游)、SpeⅠ酶切位点(下游)。25 μL PCR反应体系包括:10×PCR Buffer 2.5 μL、dNTP Mixture 2.5 μL、Primer1 0.5 μL、Primer2 0.5 μL、DNA模板1 μL、Taq 0.2 μL、H2O 17.8 μL。反应过程为:94 ℃预变性10 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃终延伸10 min。反应产物于4 ℃保存。根据测序结果,通过水稻基因组数据库对测序基因序列进行比对,以验证所测基因序列是否为目的基因。
1.2.4 超表达载体的构建及遗传转化 确定测序正确以后,将pCAMBIA1302表达载体与含目的片段的质粒用相应的限制性内切酶进行双酶切,将目的基因片段与表达载体连接,送至华大生物公司进行测序。根据片段的测序结果,通过NCBI对测序基因进行序列比对验证其正确性。将含有目的基因的超表达载体转入农杆菌EHA105中,并通过侵染东农427水稻愈伤组织完成遗传转化[11],并筛选阳性转基因植株。
1.2.5 PCR鉴定阳性OsAMTR310转基因植株 当植株生长正常后,从幼嫩叶片中通过CTAB法提取DNA进行PCR鉴定。根据pCAMBIA1302载体上的抗性筛选基因序列设计PCR检测引物hygF (5′-GTCTCCGACCTGATGCAGCTCTCGG-3′),hygR(5′-GTCCGTCAGGACATTGTTGGAG-3′),以上述DNA为模板、25 μL体系进行PCR反应,反应程序为:94 ℃预变性10 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃终延伸10 min。反应产物于4 ℃保存。PCR反应产物用1.5%琼脂糖凝胶电泳检测。
1.2.6 蛋白提取与免疫印迹分析 转基因植株与正常对照植株生长35 d左右时,取正常生长的叶子并迅速置于液氮中保存;之后干旱处理相应的转基因植株与对照植株24 h,并取叶片置于液氮中保存。植物总蛋白提取采用生工生物工程(上海)公司的植物总蛋白提取试剂盒,提取后的蛋白在-80 ℃保存。蛋白质免疫印迹的试验过程:SDS-PAGE电泳后,取出胶,按照负极到正极的方向,将滤纸、凝胶、NC膜、滤纸按顺序放好,放在电泳槽中,接通电源,恒压反应90 min;反应结束后用丽春红染色2~3 min,剪下所需条带部分;用新鲜配制的5%的脱脂乳37 ℃封闭2 h;一抗4 ℃孵育过夜;将NC膜放在TBS/T缓冲液中清洗。加入二抗,37 ℃孵育1 h;将NC膜在TBS/T中洗3次,每次10 min;将NC膜放入化学发光成像仪中,调整曝光时间直到得到清晰的目的条带[12]。
1.2.7 Real-time PCR分析转基因水稻在干旱胁迫下的表达模式 分别取胁迫处理后的阴性对照植株和超表达植株的新鲜叶片并提取其RNA,试验设置3次重复。根据OsAMTR310的CDS序列,用Primer premier 5.0软件根据转录序列设计引物OsAMTR310qF(5′-AAGAAAATGGGTTCAAGGGGC-3′),OsAMTR310qR(5′-TTCCAAAAGGAGTGGTAGAAGGGT-3′),用NCBI的Blast程序在水稻基因组数据库中验证这2个基因的扩增特异性。以Actin1 作为内参基因[13]。Real-time PCR 在Roche LightCycler 2.10中进行,PCR反应程序为:95 ℃ 5 min;95 ℃ 10 s,60 ℃ 20 s,72 ℃ 20 s,45个循环。用2-ΔΔCT法计算基因的表达量,其中ΔΔCT = (CT目的基因 -CT内参基因)处理样品-(CT目的基因-CT内参基因)对照样品[14]。试验设3次重复。
1.2.8 转基因植株的生理指标测定 测定干旱胁迫后对照植株与转基因植株的存活率,试验设置3次重复;并且分别测定胁迫处理前后对照植株和转基因植株的游离脯氨酸含量。水稻叶片中的脯氨酸含量通过酸性茚三酮法测定[15]。
2 结果与分析
2.1 OsAMTR310基因的克隆与序列分析
OsAMTR310基因cDNA全长为1 873 bp,编码区全长1 533 bp[8]。根据在Phytozome数据库中比对出的OsAMTR310基因的CDS序列设计了一对引物OsAMTR310F和OsAMTR310R,并以水稻幼苗叶片的cDNA为模板进行PCR扩增,获得了一条1 500 bp左右的片段(图1),将其与pMD18-T载体连接,转入大肠杆菌DH5α后将菌液送公司测序,确认其片段长度为1 533 bp,与笔者在Phytozome数据库中比对出的OsAMTR310基因的开放阅读框(ORF)长度一致,起始密码子为ATG,终止密码子为TAG。
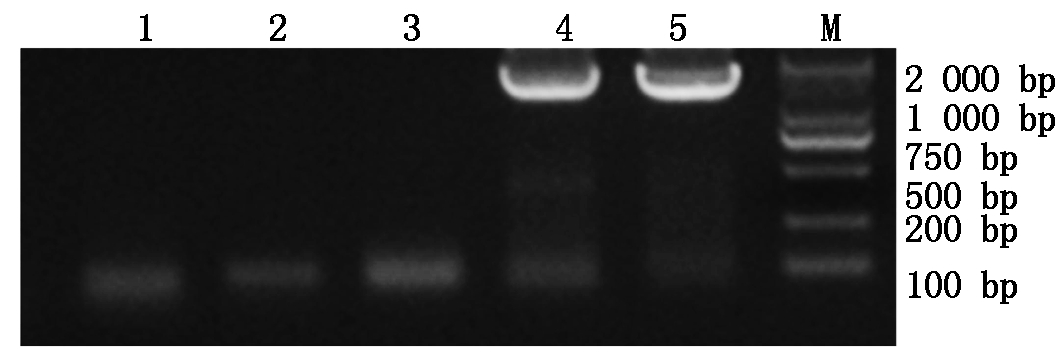
M.DL2000;1.无模板无引物的阴性对照;2.有模板无引物的阴性对照;3.无模板有引物的阴性对照;4~5.OsAMTR310的PCR产物。
M.DL2000;1.Negative control without template and primers;2.Negative control without primers with template;3.Negative control without template with primers;4-5.PCR products of OsAMTR310.
图1 OsAMTR310基因CDS序列PCR扩增
Fig.1 PCR amplification of CDS sequence of OsAMTR310
2.2 OsAMTR310的蛋白质生物信息学分析
OsAMTR310编码的蛋白质产物是一个4-氨基丁酸酯-丙酮酸转氨酶,包括510个氨基酸,分子量为56 ku,推测等电点为6.34,蛋白质稳定系数为49.76。在与其他水稻转氨酶Ⅲ家族成员的序列比对后,预测OsAMTR310具有3个保守的结构域,一个4-氨基丁酸酯-丙酮酸转氨酶结构域,一个腺苷甲硫氨酸-8-氨基-7-氧代壬酸氨基转移酶结构域和一个乙酰鸟氨酸氨基转移酶家族的共有保守结构域。
2.3 转基因植株的分子鉴定
OsAMTR310基因通过农杆菌介导法转入到粳稻品种东农427中,在第1轮筛选中,根据转化株的潮霉素抗性进行筛选,总共选择了40个潜在的转基因植株,然后转移到温室土壤中。当植物达到约12 cm时,通过PCR扩增进行第2次筛选。结果表明,9个株系显示PCR阳性(图2)。所有检测中,非转基因的东农427均为PCR阴性。对这些转基因株系用GFP抗体和his抗体进行了蛋白质免疫印迹分析,结果显示,3个转基因株系S2、 S4和 S7的OsAMTR310-GFP蛋白在转基因水稻中高度表达(图3)。对这3个转基因水稻株系进行了进一步的分子鉴定,在干旱环境下调查了3个转基因植株的表达模式。如图4所示,OsAMTR310的表达明显受干旱诱导,其在干旱条件下的相对基因表达量是正常条件下的3.0倍,而且在转基因株系中的相对表达水平要显著高于野生型水稻,这个结果揭示了OsAMTR310表达的特异性和对环境压力做出的应答。
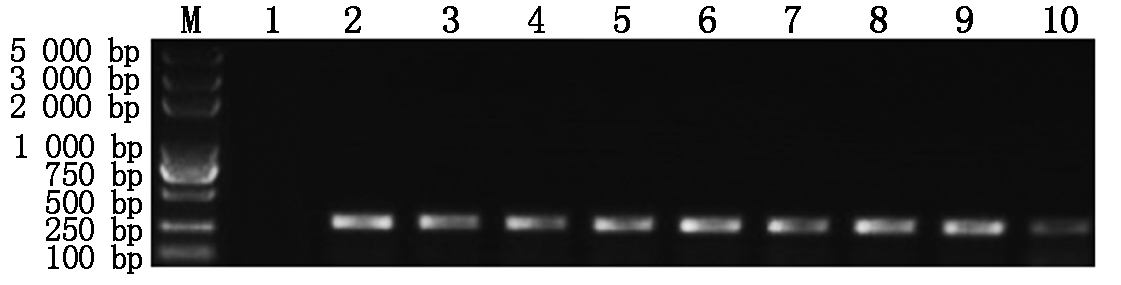
M.DL5000;1.阴性东农427;2~10.转基因株系。
M.DL5000;1.Negative Dongnong 427;2-10.Transgenic lines.
图2 OsAMTR310转基因水稻的PCR鉴定
Fig.2 Identification of transgenic rice of OsAMTR310 by PCR amplification
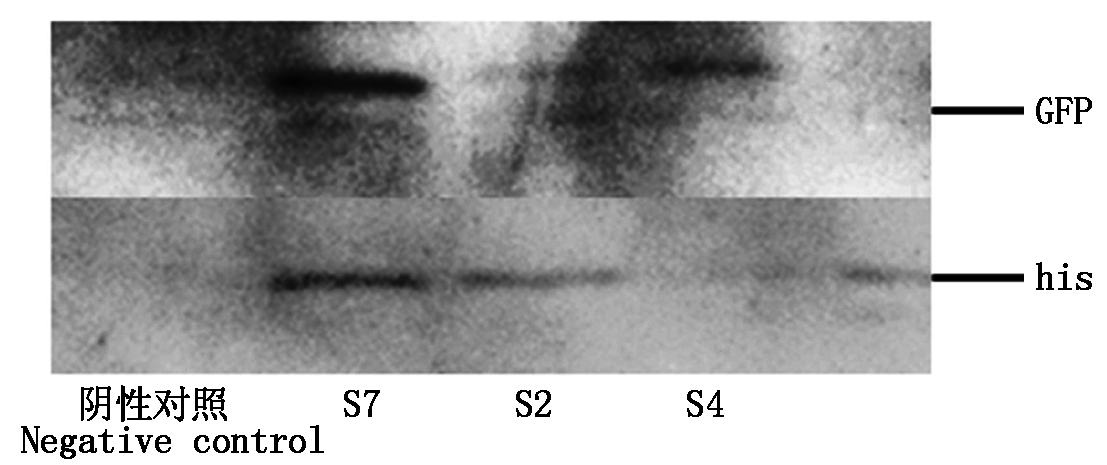
图3 OsAMTR310-GFP/his融合蛋白质的免疫印迹分析
Fig.3 Immunoblot analysis of OsAMTR310-GFP/his fusion protein
2.4 转基因植株的生理鉴定
分别取转基因与野生型水稻幼苗在正常生长条件和干旱胁迫条件下的叶片,用比色法测定叶片中游离脯氨酸的含量(以鲜质量计),结果表明,在正常条件下,转基因和野生型水稻之间的游离脯氨酸含量并不明显,然而,经过干旱胁迫后,转基因水稻的脯氨酸水平是野生型水稻的1.5~2.0倍(图5),表明OsAMTR310调节干旱胁迫下的水稻幼苗中游离脯氨酸的积累。
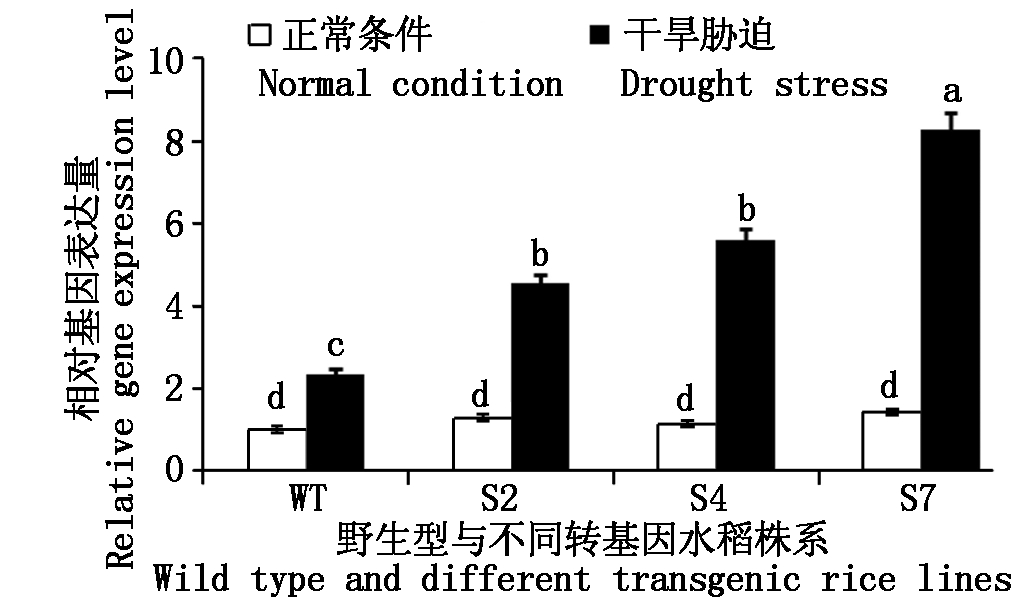
不同字母表示差异显著(P<0.05)。图5-6同。
Different letters indicate significant difference (P <0.05),according to ANOVA-protected LSD 0.05 test. The same as Fig.5-6.
图4 干旱条件下野生型水稻与转基因水稻OsAMTR310基因的实时定量分析
Fig.4 Real-time PCR assay of OsAMTR310 gene of wild type and transgenic rice under normal and drought stress
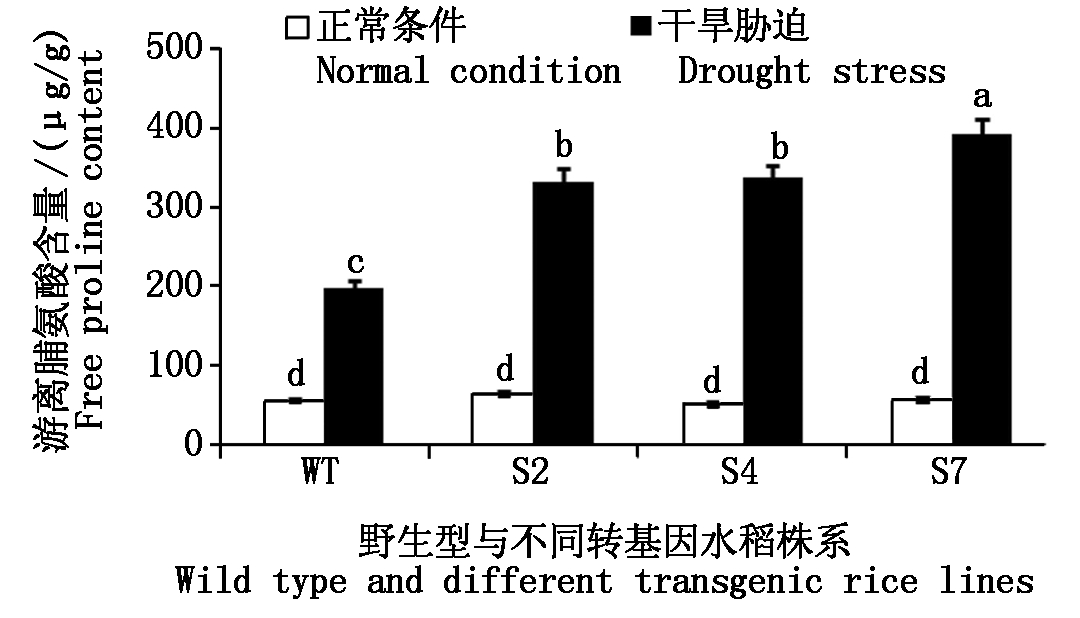
图5 野生型水稻与转基因水稻叶片脯氨酸含量测定
Fig.5 Determination of proline content of wild type and transgenic rice
2.5 转基因植株的表型分析
对3个转基因株系在正常生长条件下与干旱胁迫条件下的表型与野生型水稻做了比较,并进行了初步的耐旱性试验。野生型和OsAMTR310转基因水稻均在28 ℃下生长21 d,并经受干旱处理21 d,然后将它们转移到正常的营养液中,观察其生长,结果显示,经过干旱处理后,大部分的野生型水稻均死亡,存活率仅为38%;相比之下,大部分的OsAMTR310转基因水稻植株呈现健康的形态,并在营养液里恢复生长,图6显示,3个OsAMTR310转基因株系的存活率分别为60.9%,66.8%,72.5%,是野生型水稻的1.5~2.0倍。
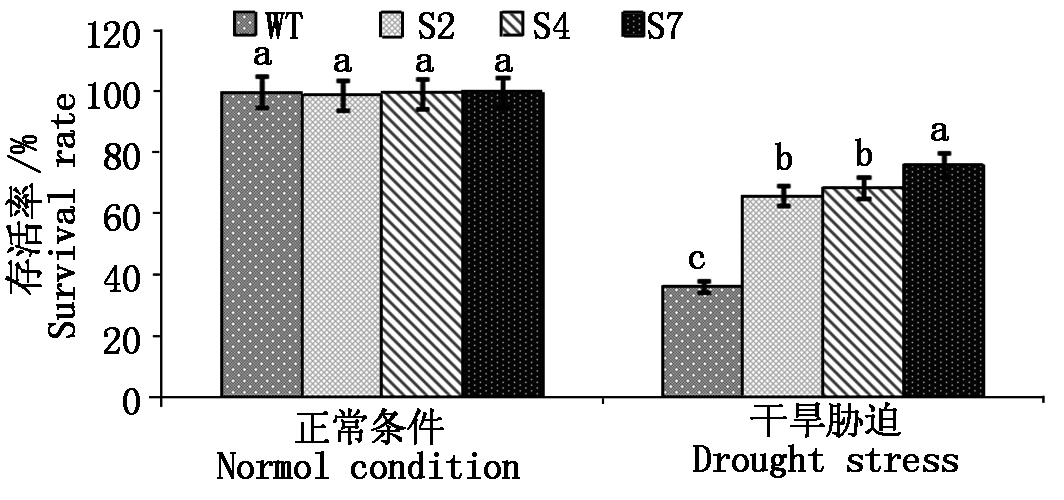
图6 干旱条件下野生型水稻与OsAMTR310转基因水稻的存活率测定
Fig.6 Survival rate of wild type and OsAMTR310 transgenic rice under normal and drought stress
3 结论与讨论
OsAMTR310基因编码的蛋白质隶属于乙酰鸟氨酸氨基转移酶家族,该家族是依赖于磷酸吡哆醛(PLP)的天冬氨酸氨基转移酶总科,其主要包括鸟氨酸转氨酶、乙酰鸟氨酸转氨酶、丙氨酸-乙醛酸氨基转移酶、二烷基甘氨酸脱羧酶、4-氨基丁酸转氨酶、β-丙氨酸-丙酮酸氨基转移酶、腺苷甲硫氨酸-8-氨基-7-氧代壬酸酯氨基转移酶和谷氨酸-1-半醛2,1-氨基变位酶。这个总科的所有酶均作用于碱性氨基酸,其衍生物涉及氨基转移或脱羧。而OsAMTR310蛋白作用之一是作为一种腺苷甲硫氨酸-8-氨基-7-氧代壬酸酯转氨酶(其他名称包括7,8-二氨基脂肪酸氨基转移酶,DAPA氨基转移酶)[16]。前人已经通过试验证明,这种酶是生物素生物合成途径中的辅助因子和辅基,而转氨酶与生物素常与植物抵抗非生物胁迫有关[17],暗示了OsAMTR310基因编码的产物可能参与了非生物胁迫抵抗。
各种各样的分子生物学手段比如转基因用来鉴定由各种非生物胁迫诱导的基因,在胁迫条件下,被诱导的基因可以通过产生应激蛋白来保护细胞免受胁迫危害[18]。前人研究表明,转氨酶在水稻中发挥了多重的关键作用,尤其是提高非生物胁迫耐性。比如,水稻谷氨酸转氨酶基因OsOAT作为胁迫应答转录因子SNAC2的靶标基因,其在水稻中的表达随着干旱和氧化胁迫而增加,表明OsOAT基因在提高植物胁迫忍耐性中起了新的作用[19];转氨酶基因OsAMTR1对非生物胁迫有应答,可以提高转基因拟南芥的非生物胁迫抗性,而且可以提高转基因植株的发芽率,根的发育和植株的鲜质量[20-21]。研究中心在前期工作中已经通过生物信息学分析与实时定量PCR验证了OsAMTR310基因对干旱胁迫有响应,在本研究,通过RT-PCR法将OsAMTR310的编码序列从水稻总cDNA中克隆出来,并且构建了pCAMBIA1302-OsAMTR310超表达载体,并通过农杆菌介导法将其转入到水稻中,进一步验证了OsAMTR310基因的功能。首先通过PCR与抗性筛选了阳性转基因水稻苗,并且根据蛋白质免疫印迹法确定了OsAMTR310在转基因水稻中正常表达。在表型鉴定试验中,在正常条件下,OsAMTR310转基因植株与野生型植株的生长状态没有明显的差别;而在干旱胁迫下,OsAMTR310转基因植株明显比野生型植株具有更强的耐旱能力。植物对环境胁迫的适应通常与代谢调节有关,例如脯氨酸的积累。脯氨酸在植物调节自身渗透压,抵抗环境胁迫的内稳态调节中起中心作用,所以,植物的脯氨酸水平是衡量其非生物胁迫耐性的重要生理指标[22]。在干旱胁迫下,OsAMTR310转基因植株叶片中的脯氨酸含量显著高于野生型植株,说明OsAMTR310转基因植株具有更高的耐旱性,并且暗示了OsAMTR310参与了水稻脯氨酸信号转导与生物合成途径。
综上所述,通过对水稻乙酰鸟氨酸氨基转移酶家族基因OsAMTR310的克隆和对转基因植株的功能分析,结果发现,OsAMTR310提高了水稻干旱胁迫耐性,并可提高转基因植株在干旱条件下的游离脯氨酸含量,表明OsAMTR310在水稻抗旱胁迫中发挥重要功能。
参考文献:
[1] Yin X,Cui Y,Wang M,et al. Overexpression of a novel MYB-related transcription factor,OsMYBR1,confers improved drought tolerance and decreased ABA sensitivity in rice [J]. Biochem Biophys Res Commun,2017,490(4):1355-1361.
[2] Li J J,Li Y,Yin Z G,et al. OsASR5 enhances drought tolerance through a stomatal closure pathway associated with ABA and H2O2 signalling in rice [J]. Plant Biotechnology Journal,2017,15(2):183-196.
[3] Hwang B Y,Cho B K,Yun H,et al. Revisit of aminotransferase in the genomic era and its application to biocatalysis [J]. Journal Of Molecular Catalysis B-Enzymatic,2005,37(1):47-55.
[4] Liepman A H,Olsen L I. Genomic analysis of aminotransferases in Arabidopsis thaliana[J]. Critical Reviews in Plant Sciences,2004,23(1):73-89.
[5] Givan C V. Aminotransferases in higher plants [J]. The biochemistry of plants ,1980,5:329-357.
[6] Reumann S. The structural properties of plant peroxisomes and their metabolic significance[J]. Biological Chemistry,2000,381(8):639-648.
[7] Yang Q Q,Zhang C Q,Chan M L,et al. Biofortification of rice with the essential amino acid lysine:molecular characterization,nutritional evaluation,and field performance[J]. Journal of Experimental Botany,2016,67(14):4285-4296.
[8] Sun J,Xie D W,Zhao H W,et al. Genome-wide identification of the class Ⅲ aminotransferase gene family in rice and expression analysis under abiotic stress[J]. Genes & Genomics,2013,35(5):597-608.
[9] Wu W X,Zheng X M,Chen D B,et al. OsCOL16,encoding a CONSTANS-like protein,represses flowering by up regulating Ghd7 expression in rice[J]. Plant Science,2017,260:60-69.
[10] Huang X Y,Deng F L,Yamaji N,et al. A heavy metal P-type ATPase OsHMA4 prevents copper accumulation in rice grain[J]. Nature Communications,2016,7:12138.
[11] Hiei Y,Ohta S,Komari T,et al. Efficient transformation of rice (Oryza sativa L.) mediated by Agrobacterium and sequence analysis of the boundaries of the T-DNA[J]. The Plant Journal :for Cell and Molecular Biology,1994,6(2):271-282.
[12] Tuteja N,Sahoo R K,Garg B,et al. OsSUV3 dual helicase functions in salinity stress tolerance by maintaining photosynthesis and antioxidant machinery in rice (Oryza sativa L. cv. IR64) [J]. Plant Journal,2013,76(1):115-127.
[13] Siahpoosh M R,Dehghanian E,Kamgar A. Drought tolerance evaluation of bread wheat genotypes using water use efficiency,evapotranspiration efficiency,and drought susceptibility index[J]. Crop Science,2011,51(3):1198-1204.
[14] Livak K J,Schmittgen T D. Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25(4):402-408.
[15] Wang F,Tong W,Zhu H,et al. A novel Cys2/His2 zinc finger protein gene from sweetpotato,IbZFP1,is involved in salt and drought tolerance in transgenic Arabidopsis [J]. Planta,2016,243(3):783-797.
[16] Menta P K,Hale T I,Chrisien P.Evolutionary relationships among aminotransferases [J]. The FEBS Journal,1989,186(1-2):249-253.
[17] Roosens N H C J,Thu T T,Iskandar H M,et al. Isolation of the ornithine-δ-aminotransferase cDNA and effect of salt stress on its expression in Arabidopsis thaliana [J]. Plant physiology,1998,117(1):263-271.
[18] Nakashima K,Yamaguchi-Shinozaki K,Shinozaki K. The transcriptional regulatory network in the drought response and its crosstalk in abiotic stress responses including drought,cold,and heat[J]. Frontiers in Plant Science,2014,5:170.
[19] You J,Hu H,Xiong L. An ornithine delta-aminotransferase gene OsOAT confers drought and oxidative stress tolerance in rice [J]. Plant Sci,2012,197 :59-69.
[20] Dansana P K,Kothari K S,Vij S,et al. OsiSAP1 overexpression improves water-deficit stress tolerance in transgenic rice by affecting expression of endogenous stress-related genes[J]. Plant Cell Reports,2014,33(9):1425-1440.
[21] Kothari K S,Dansana P K,Giri J A. Rice stress associated protein 1 (OsSAP1) interacts with aminotransferase (OsAMTR1) and Pathogenesis-Related 1a protein (OsSCP) and regulates abiotic stress responses[J]. Frontiers in Plant Science,2016,7:1057.
[22] Zhang X,Zhang B,Li M J,et al. OsMSR15 encoding a rice C2H2-type zinc finger protein confers enhanced drought tolerance in transgenic Arabidopsis[J]. Journal of Plant Biology,2016,59(3):271-281.