MADS-box转录因子广泛存在于动物、植物和真菌中[1-3],在植物生长发育的多个方面起重要作用,是研究最广泛的转录因子家族之一[4]。MADS-box转录因子在N端具有一个高度保守的MADS结构域(MADS-box domain),约由58~60个氨基酸组成,负责识别和绑定靶基因中调控区域的CArG盒[CC(A/T)6GG][5]。根据序列结构,MADS-box转录因子可以分成type Ⅰ和type Ⅱ两大类。其中,type Ⅰ编码一个SRF-like类型的MADS域,结构简单,仅含有1~2个外显子,可进一步划分为Mα、Mβ、Mγ 3个组,其功能仍不清楚[1,6-9]。type Ⅱ编码一个MEF2-like和MIKC-type类型的MADS域,具有半保守的K盒(Keratin-like domain)和保守型较差的Ⅰ盒(Intervening domain)和C盒(C-terminal region),可进一步划分为MIKCC和MIKC* 2个组,与MIKCC组相比,MIKC*组有更不保守的K盒和更长的I盒,在拟南芥和水稻中Mδ组相当于MIKC*组[6-7,10]。MIKCC组基因可进一步划分为12个亚组,即AG、AGL6、AGL12、AP3/PI、GGM13、TM3、STMADS11、AGL2、AGL17、AP1/SQUA、AGL15和FLC[2]。随着研究的不断深入,MIKCC被重新分成13个亚组,即AG(C类基因)、AGL6、AGL12、AP3-PI(B类基因)、Bs、SOC1、SVP、SEP(E类基因)、AGL17、AP1-FUL(A类基因)、AGL15、FLC和TM8[9,11-16]。大量研究发现,MADS-box基因参与许多重要的生理和发育过程,如调节花器官和果实发育以及控制开花时间和配子体细胞分裂[4,17-19]。调控光合作用和营养代谢以及参与激素信号转导等途径,与逆境胁迫相关[20-22]。
研究拟南芥和金鱼草等模式植物发现,MADS-box转录因子参与生殖分生组织属性决定和花器官发育,提出经典的ABCDE花发育模型。A、E类基因调控萼片发育,A、B、E类基因共同调控花瓣发育,B、C、E类基因共同调控雄蕊发育,C、E类基因调控心皮发育,D、E类基因调控胚珠发育[16,23]。ABCDE花发育模型相关基因,除了AP2(A类基因)之外,其他都属于MADS-box家族基因[24]。
目前,很多甜菜品种如红叶甜菜、红甜菜等,越来越多用于绿化与装饰,但观赏甜菜在栽培绿化过程中容易过早抽薹,同时易受到各种非生物胁迫的影响,严重影响了其观赏寿命和观赏效果。MADS-box转录因子参与花器官鉴定、生长发育及影响种子发育,这对推迟观赏甜菜开花进而延长观赏时限、改变育性及提升整体观赏价值、提高观赏甜菜逆境生理等多个方面均有重要作用。此外,石竹目观赏花卉众多,如香石竹、石竹和须苞石竹等,但是却缺乏高质量的基因和转录组数据,系统地研究MADS-box基因比较困难,改变、提升这些观赏花卉的观赏效果与抗性,只能借助于甜菜已发表的高质量的基因组和转录组数据。此外,石竹目属于基部双子叶植物类群,对其MADS-box家族的系统研究,也可进一步阐明MADS-box家族基因的进化。本研究通过生物信息学分析手段,基于已公开的基因组及RNA-seq数据[25-27],对甜菜MADS-box转录因子进行了全基因组鉴定,以期为进一步解析MADS-box转录因子功能,以及通过基因工程手段改良观赏甜菜观赏性状和提高逆境抗性奠定基础。同时为石竹目观赏花卉MADS-box基因的鉴定、进化及基因功能研究提供参考。
1 材料和方法
1.1 试验材料
拟南芥MADS-box基因家族的氨基酸序列来自TAIR(http://www.arabidopsis.org/);甜菜KWS2320参考基因组氨基酸序列、CDS序列和基因组序列均来源于AnnoBeet(http://genomforschung.uni-bielefeld.de/en/projects/annobeet);MADS-box家族基因Pfam种子文件:SRF-TF结构域信息(PF00319)来源于Pfam数据库(http://pfam.xfam.org/);甜菜RNA-seq数据来自SRA(SRX287608-287615;SRX647324;SRX647712;SRX647714) (https://www.ncbi.nlm.nih.gov/sra/)。
其中,SRX287608-287615包括叶片、直根、花序、幼苗和种子RNA-seq数据,SRX647712为热胁迫幼叶,SRX647714为盐胁迫幼叶,SRX647324为对照幼叶[26]。RNA-seq测序用KWS2320植株生长于沙滩,生长温度21 ℃,光照时间10 h/14 h,光照强度80~120 μmol/(m2·s),相对湿度60%。测序样品为花序、直根、叶片、种子,幼苗(20 ℃培养2 d的出芽苗)[25,27]。盐胁迫处理为:植株全营养液水培,23 d苗龄时转入含50 mmol/L NaCl的全营养液,每天增加50 mmol/L NaCl浓度,逐渐增加NaCl浓度到300 mmol/L,此后保持300 mmol/L NaCl全营养液胁迫2 d,于第30天采取幼叶样品(顶端第3,4片幼叶不含叶中脉)。热胁迫为:植株全营养液水培30 d,幼叶样品采摘前进行35 ℃高温胁迫3 h。对照幼叶取材于全营养水培30 d的植株。所有样品使用Nucleospin RNA Plant kit试剂盒(Macherey-Nagel,德国)提取RNA,利用Hiseq 1500测序仪进行双末端测序[25,27]。
1.2 试验方法
1.2.1 甜菜MADS-box家族基因的鉴定 使用hmm和Blast同源性搜索2种方法来鉴定MADS-box转录因子,以PF00319为种子文件,借助HMMER 3.0软件在甜菜氨基酸序列数据库中进行同源对比搜索,参数设置为默认参数[5,10]。以拟南芥MADS-box氨基酸序列为种子序列,借助在线基因组BlastP在线工具再次搜索。去除重复序列后,利用Pfam和SMART(http://smart.embl-heidelberg.de/)对候选基因进一步确认,最终获得甜菜MADS-box家族基因。
1.2.2 甜菜MADS-box家族基因的生物信息分析 甜菜MADS-box家族基因内含子、外显子和基因组定位信息均来自于甜菜基因组数据库,利用在线软件GSDS2.0(http://gsds.cbi.pku.edu.cn/index.php)绘制基因结构图,运用MapInspect工具绘制染色体定位图;利用在线软件MEME(http://meme-suite.org/tools/meme)对甜菜MADS-box蛋白进行基序预测,参数中预测数目设置为10,其余参数均为默认设置;利用MEGA 5.0软件对甜菜MADS-box家族蛋白序列与拟南芥MADS-box蛋白序列进行比对,用邻位(Neighbor-Joining)算法构建系统发生树,校验参数Bootstrap重复1 000次;利用Plant-mPLoc server在线软件(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)进行各MADS-box基因的亚细胞定位分析;利用ProtParam在线程序(http://web.expasy.org/protparam/)预测氨基酸序列分子量及等电点[4,28]。
1.2.3 甜菜MADS-box家族基因表达分析 利用RNA-seq数据分析甜菜MADS-box基因在不同组织(幼苗、直根、幼叶、叶、花序、种子)中的表达情况以及各MADS-box基因在盐胁迫、热胁迫下幼叶中的表达变化。具体方法:用HISAT2将reads比对到参考基因组上,用StringTie计算基因表达量,根据各基因表达量在各组织中的结果利用R语言作热图。计算热、盐胁迫下各基因相对表达量,相对表达量=log2(胁迫组表达量/对照组表达量),计算结果作热图[27]。
1.2.4 甜菜MADS-box蛋白功能联系网络分析 利用String蛋白互作数据库(http://string-db.org/)中特异性比较高的蛋白模式对34个MADS蛋白的互作网络进行分析。此外,将BvMADS19、BvMADS32蛋白序列提交至STRING网站,预测BvMADS19、BvMADS32蛋白共表达网络。所有操作物种参数均选择模式植物拟南芥[29-30]。
2 结果与分析
2.1 甜菜MADS-box家族基因的鉴定与系统发育分析
共筛选得到34个甜菜MADS-box基因,MADS-box转录因子序列为66~1 061个氨基酸长度,分子量为7.63~119.43 ku,等电点为5.44~11.00,亚细胞定位显示所有MADS-box基因均定位于细胞核上(表1),表明虽然MADS-box转录因子理化性质差异较大,但亚细胞定位十分保守。聚类分析发现,MADS-box转录因子可分为type Ⅰ和type Ⅱ两大类,其中,type Ⅰ成员7个和type Ⅱ成员27个。根据进化关系,type Ⅰ进一步分为Mα(3)、Mβ(1)、Mγ(3)3个组(图1);type Ⅱ进一步分为MIKCC(22)和MIKC*(5)2个组,MIKCC组可进一步分为AG(2)、AGL12(2)、AP3-PI(4)、Bs(2)、SOC1(1)、SVP(1)、SEP(3)、AGL17(5)、AP1-FUL(1)和FLC(1) 10个亚组,无AGL6、AGL15、TM8亚组成员被发现(图2)。
表1 甜菜MADS-box家族基因信息
Tab.1 The information of MADS-box genes in Beta vulgaris

基因基因号染色体定位氨基酸长度分子量/ku等电点亚细胞定位亚家族GeneGeneIDChr.locationAAlengthMWPISubcellularlocationSubfamilyBvMADS1Bv6g151560_ksif.t1Chr624027.648.91细胞核MIKCcBvMADS2Bv6g151560_ksif.t2Chr623927.518.70细胞核MIKCcBvMADS3Bv5g114640_zztx.t1Chr525229.118.61细胞核MIKCcBvMADS4Bv1ug019780_iszi.t1Chr126329.827.23细胞核MIKCcBvMADS5Bv1g012680_hnhs.t1Chr1728.389.86细胞核MIKCcBvMADS6Bv8g189780_esnp.t1Chr8859.869.93细胞核MIKCcBvMADS7Bv2g028730_mpsx.t1Chr225128.288.56细胞核MIKCcBvMADS8Bv6g151580_wfgt.t1Chr6667.6710.92细胞核MIKCcBvMADS9Bv9g224080_wdgq.t1Chr921725.238.43细胞核MIKCcBvMADS10Bv9g224080_wdgq.t2Chr921524.978.76细胞核MIKCcBvMADS11Bv3g062200_hycw.t1Chr325528.766.14细胞核MIKCcBvMADS12Bv1g005260_mnyn.t1Chr1697.6311.00细胞核MIKCcBvMADS13Bv9g224090_heoc.t1Chr923827.419.87细胞核MIKCcBvMADS14Bv6g134730_nuri.t1Chr622725.665.44细胞核MIKCcBvMADS15Bv9g223640_mryh.t1Chr921124.149.50细胞核MIKCcBvMADS16Bv9g223640_mryh.t2Chr921925.099.48细胞核MIKCcBvMADS17Bv6g130780_qrxi.t1Chr623527.199.43细胞核MIKCcBvMADS18Bv1g006360_gdiw.t1Chr122526.109.21细胞核MIKCc
表1(续)
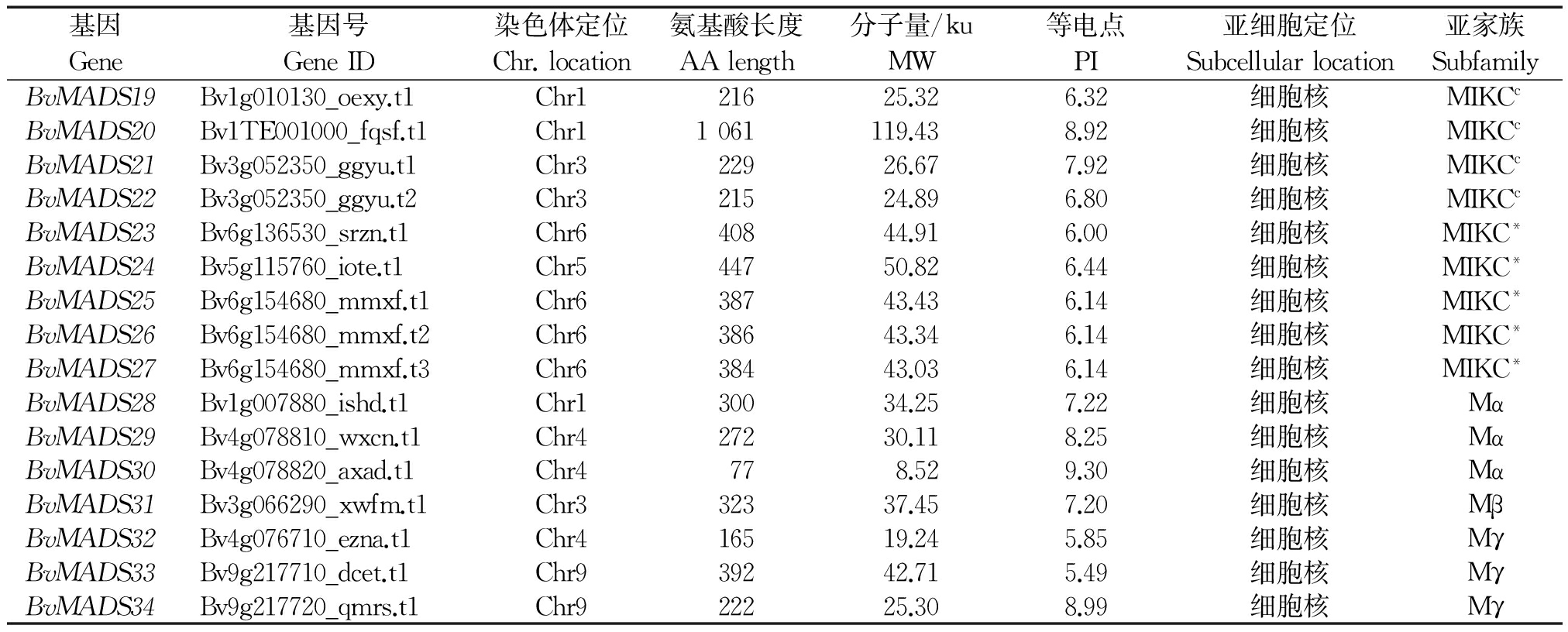
基因基因号染色体定位氨基酸长度分子量/ku等电点亚细胞定位亚家族GeneGeneIDChr.locationAAlengthMWPISubcellularlocationSubfamilyBvMADS19Bv1g010130_oexy.t1Chr121625.326.32细胞核MIKCcBvMADS20Bv1TE001000_fqsf.t1Chr11061119.438.92细胞核MIKCcBvMADS21Bv3g052350_ggyu.t1Chr322926.677.92细胞核MIKCcBvMADS22Bv3g052350_ggyu.t2Chr321524.896.80细胞核MIKCcBvMADS23Bv6g136530_srzn.t1Chr640844.916.00细胞核MIKC*BvMADS24Bv5g115760_iote.t1Chr544750.826.44细胞核MIKC*BvMADS25Bv6g154680_mmxf.t1Chr638743.436.14细胞核MIKC*BvMADS26Bv6g154680_mmxf.t2Chr638643.346.14细胞核MIKC*BvMADS27Bv6g154680_mmxf.t3Chr638443.036.14细胞核MIKC*BvMADS28Bv1g007880_ishd.t1Chr130034.257.22细胞核MαBvMADS29Bv4g078810_wxcn.t1Chr427230.118.25细胞核MαBvMADS30Bv4g078820_axad.t1Chr4778.529.30细胞核MαBvMADS31Bv3g066290_xwfm.t1Chr332337.457.20细胞核MβBvMADS32Bv4g076710_ezna.t1Chr416519.245.85细胞核MγBvMADS33Bv9g217710_dcet.t1Chr939242.715.49细胞核MγBvMADS34Bv9g217720_qmrs.t1Chr922225.308.99细胞核Mγ
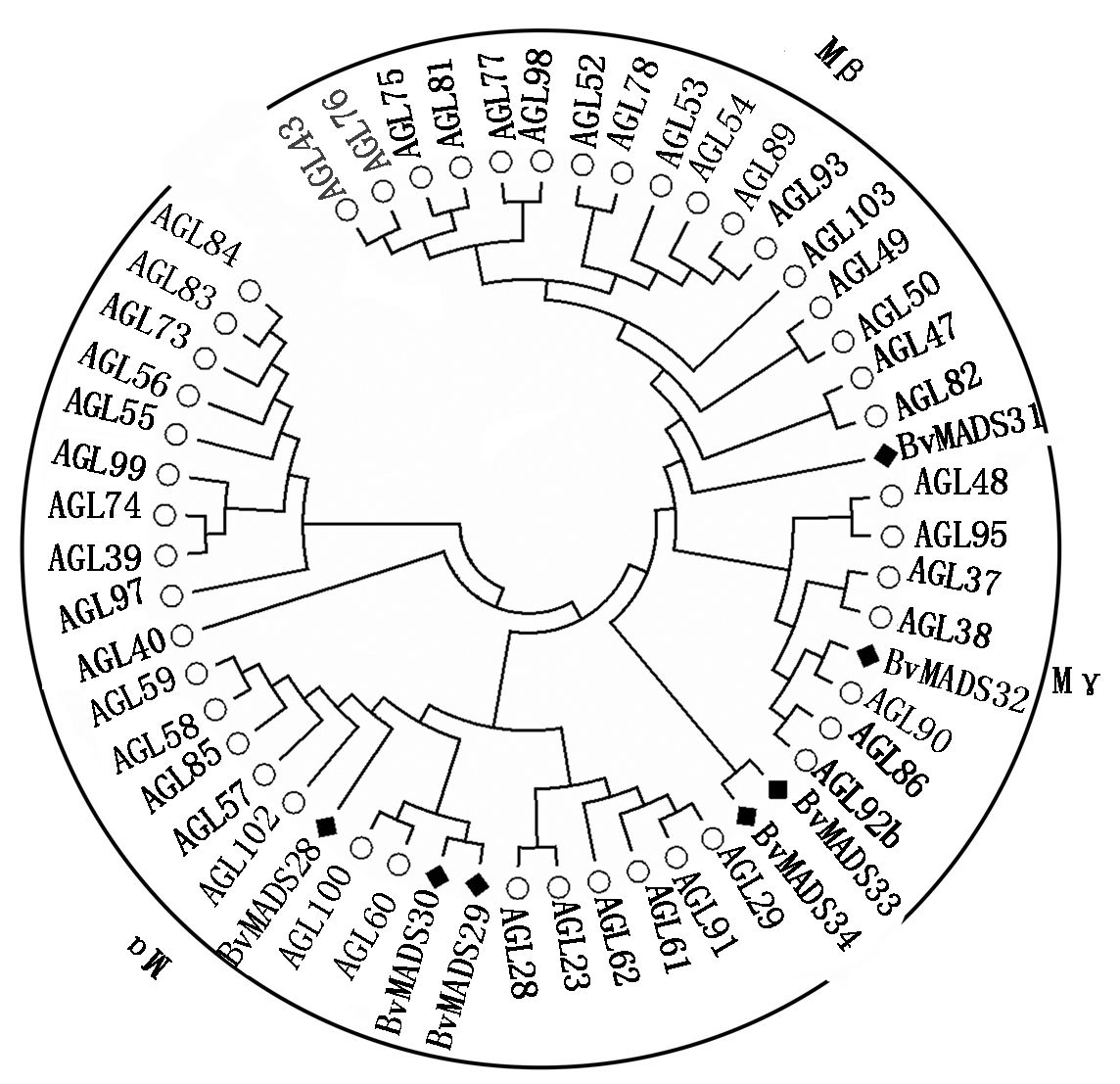
图1 甜菜和拟南芥MADS-box蛋白系统发生树(typeⅠ)
Fig.1 Phylogenetic tree of beet and Arabidopsis MADS-box proteins(typeⅠ)
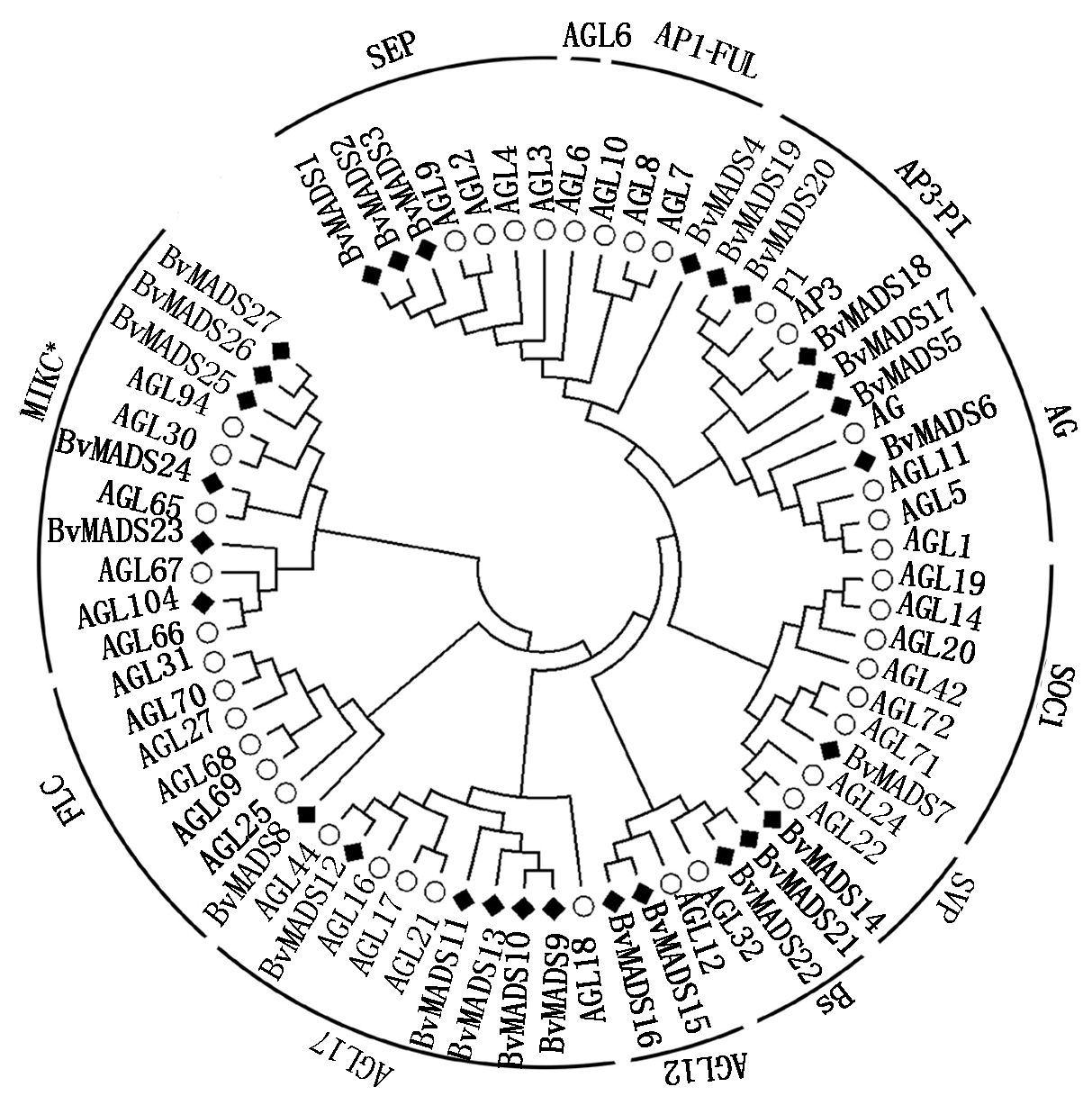
图2 甜菜和拟南芥MADS-box蛋白系统发生树(type Ⅱ)
Fig.2 Phylogenetic tree of beet and Arabidopsis MADS-box proteins(type Ⅱ)
2.2 甜菜MADS-box基因内含子和外显子结构分析
基因结构分析结果显示(图3),甜菜MADS-box type Ⅱ基因外显子数目具有高变异性(1~13个外显子),其中,AP3-PI亚组的BvMADS20具有13个外显子,数量最多,区别于该亚组其他成员(外显子为7个);AG亚组和FLC亚组含1~2外显子,数量较少;AGL17亚组的BvMADS12只具有1个外显子,区别于亚组内其他成员(含有7~8个外显子)。类似地,MIKC*组内成员外显子含量普遍较高(不少于11个)。表明即使同一组、甚至同一亚组类,基因结构差别也很大。相对于typeⅡ基因,MADS-box typeⅠ基因结构简单,只含有1个外显子。
2.3 甜菜MADS-box基因染色体定位
染色体定位分析结果显示(图4),MADS-box基因在各染色体上呈不均匀分布,在同一条染色体上的基因呈密集簇状分布。其中,第6号染色体上分布最多,为9条;第1,9号染色体上分布次之,为7条;第3号染色体上分布4条;第4,5染色体上分别分布3,2条;第2,8染色体上各分布1条,第7号染色体无MADS-box基因分布。
2.4 甜菜MADS-box蛋白序列保守元件
分析保守元件预测结果(图5),发现Motif1为MADS盒,Motif2和Motif9为K盒,其他为未知盒。Motif1存在于所有的MADS-box蛋白序列类,为MADS-box蛋白特征motif。Motif2存在于大部分MIKCC类型的蛋白序列内,但是不存在MIKC*类型的蛋白序列内。MIKC*类型的蛋白序列除含有Motif1外,还具有其他组不具有的Motif3、4、5、7、8、9、10。Motif6仅出现在SEP亚组中,为该亚组所特有。
2.5 甜菜MADS-box基因的表达分析
RNA-seq数据分析结果显示(图6),大部分MADS-box基因优势表达于花序分生组织,且在花序分生组织中typeⅡ基因表达量高于typeⅠ,MIKCC组基因表达量普遍高于MIKC*组。进一步分析发现,表达量最显著的是typeⅡ基因成员中A、B、C、E类花发育相关基因,如SEP亚组(E类基因)的BvMADS1、BvMADS2、BvMADS3;AP1-FUL亚组(A类基因)的BvMADS4;AG亚组(C类基因)的BvMADS6,AP3-PI亚组(B类基因)的BvMADS17、BvMADS18、BvMADS19。而C类基因BvMADS5和B类基因BvMADS20表达并不是很高。除此之外,AGL12亚组基因亦有较高表达量。BvMADS3基因区别于其他2个E类基因(BvMADS1、BvMADS2),在叶、幼叶与种子中也有较高表达,表明其可能参与种子发育和叶片生理活动。BvMADS8在幼嫩组织(幼苗、幼叶)和种子中表达高于其他组织,表明可能其在分生组织、细胞增殖分化方面扮演重要角色,同时与种子成熟发育有关。BvMADS14与BvMADS8表达模式类似,但其在根中亦有较高表达,表明其可能在根的生理活动中起一定作用。BvMADS18优势表达于花序和种子,暗示其可能参与从花发育到果实成熟的全过程。MIKC*组的BvMADS23特异表达于直根,可能与直根发育有关。type Ⅰ基因普遍表达量极低,只有极个别基因在某些组织中有较高表达。如BvMADS29优势表达于幼苗和种子,该基因可能与苗期生长与种子发育密切相关。
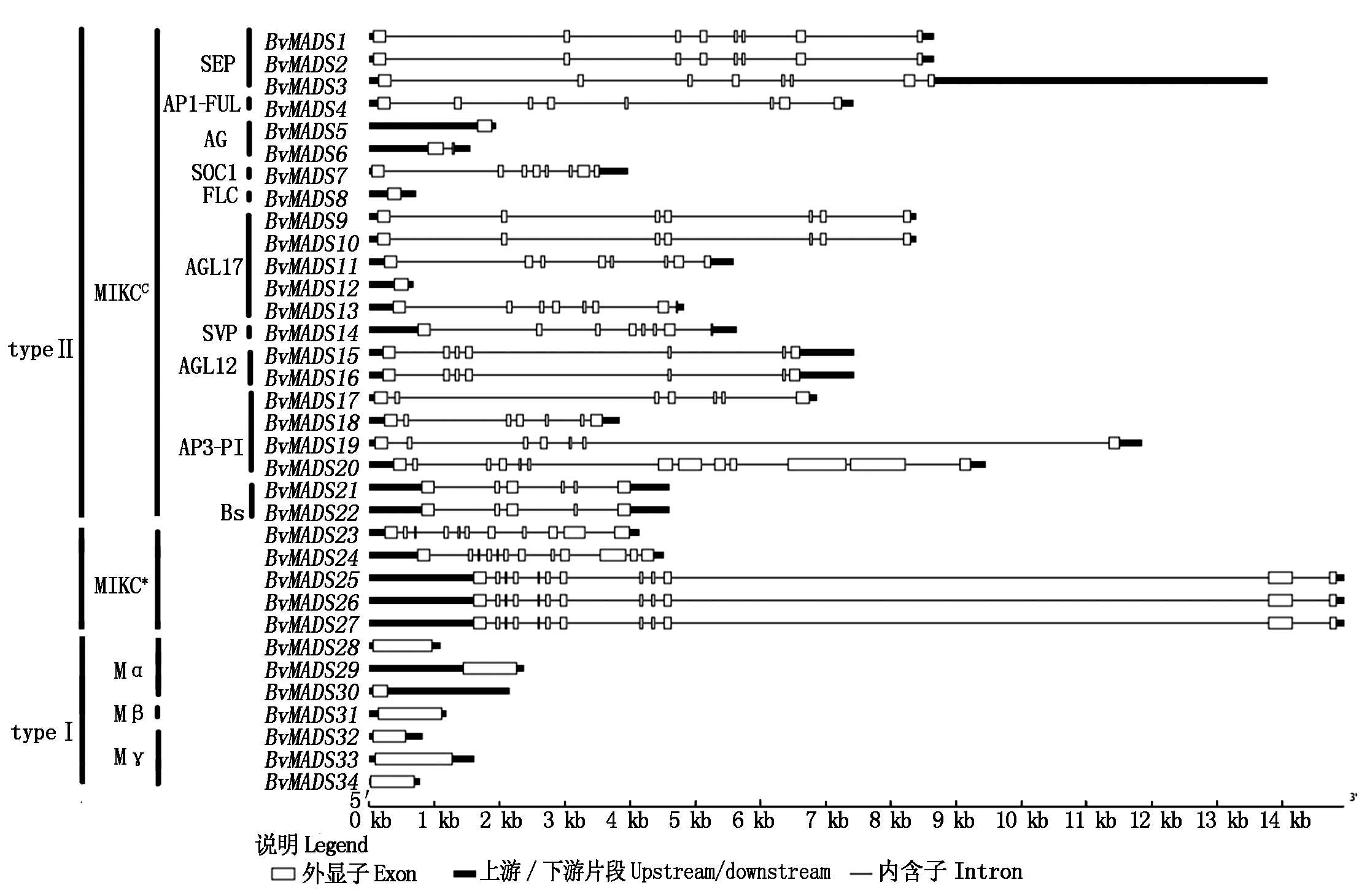
图3 甜菜MADS-box内含子和外显子结构
Fig.3 Intron and exon structure of beet MADS-box genes
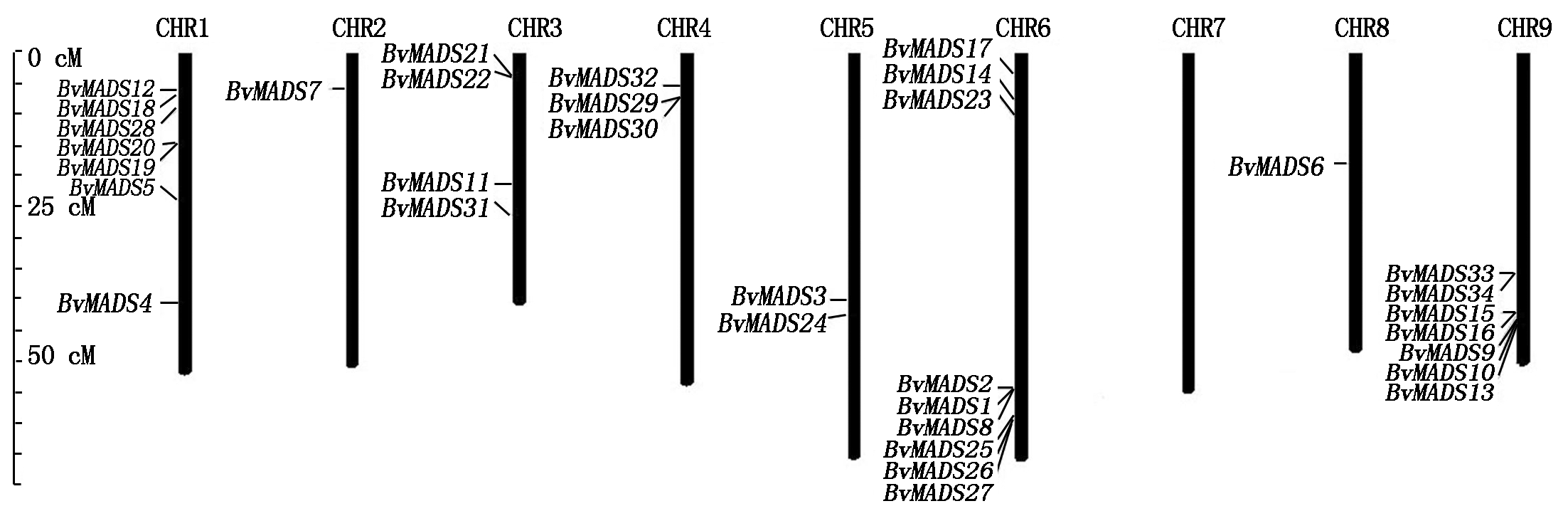
图4 甜菜MADS-box基因染色体定位
Fig.4 Chromosome location of beet MADS-box genes
分析胁迫幼叶RNA-seq发现(图6),响应盐胁迫、热胁迫的MADS-box基因极少,只有BvMADS8和BvMADS12响应盐胁迫而轻微上调,BvMADS8、BvMADS19、BvMADS32响应热胁迫而轻微上调。表明MADS-box基因也起作用于热、盐胁迫调控路径。AP3-PI亚组(BvMADS19)不仅对花发育有调控作用,也在逆境生理调控过程中起作用,Mγ组个别基因(BvMADS32)与甜菜抵御外界热胁迫密切相关。

图5 甜菜MADS-box蛋白保守元件预测
Fig.5 Conserved element prediction of beet MADS-box proteins
2.6 MADS-box蛋白功能联系预测
利用String蛋白互作数据库的蛋白(Protein)模式对甜菜MADS-box蛋白之间的功能关系进行了分析预测(图7)。从图7可以看出,typeⅡ基因形成一个联系密切的关系网,B类基因(AP3、PI)、C类基因(AG、SHP1)、E类基因(SEP2、SEP3)、AGL17亚组的(AGL16、AGL21、AGL44)以及SVP亚组的SVP形成彼此密切联系的互作网络,说明以A、B、C、D、E类基因为基础的花发育模型基因,与MADS-box基因家族内的其他基因共同作用调控植物花发育。推测甜菜中对应的MADS-box蛋白也存在类似的调控网路。此外,MIKC*组AGL104与AGL65也彼此互作,type Ⅰ基因AGL62、AGL80与AGL30存在线性互作关系。
为了探究BvMADS19与BvMADS32响应热胁迫的根源,验证RNA-seq分析结果,对这2个蛋白进行了单独的String蛋白共表达网络分析(图7)。BvMADS19共表达网络显示,BvMADS19蛋白与MADS-box蛋白AP1、AP3、AG、SEP3和SEP1存在较强的蛋白互作,与TRP-like蛋白也存在强互作关系,与MADS-box蛋白AP2存在弱强度互作。此外,与CO、LFY、UFO转录因子存在一定互作。表明MADS-box基因在控制花发育的过程中存在协同作用。BvMADS19属于AP3-PI亚组(B类),与A类(AP1、AP2)、C类(AG)以及E类(SEP1、SEP3)均发生强互作,符合ABCDE花发育模型理论。TRP-like蛋白把这些蛋白联合在一起,有强互作关系。研究发现,此基序通过形成特殊的空间结构,介导蛋白质的相互作用,并在一些重要的蛋白复合物形成中非常重要[31] ,推测TRP-like蛋白在MADS-box互作模型中起重要粘连作用。CO是光周期途径中的关键基因,位于生物钟的输出途径,参与长日照植物开花途径,受生物钟调控。此外,CO还调节P5CS2和ACS10基因,这2个基因参与脯氨酸和乙烯生物合成[32] ,推测BvMADS19通过与CO互作,也参与植物光周期调控路径,此外,其对热胁迫的响应可能是由于与CO蛋白互作,影响了脯氨酸和乙烯的合成。LFY是调控开花及花发育的关键因子,起着联系环境信号和激活下游基因的枢纽作用,激活B类基因AP3和PI表达,控制花瓣和雄蕊的形成[33-34]。UFO作为辅助因子,和LFY基因一起激活B类基因的表达[35]。BvMADS19亦属于PI基因,推测在甜菜中同样受LFY、UFO的调控,控制花瓣和雄蕊形成。
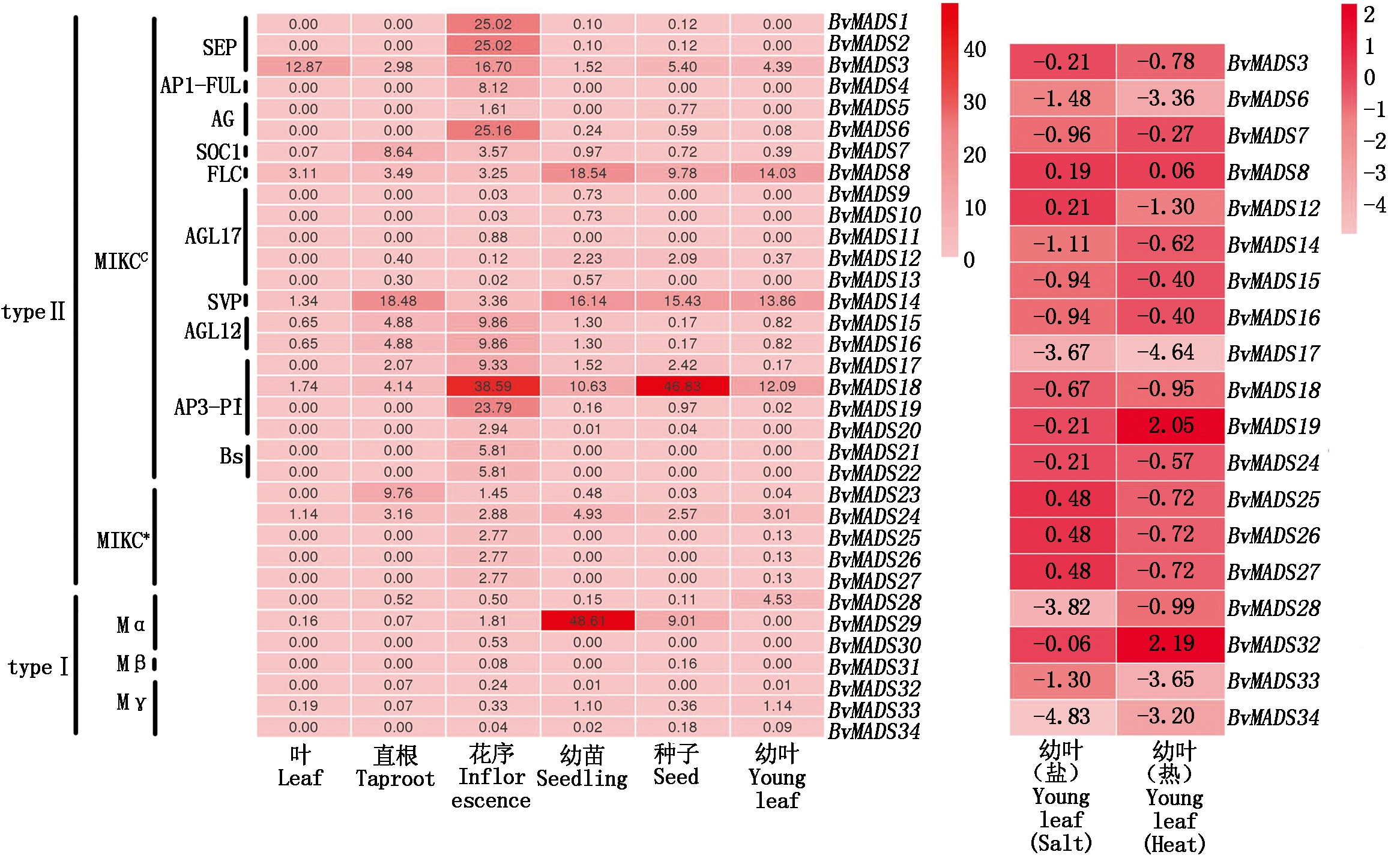
图6 甜菜MADS-box基因表达谱
Fig.6 Expression profile of MADS-box genes
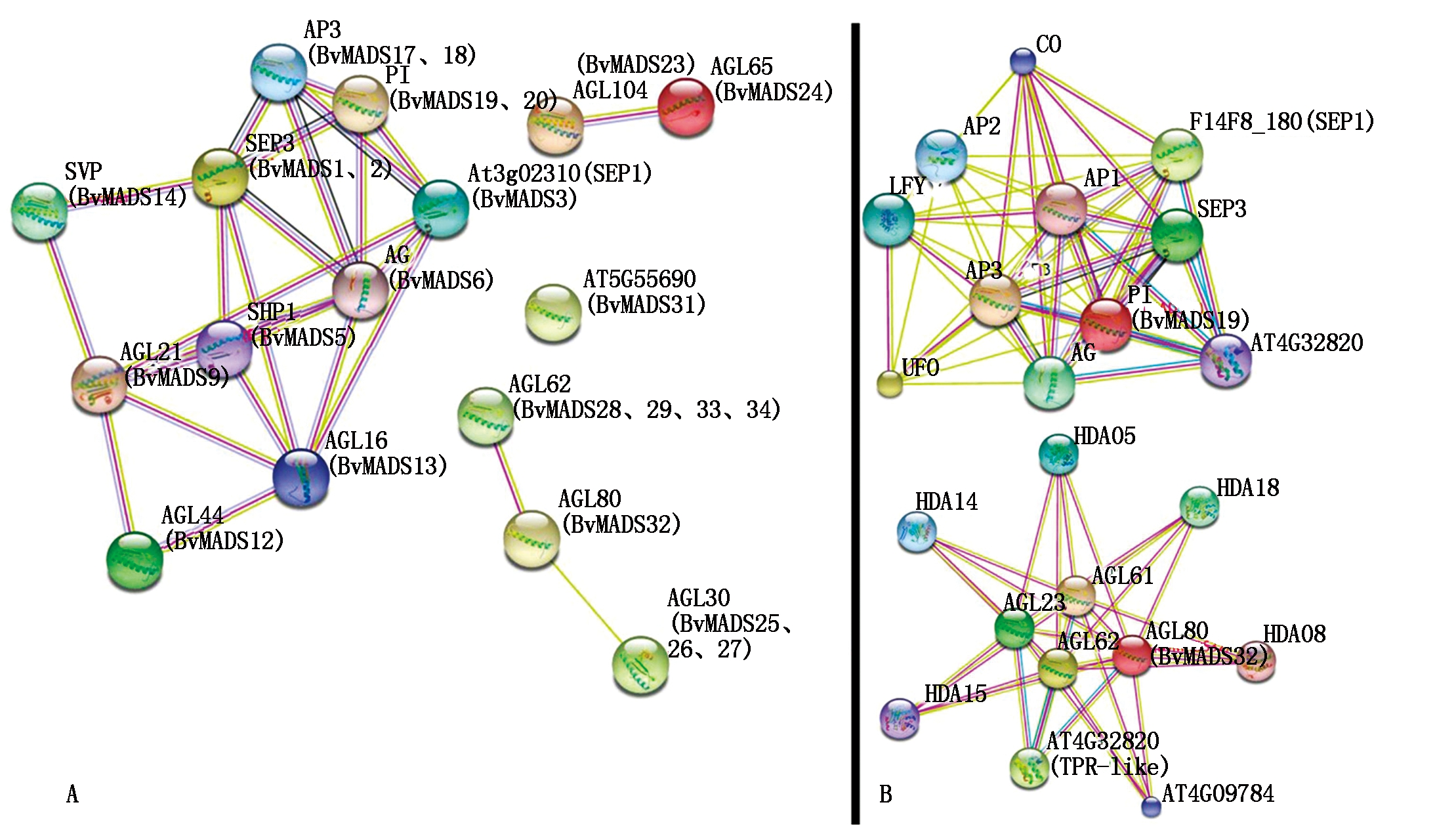
A.MADS-box蛋白功能联系网络;B.BvMADS19和MADS32蛋白共表达网络。
A.MADS-box protein function link network; B.BvMADS19 and MADS32 protein co-expression network.
图7 MADS-box蛋白功能联系网络
Fig.7 The network of MADS-box protein functional connections
Mγ组的BvMADS32与属于Mα组的AGL23、AGL61、AGL62存在蛋白互作,与组蛋白脱乙酰化酶HDA05、HDA08、HDA14、HDA15和HDA18存在蛋白互作,此外还与TRP-like蛋白以及一个未知蛋白互作(图7)。暗示type Ⅰ基因之间也存在蛋白互作,但是功能至今还不明确。在拟南芥中,HDA蛋白参与多种生物过程,包括调控盐胁迫、开花时间、免疫响应、ABA、SA、油菜素内酯和干旱响应等方面[36]。在盐、高温、低温胁迫下苹果也会改变HDA基因表达水平来抵抗胁迫[37]。结合BvMADS32表达量也受热胁迫而上调的表达谱分析,BvMADS32可能与HAD蛋白共同作用进而提高甜菜热抗性。
3 讨论
甜菜属于真双子叶植物的基部类群,对其MADS-box转录因子系统的鉴定,填补了被子植物到核心双子叶植物MADS-box转录因子无人报道的空缺。本研究共鉴定出34个甜菜MADS-box转录因子,不存在组缺失的情况,但无AGL6、AGL15、TM8亚组成员。甜菜MADS-box转录因子的数量多于低等植株江南卷柏(19个)、小立碗藓(24个)[38-40],但远少于核心双子叶植物拟南芥(108个)、杨树(105)、番茄(95)、白菜(160)[9,14,41-42]。究其原因,发现数目较多的这些物种均属高等植物,都经历过基因重复事件,包括全基因组复制、串联复制和片段复制。如白菜在进化过程中经历六倍体阶段,由全基因组复制事件产生3个亚基因组,导致白菜大量基因发生扩张[42]。相比于其他植物,甜菜进化地位相对保守,没有经历过全基因组复制,所以,MADS-box转录因子相对较少,更接近于低等物种。系统发育树结果显示,甜菜各组齐全,比低等物种亚组多,但相比于拟南芥、水稻、葡萄等高等植物,却缺失AGL6、AGL15、TM8亚组。表明在石竹目被分出来之前,MADS-box基因就完成了目前组的划分,部分亚组是在后期进化逐渐形成的。
根据系统发育树对甜菜MADS-box基因进行分类,比较基因结构和保守元件发现,即使同一亚组内基因结构也差别较大;但是MEME模体分析结构显示,同一亚组各基因翻译出的蛋白质具有类似的Motif。结合表达结果来看,同一亚组内的基因多具有一致的表达模式,但是也有部分基因表达模式展示出差异。如AG亚组的BvMADS5和BvMADS6,AP3-PI亚组的BvMADS17、BvMADS18、BvMADS19和BvMADS20。表明在进化的过程中同一起源的旁系基因在表达时空特性上发生趋异分化现象,这可能是由同一起源的旁系基因避免功能冗余,产生亚功能和新功能的内在需要导致的。
分析MADS-box基因表达谱,发现甜菜MADS-box基因主要作用于花序分生组织,但是部分MADS-box基因在幼嫩组织、种子、果实发育中发挥重要作用,如BvMADS8、BvMADS14、BvMADS18和BvMADS29。部分MADS-box基因在参与根和叶发育或生理活动,如BvMADS3、BvMADS7、BvMADS14、BvMADS23。可见,MADS-box家族基因表达于各个组织,作用广泛,对生长和发育都有重要作用。大量研究也证实了以上猜测,如番茄MADS-box基因TAGL1影响果皮角质层形成[43] ;葡萄部分MADS-box基因与胚珠败育相关[44] ;香蕉MaMADS7参与乙烯合成路径,对果实成熟有重要作用[45] ;日本梨PpMADS13-1与种子休眠相关[46] ;胁迫表达谱发现,BvMADS19、BvMADS32响应热胁迫而上调表达,表明对胁迫也有重要作用,可以提高甜菜逆境生理。类似地,在矮牵牛中也发现一个MADS-box基因PMADS9与逆境胁迫相关。
蛋白功能联系预测表明,MADS-box基因之间通过协同作用来调控花发育,这与大部分MADS-box集中在花序分生组织中优势表达RNA-seq分析结果相吻合。BvMADS19、BvMADS32热抗性功能被表达谱和蛋白共表达分析网络所证实,其抗性功能源于与HDA或CO蛋白互作,影响脯氨酸和乙烯等物质含量。本结果为进一步研究MADS-box基因的特点及其在调控观赏甜菜花器官发育、逆境抗性中的作用奠定基础。
参考文献:
[1] Alvarezbuylla E R,Liljegren S J,Pelaz S,et al.MADS-box gene evolution beyond flowers:expression in pollen,endosperm,guard cells,roots and trichomes[J].Plant Journal,2000,24(4):457-466.
[2] Becker A,Theissen G.The major clades of MADS-box genes and their role in the development and evolution of flowering plants[J].Molecular Phylogenetics and Evolution,2003,29(3):464-489.
[3] Kaufmann K,Melzer R,Theissen G.MIKC-type MADS-domain proteins:structural modularity,protein interactions and network evolution in land plants[J].Gene,2005,347(2):183-198.
[4] Tian Y,Dong Q,Ji Z,et al.Genome-wide identification and analysis of the MADS-box gene family in apple[J].Gene,2015,555(2):277-290.
[5] Saha G,Park J I,Jung H J,et al.Genome-wide identification and characterization of MADS-box family genes related to organ development and stress resistance in Brassica rapa[J].BMC Genomics,2015,16(1):178.
[6] Li C,Wang Y,Xu L,et al.Genome-Wide characterization of the MADS-box gene family in radish (Raphanus sativus L.) and assessment of its roles in flowering and floral organogenesis[J].Frontiers in Plant Science,2016,7:1390.
[7] 邱文明,徐育海,何秀娟.板栗雌雄花发育相关的MADS-box基因发掘与表达分析[J].园艺学报,2016,43(8):1593-1604.
[8] Bemer M,Gordon J,Weterings K,et al.Divergence of recently duplicated Mγ-type MADS-box genes in Petunia[J].Molecular Biology & Evolution,2010,27(2):481-495.
[9] Parenicová L,De Folter S,Kieffer M,et al.Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis:new openings to the MADS world[J].The Plant Cell,2003,15(7):1538-1551.
[10] Xu Z,Zhang Q,Sun L,et al.Genome-wide identification,characterisation and expression analysis of the MADS-box gene family in Prunus mume[J].Molecular Genetics and Genomics,2014,289(5):903-920.
[11] De Bodt S,Theissen G,Van De Peer Y.Promoter analysis of MADS-box genes in eudicots through phylogenetic footprinting[J].Molecular Biology and Evolution,2006,23(6):1293-1303.
[12] Martinez-Castilla L P,Alvarez-buylla E R.Adaptive evolution in the Arabidopsis MADS-box gene family inferred from its complete resolved phylogeny[J].Proceedings of the National Academy of Sciences of United States in America,2003,100(23):13407-13412.
[13] Kater M M,Dreni L,Colombo L.Functional conservation of MADS-box factors controlling floral organ identity in rice and Arabidopsis[J].Journal of Experimental Botany,2006,57(13):3433-3444.
[14] Leseberg C H,Li A,Kang H,et al.Genome-wide analysis of the MADS-box gene family in Populus trichocarpa[J].Gene,2006,378(1):84-94.
[15] Arora R,Agarwal P,Ray S,et al.MADS-box gene family in rice:genome-wide identification,organization and expression profiling during reproductive development and stress[J].BMC Genomics,2007,8(1):242.
[16] Díaz-Riquelme J,Lijavetzky D,Martínez-Zapater J M,et al.Genome-wide analysis of MIKCC-type MADS box genes in grapevine[J].Plant Physiology,2009,149(1):354-369.
[17] Zhang L,Xu Y,Cloning M M,et al.And chromosomal localization of two MADS-box genes in peach(Prunus persica) [J].Journal of Genetics and Genomics,2008,35(6):365-372.
[18] Martel C,Vrebalov J,Tafelmeyer P A.The tomato MADS-box transcription factor RIPENING INHIBITOR interacts with promoters involved in numerous ripening processes in a COLORLESS NONRIPENING-Dependent manner[J].Plant Physiology,2011,157(3):1568-1579.
[19] Ma H.Molecular genetic analyses of microsporogenesis and microgametogenesis in flowering plants[J].Annual Review of Plant Biology,2005,56(1):393-434.
[20] Mara C D,Irish V F.Two GATA transcription factors are downstream effectors of floral homeotic gene action in Arabidopsis[J].Plant Physiology,2008,147(2):707-718.
[21] Kaufmann K,Muino J M,Jauregui R,et al.Target genes of the MADS transcription factor SEPALLATA3:integration of developmental and hormonal pathways in the Arabidopsis flower[J].PLoS Biology,2009,7(4):854-875.
[22] 陈 璟,李名扬,闫明旭,等.矮牵牛PMADS9基因的结构特征和mRNA的表达分析[J].园艺学报,2011,38(1):108-116.
[23] 梁 芳,许申平,蒋素华,等.菊花MADS-box基因的克隆与表达载体构建[J].华北农学报,2016,31(3):25-31.
[24] 周云涛,王茂林,胡远辉,等.甘蓝型油菜MADS-box基因家族APETALA3基因编码区的克隆与序列分析[J].四川大学学报:自然科学版,2004,41(5):1091-1093.
[25] Dohm J C,Minoche A E,Holtgraewe D A,et al.The genome of the recently domesticated crop plant sugar beet (Beta vulgaris) [J].Nature,2014,505(7484):546.
[26] Minoche A E,Dohm J C,Schneider J A,et al.Exploiting single-molecule transcript sequencing for eukaryotic gene prediction[J].Genome Biology,2015,16(1):1-13.
[27] Stracke R,Holtgraewe D,Schneider J,et al.Genome-wide identification and characterisation of R2R3-MYB genes in sugar beet (Beta vulgaris)[J].BMC Plant Biology,2014,14(1):1-17.
[28] 孔维龙,胡 瑞,包满珠,等.香石竹水孔蛋白基因的克隆及表达分析[J].园艺学报,2017,44(3):515-527.
[29] 陈鸿飞,邵红霞,樊 胜,等.苹果全基因组多聚半乳糖醛酸酶基因家族的鉴定及进化分析[J].园艺学报,2016,43(10):1863-1877.
[30] 宋春晖,张 东,马娟娟,等.苹果生长素运输基因MdABCB19的克隆及其在矮化砧木中的表达分析[J].园艺学报,2017,44(3):409-421.
[31] 林 雯.TPR基序——一种介导蛋白质相互作用的结构域[J].国外医学:分子生物学分册,2000,22(5):289-293.
[32] 张 霞,唐 维,刘 嘉,等.过量表达水稻OsP5CS1和OsP5CS2基因提高烟草脯氨酸的生物合成及其非生物胁迫抗性[J].应用与环境生物学报,2014,20(4):717-722.
[33] 韩兴杰,徐玲玲,廖 亮,等.茶树LEAFY基因的克隆和表达分析[J].园艺学报,2015,42(8):1606-1616.
[34] Weigel D,Meyerowitz E M.Activation of floral homeotic genes in Arabidopsis[J].Science,1993,261(5129):1723-1726.
[35] Moyroud E,Tichtinsky G,Parcy F.The LEAFY floral regulators in angiosperms:conserved proteins with diverse roles[J].Journal of Plant Biology,2009,52(3):177-185.
[36] Song C P,Galbraith D W.AtSAP18,an orthologue of human SAP18,is involved in the regulation of salt stress and mediates transcriptional repression in Arabidopsis[J].Plant Molecular Biology,2006,60(2):241-257.
[37] 张蕊芬,刘 会,周李杰,等.苹果组蛋白脱乙酰化酶基因HDA19的生物信息学分析与胁迫响应研究[J].园艺学报,2016,43(4):613-622.
[38] Yi J M,Huang T,Huang Y,et al.Phylogenetic analysis of MADS-box gene family in physcomitrella patens[J].Plant Physiology Journal,2015,51(2):197-206.
[39] Barker E I,Ashton N W.A parsimonious model of lineage-specific expansion of MADS-box genes in Physcomitrella patens[J].Plant Cell Reports,2013,32(8):1161-1177.
[40] Kwantes M,Liebsch D,Verelst W.How MIKC*MADS-box genes originated and evidence for their conserved function throughout the evolution of vascular plant gametophytes[J].Molecular Biology & Evolution,2011,29(1):293-302.
[41] Guo X H,Chen G P,Cui B L,et al. Solanum lycopersicum agamous-like MADS-box protein AGL15-like gene,SlMBP11,confers salt stress tolerance[J].Molecular Breeding,2016,36(9):125.
[42] Duan W K,Song X M,Liu T K,et al.Genome-wide analysis of the MADS-box gene family in Brassica rapa (Chinese cabbage) [J].Molecular Genetics and Genomics,2015,290(1):239-255.
[43] Gimenez E,Dominguez E,Pineda B,et al.Transcriptional activity of the MADS box ARLEQUIN/TOMATO AGAMOUS-LIKE1 gene is required for cuticle development of tomato fruit[J].Plant Physiology,2015,168(3):1036.
[44] Wang L,Yin X J,Cheng C X,et al.Evolutionary and expression analysis of a MADS-box gene superfamily involved in ovule development of seeded and seedless grapevines[J].Molecular Genetics and Genomics,2015,290(3):825-846.
[45] Liu J H,Liu L,Li Y J,et al.Role for the banana AGAMOUS-like gene MaMADS7 in regulation of fruit ripening and quality[J].Physiologia Plantarum,2015,155(3):217-231.
[46] Saito T,Bai S L,Imai T,et al.Histone modification and signalling cascade of the dormancy-associated MADS-box gene,PpMADS13-1,in Japanese pear (Pyrus pyrifolia) during endodormancy[J].Plant Cell and Environment,2015,38(6):1157-1166.