蓖麻(Ricinus communis L.)是大戟科蓖麻属植物,蓖麻油广泛应用于国防、农业、化工、医药和机械制造等方面,具有重要的商业价值 [1]。我国是世界第二大蓖麻籽生产国,北起黑龙江,南至海南岛均有种植 [2]。与主要农作物相比,蓖麻产量相对低,这极大地限制了蓖麻产业的发展。矮秆蓖麻植株较矮,营养体小(80~150 cm),占据空间小,水分、养分运输的距离相对较短,经济系数相对高,提高了养分、水分的利用率,增强了其抗倒伏、抗旱性和耐瘠薄能力,若遇强降雨、大风等恶劣天气,矮秆蓖麻防灾抗灾能力大大增强,提高了瘠薄地、边际地的利用效率,进而提高蓖麻产量,所以蓖麻矮化育种研究成为热点。
赤霉素(GA)是一类调节植物生长的重要激素,主要在植物的顶端幼嫩部位合成,如茎尖和根尖,在生长中的种子和果实中也大量合成。GA在种子萌发、茎伸长、子叶伸展、下胚轴伸长以及成花诱导等生命活动中发挥重要作用。DELLA蛋白是GA信号转导途径的关键调控元件。它的生物学功能是负向调节GA 信号途径,抑制植物生长发育 [3-9]。DELLA蛋白的氨基酸序列中,N端的同源性不高,但均具有DELLA 和TVHYNP 2个保守的酸性结构域。一般认为,这2个结构域的突变影响蛋白与赤霉素受体GID1的结合能力 [10-11]。随着对DELLA蛋白研究的深入,DELLA基因已经在多种植物被克隆和表达分析,水稻中的SLR1基因、小麦中的RHT-1基因、玉米中的D8基因 [12]、大麦中的SLN1基因、甜樱桃中的PaGAI基因 [13]、棉花中的GhGAI3、GhGAI4、GhGAI2b、GhGAI4b基因 [14-17]以及拟南芥中的GAI、RGA、RGL1、RGL2、RGL3。Thomas等 [18]验证了DELLA蛋白和GA调控拟南芥种子萌发和花的发育。Hou等 [19]以野生型和della缺失突变体拟南芥为材料,发现在没有GA存在的条件下,DELLA结合JAZ,释放MYC2促进下游JA效应基因的表达。Boccaccini等 [20]发现DOF蛋白的DAG1和DELLA蛋白的GAI共同作用,负向调控AtGA3oxl基因,进而调控拟南芥的生长。Hussain等 [21]研究发现,DELLA与两HD-ZIP的转录因子可以调节GA信号转导,抑制拟南芥生长。大量试验证明DELLA过表达植株可以明显抑制GA的作用,显著降低株高。本试验通过克隆蓖麻DELLA基因完整阅读框并对其进行生物学信息分析,为进一步研究DELLA蛋白对蓖麻的矮化作用奠定基础。
1 材料和方法
1.1 试验材料
1.1.1 植物材料 蓖麻2129品系由内蒙古通辽市农业科学研究院提供,取自2129植株六叶期茎尖,经液氮速冻后保存于-80 ℃冰箱备用。
1.1.2 试剂及菌株 Pyrobest DNA 聚合酶、T4 DNA连接酶、DNA Marker(购自宝生物有限公司);DH5α大肠杆菌感受态细胞、pMD-18T载体(购自北京庄盟国际科技有限公司)。
1.2 试验方法
1.2.1 总RNA的提取,反转录cDNA 采用TaKaRa的RNA提取试剂盒提取蓖麻2129生长六叶期茎尖的总RNA,备用。参照TaKaRa公司的cDNA 逆转录试剂盒说明书进行逆转录。
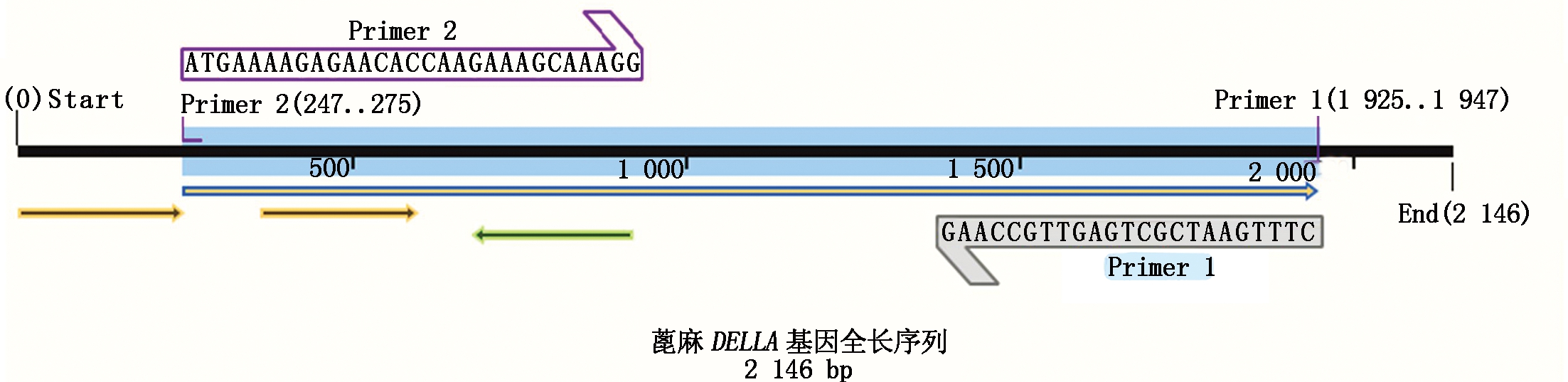
1.2.2 DELLA基因的克隆 根据NCBI上公布的蓖麻DELLA的全长序列,设计5′端引物primer2:5′-ATGAAAAGAGAACACCAAGAAAGCAAA
GG-3′,3′端引物primer1:5′-CTTTGAATCGCTG
AGTTGCCAAG-3′(图1)。以蓖麻2129六叶期茎尖的总RNA逆转录cDNA为模板进行扩增。扩增体系:cDNA 100 ng,10×PCR Buffer 5 μL,dNTP(2.5 mmol/L)3 μL,上下游引物各1 μL,Pyrobest DNA 聚合酶2.5 μL,总体积50 μL。PCR 程序为:94 ℃预变性5 min;94 ℃ 变性30 s,58 ℃退火45 s,72 ℃ 延伸2 min,28 个循环。扩增产物用1%琼脂糖凝胶电泳进行鉴定,然后用琼脂糖凝胶DNA回收试剂盒回收目的条带,连接pMD-18T载体,转化大肠杆菌DH5α感受态细胞,进行蓝白斑筛选,挑取白色单菌落,放大培养后将阳性克隆送至GENERAY公司测序。
1.2.3 DELLA基因的生物信息学分析 克隆得到的DELLA基因序列,利用在线工具ProtParam程序(http://web.Expasy.org/protparam/)预测蛋白质的分子式、分子量、等电点和不稳定系数等;TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)对蛋白质跨膜型进行预测;用SignalP(http://www.cbs.dtu.dk/services/SignalP/)程序进行蛋白质信号肽分析;用Predict-protein(http://www.Predictprotein.org)程序对蛋白质二级结构进行预测;利用Phyre 2 软件(http://www.sbg.bio.ic.ac.uk/phyre2)对蓖麻RcDELLA基因的三级结构进行预测;在线工具ProtFun 2.2 Server(http://www.cbs.dtu.dk/services/ProtFun/)进行了功能预测分析;采用Interproscan ( http: //www.Ebi.Ac.uk /interpro /scan.html)软件对RcDELLA同源序列进行保守序列和结构域分析。
The full-length sequence of DELLA gene of castor 2 146 bp
图1 引物设计
Fig.1 Primer design
2 结果与分析
2.1 蓖麻总RNA的提取
由图2可知,利用RNA提取试剂盒法提取蓖麻六叶期茎尖的总RNA,经电泳检测后28S和18S核糖体RNA亮度接近2∶1,且没有DNA 污染,紫外分光光度计检测结果显示OD260/280为1.95,说明提取的蓖麻茎尖总RNA纯度高,可以用于后续试验。
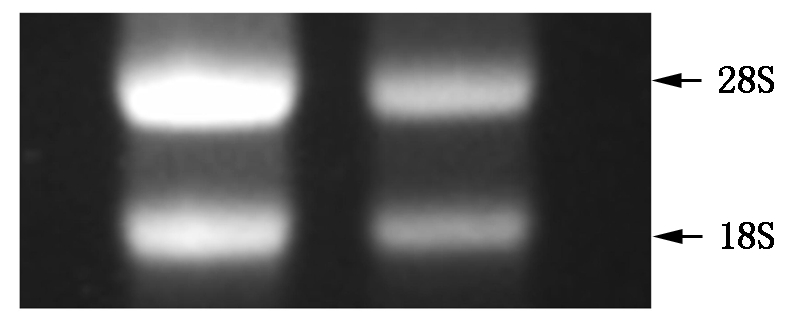
图2 蓖麻总RNA提取结果
Fig.2 Extraction of total RNA from castor
2.2 RcDELLA基因及编码的氨基酸特征分析
在已知序列的基础上对目的基因进行扩增,扩增获得DELLA完整开放阅读框全长为1 701 bp(图3),编码567个氨基酸,其中亮氨酸(Leu)最多,有61个,占10.8%;其次是丝氨酸(Ser)有57个,占10.1%;丙氨酸(Ala)有50个,占8.8%;谷氨酸(Glu)40个,占7.1%。根据ExPASyProtParam预测:RcDELLA基因编码的蛋白分子式为C2744H4296N764O860S25,分子量为62 550.40 ku,等电点为5.14,不稳定系数为49.86,被归类为不稳定蛋白,GRAVY为-0.847,为亲水性蛋白。RcDELLA中带负电荷的残留物总数为70,带正电荷的残留物总数为47,半衰期为30 h。利用SignalP进行蛋白质信号肽分析,发现RcDELLA序列含有信号肽,为分泌蛋白。利用 TMHMM Server v. 2.0 网站对RcDELLA的跨膜结构域进行分析,结果表明RcDELLA蛋白质无跨膜结构域,这与理化性质分析此蛋白为亲水性蛋白的结果相一致。
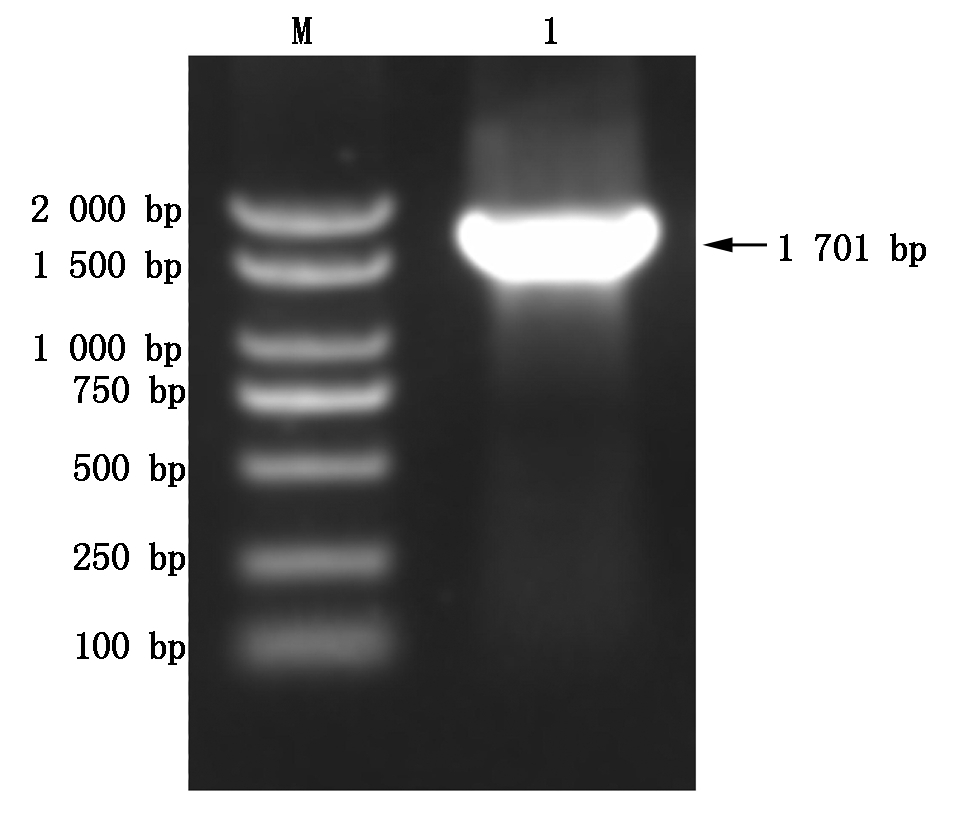
M.DL2000 Marker; 1.PCR扩增产物。
M.DL2000 Marker; 1.PCR product.
图3 RcDELLA扩增电泳图
Fig.3 Electrophoresis results of RcDELLA PCR
2.3 DELLA蛋白的二级结构和三级结构预测
利用Predict-protein对DELLA蛋白二级结构进行预测,其中α-螺旋占40.4%,β-折叠占8.6%,无规则卷曲占51.0%。利用Phyre 2网站对蓖麻DELLA基因编码的蛋白质进行三级结构预测,结果见图4。
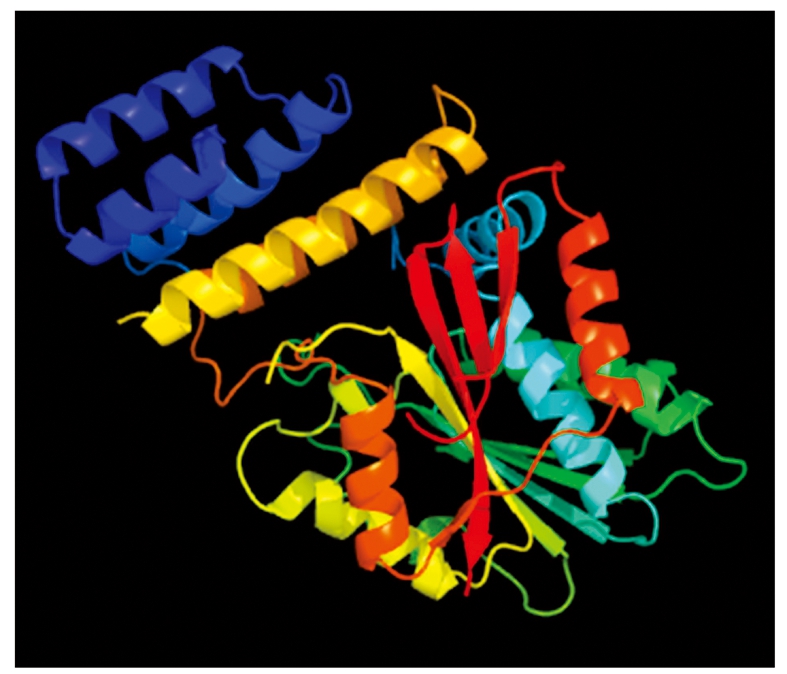
图4 RcDELLA蛋白的三级结构预测
Fig.4 Prediction of the tertiary structure of RcDELLA
2.4 RcDELLA同源性和系统进化分析
利用DNAMAN软件对蓖麻RcDELLA与柑橘(Citrus sinensis,XP_006482132.1)、木本棉(Gossypium arboreum,XP_017606442.1)、巴西橡胶树(Hevea brasiliensis,ALG02536.1)、麻疯树(Jatropha curcas,XP_012077792.2)、核桃(Juglans regia,XP_018816848.1)、苹果(Malus domestica,NP_001315916.1 )、芍药(Paeonia lactiflora,ANA05341.1)、胡杨(Populus euphratica,XP_011002785.1 )、桃(Prunus persica,XP_007214956.1 )、中国李(Prunus salicina,AQQ12221.1 )、葡萄(Vitis vinifera,XP_002266267.1 )、金丝小枣(Ziziphus jujuba,XP_015872191.1 )12种DELLA氨基酸序列进行同源性比对(图5),其中与巴西橡胶树相似性为73.87%,与柑橘相似性为74.49%,13个氨基酸序列N端的保守性不高,但均具有DELLA 和TVHYNP 2个保守的酸性结构域。
利用MEGA 7.0 软件构建系统进化树(图6),结果表明,RcDELLA先与大戟科巴西橡胶树(ALG02536.1)和麻疯树(XP_012077792.2)形成分支,结果符合进化关系,与同源性比对结果一致。
2.5 RcDELLA蛋白的功能预测分析及保守序列和结构域分析
利用在线工具ProtFun 2.2 Server进行了功能预测分析,发现此基因的主要功能是转运结合(表1)。

向右箭头为DELLA保守结构域;向左箭头为TVHYNP保守结构域;五角星为蓖麻DELLA氨基酸序列。Ps_DELLA.中国李;Hb_DELLA.巴西橡胶树;Ga_DELLA.木本棉;Cs_DELLA.柑橘;Jr_DELLA.核桃;Pp_DELLA.桃;Pe_DELLA.胡杨;Pl_DELLA.芍药;Md_DELLA.苹果;Vv_DELLA.葡萄;Rc_DELLA.蓖麻;Zj_DELLA.金丝小枣;Jc_DELLA.麻疯树。
The right arrow is the conservative domain of the DELLA; the left arrow is the TVHYNP conservative domain;five-pointed star is a castor DELLA amino acid sequence;Ps_DELLA. Prunus salicina;Hb_DELLA. Hevea brasiliensis; Ga_DELLA. Gossypium arboretum; Cs_DELLA. Citrus sinensis; Jr_DELLA.Juglans regia; Pp_DELLA.Prunus persica; Pe_DELLA.Populus euphratica; Pl_DELLA.Paeonia lactiflora; Md_DELLA.Malus domestica; Vv_DELLA.Vitis vinifera; Rc_DELLA.Ricinus communis; Zj_DELLA. Ziziphus jujube; Jc_DELLA. Jatropha curcas.
图5 不同植物DELLA基因氨基酸序列同源性比较
Fig.5 Alignment of deduced amino acid sequences of DELLA genes of different plants
利用Interproscan对RcDELLA同源序列进行保守序列和结构域分析(图7),结构域预测显示RcDELLA具有一个转录因子DELLA域和2个转录因子GRAS域,进一步证实该基因具有DELLA蛋白结构。
3 讨论
赤霉素(GA)是一类调节植物生长的重要激素,调控植物生长发育的各个阶段,包括种子的发芽、茎秆的伸长、开花时间、花与果实的成熟等许多不同的发育过程。DELLA蛋白负向调节GA 信号途径,抑制植株生长发育。当DELLA蛋白上的GA信号感知区接收到GA信号后, 这种蛋白的阻遏作用被解除,植株表现出瘦高的表型,但当DELLA结构发生变化或者过表达则不能降解或降解完全,从而使植株表现出矮化表型[22-23]。

图6 RcDELLA的进化树分析
Fig.6 Molecular phylogenic tree of RcDELLA from plants
表1 RcDELLA蛋白的功能预测分析
Tab.1 Function prediction analysis of RcDELLA protein

功能Function可能性Possibility可能率Odds生物合成的辅因子Cofactorsofbiosynthesis0.0380.530细胞外被膜Capsuleofextracellular0.0320.523细胞加工Cellprocessing0.0270.373中间代谢Intermediatemetabolism0.0410.658能量代谢Energymetabolism0.1121.242脂肪酸代谢Fattyacidmetabolism0.0171.303嘌呤嘧啶Purinepyrimidine0.0640.265转运结合Transportcombined0.8342.033

图7 RcDELLA保守序列和结构域分析
Fig.7 Analysis of conserved sequence and domain of RcDELLA
本试验克隆得到了1 701 bp的蓖麻RcDELLA蛋白基因cDNA 完整的开放阅读框序列,推测其可编码567 个氨基酸的多肽。蓖麻RcDELLA蛋白含有典型的DELLA和TVHYNP 2个保守的酸性结构域,这与NCBI上公布的其他植物的DELLA蛋白序列具有高度的同源性。通过理化性质、跨膜分析、信号肽预测和结构域分析显示:RcDELLA序列含有信号肽,为分泌蛋白,无跨膜结构域,这与理化性质分析此蛋白为亲水性蛋白结果相一致。功能预测分析表明,该蛋白的主要作用是转运结合。随着对DELLA蛋白研究的深入,它在植物生长发育过程的作用也受到越来越多的关注,利用分子生物学方法对蓖麻的RcDELLA基因进行克隆和分析,为进一步对蓖麻DELLA蛋白在植物体内作用机理的研究提供理论依据。目前,水稻、玉米、小麦等作物均已通过分子手段培育出了矮化的新品种,但在蓖麻中还未见报道。在下一步试验中,可以将RcDELLA基因转入野生蓖麻中,以期获得DELLA蛋白过表达的矮秆蓖麻表型。
参考文献:
[1] 郑 鹭, 祁建民, 陈绍军, 等. 蓖麻遗传育种进展及其在生物能源与医药综合利用潜势[J]. 中国农学通报, 2006, 22(9): 109-113.
[2] 朱国立, 何智彪, 贾娟霞, 等. 我国栽培蓖麻的生态区划及引种规律初探[J]. 内蒙古农业科技, 2012(5): 1-3.
[3] 黄桃鹏, 李媚娟, 王 睿, 等. 赤霉素生物合成及信号转导途径研究进展[J]. 植物生理学报, 2015, 51(8): 1241-1247.
[4] Zhao B, Li H, Li J, et al. Brassica napus DS-3, encoding a DELLA protein, negatively regulates stem elongation through gibberellin signaling pathway[J]. Theoretical & Applied Genetics, 2017, 130(4):727-741.
[5] Locascio A, Blázquez M A, Alabadí D. Genomic analysis of DELLA protein activity[J].Plant & Cell Physiology, 2013, 54(8):1229-1237.
[6] Yoshida H, Hirano K, Sato T, et al. DELLA protein functions as a transcriptional activator through the DNA binding of the indeterminate domain family proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(21): 7861.
[7] Li K L, Gao Z X, He H, et al. Arabidopsis DET1 represses photomorphogenesis in part by negatively regulating DELLA protein abundance in darkness[J]. MOLECULAR PLANT, 2015, 8(4): 622-630.
[8] 杜 冉,孙新蕊,钮世辉,等.油松DELLA蛋白结合GID1关键位点的鉴定和验证[J].西北植物学报,2017,37(1):32-39.
[9] Petrásek J, Friml J. Auxin transport routes in plant development[J]. Development, 2009, 136(16): 2675.
[10] Inouye S, Noguchi M, Sakaki Y, et al. Cloning and sequence analysis of cDNA for the luminescent protein aequorin[J]. Proceedings of the National Academy of Sciences of the United States of America, 1985, 82(10): 3154.
[11] Gou X P, He K, Yang H, et al. Genome-wide cloning and sequence analysis of leucine-rich repeat receptor-like protein kinase genes in Arabidopsis thaliana[J]. BMC Genomics, 2010, 11(1): 1-15.
[12] Lawit S J, Wych H M, Xu D P, et al. Maize DELLA proteins dwarf plant8 and dwarf plant9 as modulators of plant development[J]. Plant and Cell Physiology, 2010, 51(11): 1854-1868.
[13] 钟 翡, 沈欣杰, 刘 芳, 等. 甜樱桃DELLA蛋白基因PaGAI的克隆与表达分析[J]. 园艺学报, 2012, 39(1): 143-150.
[14] 陈英杰, 詹杰鹏, 王志江, 等. 棉花DELLA蛋白GhGAI2b基因的克隆和功能初步分析[J]. 石河子大学学报, 2014, 32(5): 635-640.
[15] Xiyang L, Cui B, Duan S, et al. Functional analysis of cotton DELLA protein GhGAI4a gene in Arabidopsis thaliana[J]. Journal of Shihezi University, 2013, 169(1): 137-142.
[16] Wen W, Cui B M, Yu X L, et al. Cloning and sequence analysis of the promoters of cotton DELLA protein genes GhGAI3 and GhGAI4[J]. Genomics & Applied Biology, 2010, 29(6): 1055-1063.
[17] 彭丽军, 温 玮, 李 艳, 等. 棉花DELLA蛋白GhGAI4b基因的克隆及功能初步分析[J]. 新疆农业科学, 2012, 49(3): 405-413.
[18] Thomas S G, Hu J, Dill A, et al. DELLA proteins and Gibberellin-regulated seed germination and floral development in Arabidopsis[J]. Plant Physiology, 2004, 135(2): 1008.
[19] Hou X, Li Y L, Xia K, et al. DELLAs modulate jasmonate signaling via competitive binding to JAZs[J]. Developmental Cell, 2010, 19(6): 884.
[20] Boccaccini A, Santopolo S, Capauto D, et al. The DOF protein DAG1 and the DELLA protein GAI cooperate in negatively regulating the AtGA3ox1 gene[J]. Molecular Plant, 2014, 7(9): 1486-1489.
[21] Hussain A, Peng J R. DELLA proteins and GA signalling in Arabidopsis[J]. Journal of Plant Growth Regulation, 2003, 22(2): 134-140.
[22] Peng J, Carol P, Richards D E, et al. The Arabidopsis GAI gene defines a signaling pathway that negatively regulates gibberellin responses[J]. Genes & Development, 1997, 11(23): 3194-3205.
[23] Sato T, Miyanoiri Y, Takeda M, et al. Expression and purification of a GRAS domain of SLR1, the rice DELLA protein[J]. Protein Expression & Purification, 2014, 95(3):248.