棉花体细胞胚胎发生是棉花转基因工程中的重点和难点,对其进行系统研究对促进棉花遗传工程改良具有极其重要的意义[1]。进一步研究棉花体细胞胚胎发生时相关调控因子发挥的作用,对陆地棉体细胞胚胎发生过程中特异表达的特定基因沉默或者超表达,可在一定程度上提高或降低棉花体细胞胚胎诱导率,提高棉花遗传转化的速度与规模,进而加速棉花遗传育种进程,这对于突破棉花体细胞胚胎发生对基因型的依赖,扩大种质资源创制速度和利用范围,加速棉花遗传改良具有重大意义[2-3]。棉花胚胎发生率及植株再生率低在很大程度上限制了棉花转基因工程的进程。王志才等[4]从棉花高体胚能力种子的选育、高质量外植体的制备及愈伤组织的诱导、激素和调节物质的选择、畸形胚的发生和控制等多个方面系统分析了棉花体细胞胚胎发生中多种因素。王彦霞等[5]也对影响棉花幼胚(珠)离体培养及植株建成的因素进行了系统分析。目前,中国农业科学院棉花研究所李付广研究员的科研团队已成功攻克了限制棉花体细胞胚胎发生的外部因素[6-7],例如培养基的种类和激素水平等,但是对于陆地棉体细胞胚胎诱导过程内在因素所发挥的分子机制的研究仍然较少。武秀明[8]以中棉所24的高分化率株系W10为材料,通过抑制性消减杂交法构建了棉花愈伤组织的抑制性消减杂交文库,即SSH文库(Suppression subtractive hybridization,SSH),包含368个差异表达的ESTs(Expressed sequence tags),部分ESTs与棉花体细胞胚胎发生密切相关。但是,使用常规手段对海量ESTs进行正向探索是一项相当复杂的工程。RNAi(RNA interference)干涉作为一种高效易于操作、试验结果易于显现的新技术,为研究海量ESTs提供了有效的实用方法[9-14]。当前,利用RNAi技术对特定基因的表达进行干涉已逐渐成为转基因研究的热点[15-19]。其中,以EST-SSR标记为代表的新技术的成功利用,使得我们对于植物的微观世界有了更进一步的了解[20-24]。
本研究采用RNAi技术,筛选武秀明[8]构建的SSH文库中在棉花愈伤组织分化过程中上调表达的4个ESTs[25-28]。利用pCRI1210双元表达载体,构建针对这4个ESTs的干涉载体,以棉花无菌苗下胚轴切段为受体,利用农杆菌介导法转化pCRI1210-EST干涉载体,通过在愈伤组织诱导培养基进行分化,从而研究这4个ESTs对愈伤组织诱导的影响。研究结果可为分离棉花体细胞胚胎发生相关基因提供技术支撑和理论依据。
1 材料和方法
1.1 试验材料
棉花种子由中国农业科学院棉花研究所提供,来自于中24筛选出的体细胞胚胎分化率较高的株系W10。所用载体pCRI1210、根癌农杆菌菌株LBA4404和ESTs由中国农业科学院棉花研究所李付广老师提供,购自天根生物制品公司。大肠杆菌(Escherichia coli)DH5α感受态细胞和连接酶等生物制剂,购自TaKaRa公司。其他化学药品均为国产分析纯。
1.2 引物和测序
测序工作由深圳华大基因科技有限公司完成。
1.3 试验方法
1.3.1 ESTs的选择 本研究挑选了SSH文库中4个在体细胞胚胎分化过程中上调表达的ESTs,文库编号分别为rmhcya0_000418、rmhcya0_000447、rmhcya0_000496、rmhcya0_000653(以下简称418、447、496和653),各ESTs信息如表1所示。
表1 ESTs信息表
Tab.1 The information of each ESTs

ESTs文库编号ESTsnumber长度/bpLengthORF长度/bpLength功能(SWISSPROT)Function功能(KEGG)Functionrmhcya0_000418384273蛋白激酶14蛋白激酶rmhcya0_000447515195耐乙烯蛋白2耐乙烯蛋白2rmhcya0_000496487249未知未知rmhcya0_000653552408耐盐蛋白表达蛋白
1.3.2 ESTs序列分析 利用DNAMAN软件和NCBI Blast搜索程序对各ESTs序列进行组装、分析和搜索。
1.3.3 ESTs序列的获得 根据ESTs序列信息 设计引物(表2)。ESTs扩摇后提取质粒。PCR扩增ESTs序列cds区的正向和反向片段。将扩增产物回收,与pMD19-T simple载体连接,转化大肠杆菌DH5α,菌落PCR和酶切验证后进行测序。
1.4 各ESTs干涉载体的构建
1.4.1 各ESTs正向序列和载体骨干片段的连接 利用KpnⅠ/BamHⅠ双酶切组合,分别酶切已连接正向片段的pMD19-T simple载体和pCRI1210载体,回收目标片段。将正向片段与载体骨干片段连接后转化DH5α感受态,利用质粒提取试剂盒提取质粒,然后酶切验证连接是否正确,进而确认中间连接载体A是否构建成功。
表2 各ESTs引物序列信息
Tab.2 The primer sequence information of each ESTs

ESTs编号 ESTsnumber 引物Primer引物序列(5'-3') Primersequence 引入酶切位点Restrictionsitesrmhcya0_000418418aFGGTACCTACTGGGGTTTTGGCTKpnⅠ418aRGGATCCCGAGGTACAAAACTCAACBamHⅠ418bFGAATTCCGAGGTACAAAACTCAACAGEcoRⅠ418bRGAGCTCTACTGGGGTTTTGGCTSacⅠrmhcya0_000447447aFGGTACCTCCTCTAGAGATTAGCGTGKpnⅠ447aRGGATCCCTGCAATTACACACCCBamHⅠ447bFGAATTCCTGCAATTACACACCCAGTEcoRⅠ447bRGAGCTCTCCTCTAGAGATTAGCGTGSacⅠrmhcya0_000496496aFGGTACCCAATTGTAGTAGGATTTCACAKpnⅠ496aRGGATCCTAGGAGAGGCAGTGAABamHⅠ496bFGAATTCGGTAGGAGAGGCAGTGAEcoRⅠ496bRGAGCTCCAATTGTAGTAGGATTTCACASacⅠrmhcya0_000653653aFGGTACCTATGATTCTCCACCAAGCKpnⅠ653aRGGATCCGGCATTGAAGGAGAAGBamHⅠ653bFGAATTCGGCATTGAAGGAGAAGCEcoRⅠ653bRGAGCTCTATGATTCTCCACCAAGCSacⅠ检测用intronAFintronAGATTCTCTTCTCTGCCACTCTGC -Detectionintron空载体检测135SFCACTATCCTTCGCAAGACCT -No-loaddetection1空载体检测2TRAACACATGAGCGAAACCCTA -No-loaddetection2
1.4.2 各ESTs反向序列和载体 骨干片段的连接 利用EcoRⅠ/ SacⅠ双酶切组合,分别酶切已连接反向片段的pMD19-T simple载体和上述中间载体A,回收目的片段。构建方法同上,4个ESTs干涉载体分别命名为pCRI1210-418、pCRI1210-447、pCRI1210-496和pCRI1210-653。
1.5 农杆菌介导转化棉花
1.5.1 干涉载体转化农杆菌 将空载体pCRI1210和4个干涉载体的质粒分别转化根癌农杆菌菌株LBA4404。
1.5.2 农杆菌介导转化棉花 W10株系无菌苗培养:将W10株系于培养基上无菌培养6~7 d,得到无菌苗。用手术刀切去无菌苗的叶片和根部,得到下胚轴,将其切成0.5~0.8 cm的切段,整齐摆放到无菌培养皿中。
浸染和共培养:将携带有干涉载体pCRI1210-418、pCRI1210-447、pCRI1210-496和pCRI1210-653质粒的农杆菌在LB培养基中活化并扩摇至OD600=0.3。
再将菌液倒入培养皿中,直至菌液完全淹没切段,轻轻摇动3~4 min,使菌液和切段充分接触,然后将经过浸染的切段用镊子一段一段整齐地摆放在共培养基上,切段之间保留3~5 mm的间隙,将培养皿用封口膜沿边缘封口2~3圈,然后将培养皿置于28 ℃恒温培养箱中避光培养48 h。
诱导下胚轴愈伤组织:避光培养48 h后,将下胚轴切段转移至愈伤组织诱导培养基上,野生型为对照,40 d后观察下胚轴出愈率。出愈率=出愈下胚轴/接种下胚轴×100%。3次重复。将野生型愈伤组织转接到分化培养基上诱导胚性愈伤组织。每隔40 d转移继代一次,连续继代3次。继代3次后仍没有分化的,按不分化统计。
1.5.3 PCR检测 提取转基因愈伤组织基因组DNA。PCR检测转基因阳性组织,所用引物如表2所示。
1.5.4 荧光显微镜下检测GFP荧光蛋白 用无菌镊子轻轻夹取少量发育良好的愈伤组织,轻轻压碎,慢慢盖上盖玻片制成水装片,在荧光显微镜下观察GFP荧光蛋白的表达量和分布,利用CCD 进行图像的采集。
1.6 Semi-Quantitative RT-PCR
1.6.1 总RNA的提取 利用Column Plant RNAOUT 2.0提取试剂盒提取下胚轴愈伤组织总RNA。
1.6.2 下胚轴愈伤组织总RNA的反转录 利用PrimeScriptⅡ1st Strand cDNA Synthesis Kit试剂盒反转录cDNA。
1.6.3 确定Semi-Quantitative RT-PCR指数扩增期 以转化pCRI1210-418 和pCRI1210的阳性愈伤组织以及野生型材料的cDNA为模板,进行3轮PCR扩增,循环数分别设为28,30,32,确定Semi-Quantitative RT-PCR的指数扩增期。
1.6.4 PCR扩增及电泳检测 PCR扩增30 次循环,反应结束后,将反应产物在1%的琼脂糖凝胶上检测,拍照。
1.6.5 干涉结果分析 将空载体pCRI1210的Semi-Quantitative产物进行10倍的倍比稀释,与pCRI1210-X(X=418、447、496和653)干涉后的Semi-QuantitativeRT-PCR产物进行对比,估算干涉后mRNA的降低量。
2 结果与分析
2.1 ESTs序列的获得和干涉载体的构建
以EST-418和EST-447为例,分析试验结果。
2.1.1 PCR扩增文库中各ESTs片段 从文库96孔板中挑取目标ESTs,提取质粒后PCR扩增各ESTs片段。扩增结果如图1所示,其中,图1-A、B分别为EST-418和EST-447的正向片段和反向片段的PCR扩增条带。
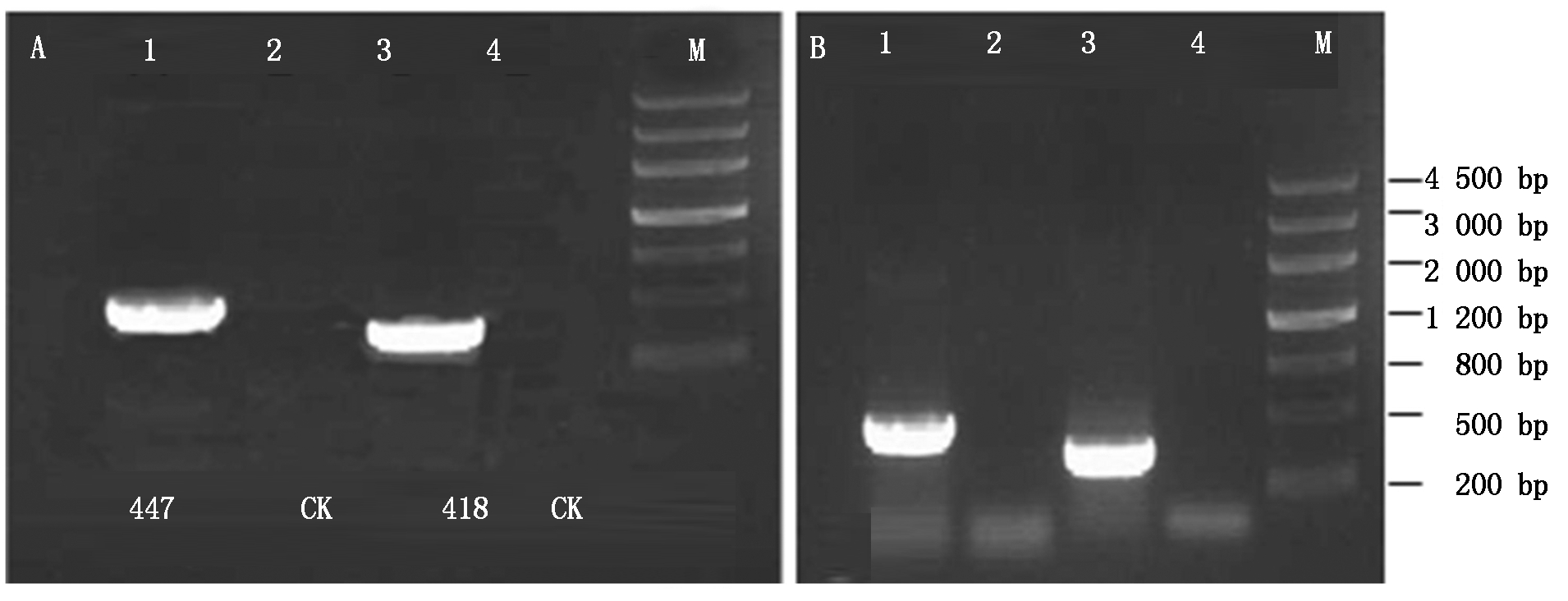
M.DNA Marker Ⅲ;A.正向片段:1.447正向扩增片段;2.447正向片段阴性对照;3.418正向扩增片段;
4.418正向片段阴性对照;B.反向片段:1.447反向扩增片段;2.447反向片段阴性对照;3.418反向扩增片段;4.418反向片段阴性对照。
M.DNA Marker Ⅲ;A.Forward:1.447 forward;2.447 forward negative control;3.418 forward;4.418 forward negative control;
B.Reverse:1.447 reverse;2.447 reverse negative control;3.418 reverse;4.418 reverse negative control.
图1 PCR扩增418和447正反向片段
Fig.1 PCR amplified 418 and 447 positive and reverse fragment
2.1.2 菌落PCR检测 各ESTs分别与pMD19-T simple载体连接,然后转化大肠杆菌感受态细胞DH5α,培养24~36 h,待培养皿上明显出现菌斑后,挑取单克隆进行菌落PCR,检测出阳性单克隆供后续试验使用。检测结果如图2所示。EST-418和EST-447扩增序列成功完成了T连接。
2.1.3 pMD19-T simple和pCRI1210上游多克隆位点的双酶切 提取EST-418和EST-447正向扩增序列和pCRI1210阳性单克隆质粒后,使用KpnⅠ/BamHⅠ双酶切,利用0.8%琼脂糖凝胶电泳对得到的酶切片段进行分离,然后使用凝胶试剂盒进行回收,结果显示,中间载体pCRI1210的上游多克隆位点和T载体均得到了十分完整的酶切,分离充分,符合试验要求(图3)。
2.1.4 中间载体PCR检测 将EST-418和EST-447的正向扩增片段和载体pCRI1210进行连接,获得中间载体pCRI1210-X(X=418、447),然后转化大肠杆菌感受态细胞DH5α,进行菌落PCR检测,检测结果如图4所示。图4显示,EST-418和EST-447的正向扩增片段均已成功连接到载体pCRI1210中。
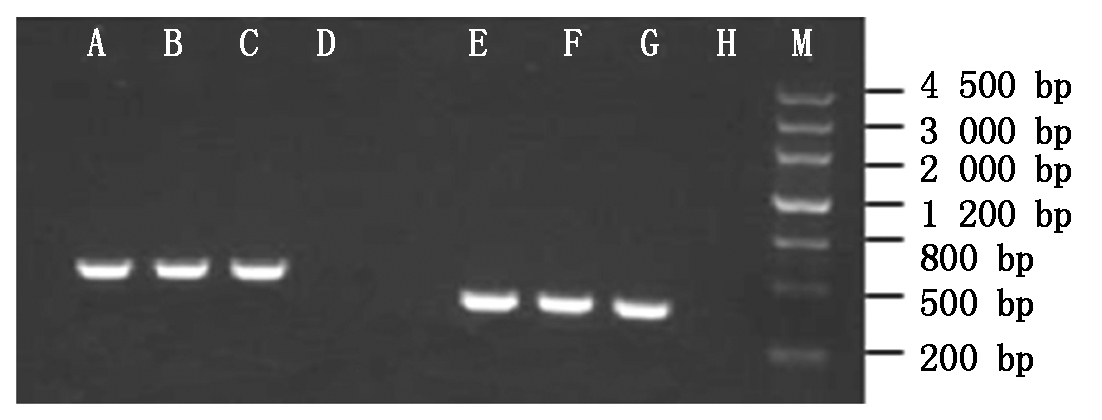
M.DNA Marker Ⅲ;A,B,C.447 3个单克隆PCR扩增条带;D.447阴性对照扩增条带;E,F,G.418 3个单克隆PCR扩增条带;H.418阴性对照扩增条带。
M.DNA Marker Ⅲ;A,B,C.Three monoclonal PCR amplification bands of 447;D.Amplification bands of 447 negative controls;E,F,G.Three monoclonal PCR amplification bands of 418;H.Amplification bands of 418 negative controls.
图2 菌落PCR检测各EST转化的pMD19-T simple载体
Fig.2 Identification of the EST transformed colonies to pMD19-T simple vector with PCR
M.DNA Marker Ⅲ;A.418;B.447;C~F.pCRI1210;G.pCRI1210 对照。
M.DNA Marker Ⅲ;A.418;B.447;C-F.pCRI1210;G.pCRI1210 control.
图3 Kpn Ⅰ/BamH Ⅰ双酶切中间载体的上游多克隆位点
Fig.3 Kpn Ⅰ/BamH Ⅰ double cleavage intermediate vector of upstream cloning site
M.DNA Marker Ⅲ;A~D.4个447不同单克隆扩增条带;E~H.4个418不同单克隆扩增条带。
M.DNA Marker Ⅲ;A-D.Four bands of 447 different monoclonal amplification;E-H.Four bands of 418 different monoclonal amplification.
图4 中间载体的菌落PCR扩增检测
Fig.4 Detection of colony of intermediate vector by PCR amplification
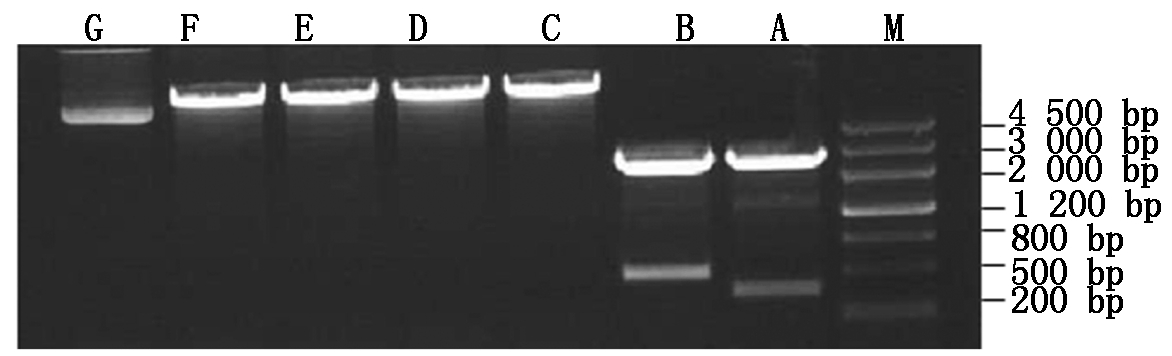
2.1.5 pCRI1210中间载体下游多克隆位点和T载体反向PCR扩增片段双酶切 将EST-418和EST-447的pCRI1210中间载体及其反向PCR扩增片段的T载体利用质粒提取试剂盒提取质粒,采用EcoRⅠ/SacⅠ双酶切组合同时进行酶切,结果如图5所示。
由图5可知,连接在T载体上的418和447反向扩增序列和载体pCRI1210下游的多克隆位点酶切效果良好,符合后续试验要求。
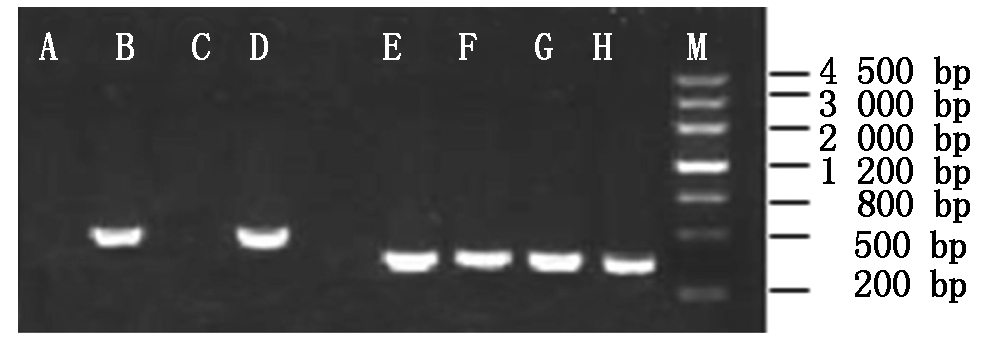
2.1.6 PCR检测目标干涉载体 将目标干涉载体转化大肠杆菌DH5α,菌落PCR检测。所用特异性检测引物AF intron(表2),检测结果如图6所示,目标干涉载体pCRI1210-X(X=418、417)构建成功,符合试验要求。

M.DNA Marker Ⅲ;A.418;B.447;C~D.EST-418下游中间载体;E.EST-418下游中间载体对照;F.EST-447下游中间载体对照;G~H.EST-447下游中间载体。
M.DNA Marker Ⅲ;A.418;B.447;C-D.EST-418 downstream intermediate carrier; E.EST-418 downstream intermediate carrier contrast;F.EST-447 downstream intermediate carrier contrast;G-H.EST-447 downstream intermediate carrier.
图5 pCRI1210中间载体下游多克隆位点和T载体反向PCR扩增片段双酶切
Fig.5 Double enzyme digestion of pCRI1210 intermediate vector downstream cloning site and T vector reverse PCR amplification fragment
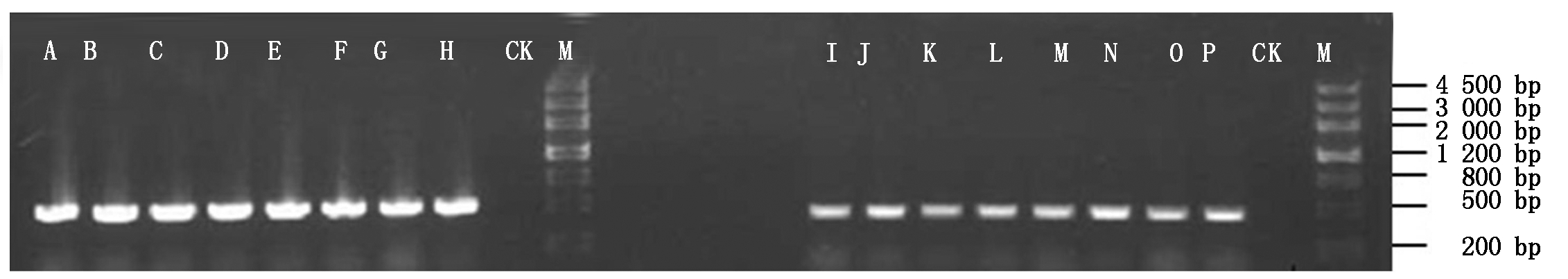
M.DNA Marker Ⅲ;CK.负对照;A~H.EST-447完整干涉载体;I~P.EST-418完整干涉载体。
M.DNA Marker Ⅲ; CK.Negative contrast; A-H.EST-447 complete interference vector; I-P.EST-418 complete interference vector.
图6 目标完整干涉载体菌落PCR扩增检测
Fig.6 Colony PCR amplification detection of target complete interference vector
2.1.7 目标干涉载体酶切验证 提取目标干涉载体pCRI1210-X(X=418、417)阳性克隆的质粒,对上游和下游扩增序列进行双酶切,由图7 可知,目标干涉载体pCRI1210-X(X=418、417)的正反片段均得到了预期的片段,证明各干涉载体均成功构建。
2.1.8 农杆菌菌落PCR法检测目标完整干涉载体 利用质粒提取试剂盒提取目的质粒,冰冻法转化根癌农杆菌LBA4404,菌落PCR法检测阳性单克隆,如图8所示。
由图8可知,各ESTs干涉载体转化农杆菌成功,可以用于浸染下胚轴外植体等后续试验。
2.2 农杆菌介导法转化棉花下胚轴外植体
2.2.1 诱导下胚轴愈伤组织 各ESTs干涉载体转化农杆菌后所浸染的下胚轴切段,部分切段12 d左右两端开始膨大,出现愈伤,40 d后达到最大值。不同ESTs干涉载体出愈率发生了变化,与对照相比,pCRI1210-418出愈率有所提高,而其他载体的出愈率出现了不同程度的下降,3次重复结果一致。
M.DNA Marker Ⅲ;A.EST-418正向扩增条带;B.EST-418反向扩增
条带;C.EST-447正向扩增条带;D.EST-447反向扩增条带。
M.DNA Marker Ⅲ; A.EST-418 positive amplification bands; B.EST-418 reverse amplification bands; C.EST-447 positive amplification bands;D.EST-447 reverse amplification bands.
图7 目标完整干涉载体酶切验证
Fig.7 Verification of target intact interference vector by enzyme digestion
由图9可知,农杆菌浸染后的下胚轴诱导40 d后,转化各EST对应的干涉载体的愈伤组织出愈率和愈伤组织的生长状态出现了明显的差异。EST-447的下胚轴切段经过培养基诱导后虽然两端也一定程度上发生了膨大,但发育非常缓慢最终未能继续诱导成愈伤组织,有的变为黑褐色后凋亡。而EST-418的愈伤组织生长旺盛且有一定光泽。
A.pCRI1210;B.pCRI1210-418干涉载体;C.pCRI1210-447干涉载体。
A.pCRI1210;B.pCRI1210-418 interference vector;C.pCRI1210-447 interference vector.
图8 农杆菌菌落PCR法检测目的完整干涉载体
Fig.8 Detection of complete interference vector by Agrobacterium tumefaciens colony PCR method
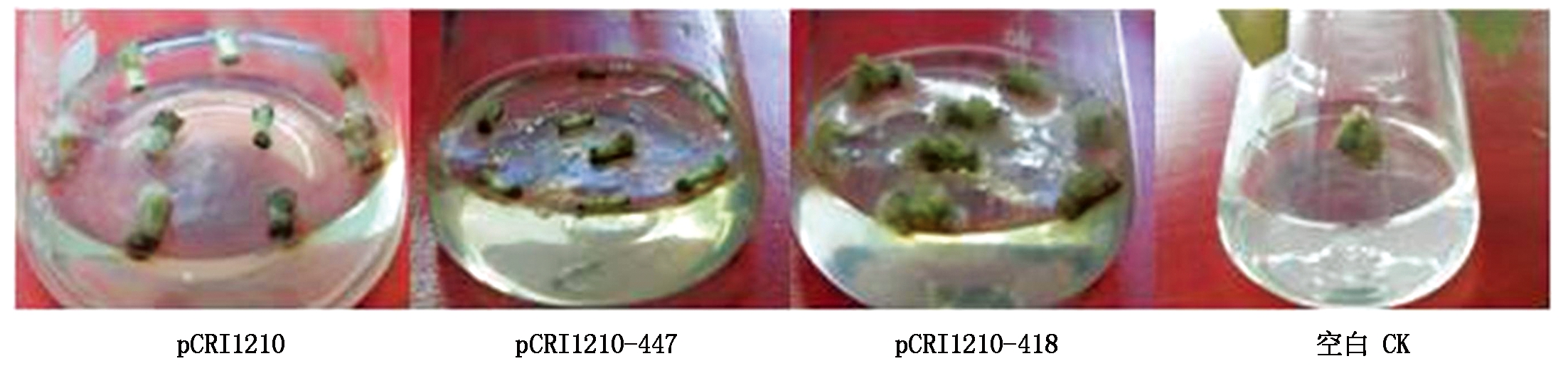
图9 诱导40 d的下胚轴愈伤组织
Fig.9 Callus induction of hypocotyl at 40 days
2.2.2 出愈率统计分析 统计各ESTs干涉载体转化的下胚轴愈伤组织的诱导情况,记录其出愈块数及其生长状态,统计其出愈率,具体情况如表3所示。
表3 各干涉载体对愈伤组织诱导的影响
Tab.3 Effect of different interference vector on induction of callus
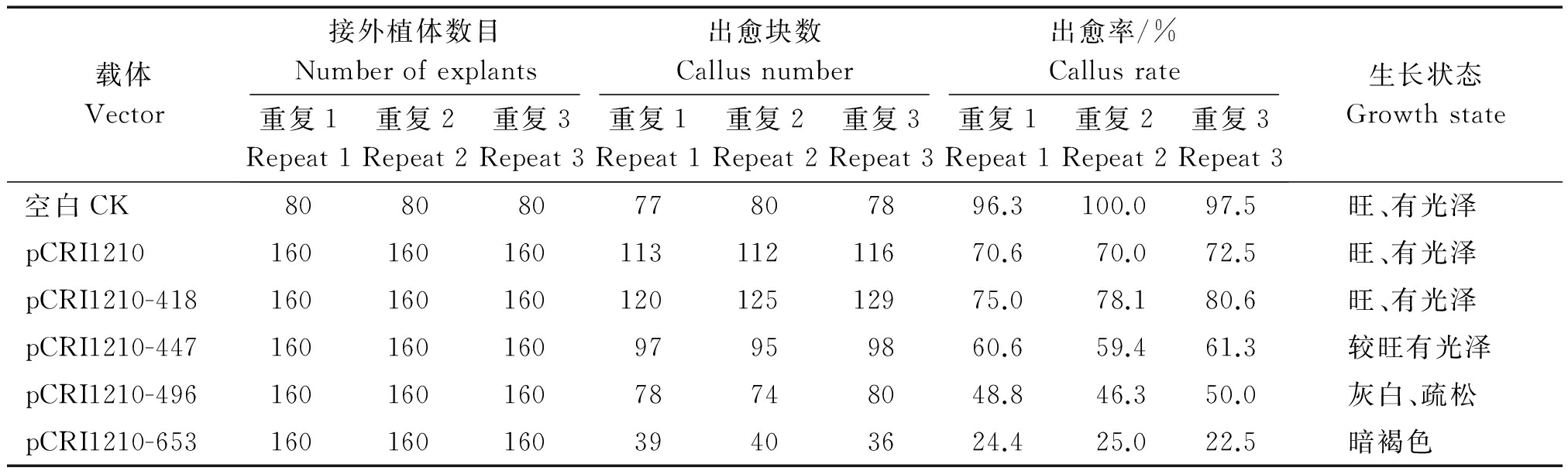
载体Vector接外植体数目Numberofexplants出愈块数Callusnumber出愈率/%Callusrate重复1Repeat1重复2Repeat2重复3Repeat3重复1Repeat1重复2Repeat2重复3Repeat3重复1Repeat1重复2Repeat2重复3Repeat3生长状态Growthstate空白CK80808077807896.3100.097.5旺、有光泽pCRI121016016016011311211670.670.072.5旺、有光泽pCRI1210-41816016016012012512975.078.180.6旺、有光泽pCRI1210-44716016016097959860.659.461.3较旺有光泽pCRI1210-49616016016078748048.846.350.0灰白、疏松pCRI1210-65316016016039403624.425.022.5暗褐色
利用统计学软件SAS 9.1,对记录的数据整理后进行显著性方差分析,结果如表4所示。
将表3,4转化绘制成图10,可直观观测各载体诱导的愈伤干涉后出愈率的变化。
由表4、图10可知,各ESTs平均值与对照pCRI210相比,在P<0.01和P<0.05水平上均存在显著性差异。其中,与对照相比,转化载体pCRI1210-418所转化的下胚轴外植体出愈率提高,而其他3个载体pCRI1210-X(X=447、496和653)所转化的下胚轴外植体的出愈率均出现不同程度的下降。其中,转化载体pCRI1210-653的下胚轴外植体出愈率最低,且其愈伤组织生长最为缓慢,生长状态也较差。
表4 显著性方差分析
Tab.4 Significant analysis of variance
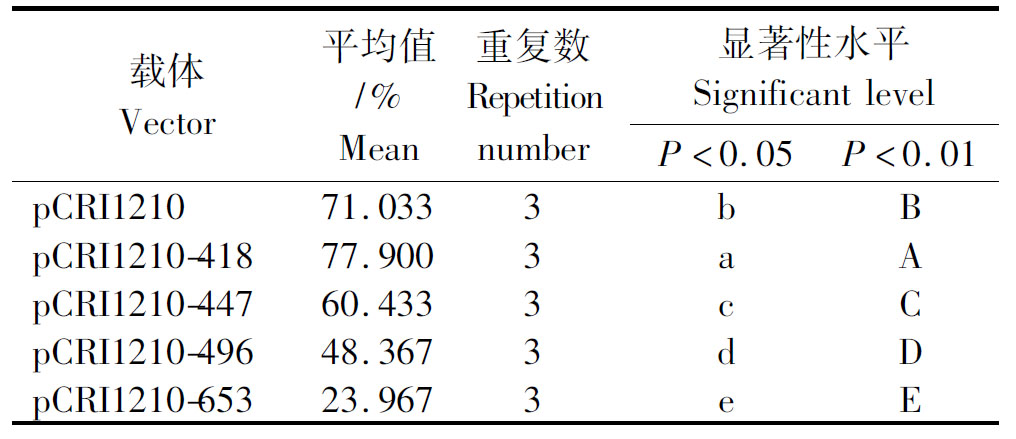
载体Vector平均值/%Mean重复数Repetitionnumber显著性水平SignificantlevelP<0.05P<0.01pCRI121071.0333bBpCRI1210-41877.9003aApCRI1210-44760.4333cCpCRI1210-49648.3673dDpCRI1210-65323.9673eE
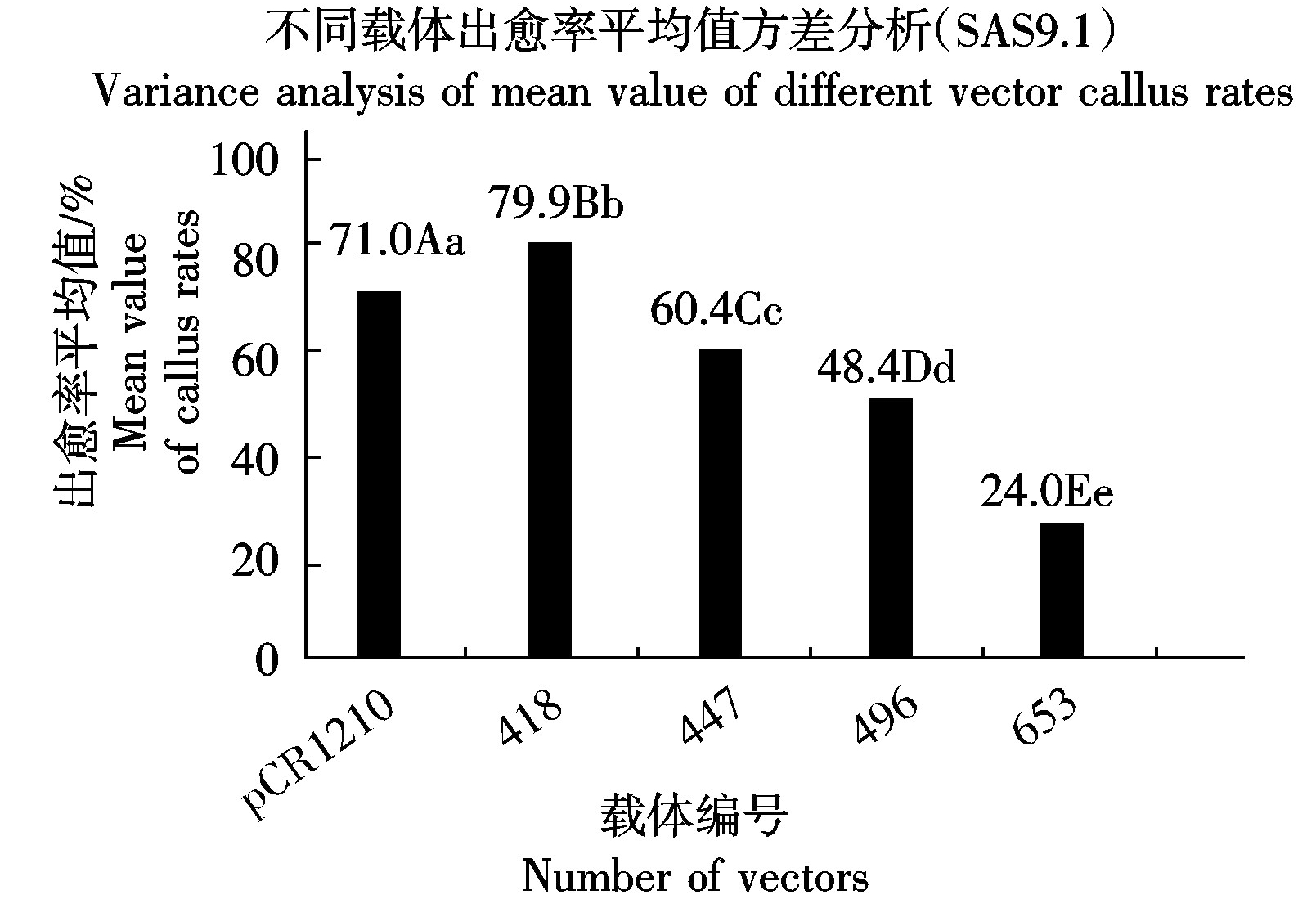
不同小写字母表示0.05水平差异显著(P<0.05);
不同大写字母表示0.01水平差异显著(P<0.01)。
Different lowercase means significant difference (P<0.05);Different capital letters mean significant difference (P<0.01)
图10 干涉后各EST干涉载体转化的愈伤组织诱导率变化
Fig.10 Change of callus induction rate after EST interference vector transformation
2.3 愈伤组织PCR和GFP荧光检测
2.3.1 PCR法检测愈伤组织基因组DNA 提取诱导40 d后的下胚轴外植体愈伤组织的基因组DNA,进行PCR检测,结果如图11所示,在相应位置均出现了目标条带,条带清晰且单一,可以确定4个干涉载体和pCRI210都成功整合进棉花愈伤组织基因组DNA中。
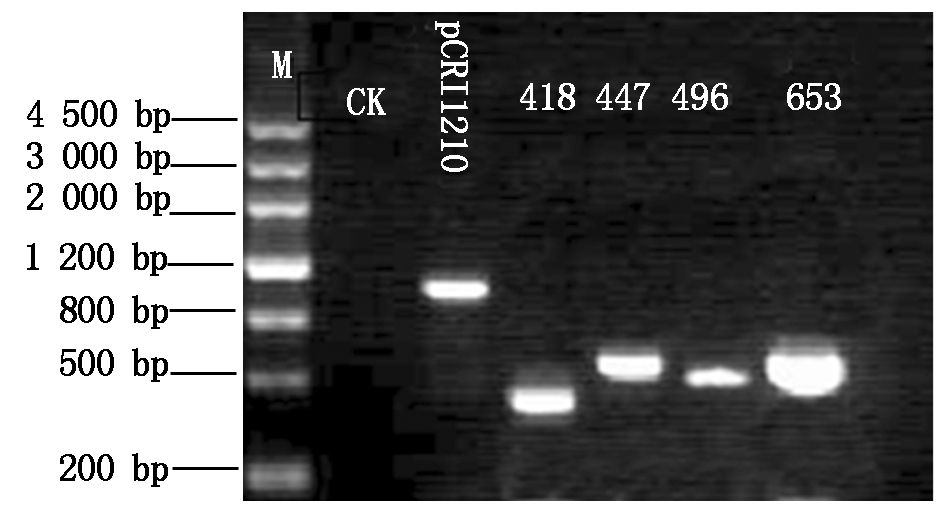
图11 PCR检测愈伤组织基因组DNA
Fig.11 PCR screening of callus genomics DNA
2.3.2 荧光检测下胚轴外植体愈伤组织GFP表达 将诱导40 d的下胚轴外植体愈伤组织,用无菌镊子轻轻夹取少量制成水装片,在荧光显微镜下检测GFP基因蛋白的表达量和分布,检测结果如图12所示。由图12可知,GFP基因成功地整合进棉花愈伤组织基因组DNA中,在细胞核和细胞膜中均得到了高效表达。
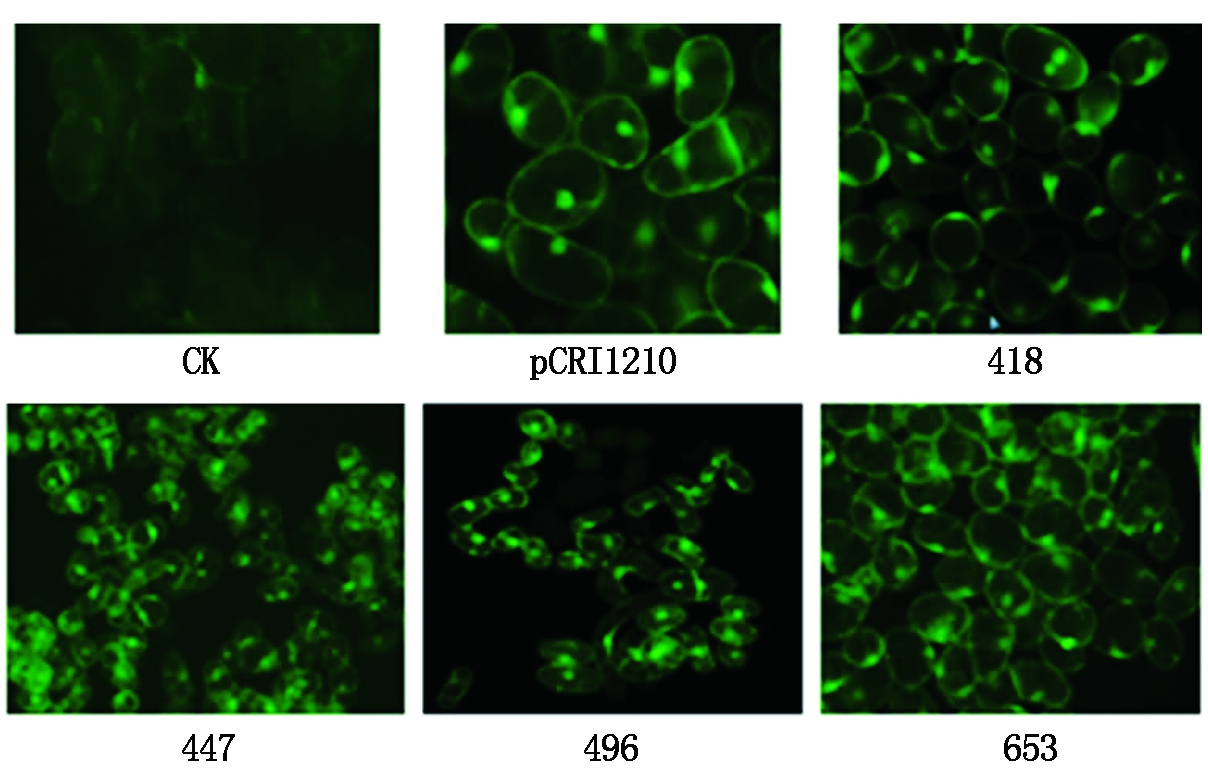
图12 GFP在棉花愈伤组织中的表达
Fig.12 Expression of GFP in cotton callus
2.4 Semi-Quantitative RT-PCR
2.4.1 RNA提取 利用天泽植物RNAout 2.0试剂盒提取诱导40 d的下胚轴外植体愈伤组织的总RNA,利用琼脂糖凝胶电泳分离,结果由图13可知,所提取的RNA条带清晰无弥散现象,可满足试验要求。
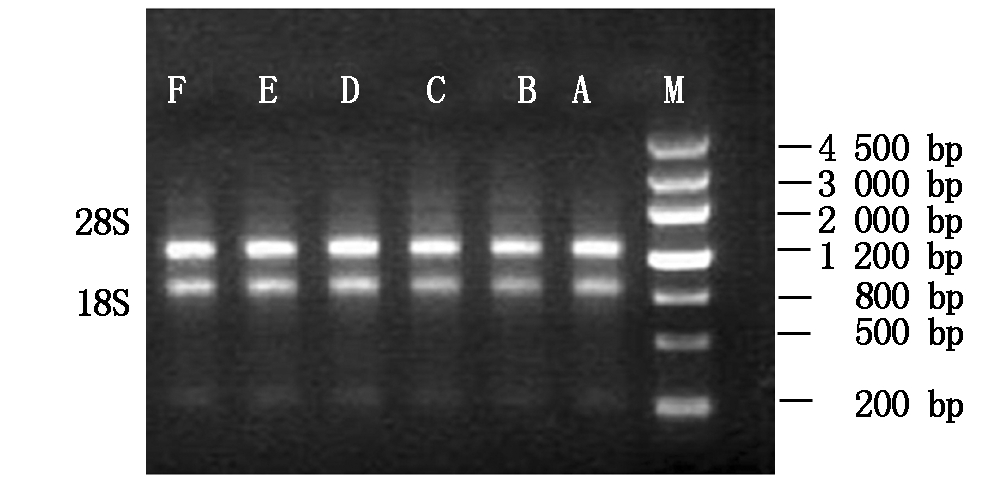
M.DNA Marker Ⅲ; A.野生型;B.pCRI1210;C.pCRI1210-418;D.pCRI1210-447; E.pCRI1210-496;F.pCRI1210-653。
M.DNA Marker Ⅲ;A.Wild type;B.pCRI1210;C.pCRI1210-418;D.pCRI1210-447;E.pCRI1210-496;F.pCRI1210-653.
图13 愈伤组织总RNA
Fig.13 Total RNA of cotton callus
2.4.2 确定Semi-Quantitative RT-PCR指数扩增期 由图14可知,循环数分别为28,30,32的扩增条带中,将扩增数设定为30,扩增产物刚好处于指数扩增期。
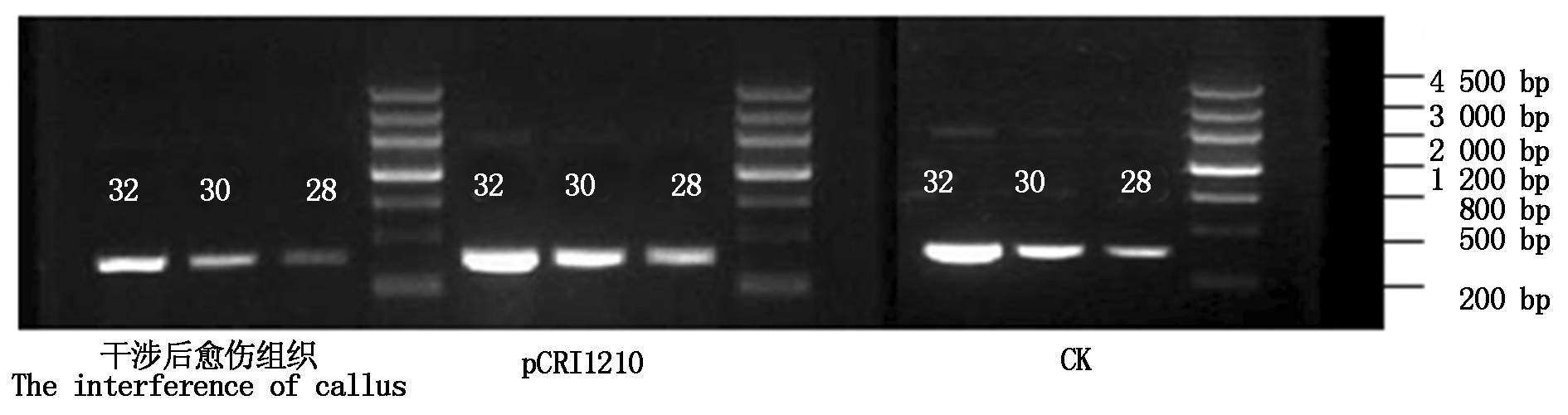
图14 半定量RT-PCR指数扩增期的确定
Fig.14 Identify index of the semi-quantitative RT-PCR amplification
2.4.3 Semi-Quantitative RT-PCR 结果 内参基因Histone3调整后,干涉后愈伤组织分别和野生型、空载体相比。由图15可知,各干涉载体对应的下胚轴外植体愈伤组织中,目标RNA的转录水平均出现了一定程度的下降。而野生型和空载体对照下胚轴外植体RNA的转录量差异不明显。
2.4.4 检测目标RNA的转录效率 采用倍比稀释法检测目标RNA的转录效率。将转化载体pCRI1210的愈伤组织进行RT-PCR扩增,对扩增产物(R1)采用10倍倍比稀释法,与pCRI1210-418干涉后的RT-PCR产物(R2)进行对比,干涉后mRNA的降低量为60%~70%,结果如图16所示。由于半定量RT-PCR的局限性,重复多次试验,结果一致。
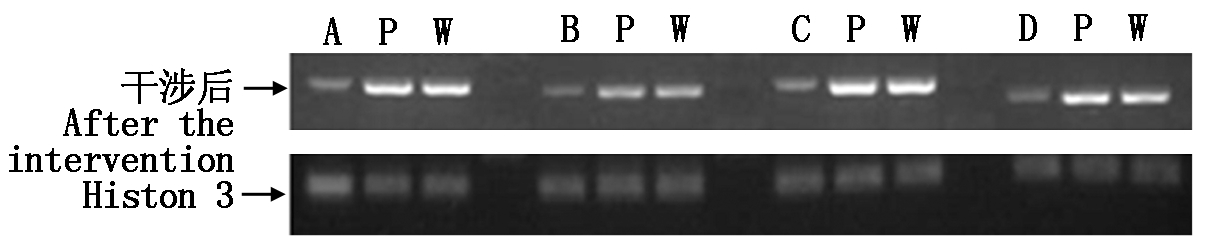
A.干涉后653;B.干涉后496;C.干涉后447;D.干涉后418;P.空载体pCRI1210;W.野生型。
A.After the intervention 653;B.After the intervention 496;C.After the intervention 447;D.After the intervention 418;P.Empty vector pCRI1210;W.Wild type.
图15 愈伤组织干涉前后表达量的差异
Fig.15 The expression before and after interference
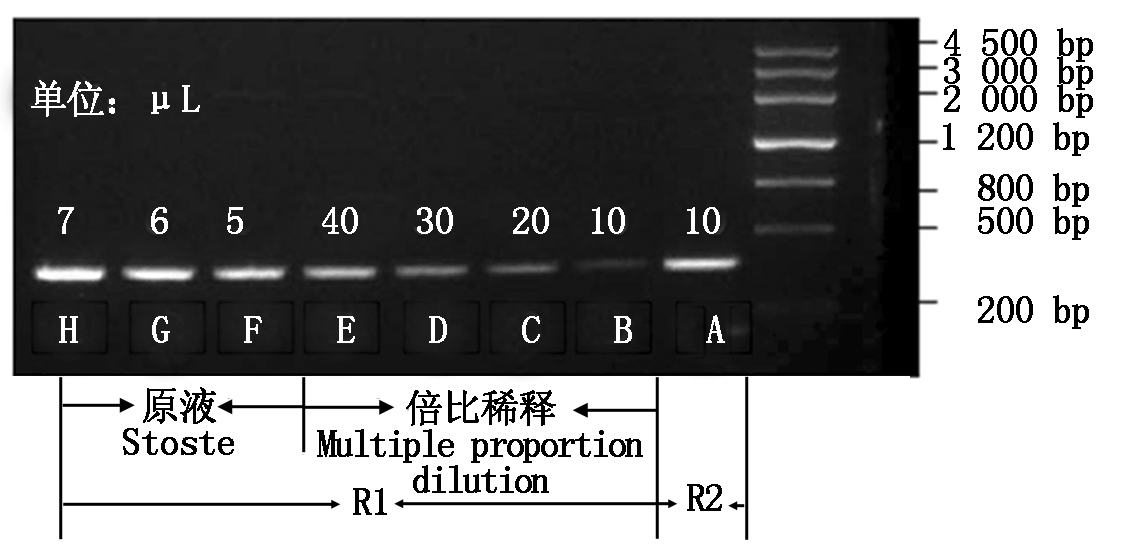
A.10 μL干涉后pCRI1210-418; B.10 μL 空载体pCRI1210;C.20 μL 空载体pCRI1210;D.30 μL 空载体pCRI1210;E.40 μL 空载体pCRI1210;F.5 μL 空载体pCRI1210原液;G.6 μL 空载体pCRI1210原液;H.7 μL 空载体pCRI1210原液。
A.10 μL pCRI1210-418 after interference;B.10 μL empty vector pCRI1210; C.20 μL empty vector pCRI1210 D.30 μL empty vector pCRI1210; E.40 μL empty vector pCRI1210 F.5 μL empty vector pCRI1210 stoste; G.6 μL empty vector pCRI1210 stoste; H.7 μL empty vector pCRI1210 stoste.
图16 估测目标RNA的转录效率
Fig.16 Estimation of transcriptionalefficiency of target RNA
3 讨论
棉花体细胞胚胎发生是一个极其复杂的过程,它既受到细胞内因素的影响,如基因、激素和调控因子等,同时也受到细胞外因素的影响。不同的棉花品种体细胞胚胎的发生能力差异十分明显,有的发生能力高达90%以上,有的发生能力几乎为0,并且同一品种不同种子之间体细胞胚胎的发生也存在一定的差异,这种差异极有可能是由基因型之间的差异造成的[29]。通过本试验可知,不同的调控因子在体细胞胚胎分化过程中发挥着不同的作用。干涉后,EST-418的出愈率提高,而EST-447、EST-496和EST-653的出愈率出现了不同程度的下降。我们大胆预测,EST-418在棉花体细胞胚胎发生过程中起抑制作用,而EST-447、EST-496和EST-653在棉花体细胞胚胎发生过程中起促进作用,SSH文库中必定还存在很多这样的调控因子。从棉花种类来分,再生能力从易到难依次为珂字棉、爱字棉、斯字棉和岱字棉。国内品种中,从易到难依次为黄河流域品种、陕西及河北的品种、长江流域的品种。外植体是影响体细胞胚胎发生的一个重要因素,不同类型的外植体分化得到体细胞胚胎的能力差异很大,其中,下胚轴最容易,子叶较差,真叶和茎最差[30]。也有研究指出,培养基的成分和培养条件(pH值、温度等)以及激素的种类(氮化物、谷氨酰胺、吲哚乙酸、细胞分裂素、2,4-二氯苯氧基乙酸等)都对体细胞胚胎发生具有重要的作用[31-32],其发生途径很好地诠释了细胞的全能性,证明植物体细胞同样具有一套完整的遗传信息。因此,为了更加深入了解细胞分化、发育、形态发生以及合子胚发育的机制,必须进一步研究植物体细胞胚胎发生机制以及真核细胞中基因表达与调控。只有深入了解体细胞胚胎发生的分子机制,将与体细胞胚胎发生相关的基因进行克隆并进行遗传转化,甚至找到棉花体细胞胚胎发生的主效基因,才能在研究棉花体细胞胚胎发生的道路上走得更远,从而使不容易发生体细胞胚胎分化的品种实现分化,创造出更多的优质、特异种质资源供育种家使用。一些无法利用传统手段来解决的问题或许可以通过遗传转化和体细胞胚胎发生的途径来解决。抑制性消减杂交文库的构建提供了大量的差异表达基因,SSR-EST、RNAi等技术的应用找到了验证这些基因的方法[33-36]。相信,随着研究工作的深入以及一些新技术新方法的使用,棉花体细胞胚胎发生的密码将会一步步被破解。
参考文献:
[1] 张朝军,李付广,张 玲.植物体细胞胚胎发生机理的研究进展[J].棉花学报,2008,20(2):141-147.
[2] 张朝军,范术丽,武芝霞,等.棉花大田植株叶柄组织培养体系的建立[J].西北植物学报,2011,31(6):1257-1263.
[3] 焦改丽,孟钊红,杨晓黎,等.光学显微及扫描电镜冷台观察陆地棉胚胎发育[J].山西农业科学,2013,41(11):1180-1182,1196.
[4] 王志才,木合热皮亚·艾尔肯,廖茂森,等.影响棉花体细胞胚胎发生和植株再生的关键因素分析[J].新疆农业科学,2011,48(1):39-44.
[5] 王彦霞,王省芬,马峙英.影响棉花幼胚(珠)离体培养及植株建成的因素分析[J].华北农学报,2006,21(S):37-40.
[6] 张朝军,王 晔,王玉芬,等.棉花叶柄分化率主基因+多基因混合遗传分析[J].棉花学报,2012,24(1):3-9.
[7] 张朝军,王 晔,王玉芬,等.大田棉花真叶叶柄组织培养特性研究[J].棉花学报,2011,23(6):587-591.
[8] 武秀明.陆地棉体细胞胚胎分化过程中特异表达基因的研究[D].北京:中国农业科学院,2008.
[9] 黄冰艳,苗利娟,张新友,等. ahFAD2基因RNA干扰体的删除筛选标记载体构建及其遗传转化[J].华北农学报,2008,23(5):13-16.
[10] 田莉莉,牛 良.GFLV CP基因RNAi载体构建及其在转基因烟草中的干涉效果[J].华北农学报,2014,29(S1):1-5.
[11] 王 钟.玉米黄质环氧化酶基因的转基因RNA干涉[D].雅安:四川农业大学,2013.
[12] 尹富仕,曾洪梅,张雨良,等.棉铃虫HaHR3基因的克隆及其植物RNA干扰载体的构建[J].棉花学报,2010,22(2):157-162.
[13] 宋春丽,马俊莲,唐 霞,等.RNA干扰技术抑制草莓FaEtr2基因表达的研究[J].华北农学报,2014,29(1):28-30.
[14] 龚 磊,王 舰,杨永智.RNA干涉转基因马铃薯对卷叶病毒抗性的研究[J].湖北农业科学,2016,55(8):1994-1997.
[15] 王彦霞,刘正杰,马峙英,等.RNA干涉技术与棉花高油育种[J].棉花学报,2011,23(2):178-183.
[16] 田 迪,刘早利,陈亚红,等.水稻细胞色素 P450 基因家族成员 CYP81A6 基因 RNAi 载体构建[J].分子植物育种,2016,14(2):370-373.
[17] 王更先,司马杨虎,张升祥,等.RNA干扰沉默BmDHS基因对家蚕生长发育的影响研究初探[J].华北农学报,2011,26(2):13-16.
[18] 包崇来,杜黎明,胡天华,等.紫红线茄转基因体系优化及SmARF8基因RNA干涉表达载体的遗传转化[J].园艺学报,2014,41(6):1105-1114.
[19] 李 磊.RNA干涉抗矮花叶病转基因玉米株系后代鉴定[D].雅安:四川农业大学,2013.
[20] 龚 磊.RNA干涉转基因马铃薯抗卷叶病毒(PLRV)抗病性研究[D].西宁:青海大学,2013.
[21] 王金彦,潘丽娟,杨庆利,等.花生EST-SSR分子标记的开发[J].华北农学报,2009,24(S):42-45.
[22] 任建东,李凤珍,许媛君,等.基于EST-SSR分子标记的青海高原以礼草属主要物种的遗传多样性分析[J].植物遗传资源学报,2016,17(4):663-670.
[23] 周 萌,李友勇,孙雪梅,等.基于 EST-SSR 标记的云南大茶树遗传多样性分析[J].华北农学报,2013,28 (S):91-96 .
[24] 胡宏霞,穆国俊,侯名语,等.河北省花生地方品种基于EST-SSR的遗传多样性及性状-标记相关分析[J].植物遗传资源学报,2013,14(6):1118-1123,1129.
[25] 包文风,王吉明,尚建立,等.基于公共数据库的西瓜EST-SSR信息分析与标记开发[J].华北农学报,2011,26(2):85-89.
[26] 盖树鹏,穆 平,张 风,等.休眠解除过程中牡丹花芽均一化酵母杂交文库的构建[J].植物遗传资源学报,2012,13(3):497-501.
[27] 蔡 健,祝 旋,谢实龙,等.利用 EST-SSR 标记和形态性状检测31 个芍药品种的遗多样性[J].华北农学报,2016,31(S):92-96.
[28] 刘佳杰,林清芳,李连国,等.蒙古沙冬青冷冻胁迫SMART cDNA文库的构建及序列分析[J].植物遗传资源学报,2011,12(5):770-774.
[29] 程文翰.棉花(Gossypium hirsutum L.)体细胞胚胎发生的生理及分子机制研究[D].石河子:石河子大学,2016.
[30] 程文翰,朱华国,李鹏飞,等.棉花多胺HPLC的测定方法优化及其在体细胞胚胎发生过程中的变化规律[J].棉花学报,2014,26(2):138-144.
[31] 张云超,杨细燕,何良荣,等.鲁棉6号体细胞胚胎发生过程及植株再生[J].华中农业大学学报,2014,33(2):22-27.
[32] 丁喜莲,曲延英,李 琼,等.六个海岛棉品系体细胞胚胎发生及再生体系的研究[J].生物技术通报,2016,32(1):97-102.
[33] 李雪姣,张 耿,顾爱侠,等.芸薹属作物EST-SNP的发掘与分析[J].植物遗传资源学报,2010,11(6):772-776.
[34] 郭九峰,孙国琴,乔慧蕾.水分胁迫柠条锦鸡儿叶片均一化全长cDNA文库的构建及EST分析[J].华北农学报,2012,27(3):67-71.
[35] 徐洪伟,杜丰平,张 鑫,等.体细胞胚胎发生相关类受体蛋白激酶(SERK)的生物信息学分析[J].吉林师范大学学报:自然科学版,2016,37(1):101-104,107.
[36] 郭家雁,张 霞,丁喜莲,等.不同处理诱导新海16号体细胞胚胎同步化发生[J].棉花学报,2017,29(4):385-392.