JAZ蛋白是茉莉素(JA)信号转导途径的关键组分,扮演着转录调节因子的作用,在植物抵御非生物逆境中发挥着非常重要的调节作用。在拟南芥中,JAZ基因能够被机械损伤、臭氧、高盐等非生物胁迫诱导表达[1-2]。AtJAZ10超表达的拟南芥植株表现出对茉莉素响应不敏感,并降低了由胁迫损伤而导致的生长抑制[3]。水稻所有JAZ基因均受到至少一种非生物胁迫(盐、干旱、低温)诱导表达[4]。大豆GsJAZ2基因在拟南芥中的超量表达,增加了拟南芥对盐碱胁迫的敏感性,即降低了拟南芥对盐碱胁迫的耐性,表明该基因参与并干扰了植物在应对盐胁迫逆境过程中的防御机制[5-6]。大多数葡萄VvJAZ基因均受JA、ABA和环境胁迫(低温、干旱和盐碱)诱导响应,并且除VvJAZ1外的其他冷胁迫响应基因同时也是干旱或者高盐胁迫响应基因[7]。
其他物种中的研究表明,JAZ类基因具有普遍的非生物胁迫诱导表达特性,在植物抵御非生物逆境伤害中可能起着重要的调节作用。在棉花中,有关JAZ基因的研究还相对较少。随着棉花基因组测序的完成,JAZ所属的TIFY家族的全基因组鉴定相继完成[8-10]。Hu等[11]发现,GhJAZ2通过与R2R3-MYB类转录因子GhMYB25-like等互作负调控棉花纤维起始发育。然而,有关棉花JAZ基因抵御逆境胁迫作用机理的研究还有待进一步深入。本研究从棉花品种中棉所36低温转录组中筛选得到了一个低温响应基因GhJAZ1(登录号KJ562212),分析了其生物信息学特征,对其组织表达特征以及激素和低温胁迫下的表达模式进行了研究,并检测了其亚细胞定位特征,为揭示GhJAZ1在棉花低温逆境响应中的作用机制奠定基础。
1 材料和方法
1.1 试验材料
供试陆地棉品种中棉所36由中国农业科学院棉花研究所提供,冀棉958由分子育种课题组种植保存。
1.2 试验方法
1.2.1 胁迫处理方法 中棉所36种子经硫酸脱绒后,直接播种于营养土中,放置在培养箱(白天28 ℃,夜晚26 ℃,湿度70%)中生长。待棉苗长到2片真叶展开时,对棉株进行胁迫处理。
激素胁迫处理:选择生长整齐健壮的棉苗,叶面喷施0.1 mmol/L 脱落酸(ABA)和0.1 mmol/L 茉莉酸甲酯(MeJA)处理0,1,3,6 h。
低温胁迫处理:将生长整齐健壮的棉苗移到4 ℃的培养箱中,处理0,3,6,12,24 h。
处理结束后,迅速剪取植株真叶,置于液氮中速冻后,-80 ℃保存。
1.2.2 GhJAZ1基因克隆 根据分子设计育种实验室中棉所36低温转录组测序获得的JAZ基因序列片段,使用Primer Premier 5.0软件设计该基因的PCR引物(表1)。提取低温处理12 h的中棉所36叶片总RNA,反转录合成第一链cDNA,以第一链cDNA为模板扩增目的基因。扩增反应采用TaKaRa公司的LA Tag酶,反应体系含TaKaRa LA Tag酶(5 U/μL)0.5 μL,10×LA PCR Buffer(Mg2+ Plus)5 μL,dNTP Mixture 8 μL,模板1 μL,上下游引物各1 μL,加ddH2O至终体积50 μL。扩增程序为:94 ℃预变性3 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸60 s,30个循环;最后72 ℃延伸10 min。
PCR产物经1%琼脂糖凝胶电泳回收,连接到pMD18-T(TaKaRa)载体上,转入大肠杆菌感受态细胞DH5α,涂含氨苄青霉素(Ampicillin)的LB琼脂板,37 ℃培养16~20 h,筛选阳性克隆,然后送测序。本试验引物由北京全式金生物技术有限公司合成,DNA聚合酶、载体购自TaKaRa,大肠杆菌DH5α感受态细胞购自天根生化科技有限公司。
1.2.3 GhJAZ1生物信息学分析 利用Clustalx 1.83、MEGA 4.1软件进行多序列比对并构建系统进化树。利用DNAMAN软件进行保守域分析。利用Expasy软件( http://www.expasy.ch/)对蛋白进行理化性质分析。利用NetPhos 2.0 Server(http://www.cbs.dtu.dk/services/Nephos/)和TMHMM(http://www.cbs.dtu.dk/server/TMHMM/)在线分析蛋白磷酸化位点和跨膜域。利用CELLO v.2.5(http://cello.life.nctu.edu.tw/)进行蛋白亚细胞定位预测。利用SOPM(http://npsa-prabi.ibcp.fr/cgi-bin/npsa automat.pl?page=/NPSA/npsa_sopm.html)在线程序对蛋白质序列进行二级结构预测。
1.2.4 GhJAZ1基因表达分析 提取中棉所36不同组织(胚根、子叶、3周龄的根、茎、叶)以及ABA、MeJA和低温处理的叶片总RNA,反转录合成第一链cDNA,以第一链cDNA为模板进行qRT-PCR分析。以陆地棉组蛋白基因Histone3作为内参基因,设计GhJAZ1特异引物GhJAZ1-qRT-F和 GhJAZ1-qRT-R(表1)在Q-TOWER定量PCR仪(Analytik Jena,Germany)上进行实时荧光定量PCR(qRT-PCR)。按照TransStart Green qPCR SuperMix试剂盒推荐的反应体系进行PCR扩增,每个样品进行3次生物学重复,采用2-ΔΔCT法对基因相对表达量进行分析。
表1 所用引物
Tab.1 Primers used in this study

引物 Primer 序列(5'-3') Sequence(5'-3') 用途 Function GhJAZ1-F1CTTCCCGTCATCATCTTTTA基因克隆GhJAZ1-R1ACTCCATTTACCCACTGTCAGhJAZ1-F2CCATGGTAATGTCTAATTTAGGGCAAAAATCTCCTGACGFP融合表达载体构建GhJAZ1-R2ACTAGTGTTGAGATCAATGTCTCTTGGCGTTTCGhJAZ1-qRT-FGCGGACAAGGTGAAGGAG实时荧光定量PCRGhJAZ1-qRT-RAGCGGTTGCAGAGGTGATHistone3-FTCAAGACTGATTTGCGTTTCCA实时荧光定量PCRHistone3-RGCGCAAAGGTTGGTGTCTTC
1.2.5 GhJAZ1亚细胞定位观察 根据GhJAZ1基因的ORF序列和pCAMBIA1302载体的多克隆位点设计引物GhJAZ1-F2和GhJAZ1-R2(表1)。用LA Tag酶扩增获得GhJAZ1的ORF序列,连接到pCAMBIA1302载体上,得GFP:GhJAZ1瞬时表达载体。将该载体利用烟草注射异源表达蛋白的方法注入烟草[12],使用带有GFP滤镜的激光共聚焦显微镜FV10-ASW(购自日本Olympus)在488 nm波长下,观察目的蛋白亚细胞定位情况。
2 结果与分析
2.1 陆地棉GhJAZ1基因全长cDNA的克隆
以中棉所36的叶片cDNA为模板进行PCR扩增,回收目的条带,克隆到pMD18-T载体上。选取3个独立的基因转化子送测序,测序结果完全一致。将测序得到的序列进行BlastX分析及ORF finder预测,最终确定出GhJAZ1的全长CDS序列,大小为810 bp(图1),上传至NCBI(GenBank登录号:KJ562212)。
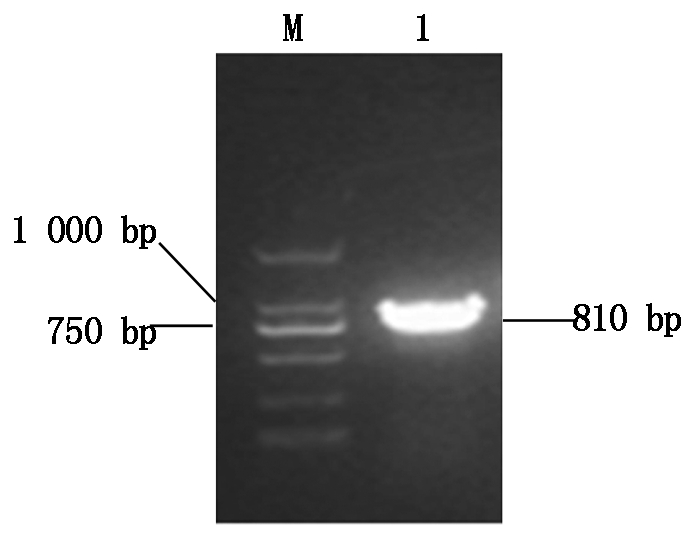
M.DL2000 Marker; 1.GhJAZ1基因。
M.DL2000; 1.GhJAZ1 gene.
图1 陆地棉GhJAZ1基因的克隆
Fig.1 Cloning of GhJAZ1 gene from Gossypium hirsutum L.
2.2 GhJAZ1生物信息学分析
根据在线软件ProtParam的分析结果显示,GhJAZ1蛋白长度为270个氨基酸残基,分子量约29.808 ku,等电点pI约为8.33。CELLO亚细胞定位预测显示,GhJAZ1蛋白定位于细胞核中。Netphos分析GhJAZ1蛋白磷酸化位点发现,整个肽链中3种磷酸化位点都存在,但以丝氨酸磷酸化位点最多,酪氨酸最少(图2)。TMHMM跨膜结构分析显示,GhJAZ1蛋白为非跨膜蛋白。

图2 GhJAZ1蛋白磷酸化位点预测
Fig.2 The predicted phosphorylation sites in GhJAZ1
用SOPM在线程序对GhJAZ1基因编码蛋白进行二级结构预测,结果显示,该蛋白的92个氨基酸可能形成α螺旋,32个氨基酸可能形成延伸带,18个氨基酸可能形成β转角,128个氨基酸可能形成无规则卷曲,组成α螺旋、延伸带、β转角和无规则卷曲的氨基酸比例分别为34.07%,11.85%,6.67%和47.41%。
根据GhJAZ1氨基酸序列在NCBI中BlastP的比对结果,GhJAZ1与可可树(EOY27299.1)、黄麻(OMO66076.1)的JAZ蛋白的相似性较高,分别为77%和76%,与拟南芥(NP_565043.1)的相似性为41%。选取可可树(EOY27299.1)、黄麻(OMO66076.1)、枣(XP_015902663.1)、葡萄(XP_002272363.2)、橡胶(AIY25007.1)、蓖麻(XP_002529200.1)、毛白杨(AKT94819.1)、拟南芥(NP_565043.1)和大豆(NP_001276248.1)9个代表性物种的JAZ氨基酸序列,与陆地棉GhJAZ1进行氨基酸序列多重比对。序列比对结果表明,不同植物JAZ在氨基酸序列上保守性较高,且都具有完整保守的功能结构序列(图3)。GhJAZ1蛋白具有一个N端NT结构域,一个TIFY保守结构域(TIFFGG模式)和一个C端极度保守的Jas结构域,属于TIFY基因家族。

GhJAZ1.棉花KJ562212;TcJAZ.可可树EOY27299.1;CoJAZ.黄麻OMO66076.1;ZjJAZ.枣XP_015902663.1;VvJAZ.葡萄XP_002272363.2;HbJAZ.橡胶AIY25007.1;PtJAZ.毛白杨AKT94819.1;RcJAZ.蓖麻XP_002529200.1;AtJAZ.拟南芥NP_565043.1;GmJAZ.大豆NP_001276248.1。虚线方框.NT结构域;双实线方框.TIFY结构域;单实线方框.Jas结构域。
GhJAZ1.Gossypium KJ562212;TcJAZ.Theobroma cacao EOY27299.1; CoJAZ.Corchorus olitorius OMO66076.1;ZjJAZ.Ziziphus jujube XP_015902663.1;VvJAZ.Vitis vinifera XP_002272363.2;HbJAZ.Hevea brasiliensis AIY25007.1;PtJAZ.Populus tomentosa AKT94819.1;RcJAZ.Ricinus communis XP_002529200.1;AtJAZ.Arabidopsis thaliana NP_565043.1;GmJAZ.Glycine max NP_001276248.1.Dash line box.NT domain; Double solid line box.TIFY domain;Single solid line box.Jas domain.
图3 不同植物JAZ氨基酸序列比较
Fig.3 Alignment of amino acid sequences of JAZs from different plant species
为研究GhJAZ1与其他物种JAZ的进化关系,用ClastalW对氨基酸序列比对后,利用MEGA 5.0软件,采用Neighbor-Joining(NJ)法构建氨基酸序列的进化树(图4),进化树分析发现,棉花GhJAZ1与可可树(EOY27299.1)和黄麻(OMO66076.1)JAZ蛋白进化距离最近,而这3个物种在经典分类学上都属被子植物门双子叶植物纲锦葵目。
2.3 GhJAZ1基因的表达分析研究
为了研究GhJAZ1基因的组织表达特征,以Histone3为内参基因,qRT-PCR检测了该基因在中棉所36萌发3 d的下胚轴和子叶以及3周龄的根、茎、叶中的表达情况。结果表明,该基因不同组织中表达量差异很大,在下胚轴中的表达量最高,茎中最低,其中下胚轴和根中该基因的表达量与子叶相比存在极显著差异(图5)。

图4 GhJAZ1与其他物种同源氨基酸序列的进化树
Fig.4 Phylogenetic tree of GhJAZ1 of G.hirsutum L.and JAZs of other species
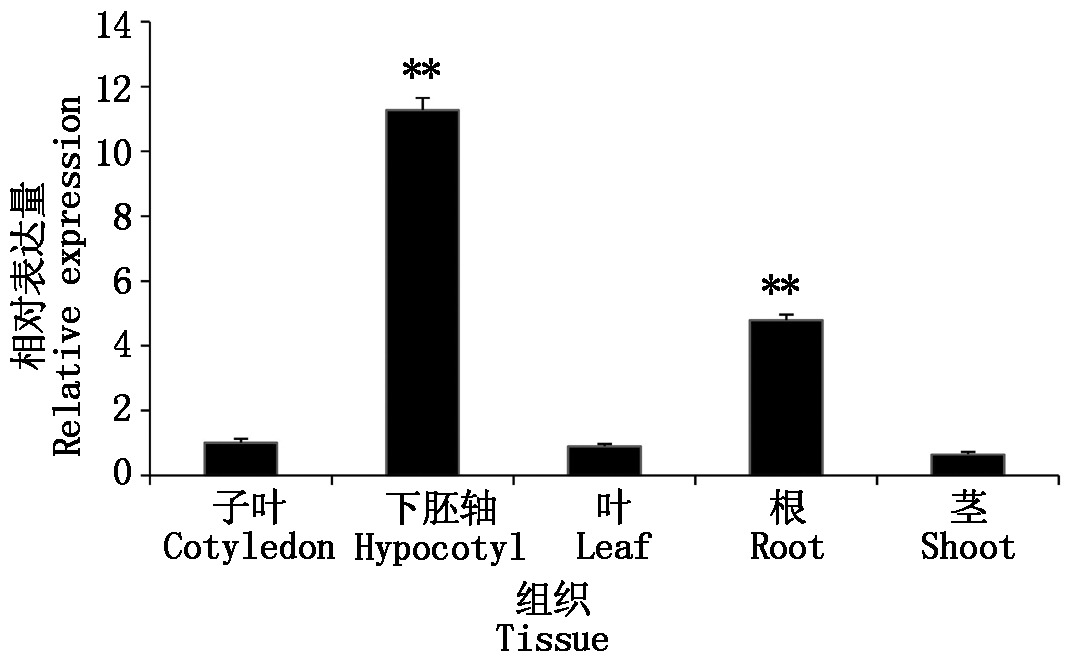
**.样本与对照(子叶)之间存在极显著差异(P<0.01)。
**.Very significant difference between treatment and control(cotyledon) (P<0.01).
图5 GhJAZ1基因的组织表达
Fig.5 Tissue expression pattern of GhJAZ1
为了了解GhJAZ1基因激素响应的表达特征,分别检测了施加0.1 mmol/L ABA和0.1 mmol/L Me-JA处理0,1,3,6 h时中棉所36幼苗叶片中GhJAZ1基因的表达情况(图6)。结果表明,ABA和MeJA处理后该基因的表达量均表现为先升高后降低的趋势,且都在处理1 h后表达量达到最高。不同的是,MeJA处理后GhJAZ1基因表达量急剧上升,1 h即达到对照的20倍。ABA处理后1 h,该基因的表达量仅为对照的2.5倍,处理后6 h,该基因的表达量反而低于对照。
为了进一步研究GhJAZ1的低温胁迫表达特性,以低温耐受能力不同的品种中棉所36和冀棉958幼苗叶片为材料,检测了在4 ℃处理0,3,6,12,24 h时GhJAZ1基因表达情况。从图6可以看出,在耐低温材料中棉所36中,该基因的表达量在受到低温胁迫后迅速升高,12 h时达到最大值,是对照的10倍。冀棉958中,该基因的表达量在低温胁迫6 h时,与对照相比变化不明显,胁迫12 h时表达量也仅是对照的2倍,低温胁迫24 h时表达量最高,为对照的5倍。可见,耐低温品种中棉所36受到低温胁迫后,GhJAZ1基因的上调表达更为显著,说明GhJAZ1基因参与了低温胁迫防御反应。
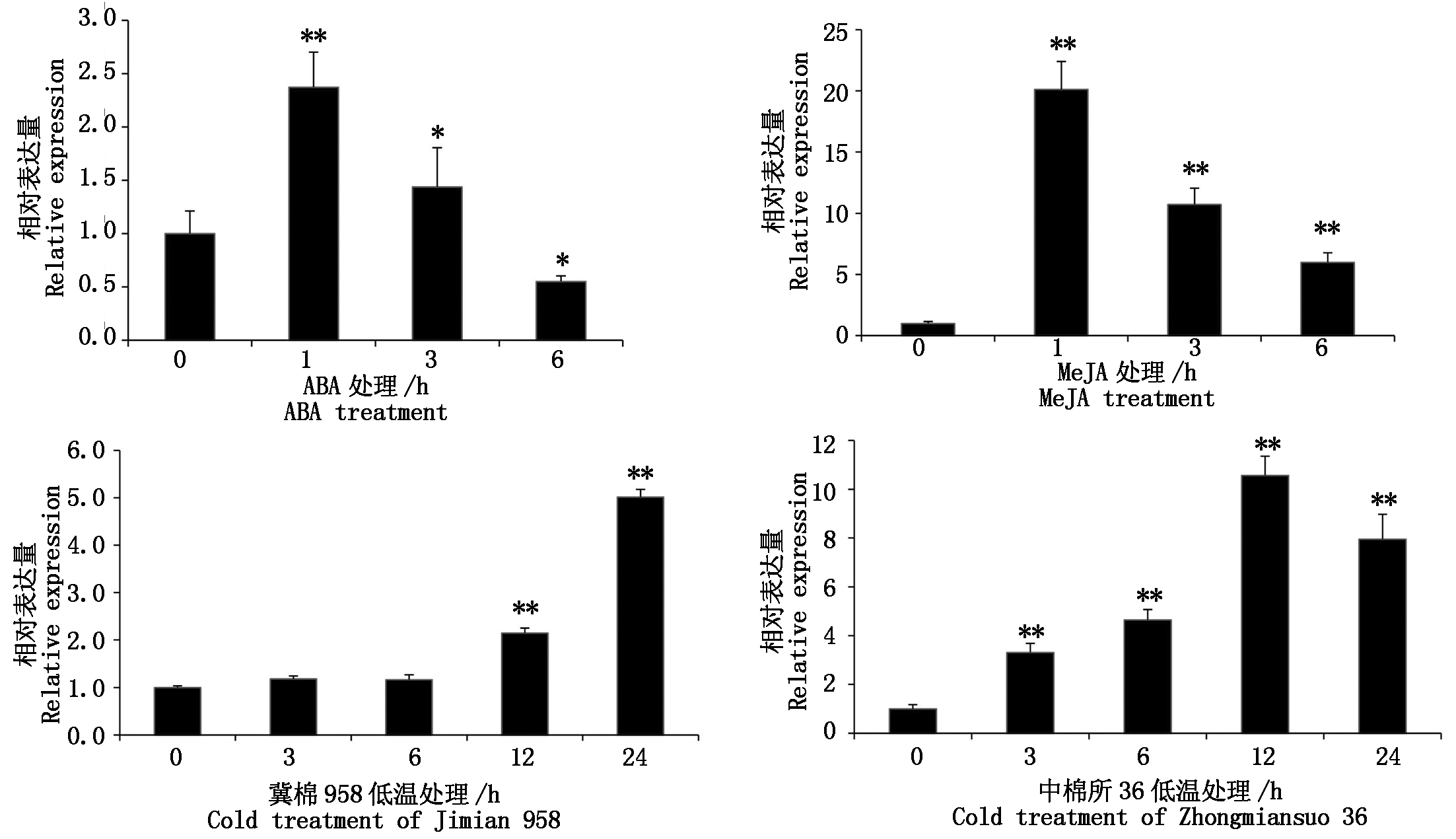
*.处理与对照(0 h)之间存在显著差异(P<0.05);**.处理与对照(0 h)之间存在极显著差异(P<0.01)。
*.Significant difference between treatment and control (0 h) (P <0.05);
**.Very significant difference between treatment and control (0 h) (P<0.01).
图6 GhJAZ1基因ABA、MeJA和低温处理的表达分析
Fig.6 Expression pattern of GhJAZ1 gene after ABA,MeJA and cold treatments
2.4 亚细胞定位
将空载质粒pCAMBIA1302-GFP和融合表达载体pCAMBIA1302-GhJAZ1-GFP注射烟草进行瞬时表达,培养2 d后,在激光共聚焦显微镜下进行观察(图7),从图7可以看出,对照的GFP空载体的绿色荧光在整个细胞都能看到,为组成型表达;GhJAZ1-GFP融合表达蛋白只在细胞核中能看到绿色荧光,表明该蛋白不具有跨膜结构,定位于细胞核内,这与生物信息分析预测的核定位信号的结果一致。
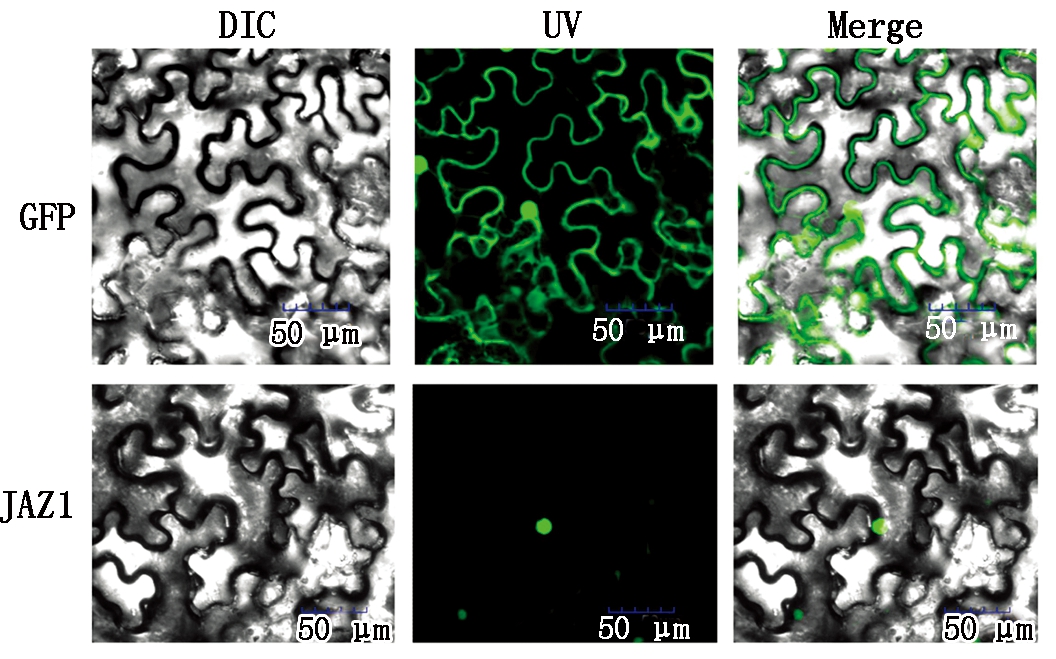
图7 GhJAZ1亚细胞定位
Fig.7 Subcellular localization of GhJAZ1
3 讨论
JAZ蛋白作为E3泛素连接蛋白降解复合体SCFCOI1的靶蛋白,是茉莉素(JA)信号转导途径的关键组分,扮演着转录调节因子的作用[3,13]。已有研究表明,JAZ基因通过与bHLH类、MYC类和ICE1等蛋白互作,在JA信号通路中发挥作用[14-16],参与植物生长发育以及逆境胁迫响应。Hu等[17]报道,拟南芥JAZ1和JAZ4能够通过抑制ICE1对下游基因的转录激活,进而抑制ICE1下游基因CBF3的表达。
棉花的JAZ基因研究还比较少。本研究克隆了陆地棉GhJAZ1基因,该基因属于植物特有的TIFY基因家族,其编码蛋白长度为270个氨基酸残基,分子量约29.808 ku,等电点pI约为8.33,是核定位蛋白。多序列比对表明,陆地棉GhJAZ1与其他植物的JAZ蛋白具有很高的相似性,都具有JAZ蛋白保守的TIFY结构域和Jas结构域。从系统进化分析可知,陆地棉与同是锦葵科的植物可可树、黄麻聚为一类,亲缘关系较近,说明基因保守性很强。
JAZs不含DNA结合结构域,必须依赖蛋白间的互作行使功能[1]。拟南芥的12个JAZ家族成员均含有3个保守结构域,即NT、TIFY(TIF[F/Y]XG)和Jas结构域(SLX2FX2KRX2RX5PY),这3个结构域都可以与其他因子形成二聚体[18-19]。其中,N末端弱保守的NT结构域可与DELLA蛋白互作[18],TIFY结构域可介导不同JAZs之间形成同源或异源二聚体[20],Jas结构域可以同MYC等很多蛋白互作[21]。已有研究表明,JAZ家族基因在植物抵御非生物胁迫中发挥着非常重要的调节作用,但不同JAZ基因参与调节逆境响应的方式可能不同[4,7]。本研究发现,GhJAZ1基因受ABA、MeJA和低温胁迫的诱导表达,但胁迫表达方式存在差异,说明GhJAZ1基因可能在逆境应答中发挥着重要调控作用,但不同逆境下该基因参与调控的信号网络及其调控的互作因子可能不同。Jas结构域中存在细胞核定位信号,使JAZs具有核定位特性[22]。本研究的亚细胞定位结果也证实,GhJAZ1定位于细胞核中。GhJAZ1编码的蛋白在细胞核中可能作为一个转录调节子,通过激活或抑制相关转录因子的活性来调控该转录因子下游基因的表达。
本研究对GhJAZ1基因进行了生物信息学分析和表达分析,可以初步推测GhJAZ1基因在棉花低温胁迫响应中发挥着重要的作用,这为后续研究基因功能和其作用机制奠定了基础。
参考文献:
[1] Vanholme B,Grunewald W,Bateman A,et al.The tify family previously known as ZIM[J].Trends in Plant Science,2007,12(6):239-244.
[2] Jiang Y,Deyholos M K.Comprehensive transcriptional profiling of NaCl-stressed Arabidopsis roots reveals novel classes of responsive genes[J].BMC Plant Biology,2006,6(1):25.
[3] Yan Y,Stolz S,Chételat A,et al.A downstream mediator in the growth repression limb of the jasmonate pathway[J].The Plant Cell,2007,19(8):2470-2483.
[4] Ye H,Du H,Tang N,et al.Identification and expression profiling analysis of TIFY family genes involved in stress and phytohormone responses in rice[J].Plant Molecular Biology,2009,71(3):291-305.
[5] Zhu D,Bai X,Luo X,et al.Identification of wild soybean (Glycine soja) TIFY family genes and their expression profiling analysis under bicarbonate stress[J].Plant Cell Reports,2013,32(2):263-272.
[6] 朱 丹,柏 锡,朱延明,等.野生大豆盐碱胁迫相关GsTIFY11b的克隆与功能分析[J].遗传,2012,34(2):230-239.
[7] Zhang Y,Gao M,Singer S D,et al.Genome-wide identification and analysis of the TIFY gene family in grape[J].PLoS One,2012,7(9):e44465.
[8] Sun Q,Wang G,Zhang X,et al.Genome-wide identification of the TIFY gene family in three cultivated Gossypium species and the expression of JAZ genes[J].Scientific Reports,2017,7:42418.
[9] Zhao G,Song Y,Wang C,et al.Genome-wide identification and functional analysis of the TIFY gene family in response to drought in cotton[J].Molecular Genetics and Genomics,2016,291(6):2173-2187.
[10] He D H,Lei Z P,Tang B S,et al.Identification and analysis of the TIFY gene family in Gossypium raimondii[J].Genetics and Molecular Research,2015,14(3):10119-10138.
[11] Hu H,He X,Tu L,et al.GhJAZ2 negatively regulates cotton fiber initiation by interacting with the R2R3-MYB transcription factor GhMYB25-like[J].The Plant Journal :for Cell and Molecular Biology,2016,88(6):921-935.
[12] Jia H G,Pang Y Q,Fang R X.Agroinoculation as a simple way to deliver a tobacco Mosaic virus based expression vector[J].Acta Bot Sin,2003,45(7):770-773.
[13] Thines B,Katsir L,Melotto M,et al.JAZ repressor proteins are targets of the SCF(COI1) complex during jasmonate signalling[J].Nature,2007,448(7154):661-665.
[14] Fonseca S,Fernández-Calvo P,Fernández G M,et al.bHLH003,bHLH013 and bHLH017 are new targets of JAZ repressors negatively regulating JA responses[J].PLoS One,2014,9(1):e86182.
[15] Goossens J,Swinnen G,Vanden Bossche R,et al.Change of a conserved amino acid in the MYC2 and MYC3 transcription factors leads to release of JAZ repression and increased activity[J].The New Phytologist,2015,206(4):1229-1237.
[16] Chini A,Fonseca S,Fernández G,et al.The JAZ family of repressors is the missing link in jasmonate signalling[J].Nature,2007,448(7154):666-671.
[17] Hu Y,Jiang L,Wang F,et al.Jasmonate regulates the inducer of cbf expression-C-repeat binding factor/DRE binding factor1 cascade and freezing tolerance in Arabidopsis[J].The Plant Cell,2013,25(8):2907-2924.
[18] Kazan K,Manners J M.JAZ repressors and the orchestration of phytohormone crosstalk[J].Trends in Plant Science,2012,17(1):22-31.
[19] 晏胜伟,孙 程,周晓今,等.玉米JAZ家族基因ZmJAZ4的克隆及功能分析[J].生物技术通报,2015,31(3):96-101.
[20] Chung H S,Howe G A.A critical role for the TIFY motif in repression of jasmonate signaling by a stabilized splice variant of the JASMONATE ZIM-domain protein JAZ10 in Arabidopsis[J].The Plant Cell,2009,21(1):131-145.
[21] 孙 程,周晓今,陈茹梅,等.植物JAZ蛋白的功能概述[J].生物技术通报,2014(6):1-8.
[22] Grunewald W,Vanholme B,Pauwels L,et al.Expression of the Arabidopsis jasmonate signalling repressor JAZ1/TIFY10A is stimulated by auxin[J].EMBO Reports,2009,10(8):923-928.