牛和羊等反刍动物以前肠发酵的方式从它们的食物中获取营养物质[1]。这种以细菌进行的前肠发酵,其产生的短链脂肪酸被胃壁吸收,为反刍动物提供能量。为了从这些微生物群体中回收必需的营养物质,反刍动物必须破坏细菌和其他微生物细胞,释放内容物,之后胃中的胃消化酶从内容物中提取营养物质[2]。细菌对于哺乳动物的消化酶普遍具有抗性,因此反刍动物募集一种抗细菌的溶菌酶来裂解这些前肠微生物[3]。
溶菌酶(Lysozyme,LYZ)是一种抑菌蛋白,它分布于生物体多种体液和组织中,包括禽类蛋清、动物分泌物(如人乳和眼泪)和多形核白细胞分泌物[4]。LYZ全称为1,4-O-溶菌酶,是一种低分子量的基础蛋白,它通过裂解细胞壁肽聚糖中N-乙酰氨基葡萄糖和N-乙酰胞壁酸之间的β-1,4-糖苷键直接溶解革兰氏阳性细菌,在分泌型免疫球蛋白A和补体的参与下,也可以间接溶解革兰氏阴性细菌[5-6]。动物LYZ分为3种:C型、G型和I型,其中溶菌酶C是唯一存在于脊椎动物、原索动物和无脊椎动物的类型,也是目前研究最深入的类型[7]。
迄今,在反刍动物中已经鉴定出14种LYZ c基因,但只有9个LYZ c基因可以编码出相应的功能蛋白,它们之间具有74.8%~97.5%的同源性[8]。胃型LYZ c基因是溶菌酶c基因家族的重要成员,它们在反刍动物的胃中高表达。普通牛胃型LYZ c基因共有4个外显子和3个内含子,编码区(Coding sequence,CDS)序列全长为444 bp,编码147个氨基酸,4个外显子对应的编码区大小分别为136,165,76,67 bp。胃型和非胃型LYZ c氨基酸序列虽然相似,但胃型LYZ c发生了适应性进化,等电点变低,能适应胃中的酸性环境,其作为反刍动物胃中的一种消化酶分解微生物,释放营养物质,最后被宿主胃壁吸收。
水牛是热带和亚热带地区重要的家养动物,在农业生产中扮演重要角色,具有役用性能好、乳质佳、耐粗饲,且饲料转化率高等特点[9]。胃型LYZ c在反刍动物饲料的转化中发挥重要的作用,但有关水牛胃型LYZ c基因的研究较少。本研究以河流型和沼泽型水牛为研究对象,对两类水牛胃型LYZ c基因编码区序列进行了变异检测和群体遗传学分析,并结合已发表的牛科物种的同源序列进行了比较基因组学和生物信息学分析,以揭示水牛胃型LYZ c基因的遗传特征及其与其他牛科物种的差异。
1 材料和方法
1.1 试验材料
本研究总样本量为224份,其中槟榔江水牛(河流型)66份,沼泽型水牛158份(包括德昌水牛65份,德宏水牛11份,盐津水牛29份,东流水牛9份,福安水牛17份,湖南滨湖水牛15份和江西滨湖水牛12份)。样品均为耳组织样,个体间不存在直接血缘关系,样品保存于酒精中低温带回实验室,置于-80 ℃冰箱保存。
1.2 基因组DNA提取
采用酚/氯仿法提取基因组DNA,经琼脂糖凝胶电泳和紫外分光光度法检测其纯度和浓度,TE缓冲液稀释为50 ng/μL,保存于4 ℃备用。
1.3 PCR引物设计和PCR扩增及测序
根据水牛胃型LYZ c基因全序列(GenBank登录号为NW_005785126)使用Primer Premier 5软件设计引物(表1),分别扩增该基因的4个外显子及其旁侧序列。
PCR反应体系为25 μL,包含ddH2O 19.2 μL,10×Buffer(Mg2+)2.5 μL,上、下游引物各0.4 μL(10 μmol/L),dNTP 0.2 μL(2.5 mmol/L),Taq酶(5 U/μL)0.3 μL,DNA模板(50 ng/μL)2.0 μL。PCR反应程序为:94 ℃预变性3 min→35个循环(94 ℃变性30 s→退火40 s→72 ℃延伸30 s)→72 ℃后延伸5 min终止反应。退火温度与延伸时间依引物而异(表1)。PCR产物以2%琼脂糖凝胶电泳检测。产物经胶回收纯化后,进行双向测序,测序引物与PCR引物相同。
表1 胃型LYZ c基因分段扩增引物信息
Tab.1 Primer information for segmented amplification of stomach LYZ c gene
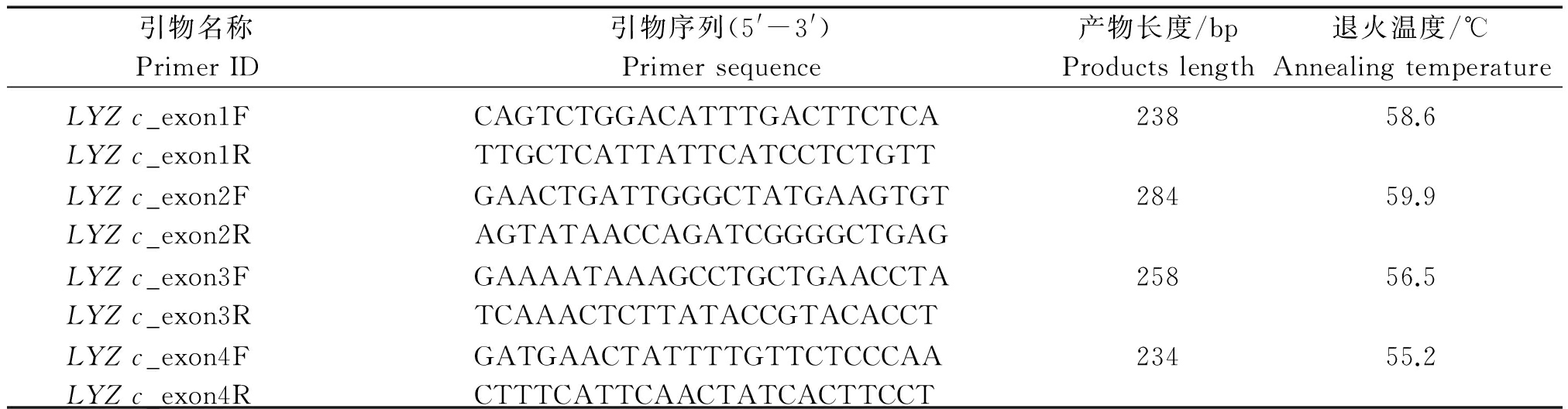
引物名称PrimerID引物序列(5'-3')Primersequence产物长度/bpProductslength退火温度/℃AnnealingtemperatureLYZc_exon1FLYZc_exon1RCAGTCTGGACATTTGACTTCTCATTGCTCATTATTCATCCTCTGTT23858.6LYZc_exon2FLYZc_exon2RGAACTGATTGGGCTATGAAGTGTAGTATAACCAGATCGGGGCTGAG28459.9LYZc_exon3FLYZc_exon3RGAAAATAAAGCCTGCTGAACCTATCAAACTCTTATACCGTACACCT25856.5LYZc_exon4FLYZc_exon4RGATGAACTATTTTGTTCTCCCAACTTTCATTCAACTATCACTTCCT23455.2
1.4 数据分析
1.4.1 核苷酸序列及变异分析 采用上述引物分别对胃型LYZ c基因4个外显子及旁侧序列进行扩增和测序,测序结果采用DNAstar软件包(DNAstar Inc.)中SeqMan 程序进行序列核对和组装,得到胃型LYZ c基因完整外显子拼接序列,拼接好的序列经ORF Finder程序(http://www.ncbi.nlm.gov/gorf/)确定开放阅读框(Open reading frame,ORF),并翻译成蛋白序列。编码区SNP位点的数量、位置由SeqMan、Mega 7软件确定和输出[10],各SNP位点的群体遗传组成用PopGen 32软件(version 1.31)计算,单倍型划分用Phase v2.1软件进行。采用PANTHER蛋白分析软件(http://www.pantherdb.org/)预测氨基酸替换对其功能的影响。
1.4.2 蛋白质理化特性及结构分析 水牛LYZ c相对分子质量和等电点、疏水性、跨膜区和信号肽采用在线软件Compute pI/Mw tool、ProtParam tool、ProtScale、TMHMM server 2.0和SignalP 4.1 Server进行预测分析;采用ProtComp 9.0程序进行LYZ c蛋白的亚细胞定位;通过NCBI数据库预测蛋白质的保守结构域;基于同源建模法利用在线服务器SWISS-MODEL预测LYZ c蛋白的三维结构。
2 结果与分析
2.1 外显子序列拼接及群体变异分析
采用PCR产物直接测序技术对胃型LYZ c的4个外显子及其旁侧序列进行测序,共得到1 014 bp的序列,由ORF Finder程序中确定其编码区序列。将获得的编码区序列提交到NCBI数据库(http://www.ncbi.nlm.nih.gov/)进行同源比对,确定为水牛胃型LYZ c基因序列。
在水牛胃型LYZ c基因编码区中共发现6个SNP位点,分别为c.87G>T、c.228G>A、c.396C>T、c.423C>T、c.424G>A和c.438C>G,其中SNP87仅存在于河流型水牛中,其他5个SNP为河流型和沼泽型水牛共享。在河流型水牛中发现的SNP87位点,G等位基因趋于纯合,处于Hardy-Weinberg平衡状态。水牛中检测到的5个共享SNP位点在两类水牛中均以杂合状态存在,等位基因频率相一致,且均处于Hardy-Weinberg不平衡状态;这些位点除SNP228外,其他4个SNP均位于第4外显子中。各SNP位点的群体遗传分析结果见表2。
表2 水牛胃型LYZ c基因的群体变异信息
Tab.2 The variation information of buffalo stomach LYZ c gene
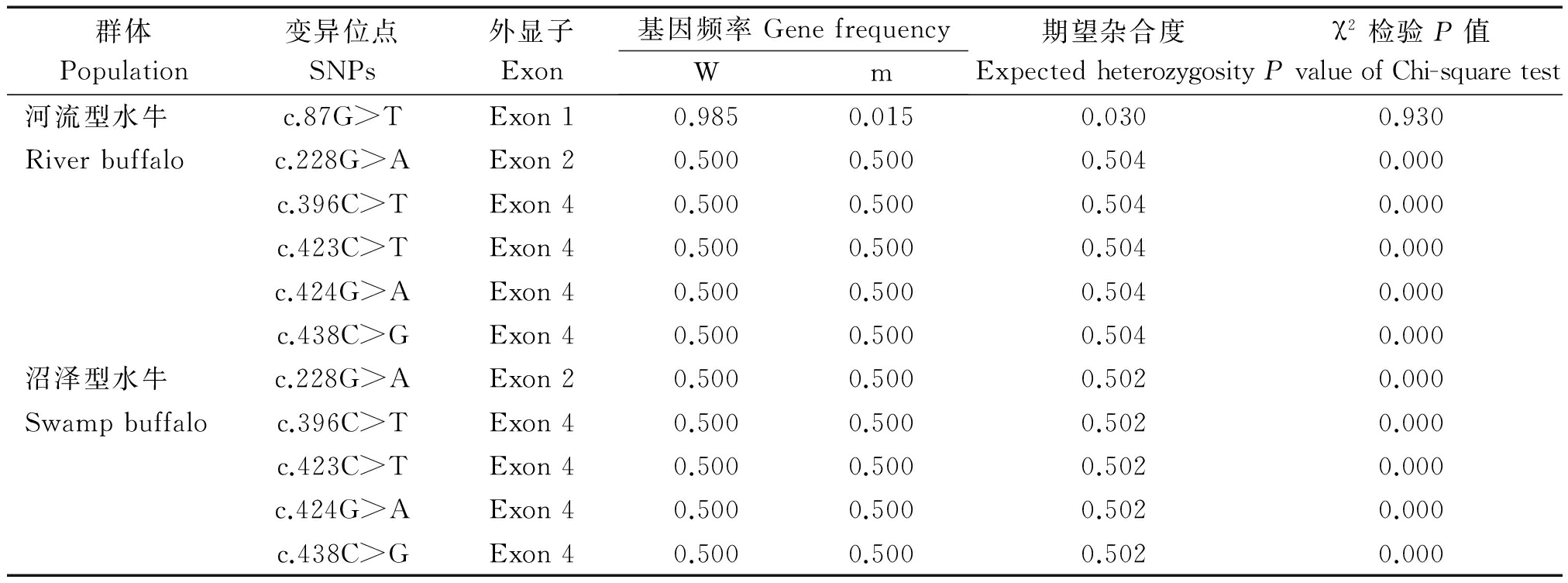
群体Population变异位点SNPs外显子Exon基因频率GenefrequencyWm期望杂合度Expectedheterozygosityχ2检验P值PvalueofChi-squaretest河流型水牛c.87G>TExon10.9850.0150.0300.930Riverbuffaloc.228G>AExon20.5000.5000.5040.000c.396C>TExon40.5000.5000.5040.000c.423C>TExon40.5000.5000.5040.000c.424G>AExon40.5000.5000.5040.000c.438C>GExon40.5000.5000.5040.000沼泽型水牛c.228G>AExon20.5000.5000.5020.000Swampbuffaloc.396C>TExon40.5000.5000.5020.000c.423C>TExon40.5000.5000.5020.000c.424G>AExon40.5000.5000.5020.000c.438C>GExon40.5000.5000.5020.000
注:W.野生型等位基因;m.突变型等位基因;P.Hardy-weinberg平衡检验概率。
Note:W.Wild-type allele;m.Mutant-type allele;P value.The probability of Hardy-weinberg equilibrium.
2.2 单倍型划分及比较基因组学分析
基于本研究水牛序列SNP位点信息,采用Phase v2.1软件进行两类水牛单倍型划分,划分出的单倍型及其频率等信息见表3。为了揭示水牛LYZ c基因的遗传特征,采用水牛LYZ c单倍型序列作为查询序列在NCBI数据库中进行同源搜索,获得以下牛科物种的胃型LYZ c基因序列进行比较分析:水牛序列1条,为:NM_001290888;普通牛序列10条,分别为:BG937730、BG938401、BG937779、BG938044、CK971540、DY056864、M26241、BG937795、M26244和BG937506;瘤牛序列1条:XM_019960836;牦牛序列1条:XM_005900300;野牛序列1条:XM_010841550;山羊序列1条:KC954667(图1)。在河流型水牛中共确定5种单倍型,即River buffalo_hap1~hap5,其中River buffalo_hap1~hap4为本研究数据确定的单倍型;River buffalo_hap1和River buffalo_hap2的期望频率高,为河流型水牛群体优势单倍型。在沼泽型水牛群体内共确定2种单倍型,即Swamp buffalo_hap1~hap2,均为本研究数据确定的单倍型,且它们的期望频率相同。河流型与沼泽型水牛间未发现共享单倍型。对已发表的其他牛科物种胃型LYZ c数据进行分析,在普通牛中确定10种单倍型,即Cattle_hap1~hap10;在瘤牛、牦牛、野牛和山羊中均确定1种单倍型,分别为Zebu_hap1、Yak_hap1、Bison_hap1和Goat_hap1。
表3 由本研究数据确定的水牛LYZ c基因各单倍型信息
Tab.3 The haplotypes information of buffalo LYZ c based on the data in this study
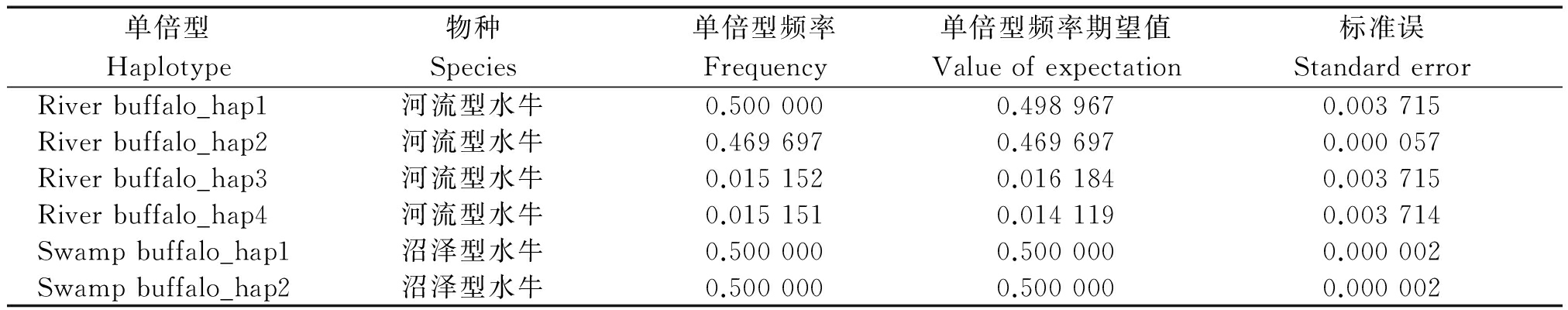
单倍型Haplotype物种Species单倍型频率Frequency单倍型频率期望值Valueofexpectation标准误StandarderrorRiverbuffalo_hap1河流型水牛0.5000000.4989670.003715Riverbuffalo_hap2河流型水牛0.4696970.4696970.000057Riverbuffalo_hap3河流型水牛0.0151520.0161840.003715Riverbuffalo_hap4河流型水牛0.0151510.0141190.003714Swampbuffalo_hap1沼泽型水牛0.5000000.5000000.000002Swampbuffalo_hap2沼泽型水牛0.5000000.5000000.000002
各物种LYZ c核苷酸、氨基酸序列比对结果见图1,2。各牛科物种胃型LYZ c基因序列长度相同,均为444 bp,编码147个氨基酸。将本研究数据与已发表的序列数据合并后,水牛的SNP位点数增加了3个,分别为c.196G>A、c.336G>C和c.351T>C。新增加的SNP位点均只存在于河流型水牛中,因此,共有4个SNP位点只存在于河流型水牛中(SNP87、SNP196、SNP336和SNP351)。水牛中异义替换为2个,分别为p.G66S和p.V142I,对应的SNP位点为SNP196和SNP424(图2)。经采用PANTHER软件预测表明,水牛中发现的p.G66S和p.V142I替换对胃型LYZ c功能没有影响。水牛与其他牛科物种的种间核苷酸差异位点为c.42、c.132和c.348,但只有c.348相应的编码氨基酸与其他牛科物种的不同(水牛:p.116Q;其他牛科物种:p.116H)。序列比对还揭示普通牛和瘤牛特有的核苷酸为c.13G和c.270T(图1,2)。
由水牛胃型LYZ c基因7种单倍型确定出3种蛋白变异体,即Buffalo 1~Buffalo 3。变异体Buffalo 1对应水牛LYZ c基因River buffalo_hap1、River buffalo_hap3、River buffalo_hap4、Swamp buffalo_hap2单倍型;而变异体Buffalo 2则对应River buffalo_hap2、Swamp buffalo_hap1单倍型、变异体Buffalo 3则对应River buffalo_hap5单倍型。变异体Buffalo 1在水牛群体中出现频率最高。由普通牛、瘤牛、牦牛、野牛和山羊LYZ c基因单倍型分别各确定了4,1,1,1和1种变异体。水牛群体中发现有特异的氨基酸替换p.V142I。所有物种在第66位均出现Ser残基。普通牛和瘤牛具有特异的氨基酸p.5V。各牛科物种间胃型LYZ c核苷酸序列差异位点较多,但其编码蛋白氨基酸序列差异较小(图1,2)。

图1 牛科物种胃型LYZ c基因单倍型序列核酸差异
Fig.1 Nucleotide differences of haplotypes detected in stomach LYZ c among Bovidae species

图2 牛科物种胃型LYZ c变异体氨基酸差异
Fig.2 Amino acid sequence differences among the variants of stomach LYZ c in Bovidae
2.3 水牛LYZ c变异体序列结构分析
水牛群体中的3种胃型LYZ c蛋白变异体的序列比较见图3。水牛胃型LYZ c 3种变异体均具有N端的信号肽,切割位点位于第18和第19氨基酸残基,其成熟肽包含129个氨基酸。生物信息学分析显示,水牛3种胃型LYZ c变异体都含有1个属于类溶菌酶超家族的LYZ1保守结构域,位于第19-145氨基酸残基处,它们均不具有跨膜结构,为分泌到胞外发挥功能的亲水蛋白。通过靶模板比对,构建水牛胃型LYZ c三维结构同源模型(图4)。结果表明,水牛胃型3种LYZ c变异体成熟肽与普通牛胃型Lysozyme C-2成熟肽氨基酸序列长度相同,一致性分别为98.45%,97.67%和98.45%。

阴影表示信号肽序列;下划线表示保守结构域。
Signal peptide sequence is highlighted as shaded region;The region underlined indicates the putative conserved domain.
图3 水牛胃型LYZ c变异体氨基酸序列及其对应的核苷酸序列
Fig.3 Amino acid sequences of buffalo stomach LYZ c variants and its corresponding nucleotide sequences
2.4 LYZ c蛋白序列差异分析
将水牛胃型LYZ c变异体氨基酸序列与已报道的普通牛胃型(S1、S2和S3)和非胃型(牛奶、肾、气管和小肠)LYZ c氨基酸序列(Ensembl database ID:ENSBTAG00000011941、ENSBTAG00000026779、ENSBTAG00000000198、ENSBTAG00000026323、ENSBTAG00000046511、ENSBTAG00000026088和ENSBTAG00000046628)进行比较[8],结果见图5。水牛胃型LYZ c变异体与普通牛3种胃型LYZ c氨基酸序列一致性较高,与非胃型LYZ c序列差异较大。水牛与普通牛胃型LYZ c在氨基酸序列上的差异主要体现在第116,144位氨基酸的组成上,这2个位点对LYZ c蛋白的等电点影响较大。水牛3种LYZ c变异体第116,144位氨基酸与普通牛S1蛋白一致,分别为Gln(PI=5.65)和Glu(PI=3.22),而普通牛S2蛋白在这2个位点分别为His和Glu,普通牛S3则分别为His(PI=7.59)和Gln。相比于普通牛非胃型LYZ c氨基酸序列,水牛与普通牛胃型LYZ c蛋白一样,缺失了非胃型LYZ c蛋白第120-121位氨基酸之间的Asp-Pro肽键。
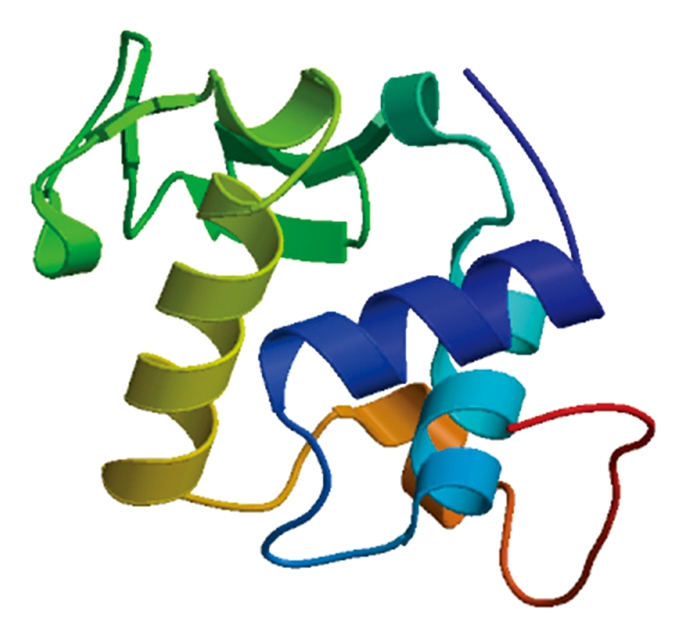
图4 水牛胃型LYZ c变异体三级结构
Fig.4 The tertiary structure of buffalo stomach LYZ c protein
水牛胃型与普通牛的胃型、非胃型LYZ c蛋白理化特性的比较见表4。胃型LYZ c蛋白的等电点较低,偏向于酸性,而非胃型LYZ c蛋白偏向于碱性。胃型LYZ c蛋白含有的正电荷和负电荷的氨基酸残基数基本相同,而非胃型LYZ c蛋白含有正电荷的氨基酸残基数普遍大于负电荷的氨基酸残基。水牛和普通牛胃型LYZ c蛋白的理化特性差异较小,但水牛胃型LYZ c蛋白的等电点更低。所有LYZ c蛋白变异体均属于稳定的亲水蛋白。

图5 LYZ c氨基酸序列比对
Fig.5 Alignment of LYZ c amino acid sequences
表4 牛科物种LYZ c蛋白理化特性差异
Tab.4 The physical and chemical properties differences in Bovidae LYZ c protein
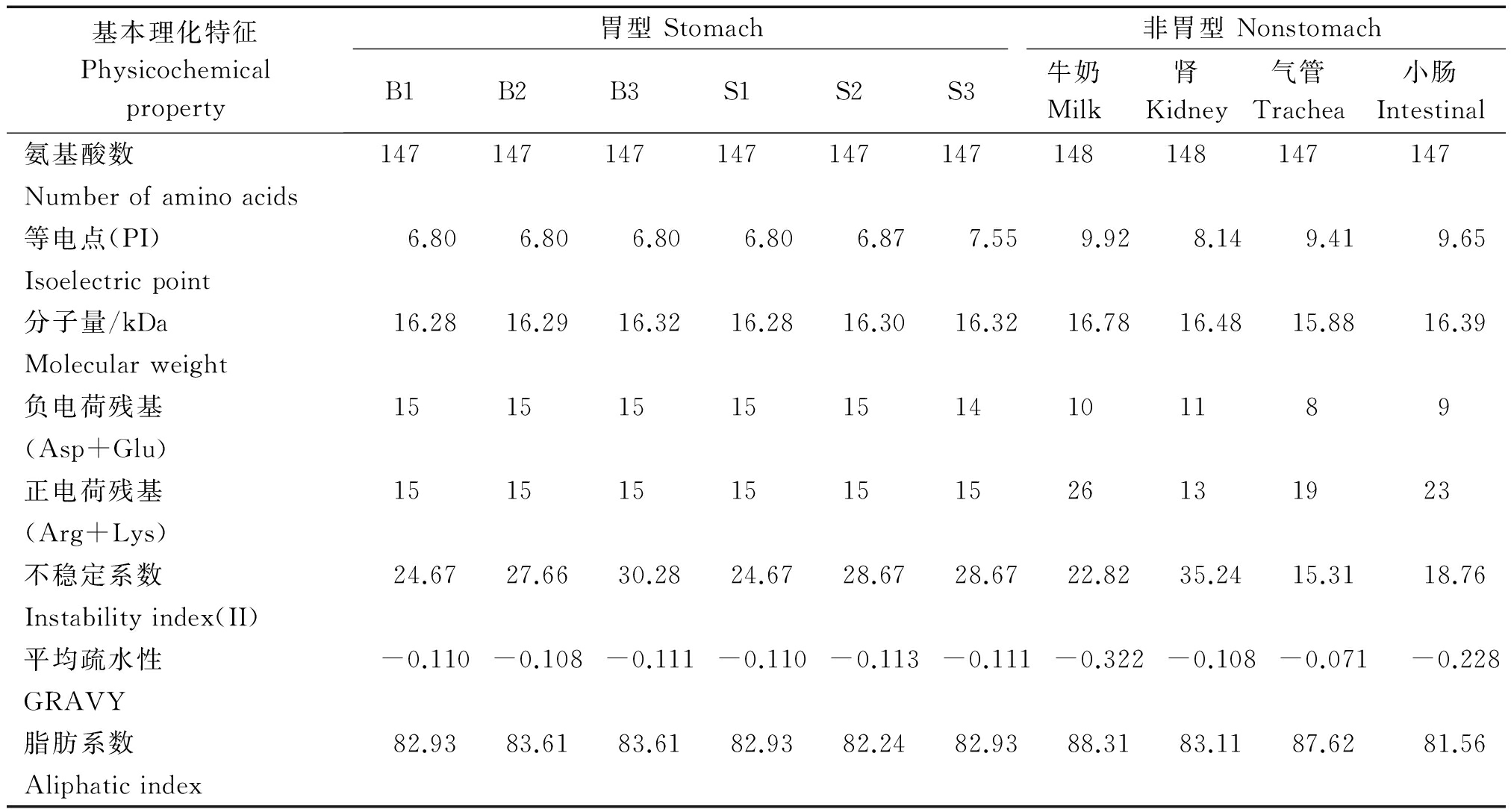
基本理化特征Physicochemicalproperty胃型Stomach非胃型NonstomachB1B2B3S1S2S3牛奶Milk肾Kidney气管Trachea小肠Intestinal氨基酸数147147147147147147148148147147Numberofaminoacids等电点(PI)6.806.806.806.806.877.559.928.149.419.65Isoelectricpoint分子量/kDa16.2816.2916.3216.2816.3016.3216.7816.4815.8816.39Molecularweight负电荷残基151515151514101189(Asp+Glu)正电荷残基15151515151526131923(Arg+Lys)不稳定系数24.6727.6630.2824.6728.6728.6722.8235.2415.3118.76Instabilityindex(II)平均疏水性-0.110-0.108-0.111-0.110-0.113-0.111-0.322-0.108-0.071-0.228GRAVY脂肪系数82.9383.6183.6182.9382.2482.9388.3183.1187.6281.56Aliphaticindex
注:B1~B3.水牛胃型LYZ c变异体;S1~S3.普通牛胃型LYZ c蛋白。
Note:B1-B3.Buffalo stomach LYZ c variants;S1-S3.Cattle stomach LYZ c proteins.
3 讨论与结论
本研究通过分段测序,获得了水牛胃型LYZ c基因的编码区序列。水牛胃型LYZ c基因编码区长为444 bp,编码蛋白具有一个18肽的N端信号肽,成熟肽包含129个氨基酸。在水牛胃型LYZ c基因编码区共发现9个SNP位点,有4个存在于外显子4中。河流型与沼泽型水牛共享5个SNP位点,共享的SNP位点具有相同的群体遗传特征。分析表明,有2个SNP位点导致了氨基酸的替换,但它们对LYZ c蛋白的功能没有影响。本研究确定胃型LYZ c基因的c.42、c.132和c.348位核苷酸为水牛与其他牛科物种相互区分的核苷酸位点,在蛋白水平上只有第348位相应的编码氨基酸与其他牛科物种不同,这些核苷酸位点可以作为水牛与其他物种间亲缘分析的分子标记。在水牛中确定胃型LYZ c蛋白存在3种变异体,河流型水牛存在变异体Buffalo 1~3,沼泽型水牛中只有变异体Buffalo 1~2。经比较分析发现,水牛中的3种LYZ c蛋白变异体在氨基酸组成上只有1~2处差异,它们在理化特性、结构上的特征基本一致,揭示它们间的功能差异较小,也提示两类水牛间LYZ c的功能差异较小。牛科物种胃型LYZ c基因编码区序列比对发现,虽然该区域SNP位点较多,但异义替换较少,LYZ c氨基酸序列一致性较高,其中普通牛和瘤牛一致性更高,这揭示牛科物种胃型LYZ c基因具有较高的功能保守性。
哺乳动物典型的溶菌酶c是在中性pH环境下发挥功能,而反刍动物胃型溶菌酶c裂解胃中细菌的细胞壁,其发挥功能必须要适应酸性pH环境,并对胃中的消化酶具有抗性[3]。在这种适应的过程中,反刍动物胃型LYZ c的氨基酸序列发生了一些适应性改变。这些改变包括赖氨酸被精氨酸残基替换,从而消除了胃中消化酶潜在的裂解位点,同时还失去了天冬氨酸-脯氨酸间的酸不稳定肽键[11-12]。胃型LYZ c的天冬氨酸残基与酰胺基的含量与非胃型LYZ c相比偏低,同时它所含有的酸不稳定氨基酸残基(天冬氨酸、天冬酰胺和谷氨酰胺)数量仅有18个,而非胃型LYZ c含有20~28个,这些特征保证了胃型LYZ c能在低pH环境下维持其功能。本研究预测水牛胃型LYZ c蛋白具有LYZ 1保守结构域,揭示它具有裂解细菌细胞壁肽聚糖中N-乙酰氨基葡萄糖和N-乙酰胞壁酸之间的β-1,4-糖苷键,消化微生物细胞壁的功能。水牛胃型LYZ c与牛奶和肾溶菌酶相比,其第120位的Asp被Glu替换,同时还缺失了121位的Pro,这样就失去了非胃型LYZ c蛋白原有的天冬氨酸-脯氨酸二肽,提高了水牛胃型LYZ c对酸性环境的耐受力[13]。水牛除了具有普通牛胃型LYZ c蛋白的特点外(酸不稳定氨基酸残基数为18个、不具有天冬氨酸-脯氨酸二肽),水牛胃型LYZ c蛋白的第116位氨基酸为Gln,其等电点为5.65,而普通牛Stomach 3在该位点上为His,等电点为7.59;同时,水牛第144位氨基酸为Glu,等电点为3.22,普通牛Stomach 3在该位点上为Gln。因此,水牛胃型LYZ c蛋白的等电点(pI=6.80)比普通牛Stomach 3蛋白(pI=7.55)的更低,这可能会导致水牛胃型LYZ c蛋白更适合酸性环境。
许多研究表明,在相同的饲喂条件下,水牛粗纤维消化率要高于其他反刍动物[14-17]。水牛对粗饲料的高消化率,可能是由于水牛瘤胃内特殊的微生物生态系统造成的[18]。其可能具有更多的纤维素分解菌、真菌游动孢子以及较低的原虫数量,导致水牛瘤胃微生物具有更高的蛋白质合成能力和氮循环利用能力[19-20]。水牛与普通牛胃型LYZ c蛋白之间最主要的差异是等电点不同,造成这种差异的原因可能与水牛瘤胃内特殊的微生物生态系统有关,推测胃型LYZ c与其瘤胃内的微生物经历了长期协同进化的过程。
本研究在水牛胃型LYZ c内共发现9个SNP位点,定义了7种单倍型,确定了3种变异体,但它们的功能差异较小。河流型与沼泽型水牛在核苷酸水平尽管存在一定差异,但两者的胃型LYZ c蛋白功能一致。水牛胃型LYZ c变异体不仅可以适应胃中的酸性环境,也可以在胃中裂解微生物细胞壁,这与其他牛科物种的胃型LYZ c蛋白具有相似性,但水牛胃型LYZ c蛋白等电点比普通牛更低,表明水牛胃型LYZ c蛋白更适应胃中酸性环境。水牛与牛科物种的胃型LYZ c蛋白氨基酸组成、结构和功能相近,揭示该基因在牛科物种中是由共同的祖先基因进化而来的,且受到了较高的选择压力。
参考文献:
[1] Chauss M,Hume I D,Hummel J.Evolutionary adaptations of ruminants and their potential relevance for modern production systems[J].Animal,2010,4(7):979-992.
[2] Steven C E,Hume L D.Contributions of microbes in vertebrate gastrointestinal tract to production and conservation of nutrients[J].Physiological Reviews,1998,78(2):393-427.
[3] Callewaert L,Michiels C W.Lysozymes in the animal kingdom[J].Journal of Biosciences,2010,35(1):127-160.
[4] Jollès P,Jollès J.What′s new in lysozyme research?Always a model system,today as yesterday[J].Molecular and Cellular Biochemistry,1984,63(2):165-189.
[5] Priyadarshini S,Kansal V K.Lysozyme activity in Buffalo milk:Effect of lactation period,parity,mastitis,season in India,pH and milk processing heat treatment[J].Asian-Australasian Journal of Animal Sciences,2002,15(6):895-899.
[6] 江伟华,朱莲莲,刘益丽,等.藏绵羊溶菌酶1基因的克隆,原核表达及生物信息学分析[J].中国畜牧兽医,2015,42(1):44-52.
[7] 刘 梅,张士璀.溶菌酶C及其生物学功能[J].生命的化学,2006,26(5):465-467.
[8] Irwin D M.Genomic organization and evolution of ruminant lysozyme c genes[J].Zoological Research,2015,36(1):1-17.
[9] 余 梅,刘建勇,赵 刚,等.补饲精料对采食蔗梢青贮水牛消化代谢的影响[J].云南农业大学学报,2013,28(4):470-475.
[10] Kumar S,Stecher G,Tamura K.MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1870-1874.
[11] Stewart C B,Schilling J W,Wilson A C.Adaptive evolution in the stomach lysozymes of foregut fermenters[J].Nature,1987,330(6146):401-404.
[12] 邓霄禹,胡明军,江明锋.反刍动物胃溶菌酶的研究进展[J].中国畜牧兽医,2012,39(9):143-147.
[13] Irwin D M.Evolution of cow nonstomach lysozyme genes[J].Genome,2004,47(6):1082-1090.
[14] Chanthakhoun V,Wanapat M,Kongmun P,et al.Comparison of ruminal fermentation characteristics and microbial population in swamp Buffalo and cattle[J].Livestock Science,2012,143(2/3):172-176.
[15] Hussain I,Cheeke P R.Evaluation of annual ryegrass straw:corn juice silage with cattle and water Buffalo:digestibility in cattle vs.Buffalo,and growth performance and subsequent lactational performance of Holstein heifers[J].Animal Feed Science and Technology,1996,57(3):195-202.
[16] Jabbari S,Eslami M,Chaji M,et al.The comparison of in vitro digestibility of wheat straw by rumen microorganism of Khuzestani buffalo and Hostein cow[J].International Conference on Biology,2010,1:266-268.
[17] 张 勤,李丽莉,韦升菊,等.尼龙袋法比较水牛与娟姗牛对象草消化能力的差异[J].中国牛业科学,2016,42(2):20-24.
[18] 张瑞云,钱朝海,顾招兵,等.反刍动物饲料营养价值评定方法及在水牛中的应用[J].中国奶牛,2016,313(5):4-9.
[19] Wanapat M.Rumen manipulation to increase the efficient use of local feed resources and productivity of ruminants in the tropics[J].Asian Australasian Journal of Animal Sciences,2000,13:59-67.
[20] 梁停停,刘文丽,许 浩,等.水牛瘤胃微生物研究进展[J].中国奶牛,2017,322(2):1-5.