二穗短柄草(Brachypodium distachyon)是一种广泛分布于温带地区的一年生单子叶植物,是禾本科(Poaceae)、早熟禾亚科(Pooideae)、短柄草族(Brachypodieae)、短柄草属(Brachypodium)的植物,与大多数重要的禾本科农作物即以小麦、大麦、燕麦和黑麦为主的麦类作物和以高丹草、苏丹草、黑麦草和狗牙根等为主的饲用作物近缘。由于二穗短柄草植株矮小,一般在20 cm以内;生活周期短;生长条件简单,对环境要求低,易于培养,自花授粉,基因组小,只有300 Mb等特点,2001年就被提议作为单子叶模式植物[1-2]。随后人们开始关注二穗短柄草并展开研究工作。2008年建立农杆菌介导的高效短柄草遗传转化体系和T-DNA插入突变体库[3]。2010年完成反复8次的二穗短柄草全基因组测序工作,研究发现二穗短柄草重复序列的大小和比例与拟南芥和水稻的相似,与麦类作物具有类似的基因组成和良好的共线性[2,4]。全基因组测序后,研究者们对短柄草进行了更深入的研究,如根的形态建成[5]、开花时间调控的分子机理[6]以及生物胁迫(病毒、真菌)和非生物胁迫(冷、热、盐和干旱等)响应机制等[7-12]。通过序列同源性来寻找并研究未报道的植物功能蛋白是研究未知植物蛋白的有效方法之一。Yordem等[13]用玉米Yellow Stripe-Like (ZmYSL) 转运蛋白序列在短柄草数据库中进行Blast搜索,发现了19个二穗短柄草YSL成员,发现这19个BdYSLs成员在系统发生树上分为4枝,其中1枝的成员具有禾草特异性,通过酵母功能互补试验证明了BdYSL具有Fe3+转运功能。
谷氨酸除了具有营养功能外,在动物的中央神经系统中还具有信号转导功能。它作为一种神经递质,由突触前膜释放,与突触后膜的谷氨酸受体结合后,诱导氨基酸门控通道的打开或激活G蛋白偶联受体[14]。谷氨酸受体包括离子型谷氨酸受体(ionotropic GluRs,iGLURs),与神经递质相关,可以形成K+、Na+和Ca2+阳离子内流通道,根据激动剂的特异性,分为3种:N-甲基-D-天冬氨酸受体(N-methyl-d-aspartate,NMDA)、红藻氨酸受体(Kainate,KAR)和α-氨基-3羟基-5甲基-4异恶唑(α-amino-3-hydroxy-5-methyl-4-isoxazolepropionate,AMPA)受体;代谢型谷氨酸受体(metabotropic,mGLURs),与G蛋白偶联促进激活第二信或iGLURs[15]。自从1989年人们发现与动物iGLURs同源的植物谷氨酸受体(Glutamate Receptor-Like genes,GLRs)[16]后,发现植物谷氨酸受体具有重要的功能,参与C/N平衡、光合作用、非生物胁迫、根的形态建成、种子萌发、花粉管生长、植物和病原菌的互作、细胞内钙动力学调控、接收和转导氨基酸信号等[14,17-19]。在氨基酸水平上,植物GLRs有6个功能域,4个跨膜域M1-M4;2个配体结合域GlnH1和GlnH2,植物与iGLuRs中的NMDA受体的同源性较高,尤其是M3结构域,序列相似性高达63%[14]。Aouini 等[20-21]在2011年用拟南芥AtGLRs氨基酸序列在番茄数据库进行Blast搜索,发现15个SlGLRs,通过系统发生分析发现其分为3枝,其中一枝是番茄特异枝,比其他枝先发生分化。在拟南芥中异源过表达SlGLR1.1和 SlGLR3.5后,发现它们可能参与Ca2+ 吸收。Ni等[22]发现水稻OsGLRs有13个成员,根据系统发生分析分为3枝即有3个亚家族,通过在HEK细胞中异源表达发现OsGLR2.1介导Glu引起水稻根细胞的Ca2+升高。综上所述,植物谷氨酸受体具有重要的生理功能,然而短柄草谷氨酸受体BdGLRs的相关研究尚未见报道,因此,笔者对短柄草的谷氨酸受体BdGLRs进行初步研究,以期为后续BdGLRs家族各个成员的生物学功能进行深入研究奠定基础,也为后续禾本科农作物和牧草遗传改良提供理论依据。
1 材料和方法
1.1 植物材料与培养
二穗短柄草由内蒙古大学生命科学学院祁智实验室保存。将二穗短柄草种子置于含5% NaClO和0.1% Tween20的消毒液中处理30 min,用无菌水清洗3次后,将表面消毒的种子置于MQA固体培养基上,4 ℃避光处理5 d后竖直置于培养室中培养至三叶期。培养室温度为23~25 ℃,湿度为60%~80%,光照周期为16 h光照/8 h黑暗,光照强度为100~120 μmol/(m2·s)。MQA培养基成分为:1%蔗糖;1%琼脂糖;大量元素为5 mmol/L KNO3,1 mmol/L H3PO4,1 mmol/L MgSO4,1 mmol/L CaCl2;微量元素为0.03 mmol/L ZnSO4,0.1 mmol/L MnSO4,0.1 mmol/L H3BO3,5 μmol/L KI,0.2 μmol/L CuSO4,0.1 μmol/L CoCl2,0.1 μmol/L Na2MoO4;Fe盐为0.1 mmol/L FeSO4,0.1 mmol/L Na2EDTA;5 mmol/L MES;BTP(Bis Tris Propane)调节pH值至5.7,高温高压灭菌。
1.2 试验方法
1.2.1 候选二穗短柄草谷氨酸受体BdGLRs序列的获得及系统发生分析 以拟南芥AtGLRs的氨基酸序列为种子序列,利用Phytozome数据库(http://www.arabidopsis.org/)中的Blast工具,搜寻可能存在的BdGLRs基因。拟南芥的20个GLRs氨基酸基因序列(表1)来自于The Arabidopsis Information Resource (TAIR)数据库(http://phytozome.jqi.doe.gov/pz/portal/htm)。BdGLRs的氨基酸序列和CDS的长度来自于PlantGDB数据库(http://www.plantgdb.org)。
表1 构建系统发生树所用的拟南芥、鼠和微生物的谷氨酸受体氨基酸序列登录号
Tab.1 Accession numbers and abbreviations for glutamate receptors in rat and Arabiopsis
基因Gene登录号Accession基因Gene登录号Accession拟南芥AtGLR3.1At2G17260ArabiopsisthalianaAtGLR3.2At4G35290AtGLR1.1At3G04110AtGLR3.3At1G42540AtGLR1.2At5G48400AtGLR3.4At1G05200AtGLR1.3At5G48410AtGLR3.5At2G32390AtGLR1.4At3G07520AtGLR3.6At3G51480AtGLR2.1At5G27100AtGLR3.7At2G32400AtGLR2.2At2G24720大白鼠RattusnorvegicusAtGLR2.3At2G24710GLURBX54655AtGLR2.4At4G31710GLURCM36420AtGLR2.5At5G11210GLURDM36421AtGLR2.6At5G11180GLUR1X17184AtGLR2.7At2G29120GLUR6Z11715AtGLR2.8At2G29110GLUR7M83552AtGLR2.9At2G29100GLUR5M83560
基于二穗短柄草、拟南芥和鼠的GLRs氨基酸序列(表1),利用BioEdit软件的ClustalW模块进行多重比较和MEGA 6.06[23]软件的Neighbor-joining算法构建系统发生树(Neighbor-joining tree)。选取参数为自展值重复数(Bootstrap replicates):1 000;模式/方法(Model/method):Poisson;空白数据处理(Gaps data treatment):Complete deletion。
1.2.2 半定量RT-PCR检测 采用TRIzol法[24]分别提取二穗短柄草幼苗根、茎、叶的总RNA,用DNA酶(TaKaRa)消化可能残留的DNA。通过超微量核酸蛋白测定仪Q5000(Quawell)测定RNA浓度,并将1 μg的总RNA通过First Strand cDNA Synthesis Kit 试剂盒(TaKaRa)反转录为cDNA。使用跨越内含子的引物EF1 184和EF1 185(表2)进行PCR,以检验cDNA中是否有DNA污染。
将合成的cDNA用超微量核酸蛋白测定仪Q5000将浓度调节为400 ng/μL;稀释10倍后取1 μL作为模板,半定量PCR 反应程序为95 ℃预变性3 min;95 ℃ 30 s,50~60 ℃ 30 s,72 ℃ 30 s,20~28个循环;72 ℃ 延伸3 min。PCR 产物用1.5%琼脂糖凝胶电泳进行分析。将看家基因EF1α 作为内参基因,通过调节模板量将根、茎、叶的看家基因片段的电泳条带调至亮度一致。独立重复3次试验,确定合适的模板量。用BdGLRs特异性基因引物(表2),选择上述合适的模板量进行扩增。通过是否有电泳条带和电泳条带的亮暗来推测BdGLRs在根茎叶的表达情况。
表2 BdGLRs的半定量和定量RT-PCR分析所用引物
Tab.2 Primer sequences used in semi-quantitative and quantitative RT-PCR analysis
引物Primer序列(5'-3')Sequence引物Primer序列(5'-3')SequenceBd800_FBd800_RCACCAGATACGTCAATGAGTGCCCAGGTTATCAGAGCCATTGCCACCTGABd820_FBd820_RGTAGGTAAGAAGCTTAGAGTCGGCTGGTGGATCAGCTGAGTCAAATGCTTBd920_FBd920_RGTTCGGCACAACTTCACCACCAATTTTCCATGTTGGCCCTTCTCAAGCCBd910_FBd910_RGTTAGTCACACCGCCACCGTTGCAATCGGCACTCTGCATTTCAGAATCBd790_FBd790_RCGAGAACTCGACGACCGGAGAGATGCCCGACAATGATATCGGCTTCCTGATCBd560_FBd560_RTCCCAATAGGGCAAGTTACCGTCTTTTATTGATAAGCTCGGCATAGCTTBd030_FBd030_RCACCGAACTCTTCAACCAACAATATCCCATCAGGTCCATTGTCTTGCATBd620_FBd620_RCGCAATTACAATGAACAGGACTAAATGTTCAAGAACCCAAACAACCACABd690_FBd690_RCTCCGGCGTCGCAGAACGGGGGTTCGGGAACGGCACGTAGTTGTAGACTBd940_FBd940_RGTACAGCTTCACCACTGATGGGTAACACTGTAGCTGTCGCTTACCACABd880_FBd880_RTGGCATCTCCAGGAGTCTAAATAGGTGATGCAGAACCCTTTGGGTCTGGGBd600_FBd600_RCCGTGTCGGCTTCTGGTCCAACCGGAACTGCCGGTACGTCGTCCTCCABd850_FBd850_RTAGCATCTCCGGTAATTCAAGTTCCATCGATGCAGAATCCCTTGGCAATBd890_FBd890_RCAGTTCGTGGGCGTCGACCCCAGCCCACCACGACGTCCGCCTCTTBd810_FBd810_RACTAGATCCGCAACAACGCATTTGAACCAGTGATATGATCCTTTTCGBd860_FBd860_RGGCTACATGACCAGCGGAGGAGATGGTGCCGGCCTGGTTGTAGCBd350_FBd350_RCAATCAAACTGGCTTGAAAATAACAGGCTTGTTTCACCTGTGGCATGATCTCBd947_FBd947_RCAGTCTGGCATTTCGATACTCACAGGCATTGCTACACTGTCTTAAAATGEF1184CCATCGATATTGCCTTGTGGEF1185GTCTGGCCATCCTTGGAGAT
1.2.3 定量RT-PCR检测 定量PCR分析的参比基因为ACTIN,引物Actin2-8_F:5′-ACGGTAACATTGT GCTCAGTGGTG-3′;Actin2-8_R:5′-CTTGGAGATCCAC ATCTGCTGGA-3′。采用TransStart Tip Green qPCR SuperMix(北京全式金公司)在德国耶拿FlexCycler多通道荧光定量PCR仪Qtower 2.2中进行PCR扩增,重复3次,引物(表2)和模板同半定量PCR分析,反应程序为:94 ℃ 30 s,55 ℃ 15 s,72 ℃15 s,25个循环,最后采用2-ΔΔCt法,对定量结果进行分析。
2 结果与分析
2.1 二穗短柄草候选谷氨酸受体BdGLRs
用AtGLRs氨基酸序列Blast搜索二穗短柄草基因组数据库,发现19个二穗短柄草候选BdGLRs,同拟南芥AtGLR2.2、AtGLR2.5、AtGLR2.7、AtGLR2.8、AtGLR3.3和AtGLR3.4有较高同源性,分布在二穗短柄草1~5号染色体上(表3);氨基酸序列长度523~1 008 Aa,cDNA长度为1 572~3 027 bp(表3)。
表3 候选二穗短柄草谷氨酸受体GLRs家族
Tab.3 The candidate GLR family members identified in Brachypodium distachyon
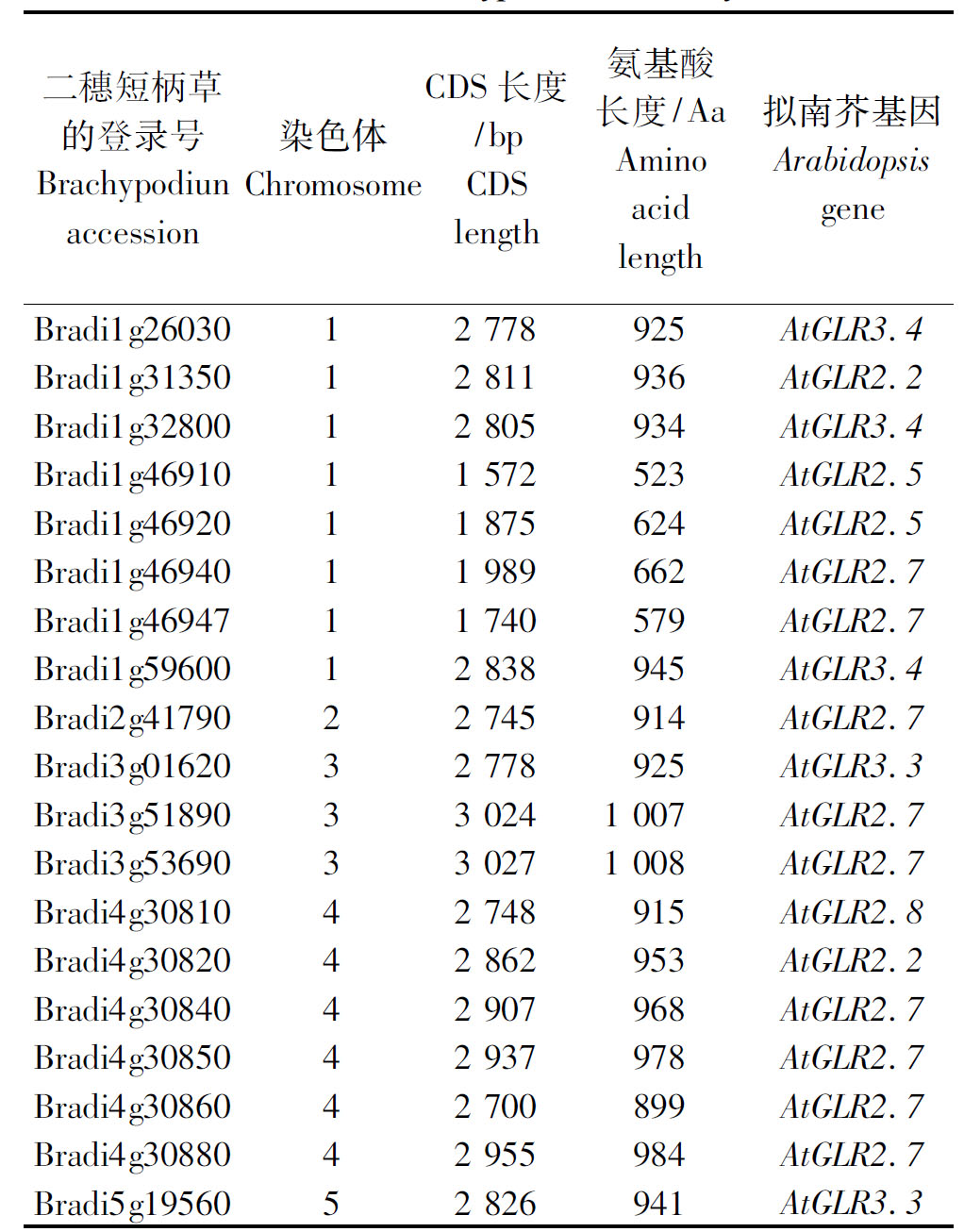
二穗短柄草的登录号Brachypodiunaccession染色体ChromosomeCDS长度/bpCDSlength氨基酸长度/AaAminoacidlength拟南芥基因ArabidopsisgeneBradi1g2603012778925AtGLR3.4Bradi1g3135012811936AtGLR2.2Bradi1g3280012805934AtGLR3.4Bradi1g4691011572523AtGLR2.5Bradi1g4692011875624AtGLR2.5Bradi1g4694011989662AtGLR2.7Bradi1g4694711740579AtGLR2.7Bradi1g5960012838945AtGLR3.4Bradi2g4179022745914AtGLR2.7Bradi3g0162032778925AtGLR3.3Bradi3g51890330241007AtGLR2.7Bradi3g53690330271008AtGLR2.7Bradi4g3081042748915AtGLR2.8Bradi4g3082042862953AtGLR2.2Bradi4g3084042907968AtGLR2.7Bradi4g3085042937978AtGLR2.7Bradi4g3086042700899AtGLR2.7Bradi4g3088042955984AtGLR2.7Bradi5g1956052826941AtGLR3.3
2.2 二穗短柄草候选谷氨酸受体BdGLRs的系统发生分析
基于二穗短柄草、拟南芥和鼠谷氨酸受体氨基酸序列构建系统发生树表明,19个 BdGLRs和20个AtGLRs聚为一大枝,先于鼠的iGLuRs分化,形成独立大枝(Clade),表明二穗短柄草和拟南芥亲缘关系较近。将BdGLRs和AtGLRs共同构建系统发生树发现,BdGLRs没有特异分枝,同AtGLRs的3个亚家族聚类在一起形成独立3枝[25]。只有AtGLRs Clade Ⅲ和BdGLR Ⅲ形成分枝的自展值高达99,其他2枝的自展值低于70,分别为4和10(图1),不适合作为BdGLRs亚家族分类的依据。

图1 二穗短柄草、拟南芥及鼠的谷氨酸受体氨基酸序列系统发生分析
Fig.1 Phylogenetic analysis based on glutamate like receptor in Brachypodium distachyon,Arabiopsis thaliana and Rattus norvegicus
由于AtGLRs和BdGLRs亲缘关系较近,相互干扰无法形成独立分枝,因此,将20个AtGLRs和19个BdGLRs分别构建系统发生树,AtGLRs分为3个亚家族,分别为AtGLR1.1-1.4、AtGLR2.1-2.9、AtGLR3.1-3.4,自展值都为100。基于BdGLRs构建的系统发生树的拓扑结构与AtGLRs的类似,也分为3枝,Clade Ⅲ和Clade Ⅰ的自展值是100,不同的是BdGLRs Clade Ⅱ的自展值是57(图2),表明BdGLRs Clade Ⅱ成员与AtGLRs变异度更大。最后根据BdGLRs与AtGLRs不同成员在系统发育树的位置以及AtGLRs的命名规则,将 BdGLRs分为3个亚家族,分别为BdGLR1.1-1.4,位于1号染色体;BdGLR2.1-2.10,位于1,2~4号染色体;BdGLR3.1-3.5,位于1,3和5号染色体(图2、表3)。
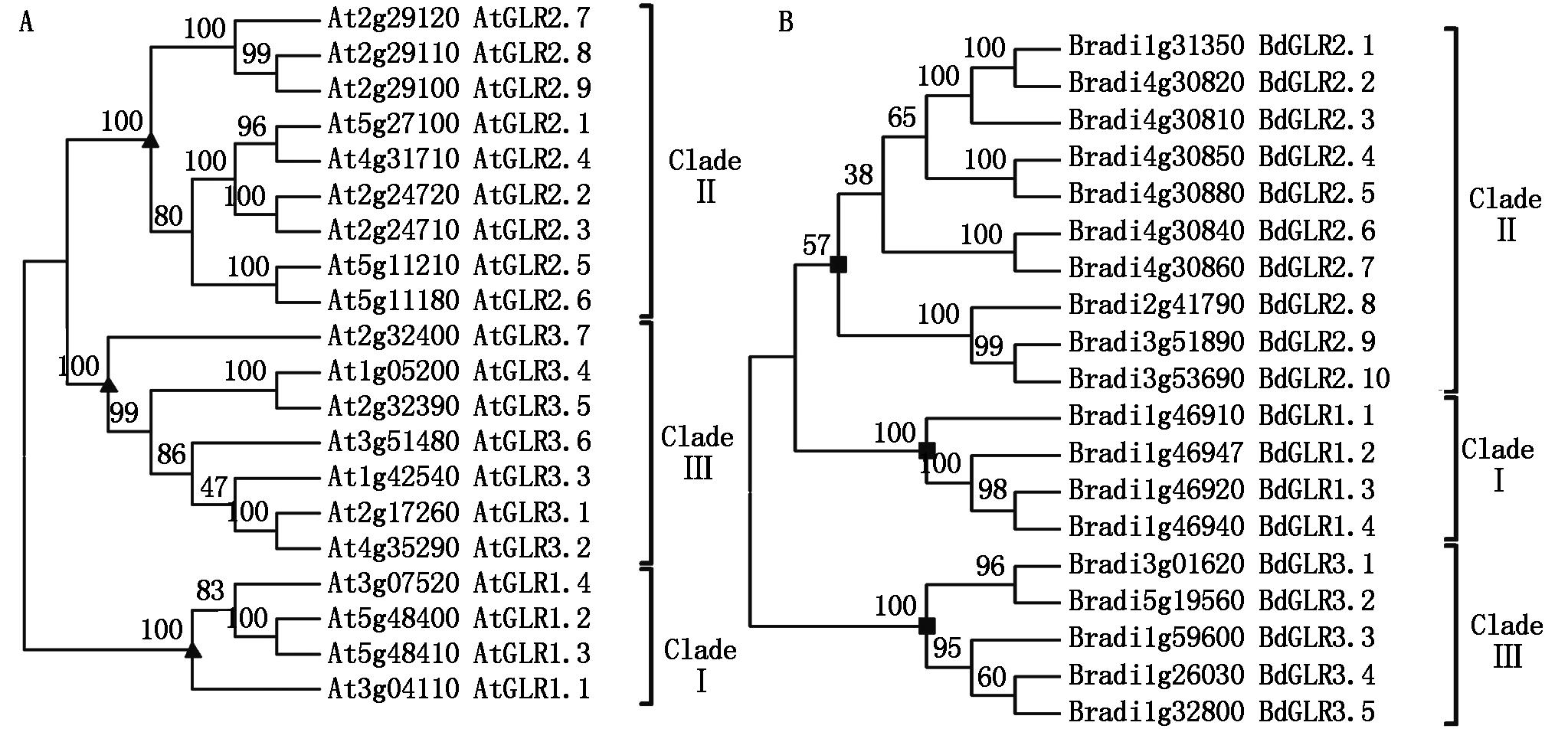
图2 拟南芥(A)和二穗短柄草GLRs(B)的系统发生分析
Fig.2 Phylogenetic analysis based on glutamate like receptor in Arabiopsis thaliana (A) and Brachypodium distachyon (B)
2.3 二穗短柄草谷氨酸受体基因表达半定量RT-PCR分析
通过半定量反转录PCR分析,发现在二穗短柄草的三叶期,除BdGLR1.4、BdGLR2.6-2.10,其余BdGLRs在根、茎、叶中均有表达。BdGLR1.4在根和茎中表达,在叶中表达量较低或不表达;BdGLR2.7在茎和叶中表达,在根中表达量较低或不表达。BdGLR2.8在根表达,在茎和叶中表达量较低或不表达; BdGLR2.6、2.9、2.10条带很弱,几乎不可见,表明其在根、茎、叶中表达量较低或不表达。虽然绝大多数BdGLRs在根、茎、叶中均有表达,但BdGLR在不同组织中表达量不同, BdGLR1.1、BdGLR1.3、BdGLR2.2和BdGLR3.1在茎中的表达量明显高于根和叶;BdGLR2.1、BdGLR2.5、BdGLR3.3和BdGLR3.4在茎和叶中的表达量明显高于根中。在19个BdGLR中只有BdGLR3.5的在根、茎、叶中表达量几乎一致(图3)。
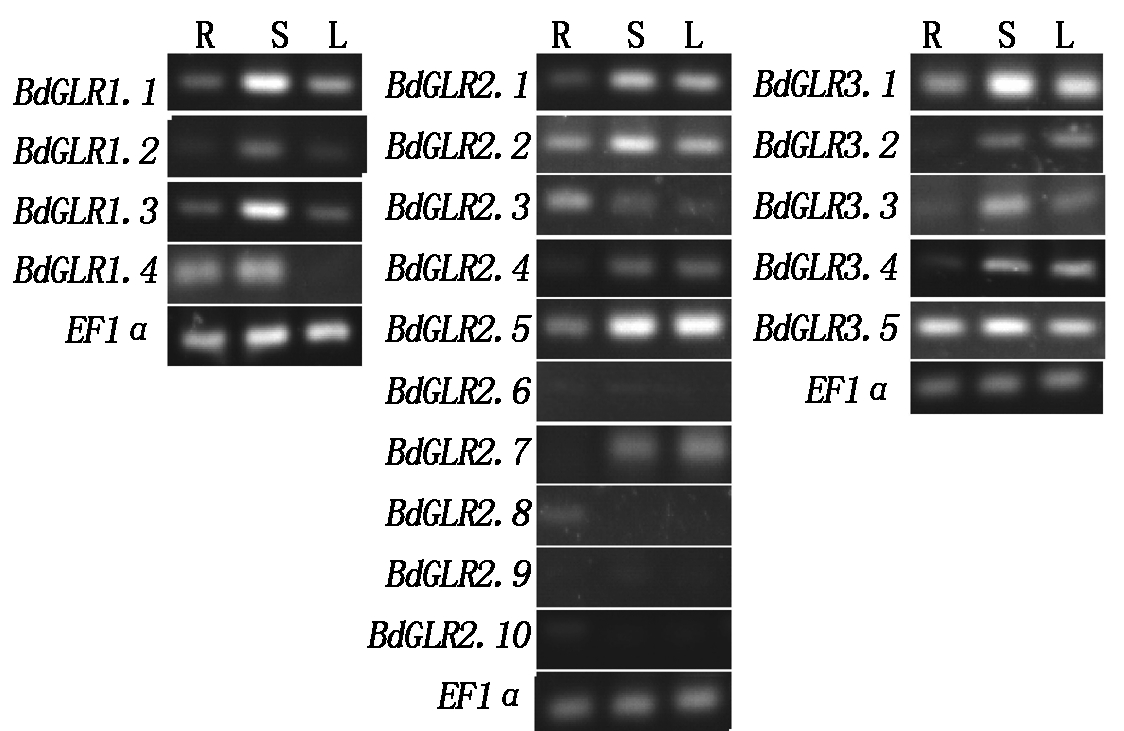
R.根;S.茎;L.叶。
R.Roots;S.Stems;L.Leaves.
图3 BdGLRs基因表达在根、茎、叶中的半定量RT-PCR分析
Fig.3 Semi-quantitative RT-PCR analysis of BdGLR gene expression
2.4 二穗短柄草谷氨酸受体基因表达定量RT-PCR分析
通过定量反转录PCR分析,BdGLRs的不同成员在植物的根、茎和叶中表达差异较大。在根中,相对于BdGLR1.1,BdGLR2.4、BdGLR2.6-2.10、BdGLR3.2-3.4的表达量较低,而BdGLR3.5的表达量较高。在茎中,BdGLR1.2、BdGLR1.4、BdGLR2.3、BdGLR2.4、BdGLR2.6-2.10、BdGLR3.2-3.4的表达量较低,BdGLR3.1的表达量较高。在叶中,BdGLR2.3、BdGLR2.4、BdGLR2.6-2.10、BdGLR 3.2-3.4的表达量较低,BdGLR2.5的表达量较高。在根、茎、叶中,绝大多数BdGLR CladeⅡ的成员表达量较低(图4)。
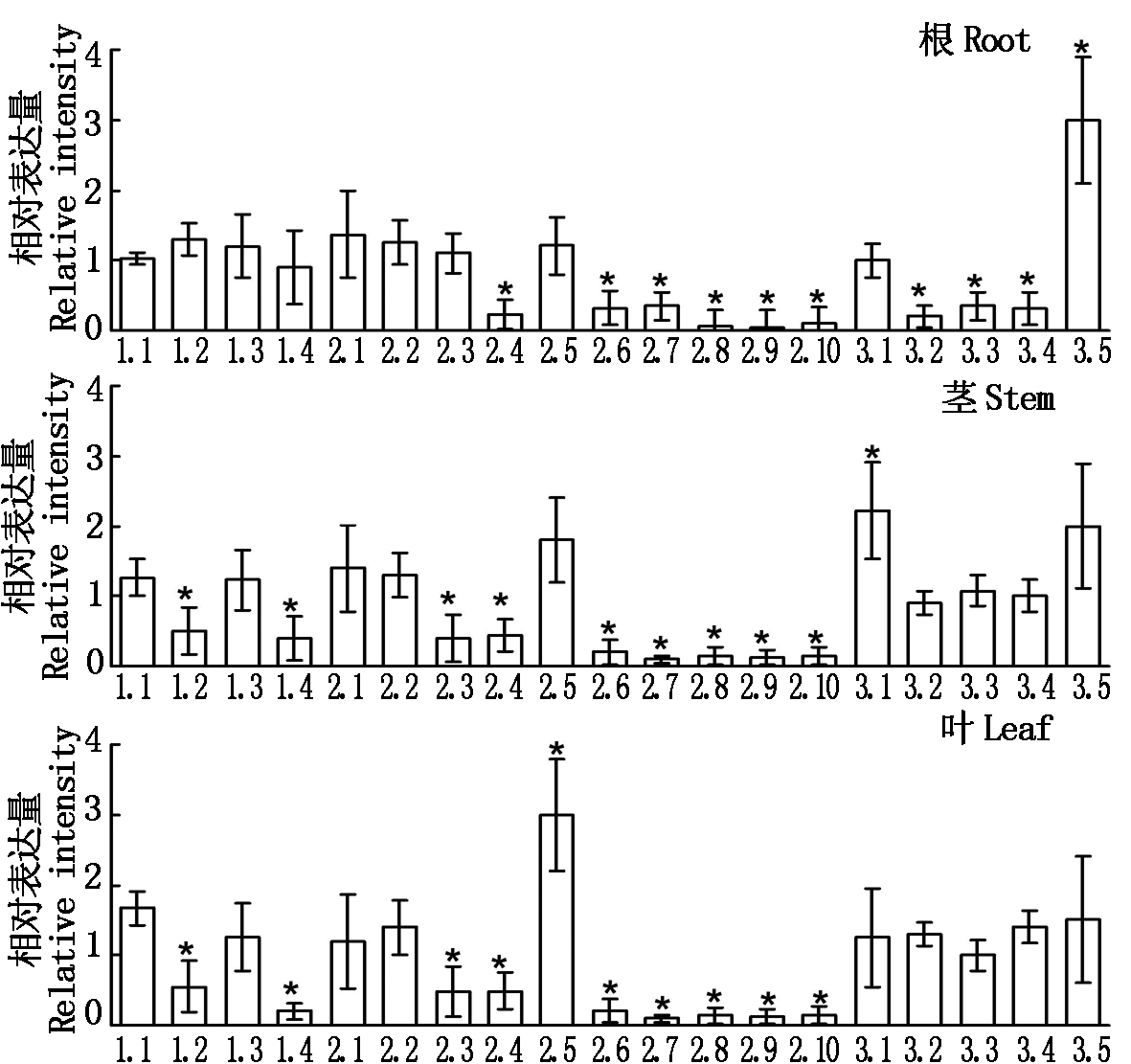
*.P<0.01。
图4 BdGLRs基因在根、茎、叶中的定量RT-PCR分析
Fig.4 Quantitative RT-PCR analysis of BdGLRs genes in the roots,stems and leaves
3 讨论
本研究是通过与拟南芥谷氨酸受体(AtGLRs)氨基酸序列的同源性来寻找模式植物短柄草谷氨酸受体(BdGLRs)。Lam等[16]于1998年首次在拟南芥中发现AtGLRs,通过序列的同源性分析,发现其与动物离子型谷氨酸受体(iGLu)和大肠杆菌(Escherichia coli)中谷氨酰胺渗透酶(Glutamine permease,GlnH)同源。Chiu等[25]将AtGLR1.1 cDNA sequence (AF079998)序列进行Blast,发现AtGLRs有20个成员,基于系统发生学分析发现AtGLRs分为3个家族20个成员,分别为AtGLR1.1-1.4、AtGLR2.1-2.9、AtGLR3.1-3.7。后续人们对AtGLRs成员深入研究,发现它们在拟南芥中起着重要作用,如AtGLR1.1参与碳氮代谢平衡[26];AtGLR3.3介导谷胱甘肽引起细胞质的[Ca2+]升高,参与先天免疫;AtGLR3.6参与根发育[18]等。Aouini 等[20]在2011年用AtGLRs氨基酸序列在番茄数据库进行Blast搜索,发现15个SlGLRs。Ni等[22]于2016年用AtGLRs氨基酸序列在水稻数据库进行Blast搜索,发现13个OsGLRs,基于系统发生分析分为3枝,分别为OsGLR1.1-1.4、OsGLR2.1-2.4、OsGLR3.1-3.5。由此可见,通过AtGLRs的序列同源性及系统发生学分析来鉴定其他植物中GLRs的成员以及成员分类是可行的。据此本研究发现BdGLRs有20个成员,分为3个亚家族。通过系统发生分析,BdGLRs和AtGLRs亲缘关系较近,在系统发生树中BdGLRs的成员分别于AtGLRs的3个不同亚家族分别聚类,只除BdGLR CladeⅢ与AtGLR CladeⅢ的自展值较高,为99外,其余2个家族分枝的自展值分布为4和10。由此推断如果根据图1对BdGLRⅠ和BdGLRⅡ进行分类可能很不准确,后续将BdGLRs和AtGLRs进行单独构建系统发生树,AtGLRs的3个亚家族与前人报道一致,3个亚家族分枝自展值都为100,结果可信。据此根据BdGLRs单独构建系统发生树的结果对BdGLRs进行分类较为可信。
本研究对BdGLRs基因表达的组织特异性进行初步研究发现,不同BdGLRs具有不同的表达模式,暗示着BdGLRs与AtGLRs类似,负责不同的功能。研究发现BdGLR2.6、2.9和2.10在二穗短柄草苗期的表达量很少,甚至几乎不表达,可能这3个基因表达具有时间特异性,在二穗短柄草的成熟期表达。其余16个BdGLRs成员虽然在苗期都表达,但不同的BdGLRs在不同的组织中表达。BdGLRs的功能很可能与AtGLRs类似,具有重要的生理功能,参与调控生物和非生物胁迫响应、根的形态建成、种子萌发、花粉管生长等。笔者后续会选择在苗期高表达且在根、茎、叶都表达的BdGLR1.1、BdGLR2.1、BdGLR3.1和BdGLR3.5进行深入研究。
参考文献:
[1] Draper J,Mur L A,Jenkins G,et al.Brachypodium distachyon.A new model system for functional genomics in grasses[J].Plant Physiology,2001,127(4):1539-1555.
[2] Brkljacic J,Grotewold E,Scholl R,et al.Brachypodium as a model for the grasses:today and the future[J].Plant Physiology,2011,157(1):3-13.
[3] Vain P,Worland B,Thole V,et al.Agrobacterium-mediated transformation of the temperate grass Brachypodium distachyon(genotype Bd21) for T-DNA insertional mutagenesis[J].Plant Biotechnology Journal,2008,6(3):236-245.
[4] Vogel J P,Garvin D F,Mockler T C,et al.Genome sequencing and analysis of the model grass Brachypodium distachyon[J].Nature,2010,463(7282):763-768.
[5] Kim C M,Dolan L.Root hair development involves asymmetric cell division in Brachypodium distachyon and symmetric division in Oryza sativa[J].The New Phytologist,2011,192(3):601-610.
[6] An Y,Guo Y,Liu C,et al.BdVIL4 regulates flowering time and branching through repressing miR156 in ambient temperature dependent way in Brachypodium distachyon[J].Plant Physiology and Biochemistry,2015,89:92-99.
[7] Chen S,Li H.Heat stress regulates the expression of genes at transcriptional and Post-Transcriptional levels,revealed by RNA-seq in Brachypodium distachyon[J].Frontiers in Plant Science,2016,7(7):2067.
[8] Bian Y,Deng X,Yan X,et al.Integrated proteomic analysis of Brachypodium distachyon roots and leaves reveals a synergistic network in the response to drought stress and recovery[J].Scientific Reports,2017,7(7):46183.
[9] Tao Y,Nadege S W,Huang C,et al.Brachypodium distachyon is a suitable host plant for study of Barley yellow dwarf virus[J].Virus Genes,2016,52(2):299-302.
[10] He Y,Zhang Y,Chen L,et al.A member of the 14-3-3 gene family in Brachypodium distachyon,BdGF14d,confers salt tolerance in transgenic tobacco plants[J].Frontiers in Plant Science,2017,8:340.
[11] Bredow M,Vanderbeld B,Walker V K.Knockdown of Ice-Binding proteins in Brachypodium distachyon demonstrates their role in freeze protection[J].PLoS One,2016,11(12):e0167941.
[12] Gill U S,Uppalapati S R,Nakashima J,et al.Characterization of Brachypodium distachyon as a nonhost model against switchgrass rust pathogen Puccinia emaculata[J].BMC Plant Biology,2015,15:113.
[13] Yordem B K,Conte S S,Ma J F,et al.Brachypodium distachyon as a new model system for understanding iron homeostasis in grasses:phylogenetic and expression analysis of Yellow Stripe-Like (YSL) transporters[J].Annals of Botany,2011,108(5):821-833.
[14] Price M B,Jelesko J,Okumoto S.Glutamate receptor homologs in plants:functions and evolutionary origins[J].Frontiers in Plant Science,2012,3:235.
[15] Flores-Soto M E,Chaparro-Huerta V,Escoto-Delgadillo M,et al.Structure and function of NMDA-type glutamate receptor subunits[J].Neurologia,2012,27(5):301-310.
[16] Lam H M,Chiu J,Hsieh M H,et al.Glutamate-receptor genes in plants[J].Nature,1998,396(677):125-126.
[17] Singh S K,Chien C T,Chang I F.The Arabidopsis glutamate receptor-like gene GLR3.6 controls root development by repressing the kip-related protein gene KRP4[J].Journal of Experimental Botany,2016,67(6):1853-1869.
[18] Li F,Wang J,Ma C,et al.Glutamate receptor-like Channel3.3 is involved in mediating glutathione-triggered cytosolic Calcium transients,transcriptional changes,and innate immunity responses in Arabidopsis[J].Plant Physiology,2013,162(3):1497-1509.
[19] Cheng Y,Tian Q Y,Zhang W H.Glutamate receptors are involved in mitigating effects of amino acids on seed germination of Arabidopsis thaliana under salt stress[J].Environmental and Experimental Botany,2016,130:68-78.
[20] Aouini A,Matsukura C,Ezura H,et al.Characterisation of 13 glutamate receptor-like genes encoded in the tomato genome by structure,phylogeny and expression profiles[J].Gene,2012,493(1):36-43.
[21] Aouini A,Hernould M,Ariizumi T,et al.Overexpression of the tomato glutamate receptor-like genes SlGLR1.1 and SlGLR3.5 hinders Ca2+utilization and promotes hypersensitivity to Na+ and K+ stresses[J].Plant Biotechnology,2012,29(3):229-235.
[22] Ni J,Yu Z,Du G,et al.Heterologous expression and functional analysis of rice Glutamate receptor like family indicates its role in glutamate triggered calcium flux in rice roots[J].Rice,2016,9(1):9.
[23] Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
[24] 张彦桃,王 欣,祁 智,等.拟南芥高亲和性钾转运体AtHAK5参与植物根对盐胁迫及ABA的反应[J].华北农学报,2014,29(6):214-219.
[25] Chiu J C,Brenner E D,Desalle R,et al.Phylogenetic and expression analysis of the glutamate-receptor-like gene family in Arabidopsis thaliana[J].Molecular Biology and Evolution,2002,19(7):1066-1082.
[26] Kang J,Turano F J.The putative glutamate receptor 1.1 (AtGLR1.1) functions as a regulator of carbon and nitrogen metabolism in Arabidopsis thaliana[J].Proceedings of the National Academy of Sciences of the United States of America,2003,100(11):6872-6877.