芍药分生组织决定基因APETALA2(AP2)的克隆及生物信息学分析
吴彦庆1,成梦琳2,赵大球2,陶 俊1,2
(1.扬州大学 动物科学与技术学院,江苏 扬州 225009;2.扬州大学 园艺与植物保护学院,江苏 扬州 225009)
摘要:花分生组织决定基因APETALA2(AP2)属于植物ABCDE模型基因,在花器官发育过程中起着重要的调控作用。为进一步了解芍药AP2基因的生物学功能,利用RACE扩增和测序技术克隆plAP2基因序列,利用生物信息学在线程序对其序列特征、蛋白结构及功能、亚细胞定位进行预测,并利用MEGA 5.0构建不同植物AP2分子进化树,最后利用qPCR检测其在内外花瓣中的差异表达情况。结果显示,克隆获得芍药AP2基因(plAP2)cDNA序列全长1 935 bp,其ORF全长为1 578 bp,编码525个氨基酸。蛋白结构与功能分析表明,plAP2蛋白为亲水性不稳定蛋白,无跨膜结构和信号肽,表明为非分泌蛋白;核定位信号位于氨基酸序列139-147(KKSRRGPRS);二级结构包括α-螺旋(24%)、β-折叠(19%)、β-转角(28%)和无规则卷曲(28%);plAP2蛋白存在8个糖基化位点和64个磷酸化位点,plAP2蛋白包含2个相同的保守结构域:AP2/(Ethylene-Responsive factors,ERF)(151-213aa和243-306aa)。亚细胞定位主要在细胞质(45.0%)中,少量分布于微体、线粒体基质间隙和溶酶体;进化树分析表明,芍药AP2基因与牡丹高度同源且亲缘关系最近;qPCR检测显示外瓣AP2表达量均极显著高于内瓣(P<0.01)。克隆出芍药AP2全长cDNA序列,系统地揭示了plAP2蛋白基本结构、功能位点区域、细胞定位以及组织表达情况,为今后深入研究plAP2基因功能提供基础素材和理论参考。
关键词:芍药;APETALA2基因;RACE;生物信息学
花器官形成作为被子植物生长发育与进化过程中的重要环节,是一个比较复杂的形态和生理生化变化的过程,并且受到多基因调控[1],因此花器官发育的分子调控机制研究受到许多学者的广泛关注。 目前,有关花器官发育的分子机制研究主要集中在拟南芥 (Arabidopsis thaliana )、金鱼草( Antirrhinum majus )、矮牵牛( Petunia hybrid )等模式植物上,植物花器官发育ABC模型在1991年被首次提出来[2],并发现FBP(Floral binding protein)7和 FBP11 基因在模式生物矮牵牛胚珠发育过程中起关键作用[3],随后ABCD模型被Colombo等[4]提出;此外,Pelaz等[5-6]在拟南芥中分离出 SEP1/2/3(SEPALLATA1/2/3) 基因,并研究发现在SEP1/2/3突变导致花器官形态发生改变,变成萼片状结构,从而提出花器官发育ABCDE模型。其中, APETALA2(AP2) 基因属于植物花器官发育ABCDE模型家族基因,在植物萼片和花瓣形成过程中起着重要的调控作用[7]。目前,在拟南芥 (Arabidopsis thaliana)、牡丹(Paeonia suffruticosa)、玉米(Zea mays)、草莓(Fragaria ananassa)、苹果(Malus domestica) 等植物上,已成功分离出 AP2 基因cDNA全长[8-12]。芍药 (Paeonia lactiflora Pall.)作为我国的传统名花,其花型优劣直接关系到芍药科植物的观赏价值和商业价值,然而芍药 AP2(plAP2) 基因的序列信息和功能机制尚不清楚。随着分子生物学技术和计算机信息时代的发展,cDNA末端快速克隆(Rapid amplification of cDNA ends,RACE)和生物信息学目前已经被广泛用于初步揭示未知基因的结构与功能[13-15]。因此,本研究以芍药托桂型品种向阳奇花的花瓣为试验材料,利用RACE克隆测序获得 plAP2 基因全长cDNA序列,在此基础上进一步利用生物信息学软件预测其序列特征、蛋白结构与功能以及基因定位等信息,并分析不同植物间 plAP2 分子进化起源关系,为今后深入研究 plAP2 基因功能提供一定的理论参考。
1 材料和方法
1.1 试验材料
试验植物:本试验选用芍药托桂型品种向阳奇花的内外花瓣(来源于扬州大学园艺与植物保护学院芍药种质资源圃)为试验材料,进行芍药 AP2(plAP2) 基因克隆;生化试剂:DL2000 Marker、RNA纯化试剂盒DNase Ⅰ、反转录试剂盒、SYBR Premix Ex Taq Ⅱ、3′-full和5′-full RACE Core Set Ver.2.0试剂盒均购于TaKaRa公司。
1.2 引物设计
由于芍药基因组信息尚未公布,参考前期芍药花色嵌合体品种金辉花瓣的无参转录组de novo测序获得的 plAP2 部分mRNA序列[16],设计3′-full和5′-full RACE扩增引物以及荧光定量引物(表1),并送上海生物工程技术服务有限公司合成。
表1 plAP2基因引物信息
Tab.1 Primer information of plAP2 gene
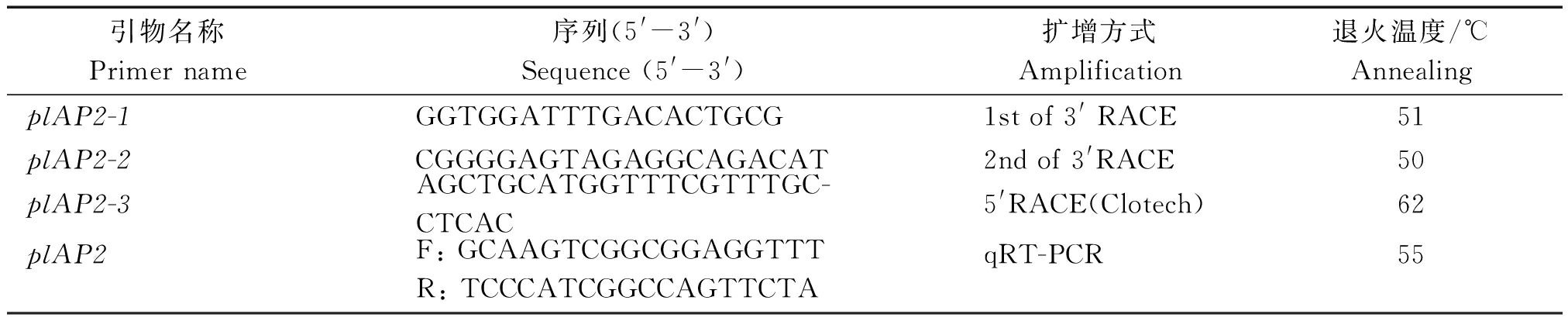
引物名称Primername序列(5'-3')Sequence(5'-3')扩增方式Amplification退火温度/℃AnnealingplAP2-1GGTGGATTTGACACTGCG1stof3'RACE51plAP2-2CGGGGAGTAGAGGCAGACAT2ndof3'RACE50plAP2-3AGCTGCATGGTTTCGTTTGC-CTCAC5'RACE(Clotech)62plAP2F:GCAAGTCGGCGGAGGTTTR:TCCCATCGGCCAGTTCTAqRT-PCR55
1.3 RNA提取及RACE克隆
根据TaKaRa MiniBEST Plant RNA Extraction Kit试剂盒(大连宝生物工程有限公司)提取方法,分别提取芍药内外花瓣总RNA。以总RNA为模板(1.0 μg),参考3′和5′-full RACE Core Set Kit试剂盒说明书分别进行3′-full和5′-full RACE扩增,2轮PCR扩增结束后将产物在1%琼脂糖凝胶上进行电泳检测。最后,PCR产物纯化回收后,将目的片段与克隆载体pMD18-T连接、转化并挑取质粒送往上海生物工程有限公司进行测序。
1.4 序列同源性分析
克隆产物经测序获得芍药 plAP2 基因cDNA序列后,利用ORF Finder软件(https://www.ncbi.nlm.nih.gov/orffinder/)预测出芍药plAP2基因编码序列(ORF)和氨基酸序列。此外,利用Blast软件对芍药plAP2基因与拟南芥、葡萄、苹果、番茄、矮牵牛、毛果杨等不同植物进行同源性分析 (http://www.ncbi.nlm.nih.gov/BLAST/),并通过DNAMAN软件生成不同植物plAP2基因的系统进化树。
1.5 蛋白理化性质与基本结构预测
利用ProtParam软件(http://web.expasy.org/protparam/)分析芍药plAP2蛋白的理化性质,ProtScale软件(http://web.expasy.org/cgi-bin/protscale/protscale.pl)预测 plAP2蛋白疏水性区域;TMHMM 2.0软件(http://www.cbs.dtu.dk/services/TMHMM-2.0/)预测plAP2蛋白跨膜螺旋结构;SignalP 4.0软件[17]分析信号肽,Antheprot 5.0 (Universite Claude Bernard,Lyon,http://www.intute.ac.uk)软件分析plAP2蛋白二级结构,其中包括α螺旋(α-helix)、β折叠(β-sheet)、β转角(β-turn)、无规则卷曲(Random coil);NLStradamus[18]分析核定位信号以及NetNES1.1[19]软件分析核运出信号。
1.6 蛋白功能位点及保守功能域分析
在真核生物中,磷酸化和糖基化是最常见的2种组蛋白翻译后修饰方式,其中糖基化主要发生于天冬酰胺(Asn)残基,而磷酸化主要发生于丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)等残基[20-21]。利用NetNGlyc软件(http://www.cbs.dtu.dk/services/NetNGlyc/)预测芍药plAP2蛋白潜在的糖基化位点以及NetPhos软件预测潜在的磷酸化位点[22],以及利用NCBI CDD软件[23]和SMART软件[24]综合分析蛋白保守功能区域。
1.7 亚细胞定位分析
基因亚细胞定位(Subcellular localization)是研究植物生长、形态发育机制的重要环节,也为初步分析与推测其基因可能发挥的生物学功能提供一定的参考信息。本研究利用TargetP[25]软件以及PSORTⅡPrediction软件(http://psort.hgc.jp/form.html)对 plAP2 基因亚细胞定位进行综合分析。
1.8 基因表达分析
利用BIO-RAD CFX96 TM Real-time System (C1000 TM Thermal Cycler) (Bio-Rad,USA)系统检测 plAP2 基因定量表达水平。25 μL定量反应体系包括12.5 μL 29 SYBR Premix Ex TaqTM,50× ROX Reference Dye Ⅱ 0.5 μL,2 μL cDNA模板,2 μL Primer引物,8 μL ddH2O。反应条件为:94 ℃预变性5 min;95 ℃变性15 s,55 ℃退火15 s,72 ℃延伸40 s,40个循环;72 ℃延伸10 min。
2 结果与分析
2.1 芍药 plAP2 基因克隆及序列分析
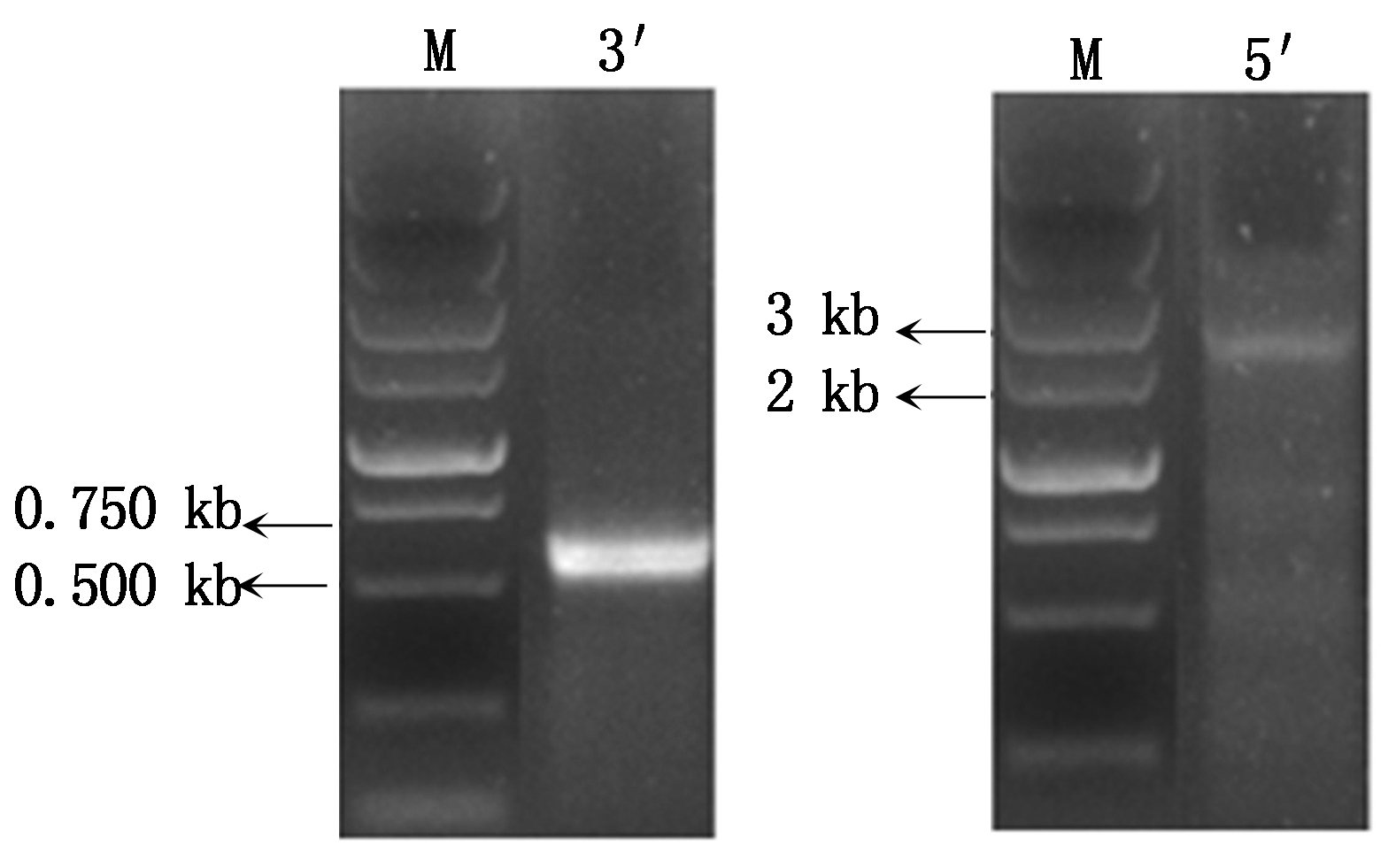
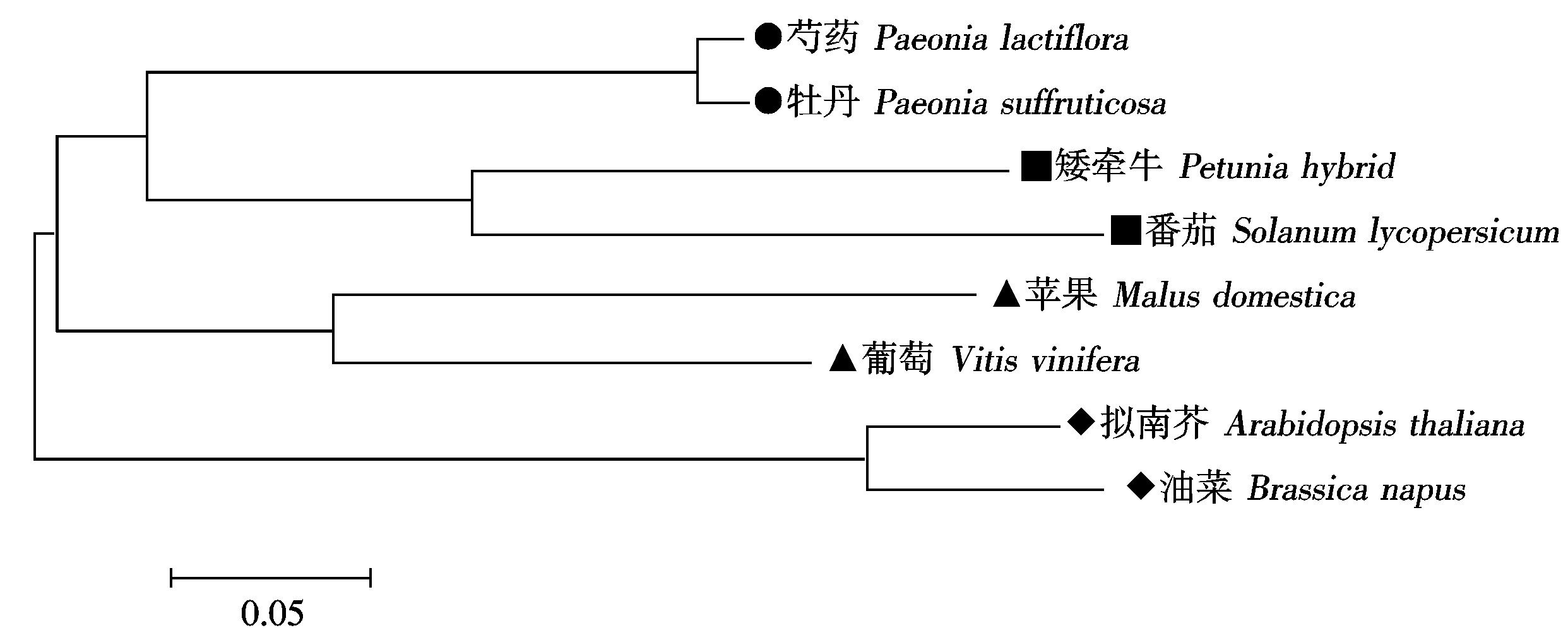
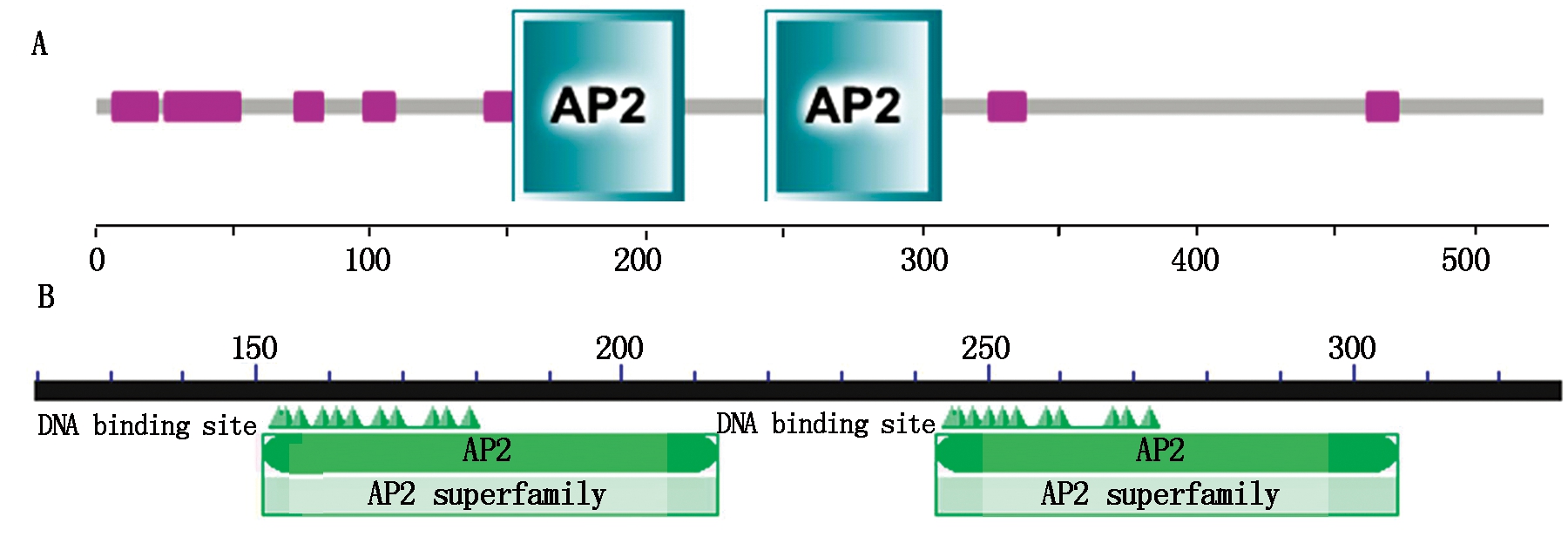
RACE扩增结果显示(图1),芍药plAP2基因5′端扩增获得2 500 bp序列,3′端扩增获得550 bp序列,经测序拼接到一条全长为1 935 bp的cDNA序列,ORF Finder分析发现该cDNA序列包括一个1 578 bp的开放阅读框(ORF),222 bp 5′端非翻译区(5′-UTR),135 bp 3′端非翻译区(3′-UTR),编码525个氨基酸(图2)。通过Blast分析发现,该基因与牡丹( Paeonia suffruticosa ,HQ222889)、拟南芥 (Arabidopsis thaliana ,NM_119856)、苹果( Malus domestica ,NM_001293950)、油菜( Brassica napus ,XM_013852206)、葡萄(Vitis vinifera,NM_001280952)、矮牵牛(Petunia hybrid ,AF132001)、番茄( Solanum lycopersicum,EU677382)同源性分别达到92.7%,45.9%,54.7%,45.7%,59.9%,58.5%,48.7%。利用MEGA 5.0构建进化树发现,芍药与牡丹聚为一类,矮牵牛和番茄聚为一类,葡萄和苹果聚为一类,拟南芥和油菜聚为一类(图3)。
2.2 蛋白结构与功能的生物信息学分析
2.2.1 基本理化性质 ProtParam程序分析蛋白理化性质结果显示,芍药plAP2蛋白分子式为C2528H3937N769O815S19,相对分子质量约为58.75 kDa,理论等电点pI为7.67,带负电荷和正电荷的氨基酸残基数分别为67,68个;plAP2蛋白脂溶指数为53.31,不稳定系数(Instability index)为51.94 (大于40),总平均亲水性(GRAVY)为-0.912,表明plAP2为不稳定、亲水性蛋白[26]。
2.2.2 跨膜螺旋与疏水性 ProtScale程序分析亲/疏水性结果显示,亲水性区域大于疏水性,并且芍药plAP2蛋白最大亲水性值处于第140位氨基酸,最大疏水性处于第45位氨基酸;TMHMM 2.0程序分析跨膜结构显示,plAP2蛋白无跨膜螺旋结构。
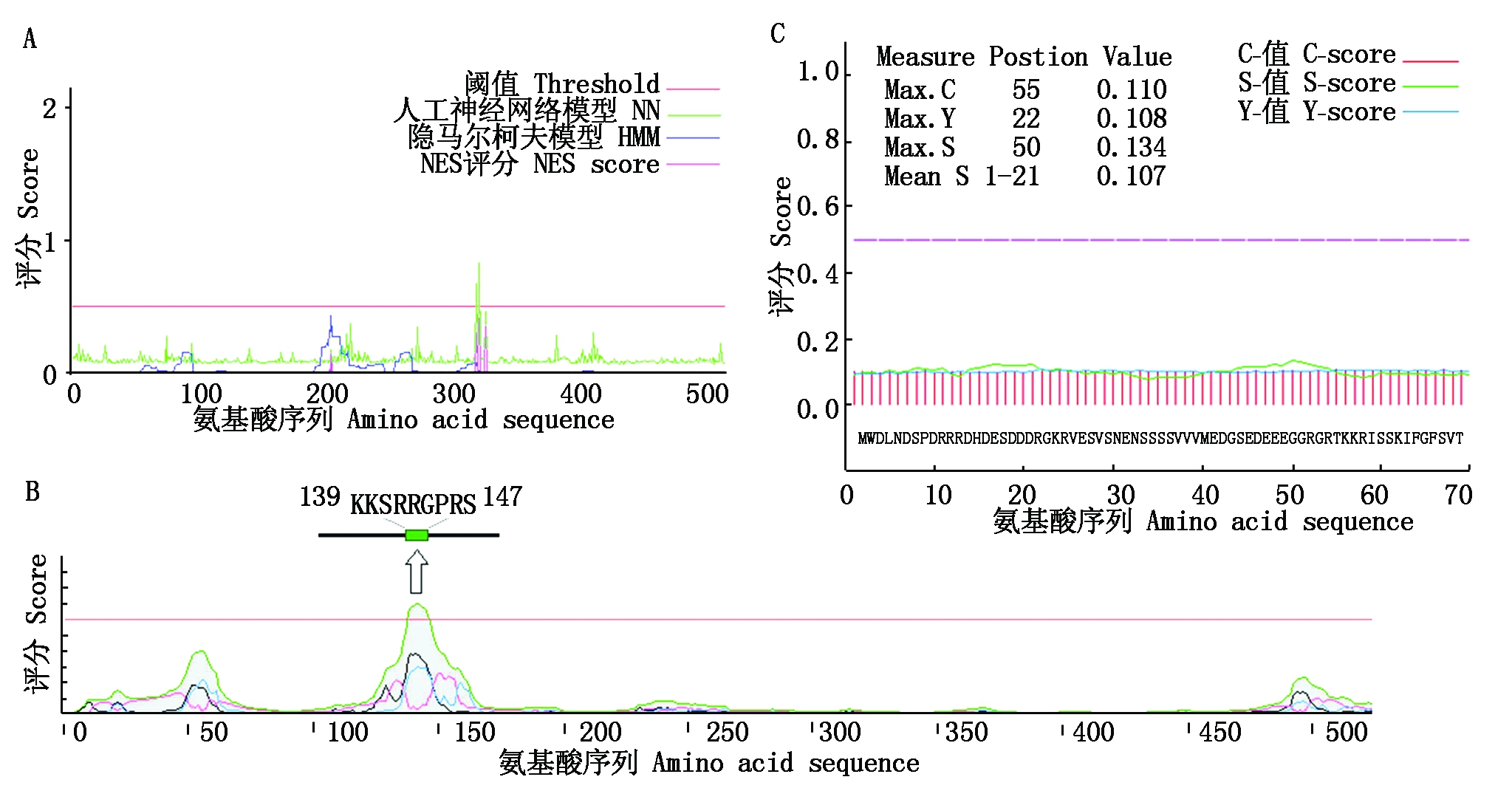
2.2.3 信号肽预测 信号肽是引导蛋白肽链进入内质网进行加工合成的一段疏水性区域(通常位于分泌蛋白N端15-35氨基酸,信号肽剪切位点有无主要根据Y最大值(Max.Y)来判断,而是否为分泌蛋白根据S平均值(Mean S)来判断,本研究结果表明(图4),Max.Y值为0.108,Mean S值为0.107(均小于0.5),表明plAP2无信号肽剪切位点,为非分泌蛋白。此外,使用NetNES 1.1 prediction预测核运出信号肽,NES评分的估计是通过HMM(隐马尔柯夫模型)和NN(人工神经网络模型)方法综合评分,结果显示plAP2蛋白不存在核运出信号肽,其次利用NLStradamus对核定位信号专门进行预测,选择隐马尔科夫模型4,阈值(Threshold)默认为0.6[18],预测结果显示(图4-B)plAP2蛋白中氨基酸序列139-147(KKSRRGPRS)为核定位信号。
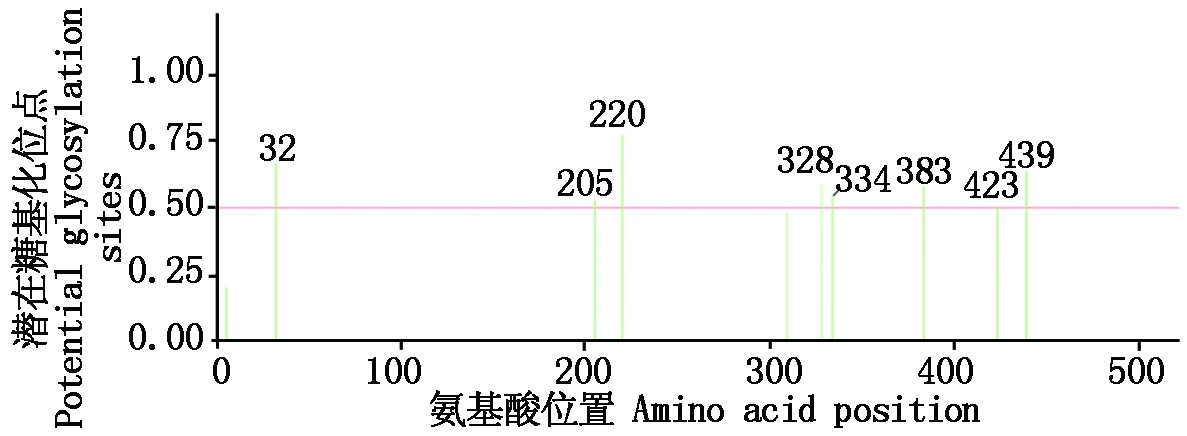
2.2.4 糖基化和磷酸化位点 NetNGlyc程序分析糖基化位点显示(图5),芍药plAP2蛋白存在8个糖基化位点,分别位于32/205/220/328/334/383/423/439号氨基酸;NetPhos软件预测磷酸化位点显示,plAP2蛋白存在64个潜在磷酸化位点,其中包括44个丝氨酸(Ser)位点、15个苏氨酸(Thr)位点和5个酪氨酸(Tyr)位点。
2.2.5 二级结构预测 Antheprot 5.0软件基于Garnier方法预测二级结构显示,plAP2蛋白中α-螺旋(α-helix)占24%,β-折叠(β-sheet)占19%,β-转角结构(β-turn)占28%,无规则卷曲(Random coil)占28%。
2.2.6 保守功能域分析 利用SMART和NCBI CDD程序综合分析保守结构域显示(图6),plAP2蛋白具有2个相同的保守结构域-AP2 domain,分别位于151-213号和243-306号氨基酸之间。研究发现,乙烯应答元件结合因子(Ethylene-responsive factors,ERF)在植物生长发育过程中起着重要作用,并且该调控元件存在于AP2结构域[27]。
2.3 亚细胞定位分析
TargetP亚细胞定位分析显示(表2),plAP2蛋白主要定位在细胞其他位置(分值:0.946),而很少分布在线粒体(分值:0.078)和信号肽分泌途径(分值:0.046)上;此外,利用PSORTⅡPrediction分析发现主要在细胞质(Cytoplasm,45.0%)中发挥生物学功能,并且在微体、线粒体基质间隙和溶酶体中也有少量分布。综合分析表明,plAP2为非分泌蛋白。
表2 plAP2蛋白的亚细胞定位分析
Tab.2 Subcellular localization analysis of plAP2 protein

名称Name长度Length线粒体靶向肽mTP分泌信号肽途径SP其他Other序列Sequence5250.0780.0460.946
2.4 芍药内外花瓣 AP2 基因差异表达分析
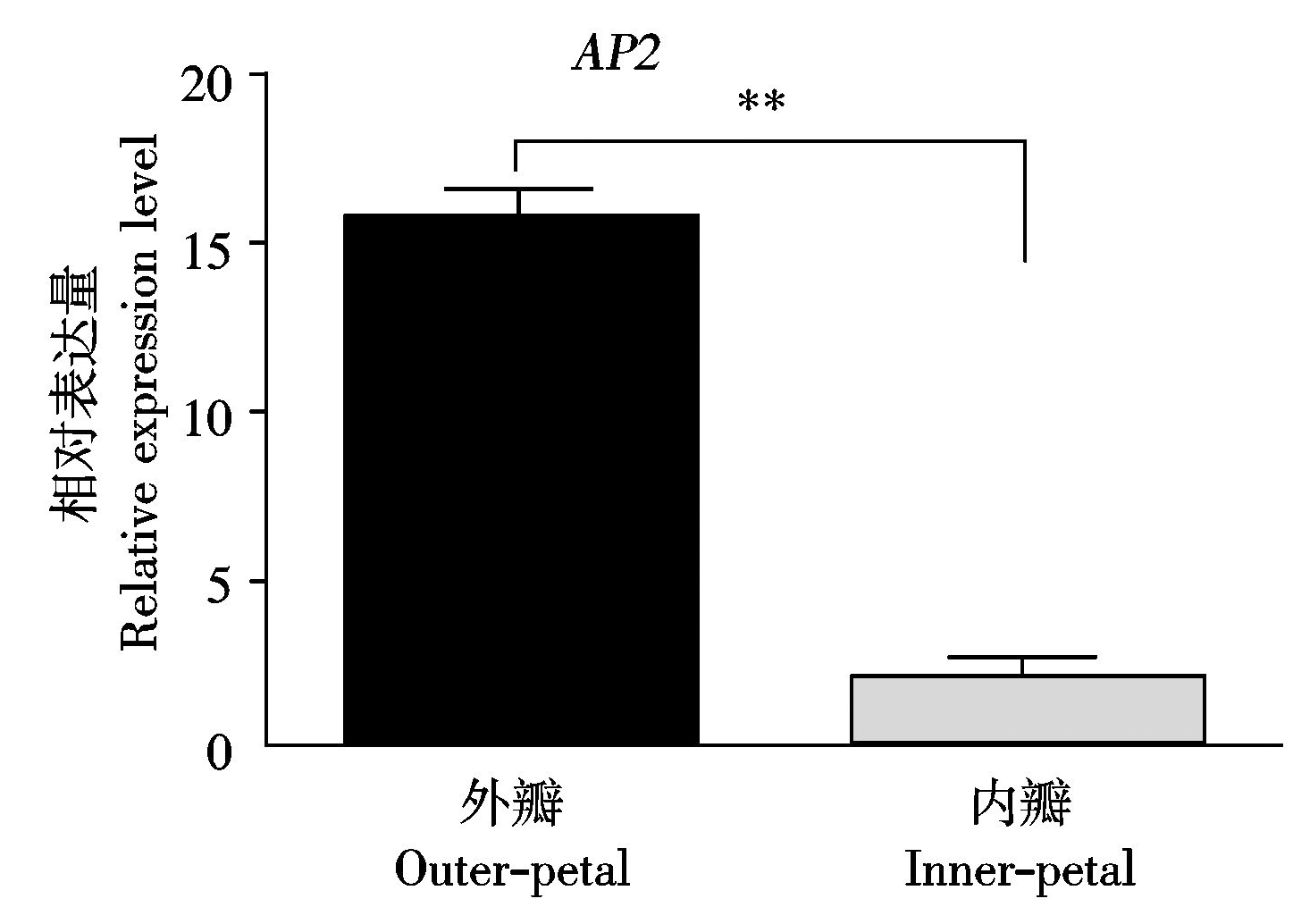
利用qPCR分析芍药花型托桂型品种向阳奇花内外花瓣(各4份)中 AP2 基因表达情况,结果显示(图7),向阳奇花外瓣 AP2 表达量均极显著高于内瓣(P<0.01)。
**.内瓣与外瓣之间AP2基因表达水平差异极显著(P<0.01)。
**.The highly significant difference of AP2 gene expression between outer-petal and inner-petal(P<0.01).
图7 芍药内外花瓣差异表达分析
Fig.7 Differential expression analysis between outer-petal and inner-petal in Paeonia lactiflora
3 讨论
目前,广泛用于解释花器官发育的分子机制主要是ABCDE模型,其中A类功能基因大多是 AP2 类转录因子基因。 AP2 基因在1994年首次在拟南芥( Arabidopsis thaliana )模式植物中被分离出[8],随后在草莓(Fragaria ananassa)、牡丹(Paeonia suffruticosa)、玉米(Zea mays)、苹果(Malus domestica)等植物中也进行了相关研究[9-12]。随着高通量测序的出现,许多物种的全基因组序列信息已经公布于GenBank数据库,如拟南芥(Arabidopsis thaliana)、玉米(Zea mays)、水稻 (Oryza sativa)、葡萄(Vitis vinifera)、苹果(Malus domestica)等物种,为基因的功能研究提供了序列参考。然而,由于芍药基因组尚未公布,芍药未知基因序列的分离与克隆陷入困境。新一代测序技术—转录组测序(RNA-Seq)的出现,可以对无参考基因组的物种进行高通量测序,因此前期对芍药花色嵌合体品种金辉花瓣进行了无参转录组de novo测序[16],获得了大量未知Unigene的部分mRNA序列,同时cDNA末端的快速扩增RACE是一种以部分的已知区域序列为起点,扩增基因转录本未知区域,从而获得 mRNA(cDNA)完整序列的有效方法,本研究参考转录组测序并运用RACE方法成功克隆出芍药AP2(plAP2)基因cDNA全长序列1 935 bp,为今后利用qPCR、过表达等技术来深入研究plAP2基因功能提供有利的素材。
研究发现,AP2类转录因子参与乙烯、脱落酸等有机酸合成信号传导途径,在植物生长、花器官发育、抗逆性等过程中发挥重要的生物学功能[28-30]。一般而言,基因发挥生物学功能与蛋白翻译后修饰位点、保守结构域密切相关。本研究利用生物信息学预测发现plAP2蛋白具有64个潜在磷酸化位点和8个糖基化位点,并且含有2个保守的结构域-AP2(也叫Ethylene-responsive factor,ERF),这与Jofuku等[8]在拟南芥中研究结果一致。此外,有关学者在烟草(Nicotiana tabacum L.)中同样分离出乙烯响应元件结合蛋白ERF结构域[31],由此表明AP2/ERF是植物中一个保守的基因家族。本研究在芍药AP2蛋白中预测出1个核定位信号肽(139-147)以及2个ERF保守结构域(151-213位和243-306位氨基酸),为今后筛选功能性SNPs位点以及利用基因沉默技术(RNAi和CRISPR/Cas9)来验证plAP2基因功能提供理论参考。此外,虽然有研究发现AP2/ERF转录因子基因家族的结构和进化在拟南芥、水稻和大豆中是相对保守的[32],但是ABCDE模型在不同双子叶植物类群中保守性差异很大[33]。研究发现,水稻ERF结构域家族基因具有XI-XIV亚组,而在拟南芥中没有发现此亚组基因[34],并且水稻中部分基因虽然与拟南芥、金鱼草等物种具有高度同源性,但是在发挥生物学功能上出现一定的区别[35]。本研究发现芍药AP2基因与牡丹高度同源(达90%以上),而与拟南芥、葡萄、矮牵牛等植物同源性较低(接近50%),由此推测花器官发育模型ABCDE的作用机制是否完全适用于芍药还有待进一步研究。
参考文献:
[1] 曾 英, 胡金勇, 李志坚.植物MADS盒基因与花器官的进化发育[J].植物生理学通讯,2001,37(4):281-287.
[2] Coen E S,Meyerowitz E M.The war of the whorls:genetic interactions controlling flower development[J].Nature,1991,353(6339):31-37.
[3] Angenent G C,Franken J,Busscher M,et al.A novel class of MADS box genes is involved in ovule development in petunia[J].The Plant Cell,1995,7(10):1569-1582.
[4] Colombo L,Franken J,Koetje E,et al.The petunia MADS box gene FBP11 determines ovule identity[J].The Plant Cell,1995,7(11):1859-1868.
[5] Pelaz S,Ditta G S,Baumann E,et al.B and C floral organ identity functions require SEPALLATA MADS-box genes[J].Nature,2000,405(6783):200-203.
[6] Pelaz S,Gustafson-Brown C,Kohalmi S E,et al.APETALA1 and SEPALLATA3 interact to promote flower development[J].The Plant Journal:for Cell and Molecular Biology,2001,26(4):385-394.
[7] Maes T,Van De Steene N,Zethof J,et al.Petunia Ap2-like genes and their role in flower and seed development[J].The Plant Cell,2001,13(2):229-244.
[8] Jofuku K D,Den Boer B G,Van Montagu M,et al.Control of arabidopsis flower and seed development by the homeotic gene APETALA2[J].The Plant Cell,1994,6(9):1211-1225.
[9] 任 磊.牡丹花器官发育相关基因的克隆与表达[D].北京: 中国林业科学研究院,2011.
[10] 宿红艳, 周盛梅, 王 磊, 等.草莓APETALA2同源基因的克隆及表达分析[J].西北植物学报,2005,25(10):1937-1942.
[11] Zhuang J,Deng D X,Yao Q H,et al.Discovery,phylogeny and expression patterns of AP2-like genes in maize[J].Plant Growth Regulation,2010,62(1):51-58.
[12] Zhuang J,Yao Q H,Xiong A S,et al.Isolation,phylogeny and expression patterns of AP2-like genes in apple(Malus×domestica Borkh) [J].Plant Molecular Biology Reporter,2011,29(1):209-216.
[13] 罗睿雄, 赵志常, 高爱平, 等.芒果4CL基因的克隆及其表达分析[J].华北农学报,2016,31(1):57-62.
[14] 雷海英, 白凤麟, 刘建霞, 等.玉米Zmcen基因的克隆,表达与生物信息学分析[J].华北农学报,2016,31(3):18-24.
[15] 侯艳霞, 汤浩茹, 林源秀, 等.基于RACE方法的草莓Faapx-c基因克隆、表达及其生物信息学分析[J].北方园艺,2016(11):88-92.
[16] Zhao D,Jiang Y,Ning C,et al.Transcriptome sequencing of a chimaera reveals coordinated expression of anthocyanin biosynthetic genes mediating yellow formation in herbaceous peony (Paeonia lactiflora Pall.) [J].BMC Genomics,2014,15(1):689.
[17] Petersen T N,Brunak S,Von Heijne G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions[J].Nature Methods,2011,8(10):785-786.
[18] Nguyen Ba A N,Pogoutse A,Provart N,et al.NLStradamus:a simple Hidden Markov Model for nuclear localization signal prediction[J].BMC Bioinformatics,2009,10(1):202.
[19] La Cour T,Kiemer L,Mølgaard A,et al.Analysis and prediction of leucine-rich nuclear export signals[J].Protein Engineering,Design & Selection,2004,17(6):527-536.
[20] Mellquist J L,Kasturi L,Spitalnik S L,et al.The amino acid following an asn-X-Ser/Thr sequon is an important determinant of N-linked core glycosylation efficiency[J].Biochemistry,1998,37(19):6833-6837.
[21] Kim J H,Lee J,Oh B,et al.Prediction of phosphorylation sites using SVMs[J].Bioinformatics,2004,20(17):3179-3184.
[22] Blom N,Gammeltoft S,Brunak S.Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J].Journal of Molecular Biology,1999,294(5):1351-1362.
[23] Marchler-Bauer A,Derbyshire M K,Gonzales N R,et al.CDD:NCBI′s conserved domain database[J].Nucleic Acids Research,2015,43:222-226.
[24] Schultz J,Milpetz F,Bork P,et al.SMART,a simple modular architecture research tool:identification of signaling domains[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(11):5857-5864.
[25] Emanuelsson O,Nielsen H,Brunak S,et al.Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J].Journal of Molecular Biology,2000,300(4):1005-1016.
[26] Wilkins M R,Gasteiger E,Bairoch A,et al.Protein identification and analysis tools in the ExPASy server[J].Methods in Molecular Biology,1999,112(112):531-552.
[27] Weigel D.The APETALA2 domain is related to a novel type of DNA binding domain[J].The Plant Cell,1995,7(4):388-389.
[28] 丰 锦, 陈信波.抗逆相关AP2/EREBP转录因子研究进展[J].生物技术通报,2011(7):1-6.
[29] 赵利峰, 柴团耀.AP2/EREBP转录因子在植物发育和胁迫应答中的作用[J].植物学通报,2008,25(1):89-101.
[30] 韩志萍, 安利佳, 侯和胜.AP2/EREBP转录因子的结构与功能[J].中国农学通报,2006,22(2):33-38.
[31] Ohme-Takagi M,Shinshi H.Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element[J].The Plant Cell,1995,7(2):173-182.
[32] Zhuang J,Anyia A,Vidmar J,et al.Discovery and expression assessment of the AP2-like genes in Hordeum vulgare[J].Acta Physiologiae Plantarum,2011,33(5):1639-1649.
[33] 张 剑, 徐桂霞, 薛皓月, 等.植物进化发育生物学的形成与研究进展[J].植物学通报,2007,24(1):1-30.
[34] 张计育, 王庆菊, 郭忠仁.植物AP2/ERF类转录因子研究进展[J].遗传,2012,4(7):835-884.
[35] Kyozuka J,Kobayashi T,Morita M,et al.Spatially and temporally regulated expression of rice MADS box genes with similarity to Arabidopsis class A,B and C genes[J].Plant & Cell Physiology,2000,41(6):710-718.
Cloning and Bioinformatics Analysis of APETALA2 (AP2) in Paeonia lactiflora
WU Yanqing1,CHENG Menglin2,ZHAO Daqiu2,TAO Jun1,2
(1.College of Animal Science and Technology,Yangzhou University,Yangzhou 225009,China;2.School of Horticulture and Plant Protection,Yangzhou University,Yangzhou 225009,China)
Abstract:Flower-meristem-identity geneAPETALA2(AP2) belonging to ABCDE model plays a role in regulating the development of plant floral organ.In order to understand the biological function of P.lactiflora AP2 gene,the plAP2 gene sequence of Paeonia lactiflora was cloned using RACE and sequencing technique technology,meanwhile the sequence signature,protein structure and function,subcellular localization were analyzed by bioinformatics methods.Molecular phylogenetic tree of AP2 from different plants were constructed by MEGA 5.0 software.Finally,we detected the differential expression between inner-petal and outer-petal by qPCR.Results showed,the full-length cDNA sequence of plAP2 gene was 1 935 bp and consisted of a 1 578 bp open reading frame (ORF) encoding 525 amino acid proteins.Analysis of protein structure and function showed,plAP2 was a hydrophilic and unstable protein,meanwhile plAP2 was also non-secretory protein without signal peptide and transmembrane structure.Nuclear localization signal located between 139th and 147th amino acid (KKSRRGPRS).The secondary structure of plAP2 protein contained 24% α-helices,19% β-sheet,28% β-turn and 28% random coil.plAP2 protein had eight glycosylation sites and sixty-four phosphorylation sites.plAP2 protein had two same conservative domain:AP2/ Ethylene- Responsive factors (ERF), which was located in the region between 151 and 213,between 243 and 306,respectively.Prediction of subcellular localization showed that plAP2 located mostly in cytoplasm (45.0%),rarely in microbody, mitochondrial matrix and lysosome.Analysis of evolutionary tree showed that the AP2 of Paeonia lactiflora were highly homologous and close to Paeonia suffruticosa.qPCR showed the expression of AP2 in outer-petal was extremely significant than that in inner-petal (P<0.01).In this study,we cloned the complete cDNA sequence of AP2 gene in Paeonia lactiflora and systematically revealed the basic protein structure,functional sites,domains,cellular localization and expression level,which will provide basic materials and theoretical references for further studying plAP2 gene function.
Key words:Paeonia lactiflora; APETALA2 gene; RACE; Bioinformatics
收稿日期:2017-04-11
基金项目:国家自然科学基金项目(31372097;31400592);江苏省农业科技自主创新资金项目(CX(12)2019)
作者简介:吴彦庆(1988-),男,江苏扬州人,博士,主要从事观赏园艺研究。
通讯作者:陶 俊(1966-),男,江苏扬州人,教授,博士,主要从事观赏植物栽培生理、遗传育种与分子生物学研究。
中图分类号:Q78; S682.03
文献标识码:A
文章编号:1000-7091(2017)03-0058-07
doi:10.7668/hbnxb.2017.03.009