松墨天牛(Monochamus alternatus),又名松天牛、松褐天牛,鞘翅目( Coleoptera),天牛科( Cerambycidae),墨天牛属( Monocham us),是林业上重要的蛀干害虫,能危害多种松树,幼虫钻蛀树干和大枝条,取食其韧皮部与木质部,同时其成虫体内能携带成千上万的松材线虫,这些松材线虫对松树能产生极其严重的病害—松材线虫病,严重影响生态与经济[1-2]。
Hox基因,即同源异形盒基因(Homeobox gene),是生物发育调控基因中的一个古老的基因簇[3-4]。在生物体的体轴建立、胚胎发生、器官组织发育方面起着决定性的作用[5-7]。在Hox基因中都包含有一个保守的Hox功能结构域,主要功能是与DNA相结合[8]。Homothorax基因(Hth),是在对果蝇Hox基因的辅因子extradenticle研究时发现的另一种辅因子[9-10]。作为Hox基因的辅因子,该基因同样也包含Hox保守功能域,在对果蝇Hth的研究中,发现该基因在胚胎及成虫发育中扮演重要的角色[11-12]。Hth是许多组织器官正常发育所必需的,是触角发育的决定因子[13],调控足与翅的末端形成[14-15],对于眼睛发育具有一定的负调控作用[16];在许多发育的进程中,参与转录的激活[17-18],既可以激活靶标基因又可以抑制靶标基因[19],在许多细胞类型中表达、变异会对在胚胎及成虫的发育具有巨大的影响,这也因此证实了Hth调控许多靶标基因,比如激活诱导蜕皮激素相关的靶基因[20]。
松墨天牛生长发育都离不开Hox基因的调控,笔者推测,Hth基因作为Hox基因的辅因子,在松墨天牛生理活动中发挥重要作用。本研究克隆到了Homothorax基因,首次对其进行生物信息学分析及表达分析,以期为阐明该基因在对松墨天牛生长发育中的调控作用提供参考。
1 材料和方法
1.1 试验材料
1.1.1 供试昆虫 松墨天牛幼虫由江苏林科院馈赠,用人工饲料在避光的人工气候箱内饲养至蛹、成虫,饲养温度为25 ℃,相对湿度为70%~ 75%。
1.1.2 主要试剂 E.Z.N.A.TM Total RNA Kit Ⅱ总 RNA提取试剂盒购自 OMEGA公司,PrimeScript® RT reagent Kit With gDNA Eraser 反转录试剂盒和SYBR® Premix Ex TaqTM实时荧光定量试剂盒购自 TaKaRa 公司。
1.2 试验方法
1.2.1 样品处理、RNA的提取及 cDNA的合成 选取20头2日龄成虫、30头3龄幼虫进行清洗、消毒、麻醉后,解剖并获取成虫触角、头(去触角)、前胸、具翅胸节、足、鞘翅、马氏管、中肠、卵巢,幼虫脂肪体、马氏管、头、体壁,用磷酸盐缓冲液冲洗后,将样本分别置于无菌去酶的EP管中,立即液氮冷冻,放入-70 ℃低温冰箱中保存备用。
用E.Z.N.ATM Total RNA Kit Ⅱ总RNA提取试剂盒提取总RNA,具体操作按该试剂盒提供提取说明书进行。经琼脂糖凝胶电泳与微量紫外分光光度计(Nanodrop 2000)检测合格后置于-70 ℃保存备用。按PrimeScript® RT reagent Kit With gDNA Eraser 反转录试剂盒提供说明书操作合成第一链cDNA,用于RT-qPCR的反应模板,置于-20 ℃保存备用。
1.2.2 基因克隆 已构建好的松墨天牛cDNA文库中克隆出Homothorax基因完整编码序列[21]。
1.2.3 生物信息学分析 用 NCBI 在线工具 ORF finder(http;//www.ncbi.nlm.nih.gov/gorf.html )搜索开放阅读框,ProtScale 程序分析疏水性,SignalP 4.0 Server(http://www.cbs.dtu.dk/services/Signalp/ )预测信号肽,NetPhos(http://www.cbs.dtu.dk/services/NetPhos/ )预测磷酸化修饰位点,Ne-tNGlye软件分析N-糖基化修饰(http://www.cbs.dtu.dk/ser-vices/NetNGlyc/),亚细胞定位( http://ipsort.hgc.ip/),TMHMM 分析蛋白质跨膜结构域(http://www.cbs.dtu.dk/services/TMH-MM/),SMART 软件(http://smart.embl-heidel berg.de/)分析蛋白质功能结构域,COILS Server(http://embnet.vital-it.ch/software/COILS form.html)预测卷曲螺旋,SOPMA 软件( http:// npsa-pbil.ibcp. fr/cgi-bin/secpred_sopma.pl)预测二级结构,SWISSMODEL 软件(http://beta.swiss-model.expasy.o-rg/)预测三级结构和同源建模。从NCBI数据库中检索昆虫Hth基因编码序列,并下载昆虫的Hth基因编码序列(表1),用于多序列同源性比对(DNAMAN软件),系统发育树(NJ法)用Clustal X和MEGA 4.0软件构建。
1.2.4 基因表达 用RT-qPCR( SYBR Green) 检测基因的表达量。所用引物:上游:5′-CGGACTCAACATCACACAC-3′;下游:5′-GGACACCACTTGCCCATAG-3′。反应程序:95 ℃ 预变性 5 min; 95 ℃ 10 s,60 ℃ 20 s, 进行40 个循环。在LightCycler480 荧光定量 PCR 仪上进行RT-qPCR反应,每个试验样品设 3 次重复,反应结束后采集目标基因的 Ct 值和内参基因(β-actin)的 Ct 平均值,采用 2-ΔΔCt相对定量法计算相对表达量。用 SPSS 18.0 软件的 Duncan′s单因素方差分析法(ANOVA)对荧光定量所得数据进行统计分析。
2 结果与分析
2.1 基因序列及其编码氨基酸序列分析
从松墨天牛cDNA文库中筛选一条具有完整 ORF 的序列,在 NCBI 的核酸序列数据库中进行Blast 程序同源性比对,鉴定为松墨天牛Homothorax基因, 命名为 MaHth(GenBank:KX364396 ),其开放阅读框长度为1 347 bp,共编码 448个氨基酸(图1),推测的编码蛋白分子量为 48.99 kDa, 等电点pI为 4.66,不稳定系数为53.82,脂肪系数为62.75。用ProtScale对蛋白进行疏水性分析,发现该蛋白氨基酸大部分为亲水性的,说明此蛋白为亲水蛋白。信号肽预测结果表明该蛋白无信号肽;跨膜结构预测结果表明该蛋白为跨膜蛋白,有1个由外到内、2个从内到外的螺旋结构;磷酸化修饰位点预测结果为:有丝氨酸磷酸化位点22个,苏氨酸磷酸化位点4个,酪氨酸磷酸化位点4个, N-糖基化修饰位点1个。预测其存在2个独立的核定位信息:KRDK、KKNQKKR。
表1 参与Hth氨基酸序列比对的16种昆虫
Tab.1 Sixteen species of insects participating in Hth amino acid sequence alignment
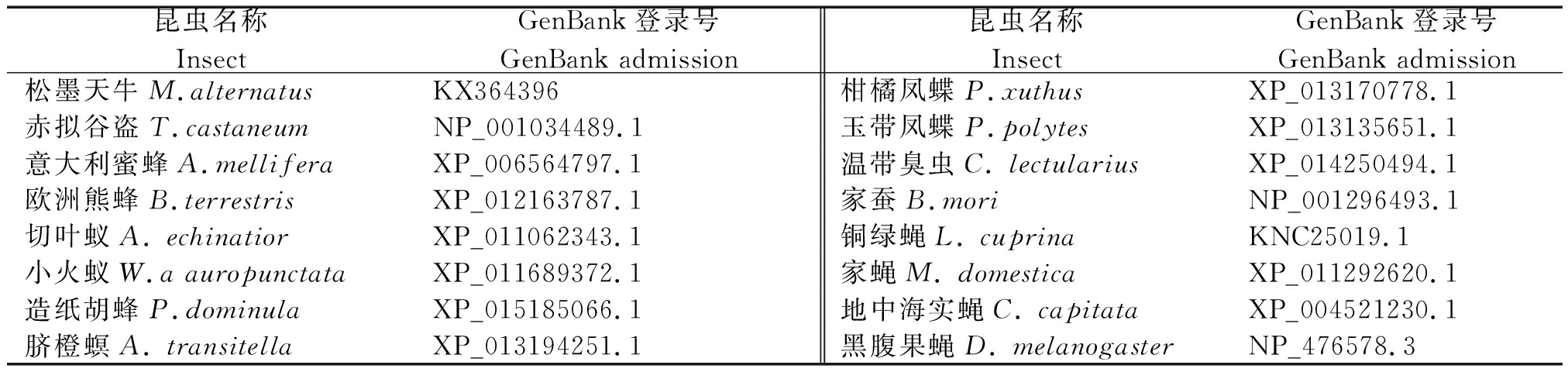
昆虫名称InsectGenBank登录号GenBank admission昆虫名称InsectGenBank登录号GenBank admission松墨天牛M.alternatusKX364396柑橘凤蝶P.xuthusXP_013170778.1赤拟谷盗T.castaneumNP_001034489.1玉带凤蝶P.polytesXP_013135651.1意大利蜜蜂A.melliferaXP_006564797.1温带臭虫C. lectulariusXP_014250494.1欧洲熊蜂B.terrestrisXP_012163787.1家蚕B.moriNP_001296493.1切叶蚁A. echinatiorXP_011062343.1铜绿蝇L. cuprinaKNC25019.1小火蚁W.a auropunctataXP_011689372.1家蝇M. domesticaXP_011292620.1造纸胡蜂P.dominulaXP_015185066.1地中海实蝇C. capitataXP_004521230.1脐橙螟A. transitellaXP_013194251.1黑腹果蝇D. melanogasterNP_476578.3

丝氨酸(Ser)、苏氨酸(Thr)、酪氨酸(Tyr)磷酸化位点分别以黑体加粗、方形、三角形标注;N-糖基化位点以圆形标注;核定位信号用方框标注;2个低复杂度区域用阴影与横线标注;Hox保守区用波浪线标注;终止密码子TGA用星号标注。
Serine phosphorylation sites,Threonine phosphorylation sites,Tyrosine phosphorylation sites, N-Glycosylation sites and nucleus localization signal are underlined with bold,square,triangles, round, box respectively;Two Low complexity region and conserved domain are highlighted in shadow with horizontal line and wavy lines, respectively;Stop codon is marked with an asterisk.
图1 MaHth 基因ORF序列全长及预测的氨基酸序列
Fig.1 ORF and its predicted amino acid sequences of MaHth
2.2 功能结构域及其亚细胞定位
通过SMART软件对MaHth蛋白进行功能结构域分析,该蛋白有2个低复杂度区域(Low complexity regions,LCRs),即TDARAATDAARR(284-295位)和GEGTGEEDDDT(318-328位),在333-398位氨基酸处为Hox结构域(图2)。通过PSORT软件在线预测MaHth蛋白在细胞中的位置,结果表明,定位在细胞核的可能性最大,为97%,据此推测MaHth蛋白定位于细胞核中。
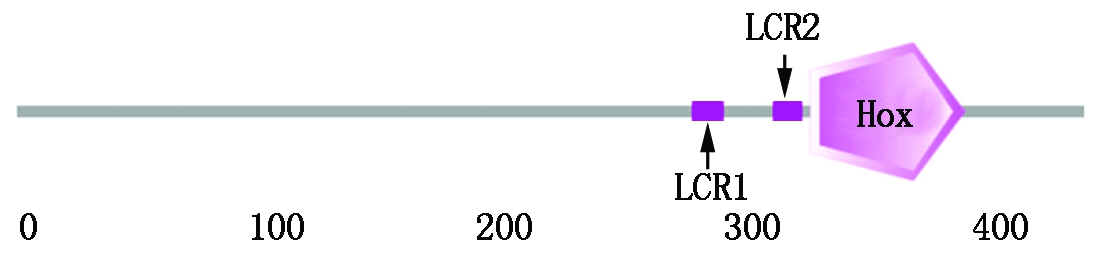
LCR1与LCR2分别表示2个低复杂度区域。
LCR1 and LCR2 represent two low complexity regions,respectively.
图2 MaHth蛋白结构域的预测
Fig.2 Predicted domains in MaHth
2.3 二级和三级结构
由软件SOPMA对MaHth蛋白进行二级结构预测,结果显示该蛋白的二级结构由α螺旋、无规则卷曲、β转角与延伸链组成,分别占28.12%,50.22%,11.16%,10.49%。由此可推测MaHth蛋白的主要二级结构元件为α螺旋和无规则卷曲。运用SWISS-MODEL进行同源建模,结果同样显示此基因编码蛋白主要由α螺旋与无规则卷曲组成,该预测结果与其二级结构预测的结果一致。由预测的三级结构可知,MaHth蛋白有3个螺旋,被2个β转角相连,构成以α螺旋-β转角-α螺旋为基元的结构(图3)。
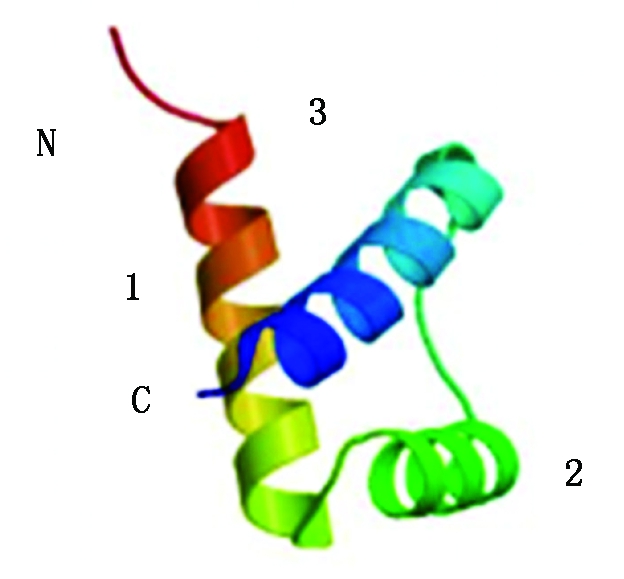
图中1、2、3表示3个α螺旋;N、C表示蛋白N端与C端。
The three alpha helices are numbered; N and C indicate N-terminal and C-terminal ends of the protein, respectively.
图3 MaHth三维结构预测
Fig.3 Three-dimensional structure of MaHth
2.4 同源序列比对及系统发育树
MaHth与赤拟谷盗Hth蛋白同源性最高,为97%;与地中海实蝇、黑腹果蝇、铜绿蝇、家蝇4种双翅目昆虫同源性在70%~80%;与柑橘凤蝶等10种昆虫同源性在80%~90%(图4)。基于MEGA 4.0软件构建的系统发育树显示:松墨天牛与赤拟谷盗处于同一分支,与同源性分析的结果一致(图5)。


图4 MaHth与15种昆虫Hth氨基酸序列同源性比对
Fig.4 Amino acid sequence alignment of Hth from M.alternatus and other insects
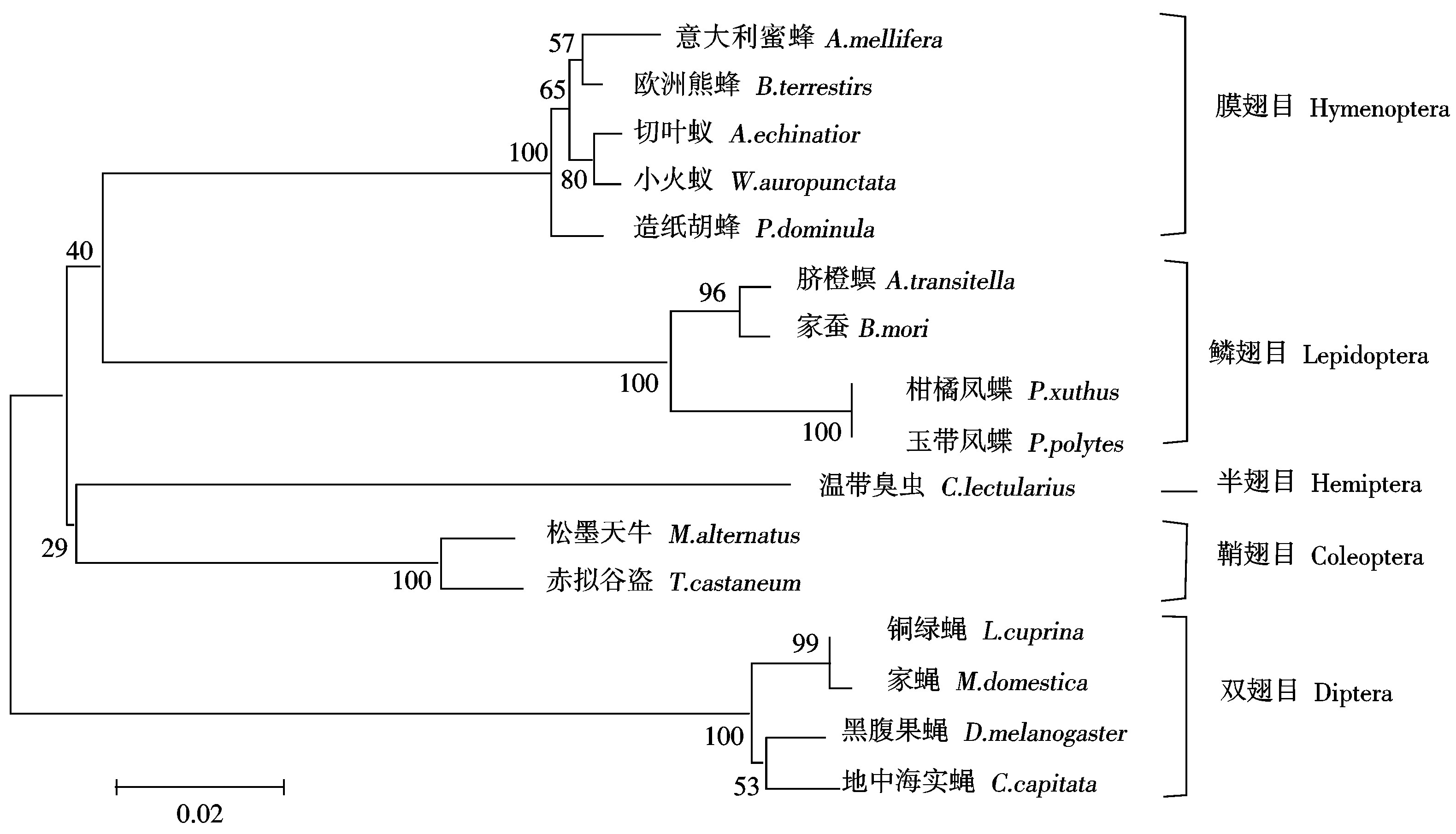
图5 基于昆虫Hth氨基酸序列构建的系统发育树
Fig.5 Phylogenetic relationships based on Hth amino acids from insects
2.5 MaHth基因的表达
MaHth基因在松墨天牛各虫态、组织、部位中广泛表达。在卵中表达量最高,成虫表达量最低,幼虫与蛹的表达量分别为卵的0.76,0.95倍,卵与幼虫、成虫之间基因表达差异显著(P<0.05),与蛹表达差异不显著(P>0.05)(图6)。幼虫各组织中,MaHth在头部表达量最高,为对照表达量的13倍,体壁次之,为对照的3.18倍,脂肪体、马氏管的表达量分别是对照的1.88,0.91倍,幼虫各组织之间MaHth基因表达均有显著差异(P<0.05)(图7)。在成虫中,MaHth在马氏管与头中表达量相对较高,分别为对照的2.87,2.75倍,两者差异不显著(P>0.05),在足、翅、触角中的表达量低于对照,分别为对照的0.74,0.19,0.07倍,三者表现差异显著(P<0.05),在前胸、具翅胸、中肠、卵巢中的表达量分别是对照的1.1,1.32,1.72,1.41倍(图8)。
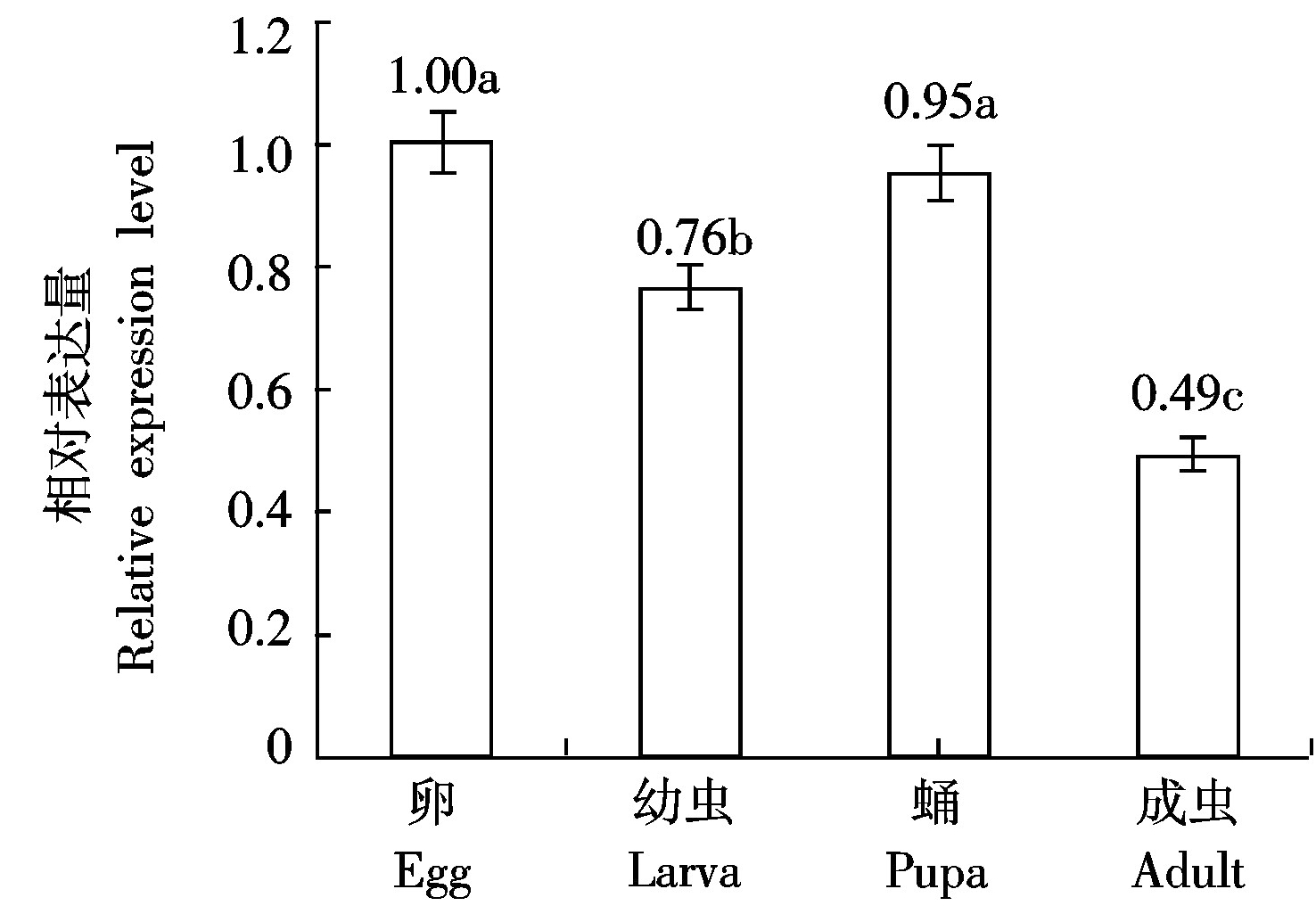
图中数据为平均数±标准误;柱上不同字母表示
差异显著(P<0.05);图7-8同。
Data in the figure are represented as mean±SE;Different letters mean
significant difference on the bar(P<0.05);The same as Fig.7-8.
图6 MaHth基因在松墨天牛各虫态中的表达
Fig.6 The expression level of MaHth indevelopment stages of M.alternatus

图7 MaHth基因在松墨天牛幼虫中的表达
Fig.7 The expression level of MaHth inthe larvae of M.alternatus
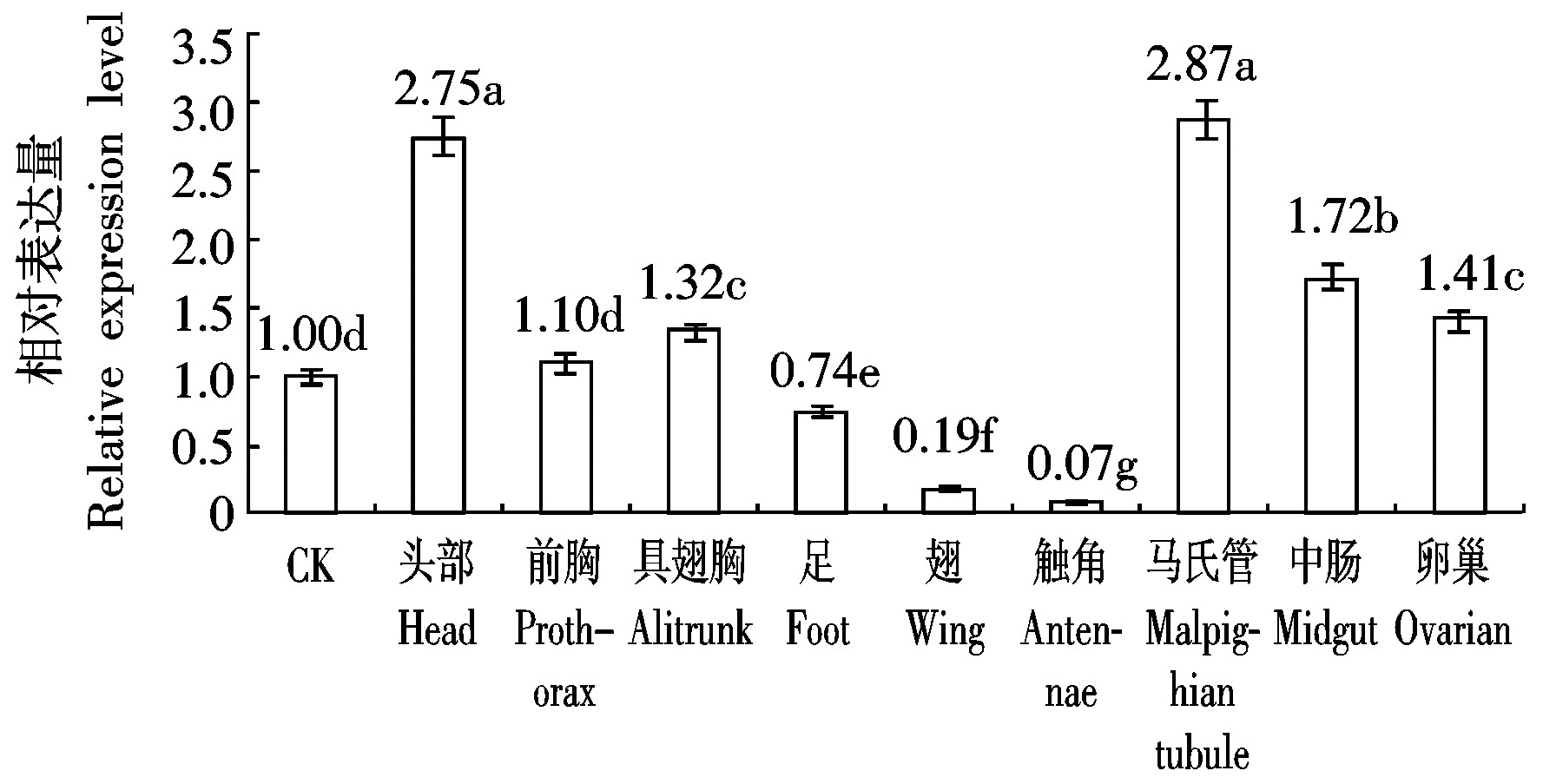
图8 MaHth基因在松墨天牛成虫中的表达
Fig.8 The expression level of MaHth inthe adults of M.alternatus
3 讨论
Hox基因属于同源异型盒家族成员之一,在大多数Hox基因中,会包含一段约180个核苷酸的同源异型盒,可以转录出约60个氨基酸序列,称为Hox保守区段 [22]。Hth是Hox基因辅因子之一,属于Meis家族,该家族也有大概60个氨基酸序列的Hox保守区。本研究发现,16种昆虫Hth蛋白都有相同序列的Hox保守结构域,即66个氨基酸序列的同源异型盒,且在Hox结构域里有3个螺旋,在第一个螺旋与第2个螺旋之间有proline(P)-tryosin(Y)-tryosin(P)3个氨基酸延伸环[23];通过MaHth蛋白三级结构预测发现,MaHth也是由3个螺旋构成,在第1个与第2个螺旋中也找到了该延伸环。因此,MaHth属于Meis家族成员。
在大部分真核生物蛋白中会存在低复杂度区域(Low-complexity regions,LCRs)[24-26],低复杂度区域包含单个氨基酸或者是一段短的氨基酸重复[27],许多LCRs是高度不稳定的,或丢掉,或重组[28]。在本研究中,MaHth蛋白有2个LCRs,分别位于284-295位氨基酸(TDARAATDAARR)和318-328位氨基酸(GEGTGEEDDDT),它们的存在可能会促进自身表型的变化来适应环境的变化[29],同时也可能会促进新蛋白序列的形成[27]。
蛋白质经折叠、修饰、组装成熟后必须要分拣到特定的细胞部位才能真正发挥其特有的生物学功能,其亚细胞定位信息会暗示蛋白质的生物学功能[30]。Jaw等[31]认为在Hth蛋白序列中可能存在2个独立的核定位信号:KRDK、KKNQKKR。我们的研究发现MaHth蛋白同样含有这2个独立的核定位信号,分别定位于73-76位氨基酸,331-337位氨基酸,由此我们认为MaHth蛋白定位于细胞核内。研究核定位信号一方面可以帮助揭示新的大分子物质核转运机制,另一方面也有助于发现一些蛋白质的新功能[32]。
松墨天牛MaHth在卵、蛹中的表达量高于幼虫、成虫,推测MaHth与胚胎期、蛹期中器官组织形成与分化有关;在幼虫中,MaHth头部表达量最高,为对照的13倍,体壁为对照的3.18倍,推测MaHth与松墨天牛头部生长、体壁的生成相关;在成虫中,MaHth在马氏管中表达量最高,为对照的2.87倍,而在幼虫体内马氏管的表达量相对较低,可能由于在蛹期里马氏管不断进行更新改造[30],所以推测该基因与马氏管的发育有一定关系。在松墨天牛的一些附肢,如足、翅、触角,MaHth表达量相对较低,而在其他一些昆虫体内证实,作为Hox基因辅助因子的Hth基因与足的近端、翅、触角[13-15]的发育也相关,但是本研究对这些附肢的基因表达分析,并没有得到在这些部位大量表达的结果,可能相比于成虫,MaHth基因对卵与蛹期的调控作用更加明显,MaHth基因对这些附肢的调控作用主要可能作用于蛹。
[1] 张心团,赵和平,樊美珍,等.松墨天牛生物学特性的研究进展(综述) [J].安徽农业大学学报, 2004, 31(2):156-157.
[2] 王玲萍.松墨天牛生物学特性的研究[J].福建林业科技, 2004, 31(3):23-26.
[3] Mcginnis W, Krumlauf R. Homeobox genes and axial patterning[J]. Cell, 1992, 68(2):283-302.
[4] Heffer A, Pick L. Conservation and variation in Hox genes:how insect models pioneered the evo-devo field[J]. Annual Review of Entomology, 2013, 58(1):161-179.
[5] Pearson J C, Lemons D, Mcginnis W. Modulating Hox gene functions during animal body patterning[J]. Nature Reviews Genetics, 2005, 6(12):893-904.
[6] Mcginnis W, Garber R, Wirz J, et al. A homologous protein-coding sequence in Drosophila homeotic genes and its conservation in other metazoans[J]. Cell, 1984, 37(2):403-408.
[7] Kumar R, Chotaliya M, Vuppala S, et al. Role of homothorax in region specific regulation of deformed in embryonic neuroblasts[J]. Mechanisms of Development, 2015, 138(Pt 2):190-197.
[8] Bürglin T, Affolter M. Homeodomain proteins:an update[J]. Chromosoma, 2016, 125(3):497-521.
[9] Potts M, Wang D, Cameron S. Trithorax, Hox, and TALE-class homeodomain proteins ensure cell survival through repression of the BH3-only gene egl-1[J]. Developmental Biology, 2009, 329(2):374-385.
[10] Wernet M, Desplan C. Homothorax and extradenticle alter the transcription factor network in Drosophila ommatidia at the dorsal rim of the retina[J]. Development,2014,141(4):918-928.
[11] Smith F, Angelini D, Jockusch E. A functional genetic analysis in flour beetles (Tenebrionidae) reveals an antennal identity specification mechanism active during metamorphosis in Holometabola[J]. Mechanisms of Development, 2014, 132(3):13-27.
[12] Smith F, Jockusch E. Hox genes require homothorax and extradenticle for body wall identity specification but not for appendage identity specification during metamorphosis of Tribolium castaneum[J]. Developmental Biology, 2014, 395(1):182-197.
[13] Casares F, Mann R. Control of antennal versus leg development in Drosophila[J]. Nature, 1998, 392(6677):723-726.
[14] Abu-Shaar M, Mann R. Generation of multiple antagonistic domains along the proximodistal axis during Drosophila leg development[J]. Development, 1998, 125(19):3821-3830.
[15] Wu J, Cohen S. Proximodistal axis formation in the Drosophila leg:subdivision into proximal and distal domains by Homothorax and Distal-less[J]. Development,1999,126(1):109-117.
[16] Pai C, Kuo T, Jaw T, et al. The homothorax homeoprotein activates the nuclear localization of another homeoprotein, extradenticle, and suppresses eye development in Drosophila[J]. Genes & Development, 1998, 12(3):435-446.
[17] Inbal A, Halachmi N, Dibner C, et al. Genetic evidence for the transcriptional-activating function of Homothorax during adult fly development[J]. Development, 2001, 128(18):3405-3413.
[18] Corsetti E, Azpiazu N. Functional dissection of the splice variants of the Drosophila gene homothorax (hth)[J]. Developmental Biology, 2013, 384(1):72-82.
[19] Kobayashi M, Fujioka M, Tolkunova E, et al. Engrailed cooperates with extradenticle and homothorax to repress target genes in Drosophila[J]. Development, 2003, 130(4):741-751.
[20] Cohen L, Salzberg A. Chromosomal binding sites of the homeotic cofactor Homothorax[J]. Molecular Genetics and Genomics,2008,280(1):73-81.
[21] 韦春梅,罗淋淋,吴华俊,等.松墨天牛幼虫cDNA文库的构建及EST分析[J].基因组学与应用生物学, 2014, 33(1):113-120.
[22] Glazov E, Pheasant M, Mcgraw E, et al. Ultraconserved elements in insect genomes:a highly conserved intronic sequence implicated in the control of homothorax mRNA splicing[J]. Genome Research, 2005, 15(6):800-808.
[23] Moens C B, Selleri L. Hox eofactors invertebrate development[J]. Developmental Biology,2006,291:193-206.
[24] María V A,Becerra A, Hernández-Morales R,et al.Low complexity regions (LCRs) contribute to the hypervariability of the HIV-1 gp120 protein[J].Journal of Theoretical Biology, 2013, 338(37):80-86.
[25] Lenz C,Haerty W,Golding G B.Increased substitution rates surrounding low-complexity regions within primate proteins[J].Genome Biology & Evolution, 2014, 6(3):655-665.
[26] Battistuzzi F, Schneider K, Spencer M, et al. Profiles of low complexity regions in Apicomplexa[J]. BMC Evolutionary Biology, 2016, 16(1):47.
[27] Toll-Riera M, Radó-Trilla N, Martys F, et al. Role of low-complexity sequences in the formation of novel protein coding sequences[J]. Molecular Biology and Evolution, 2012, 29(3):883-886.
[28] Haerty W,Golding G B.Increased polymorphism near low-complexity sequences across the genomes of plasmodium falciparum isolates[J].Genome Biology & Evolution, 2011, 3(3):539-550.
[29] Ramamoorthy S , Garapati H S, Mishra R K.Length and sequence dependent accumulation of simple sequence repeats in vertebrates: Potential role in genome organization and regulation[J].Gene, 2014, 551(2):167-175.
[30] 吴 静.拟黑多刺蚁Homothorax基因的克隆及其在个体发育的mRNA水平表达的定量研究[D].西安:陕西师范大学, 2010.
[31] Jaw T, You L, Knoepfler P, et al. Direct interaction of two homeoproteins, homothorax and extradenticle, is essential for EXD nuclear localization and function[J]. Mechanisms of Development, 2000, 91(1/2):279-291.
[32] Yamamoto T M,Wang L,Fisher L A ,et al.Regulation of greatwall kinase by protein stabilization and nuclear localization[J].Cell Cycle, 2014, 13(22):3565-3575.