枇杷(Eriobotrya japonica Lindl.)是亚热带果树中比较重要的一种,喜温暖气候和肥水湿润的土壤,在我国有着丰富的种植资源和优良品种[1]。枇杷对干旱有一定的耐受性,但在生长过程中易发生花期、幼果期和果实成熟期旱害,导致枇杷的坐果率和产量严重降低[2-3]。而干旱是不可避免的,它是影响植物生长发育最重要的非生物因素之一[4]。关于枇杷干旱的研究在生理生化方面较多[5],罗华建等[2]用盆栽的方法证明:干旱抑制枇杷的生长和光合作用;也有研究表明,轻度的干旱胁迫有助于诱导枇杷花芽分化,使果实成熟期提前,对枇杷的生长有促进作用[6-7]。但由于枇杷基因组背景信息缺乏,尚未见有关枇杷叶片转录组测序等方面的研究报道,其抗逆机理的深入研究及抗旱分子标记开发等发展相对滞后。
转录组测序(RNA sequencing)利用第2代高通量测序技术进行cDNA测序,能全面快速地获得某一状态下的全部转录本信息,可以用于新转录本预测[8]。对于缺乏基因组信息的物种而言,采用转录组测序技术可获得大量的转录本信息,从中发掘重要的功能基因,是揭示植物优良性状的重要研究手段[9]。转录组技术还可以研究不同器官、不同环境胁迫下基因表达的差异,过去10年里,上千个基因和数十条代谢和信号通路被证明与植物的耐旱性有关,拟南芥[10]、水稻[11]和短柄草[12]中的许多基因在转录水平都被证实参与干旱的调控。而参与植物光合作用、ABA合成、信号转导、渗透物质的生物合成和转录因子(WRKY、bZIP家族等)调控等的基因在干旱胁迫下表达异常,表明植物对水分刺激应答的过程受众多分子和细胞通路的调控[13]。但在其他植物中,类似的研究非常有限,植物对干旱耐性的分子机制在很大程度上还是未知的。
本研究利用Illumina HiSeq高通量测序技术对干旱前后的枇杷叶片转录组进行研究,结合生物信息学方法对所获得的差异表达基因(DEGs,Differently expressed genes)进行功能的注释、分类和代谢途径分析,在转录水平上研究干旱胁迫下枇杷关键基因的表达,挖掘与干旱相关的基因,为枇杷抗旱相关基因的克隆以及功能分析和抗旱育种等研究提供理论基础。
1 材料和方法
1.1 试验材料
试验材料为四川龙泉驿区长势良好的3年生大五星枇杷嫁接苗,定植于口径40 cm、高30 cm的花盆中,每盆种植1株。所用土壤为菜园土和沙壤土适量混合,并加入10%腐熟的有机肥和0.1%的复合肥,后期处理过程中不再施肥。待驯化后选取高度、长势一致的材料进行干旱胁迫控制性试验。试验于2015年6-9月在四川农业大学农场进行。试验开始时将材料移至大棚,每盆充分灌溉,使土壤含水量一致,停止灌溉后采用自然耗水进行干旱胁迫处理。
1.2 试验方法
1.2.1 试验处理 以土壤含水量(占田间持水量的百分数)设置正常供水(对照,田间持水量的61%~65%)、轻度干旱胁迫(田间持水量的50%~54%)、中度干旱胁迫(田间持水量的39%~43%)和重度干旱胁迫(田间持水量的28%~32%)4个水分处理。将轻度、中度和重度3个胁迫的叶片取样后混合和对照叶片液氮速冻后于-70 ℃贮存,用于RNA的提取。
1.2.2 总RNA的提取与测序 采用TRIzol法提取总RNA,用Agilent 2100 Technologies检测样品RNA的完整性和浓度,用带有Oligo(dT)的磁珠富集mRNA,再加入缓冲液将其随机打断成片段,以mRNA片段为模板用随机引物合成cDNA第一链,再加入缓冲液、dNTPs、RNase H和DNA Polymerase Ⅰ合成cDNA第二链,将洗脱纯化后的双链cDNA进行末端修复、加碱基A、加测序接头处理,然后经琼脂糖凝胶电泳回收目的大小片段最后再进行PCR扩增,完成整个文库制备后用Illumina HiSeq进行测序,测序策略为PE100。
1.2.3 测序数据的组装和功能注释 采用Trinity(20140717版)[14]将除去接头序列、两端低质量序列和低度复杂序列的高质量序列数据(Clean reads)组装出全长转录本。对组装出来的转录本利用TransDecoder(20140717版)鉴定编码区域,预测开放阅读框ORF(Opening reading fragment)。针对预测出来的ORF和Contig序列用Trinotate(20131110版)对其进行功能注释,包括与同源性搜索(NCBI-Blast)、蛋白质结构域鉴定(HMMER/PFAM)、蛋白质信号预测(SingalP/TmHMM)、Uniprot(Universal Protein:Swiss-Prot、TrEMBL和PIR-PSD)、Blast2GO软件进行的GO(Gene Ontology)、COG(Cluster of Orthologous Groups)和KEGG(Kyoto Encyclopedia of Genes and Genomes)进行序列比对,获得基因的功能注释信息。
1.2.4 差异基因筛选及富集分析 基因表达量的计算使用标准化转录本数(RPKM)法,选取|log2Ratio|≥1和q<0.05的基因作为DEGs。应用超几何检验,以q<0.05 为阈值,筛选出在DEGs中显著富集的条目和通路,分析DEGs行使的主要生物学功能和代谢通路。
1.2.5 实时荧光定量qRT-PCR 利用TRIzol试剂提取RNA后,采用Aidlab公司反转录试剂盒(TUREscript 1st Stand cDNA SYNTHESIS Kit)进行反转录操作,选取测序结果中表达数量较多且在各条目和通路中显著富集的8个差异基因,依据基因的核酸序列设计荧光定量引物,采用20 μL反应体系,对荧光定量PCR的引物浓度、退火温度进行优化,得到最佳的荧光定量PCR,每个样品3个重复,引物见表1。采用2-ΔΔCT法计算各基因相对表达量。
表1 荧光定量PCR引物
Tab.1 Primers used to fluorescent quantitative PCR
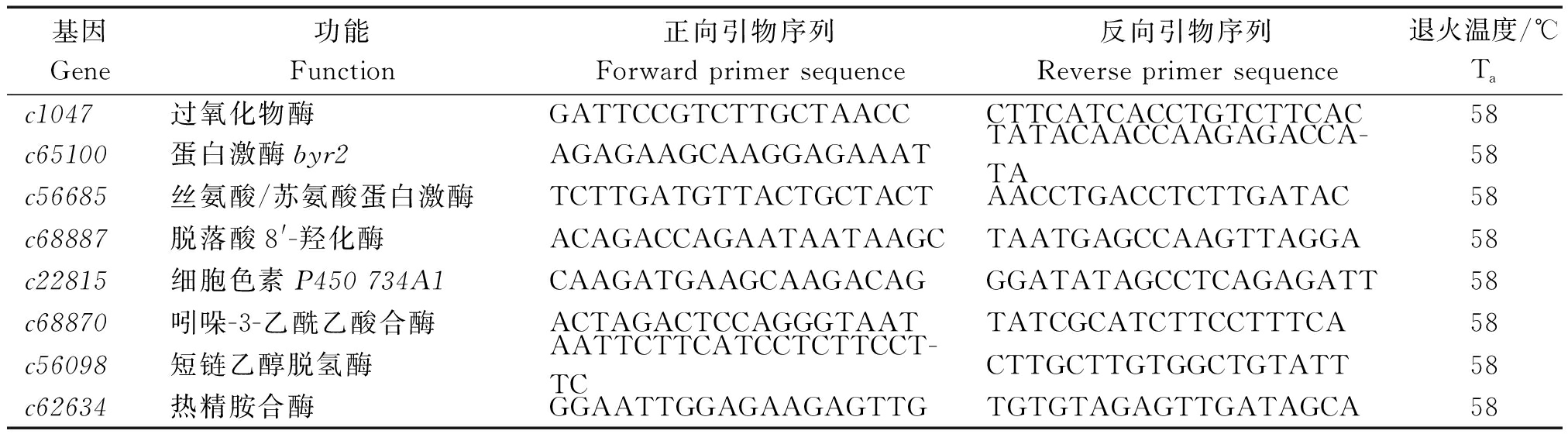
基因Gene功能Function正向引物序列Forward primer sequence反向引物序列Reverse primer sequence退火温度/℃Tac1047过氧化物酶GATTCCGTCTTGCTAACCCTTCATCACCTGTCTTCAC58c65100蛋白激酶byr2AGAGAAGCAAGGAGAAATTATACAACCAAGAGACCA-TA58c56685丝氨酸/苏氨酸蛋白激酶TCTTGATGTTACTGCTACTAACCTGACCTCTTGATAC58c68887脱落酸8'-羟化酶ACAGACCAGAATAATAAGCTAATGAGCCAAGTTAGGA58c22815细胞色素P450 734A1CAAGATGAAGCAAGACAGGGATATAGCCTCAGAGATT58c68870吲哚-3-乙酰乙酸合酶ACTAGACTCCAGGGTAATTATCGCATCTTCCTTTCA58c56098短链乙醇脱氢酶AATTCTTCATCCTCTTCCT-TCCTTGCTTGTGGCTGTATT58c62634热精胺合酶GGAATTGGAGAAGAGTTGTGTGTAGAGTTGATAGCA58
2 结果与分析
2.1 测序结果和注释数据
枇杷叶片通过新一代高通量测序技术进行转录组测序,共获得114 021 518个原始reads,过滤掉接头污染、低质量、含N比例大于50%的序列后,得到90 517 644个干净reads。碱基Q30为92.5%,表明测序的数据量和质量都比较高。利用Trinity将干净reads进行从头组装形成转录本,共发现88 530条转录本,平均长度740.64 bp,N50长度为1 189 bp,其中最长的转录本长度为10 189 bp,最短的长度为201 bp。结果表明,长度在200~400 bp的转录本数目最多,为41 141条,占所有转录本的46.47%(表2)。
组装出的转录本共预测到41 748条ORF,占所有转录本总数的47.16%,ORF平均长度为891.54 bp,N50长度为1 137 bp,长度400~600 bp所占的比例最高。将Contigs和ORF序列注释到Uniprot、GO和KEGG数据库中,对注释到每个库中的数目进行统计,BlastX数据库注释的最多,为22 039条,占所有转录本的24.89%;BlastP数据库注释的数量为16 352条;KEGG数据库注释的数量为5 484条,占6.19%;注释到GO数据库的有20 912条。
表2 枇杷叶片转录组Transcript和ORF长度统计
Tab.2 Length range data for Transcript and ORF in the transcriptome of loquat leaves
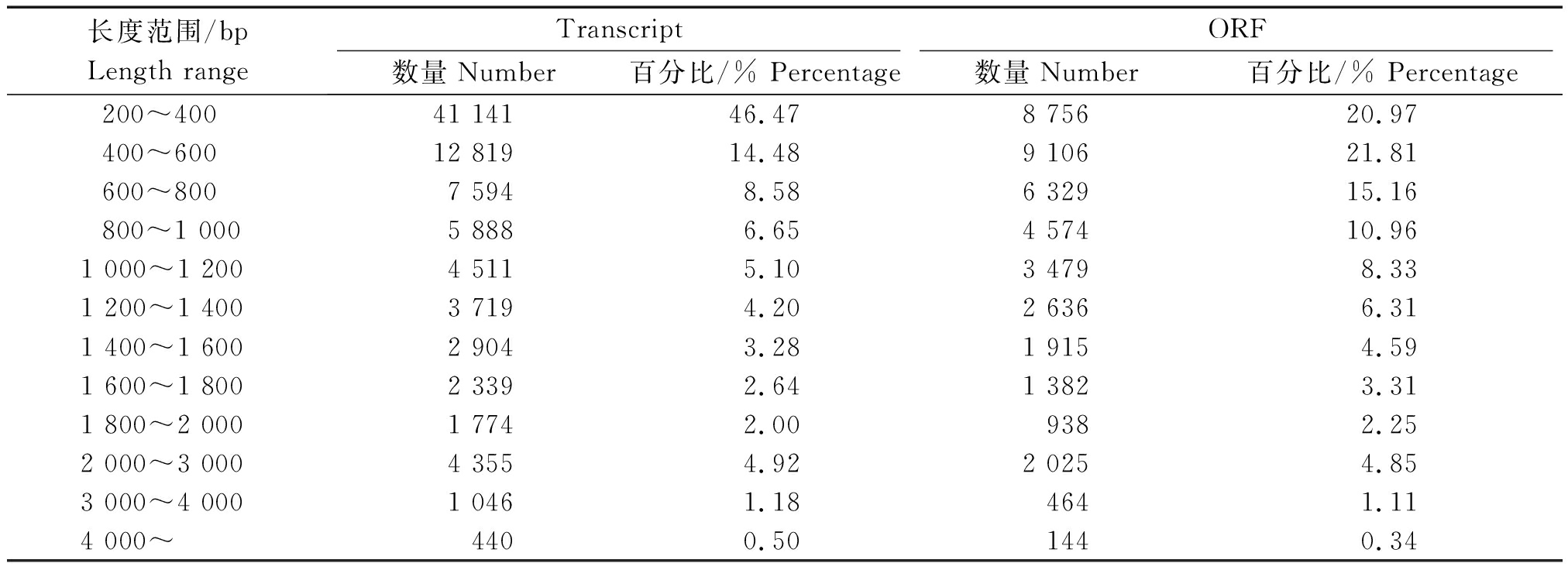
长度范围/bpLength rangeTranscriptORF数量Number百分比/% Percentage数量Number百分比/% Percentage200~40041 14146.478 75620.97400~60012 81914.489 10621.81600~8007 5948.586 32915.16800~1 0005 8886.654 57410.961 000~1 2004 5115.103 4798.331 200~1 4003 7194.202 6366.311 400~1 6002 9043.281 9154.591 600~1 8002 3392.641 3823.311 800~2 0001 7742.009382.252 000~3 0004 3554.922 0254.853 000~4 0001 0461.184641.114 000~4400.501440.34
2.2 COG功能分类
COG分类结果表明,13 285条转录本具有功能注释信息且被注释到24个功能类别。涵盖基因最多的是一般功能预测(18.14%),其次是翻译后修饰、蛋白质折叠和分子伴侣(10.65%),翻译、核糖体结构和生物合成(10.64%),复制、重组和修复(9.73%),碳水化合物运输与代谢(7.19%),信号转导机制(6.86%)和能量产生与转化(6.57%)。所有类别中,细胞运动(0.11%)和核酸结构(0.01%)的数量最少(图1)。
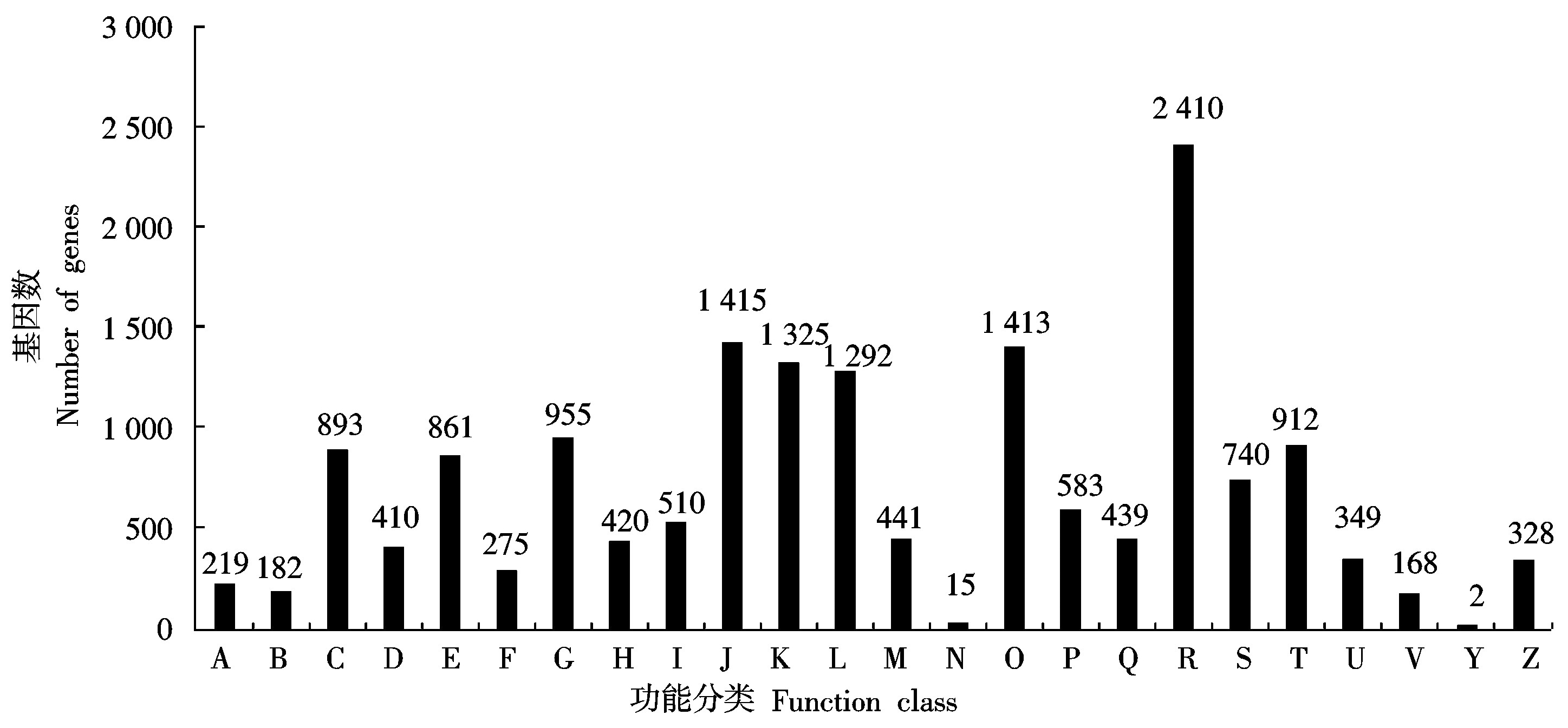
A.加工与修饰RNA ;B.染色体结构与变化;C.能量产生与转化;D.细胞周期调控与分裂、染色体重排;E.氨基酸运输与代谢;F.核苷酸运输与代谢;G.碳水化合物运输与代谢;H.辅酶运输与代谢;I.脂质运输与代谢;J.翻译、核糖体结构与生物合成;K.转录;L.复制、重组与修复;M.细胞壁、膜生物合成Cell;N.细胞运动;O.翻译后修饰、蛋白质折叠和分子伴侣;P.无机离子运输与代谢;Q.次生代谢物合成、运输和代谢;R.一般功能预测;S.未知功能;T.信号转导机制;U.细胞内转运、分泌和囊泡运输;V.防御机制;Y.核酸结构;Z.细胞骨架。
A.Processing and modification;B.Chromatin structure and dynamics;C.Energy production and conversion;D.Cell cycle control, cell division, chromosome partitioning;E.Amino acid transport and metabolism;F.Nucleotide transport and metabolism;G.Carbohydrate transport and metabolism;H.Coenzyme transport and metabolism;I.Lipid transport and metabolism;J.Translation, ribosomal structure and biogenesis;K.Transcription;L.Replication, recombination and repair;M.Wall/membrane/envelope biogenesis;N.Cell motility;O.Posttranslational modification, protein turnover, chaperones;P.Inorganic ion transport and metabolism;Q.Secondary metabolites biosynthesis, transport and catabolism;R.General function prediction only;S.Function unknown;T.Signal transduction mechanisms;U.Intracellular trafficking, secretion, and vesicular transport;V.Defense mechanisms;Y.Nuclear structure;Z.Cytoskeleton.
图1 枇杷叶片转录组COG分析
Fig.1 COG analysis of loquat leaves transcriptome
2.3 差异基因的GO分类和富集分析
通过计算基因的RPKM值,将两样品间RPKM的log2倍数绝对值大于1和q<0.05的基因作为差异基因,共筛选出25 197个差异表达的基因,其中上调基因13 482个,下调基因11 715个。54个GO分类注释结果(图2)表明:生物学过程分类中,注释数量最多的是细胞进程(上调基因为8.23%,下调基因为8.84%)和代谢途径(上调基因7.40%,下调基因8.0%)。在细胞组分中,细胞和细胞部分拥有的基因数最多(上调基因均为10.08%,下调基因均为11.94%)。分子功能中结合作用(上调基因分别为7.55%,下调基因为9.51%)的数量最多。
通过显著富集计算,生物学过程中有345条GO条目显著富集,细胞组分有87条,分子功能中有173条。其中分子功能中富集差异基因最多的前3条GO条目分别是催化活性、转移酶活性和水解酶活性(表3)。
表3 分子功能中DEGs富集最多的前10 类GO注释
Tab.3 Top 10 GO annotations with DGEs enrichment in molecular functional
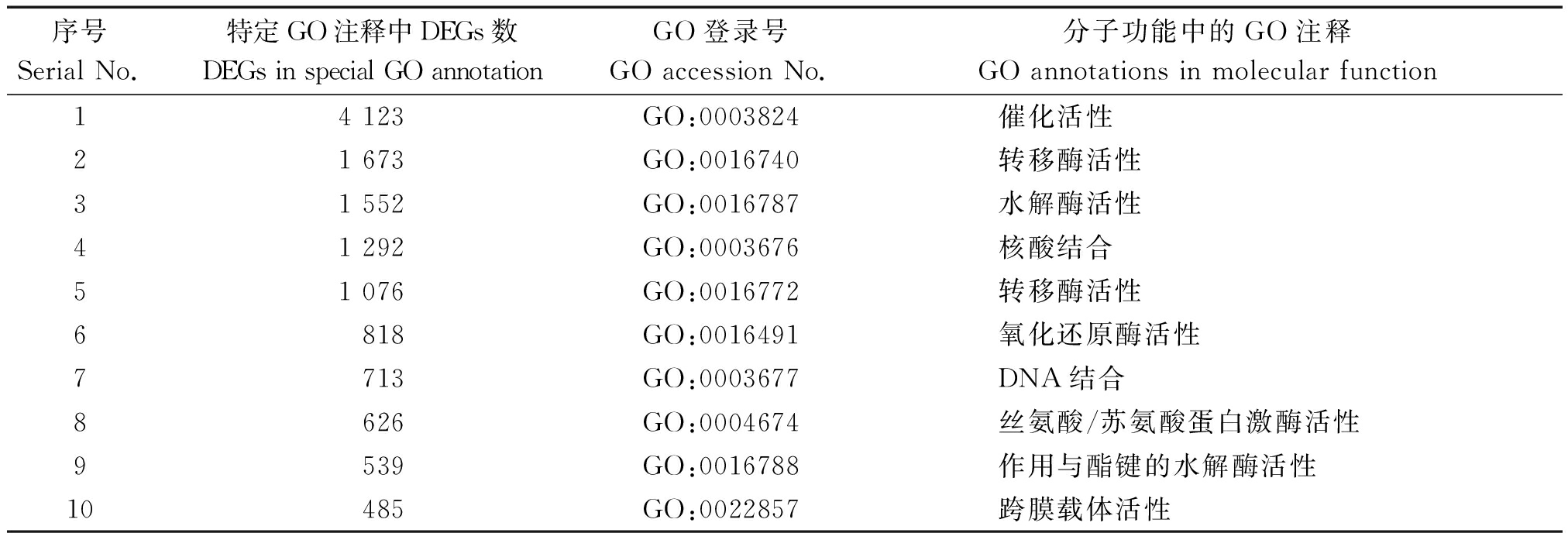
序号Serial No.特定GO注释中DEGs数DEGs in special GO annotationGO登录号GO accession No.分子功能中的GO注释GO annotations in molecular function14 123GO:0003824催化活性21 673GO:0016740转移酶活性31 552GO:0016787水解酶活性41 292GO:0003676核酸结合51 076GO:0016772转移酶活性6818GO:0016491氧化还原酶活性7713GO:0003677DNA结合8626GO:0004674丝氨酸/苏氨酸蛋白激酶活性9539GO:0016788作用与酯键的水解酶活性10485GO:0022857跨膜载体活性
2.4 代谢通路分析
KEGG分析结果表明,1 618个DEGs在291个标准代谢通路中得到注释。对KEGG中每个代谢通路用超几何检验进行富集分析,找出DEGs显著性富集的30条代谢通路(表4),主要有:代谢途径、次生代谢物的二次代谢、不同生境微生物代谢作用和核糖体。这些途径为研究枇杷干旱胁迫的具体过程、功能和途径提供了珍贵的资源。其中,与激素代谢相关的3条代谢途径:双萜类、油菜素类固醇和类胡萝卜素生物合成途径中的差异基因呈现出较为一致的表达。双萜类生物合成途径的13个基因中有6个贝壳杉烯酸氧化酶和4个贝壳杉烯氧化酶基因均下调表达。类胡萝卜素生物合成途径共14个DEGs,其中有2个9-顺式环氧类胡萝卜素双加氧酶基因和10个上调表达的脱落酸(ABA)-8′羟化酶基因。油菜素内固醇生物合成中DEGs共14个,12个均表达下调,其中有9个基因为CYP734A1。

图2 干旱胁迫下枇杷叶片DEGs的GO分类
Tab.2 Go categories of DEGs of loquat leaves under drought stress
表4 DEGs富集的30条KEGG通路
Tab.4 30 KEGG Pathways with DEGs enrichment
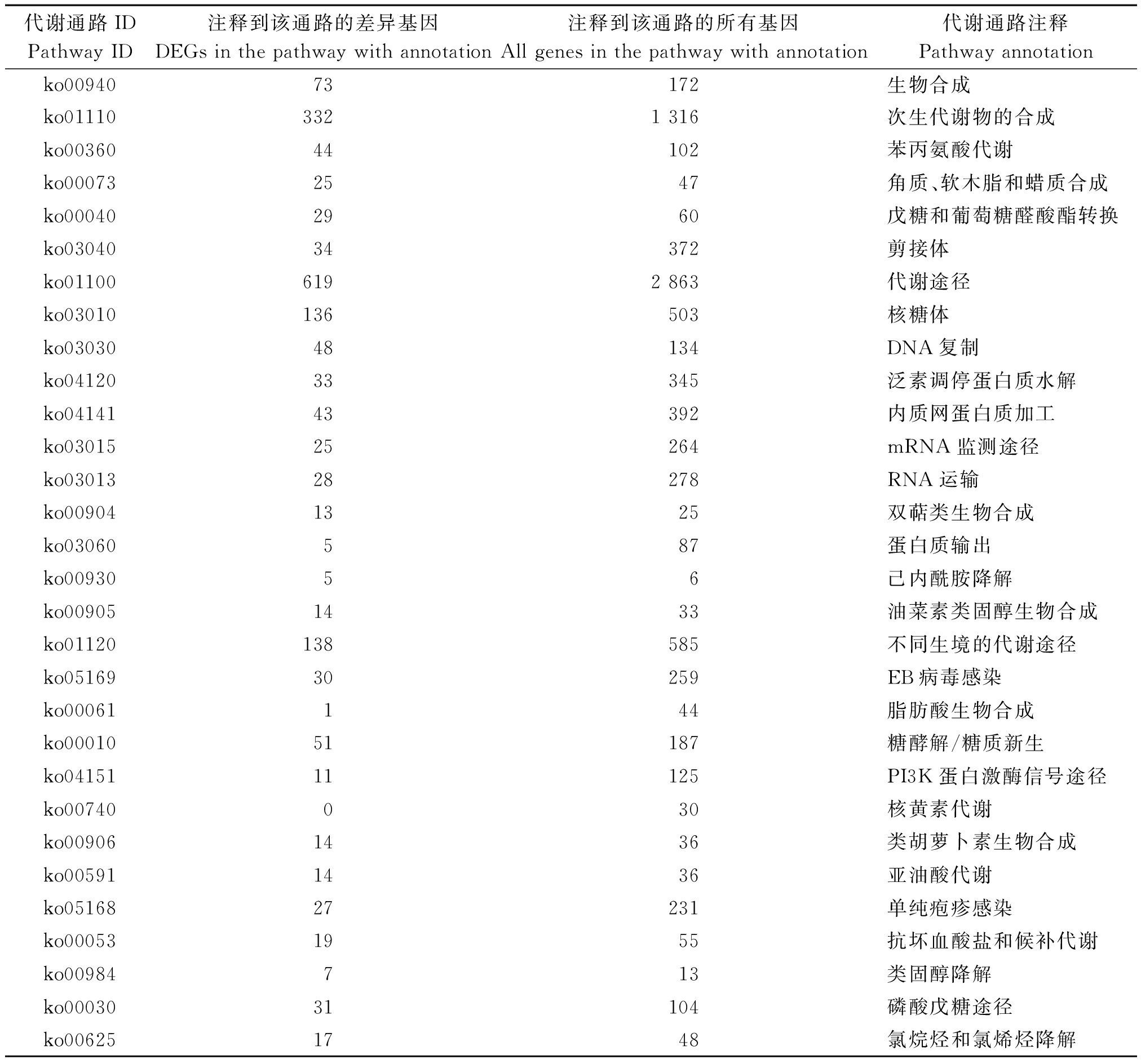
代谢通路IDPathway ID注释到该通路的差异基因DEGs in the pathway with annotation注释到该通路的所有基因All genes in the pathway with annotation代谢通路注释Pathway annotationko0094073172生物合成ko011103321 316次生代谢物的合成ko0036044102苯丙氨酸代谢ko000732547角质、软木脂和蜡质合成ko000402960戊糖和葡萄糖醛酸酯转换ko0304034372剪接体ko011006192 863代谢途径ko03010136503核糖体ko0303048134DNA复制ko0412033345泛素调停蛋白质水解ko0414143392内质网蛋白质加工ko0301525264mRNA监测途径ko0301328278RNA运输ko009041325双萜类生物合成ko03060587蛋白质输出ko0093056己内酰胺降解ko009051433油菜素类固醇生物合成ko01120138585不同生境的代谢途径ko0516930259EB病毒感染ko00061144脂肪酸生物合成ko0001051187糖酵解/糖质新生ko0415111125PI3K蛋白激酶信号途径ko00740030核黄素代谢ko009061436类胡萝卜素生物合成ko005911436亚油酸代谢ko0516827231单纯疱疹感染ko000531955抗坏血酸盐和候补代谢ko00984713类固醇降解ko0003031104磷酸戊糖途径ko006251748氯烷烃和氯烯烃降解
2.5 差异基因及其实时荧光定量验证
通过功能分析,除了部分未知功能的基因外,可能与干旱胁迫响应相关的基因主要有信号转导相关基因:促分裂原活化蛋白激酶(MAPK)、富含亮氨酸重复序列类受体蛋白激酶(LRR-RLKs)、G-type-S受体蛋白、ERF、MYB、WRKY转录因子和热激蛋白等;抗氧化酶基因:过氧化氢酶、谷胱甘肽过氧化物酶、过氧化物酶;渗透调节物质合成相关基因:海藻糖-6-磷酸合酶、乙醛脱氢酶、谷氨酸脱氢酶;激素合成相关基因:细胞色素P450家族、贝壳杉烯酸氧化酶、贝壳杉烯氧化酶、黄烷酮醇-4-还原酶、三角状五肽重复(PPR)蛋白等。
选择了差异表达数量丰富且在GO分类和KEGG代谢通路中都显著富集的8个差异基因(表1)进行实时荧光定量分析,验证这8个基因在叶片中的表达情况(图3),其中的6个基因c1047(过氧化物酶-上调)、c65100(蛋白激酶byr2-上调)、c56685(丝氨酸苏氨酸蛋白激酶-上调)、c68887(脱落酸8′-羟化酶-上调)、c22815(细胞色素P450 734A1-下调)和c68870(吲哚-3-乙酰乙酸合酶-上调)的表达结果与转录组测序一致,这也证明了测序方法的正确性。
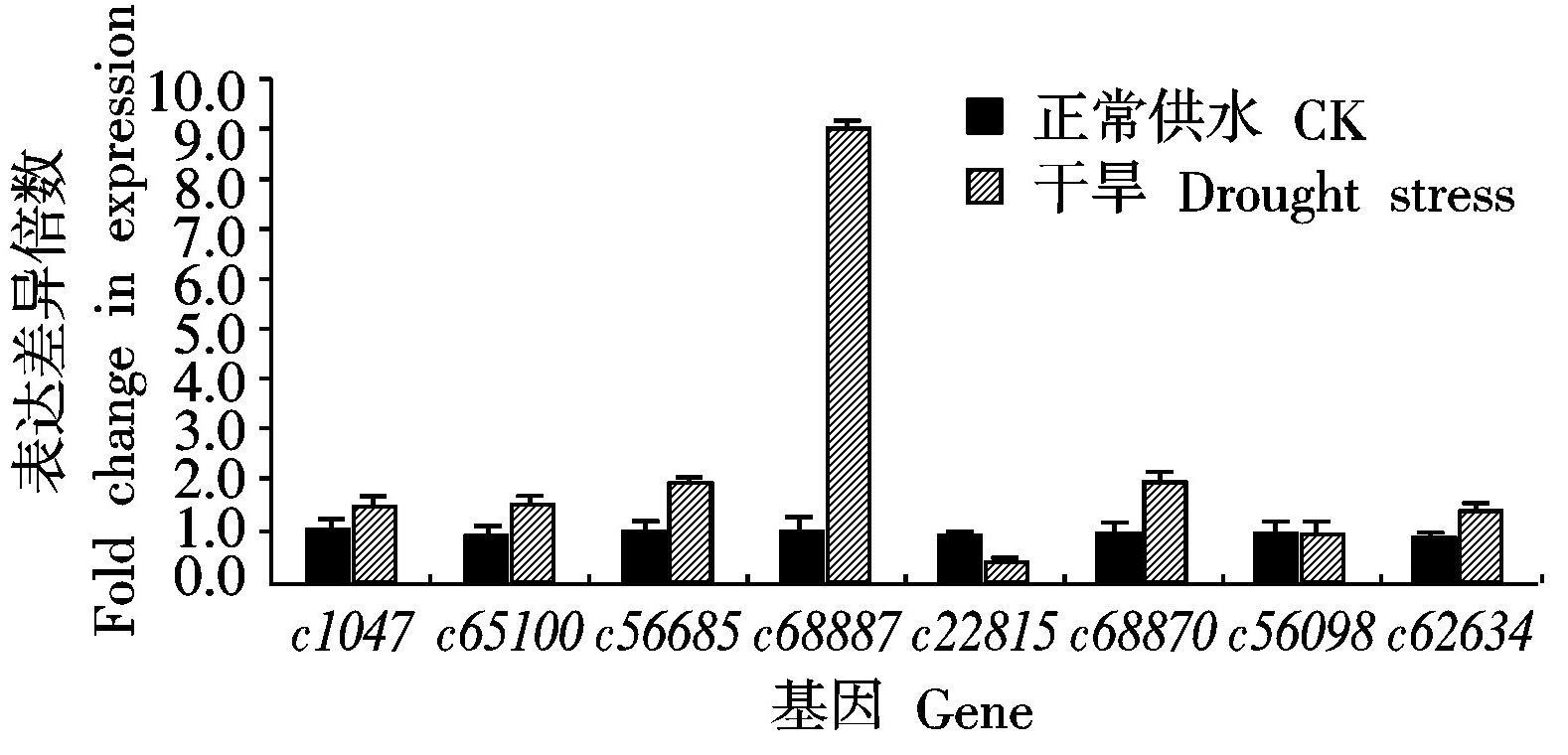
图3 八个差异表达基因的定量PCR表达
Fig. 3 Real-time PCR analysis of the transcript
levels of 8 differently expressed genes
3 讨论
抗旱性一个复杂的多基因特征[15],高通量测序能够为植物基因表达的全面分析提供合理的数据资源,尤其适合没有参考基因组信息的物种[16]。本研究采用高通量测序共获得枇杷叶片转录本88 530条,预测出41 748条ORF序列,将测序结果与Uniprot、GO和KEGG等数据库进行比对。通过筛选,得到25 197个DEGs(P<0.01),然而有大量的DEGs未得到注释且未被报道过,这很大程度上丰富了枇杷的基因信息资源库[17-18]。注释结果表明,大部分差异表达的基因参与了如催化酶、转移酶和水解酶活性的调节,大分子物质的代谢,渗透调节物质合成,信号转导和激素水平的调节。
受体蛋白激酶(RLKs)是一组膜蛋白,尽管其功能和详细的分子机制还不清楚,但RLK基因转录变化表明他们可能在干旱中起到重要的作用[19]。有研究表明,蛋白激酶信号基因在干旱时被诱导[20]。水稻受体蛋白激酶4在胁迫中被发现上调表达[11]。本研究中的丝氨酸/苏氨酸蛋白激酶、LRR-RLKs等大部分基因均不同程度的下调,MAPK则表现为上调,说明这几类蛋白激酶对干旱胁迫应答调控起不同的作用。蔡国华[21]的研究结果表明,在干旱条件下,过表达玉米MAPKK在拟南芥中通过ROS和依赖ABA途径增强了对干旱胁迫的抗性,另有研究表明,一些RLKs,如拟南芥RPK1在干旱胁迫时与ABA和BR(油菜素内固醇)信号通路有联系,而不同受体蛋白类型在信号通路中的作用还有待进一步探讨[22]。
参与胁迫信号传递网络的转录因子,尤其是ERF、WRKY、MYB等家族是信号转导通路上的关键调控因子,对干旱胁迫响应和水分刺激应答发挥了重要调节作用[23]。ERF 类转录因子是AP2/EREBP 转录因子家族的一个亚家族,通过参与乙烯、ABA、茉莉酸等多种信号转导途径,在非生物胁迫应答调控中发挥重要的作用,一些ERFs在转基因植物中能加强对干旱的耐受性,过表达的ERF类转录因子WXP1、WXP2可以增加叶表皮蜡质的积累,显著减少叶片脱水、提高植株对干旱胁迫的耐受性[24]。也有研究表明,在耐旱玉米基因型中的转录因子MYB33和MYB101能增强玉米的耐旱性[25],在柑橘胚珠的研究中与信号传递相关的基因WRKY被验证在胁迫发生时表达下调[26]。本试验中ERF、WRKY、MYB是干旱胁迫处理下数量丰富、表达量变化最显著的几类转录因子,下调显著的主要是ERF类转录因子,上调显著的是MYB相关家族编码基因,说明这些转录因子对干旱胁迫的应答显著,这与Chen等[27]的研究结果一致,原因可能是通过上调基因来实现叶片对水分刺激的应答调节,通过下调基因数量的平衡来实现叶片对干旱胁迫的响应[28]。
ABA的生物合成和分解代谢率决定ABA的积累以及对胁迫的反应强度,尽管有各种各样的途径合成ABA,但类胡萝卜素生物合成途径在被子植物中是唯一已经确定的途径[29]。本研究中类胡萝卜素生物合成通路中的10个ABA 8′-羟化酶上调表达,而ABA 8′-羟化酶属于细胞色素P450的CYP707A家族,它是合成植物激素ABA的前体[30]且CYP707A 8′-羟基化基因在拟南芥的研究中参与抗旱[31]、种子休眠和萌发[32]。ABA 8′-羟化酶可能参与合成类胡萝卜素,并积累额外的ABA以响应胁迫。ABA与赤霉素(GA)有拮抗作用,在干旱胁迫下,激素的比值会发生不同程度的改变,有研究表明,GA3/ABA值能体现玉米萌发期适应胁迫的能力[33]。在本研究中,双萜类生物合成途径中的GA合成上游的2种关键酶均下调表达:贝壳杉烯酸氧化酶和贝壳杉烯氧化酶,这可能与ABA 8′-羟化酶基因的上调表达相互平衡,共同响应干旱,但二者的具体变化情况还不明确。油菜素内酯(BRs)是一种植物甾醇类激素,有研究发现,油菜素内酯能提高拟南芥幼苗忍耐低温的能力,还能增加植物的环境抗逆性[34]。而本研究中,油菜素内固醇生物合成途径中下调基因中有9个都是细胞色素P450家族的CYP734A1基因,而拟南芥中的CYP734A1能够使体内BR失活[35],说明此时植物可能通过降低CYP734A1基因的表达量来调节BR的活性以应对干旱胁迫,关于CYP734A1基因和其他未知功能基因与植物响应干旱之间的具体关系都还有待更深入的研究。
[1] Carbonell F B, Barrachina A C, Roig A V, et al. Effects of irrigation water quality on loquat plant nutrition:Sensitivity of loquat plant to salinity[J]. Journal of Plant Nutrition, 1997, 20(1):119-130.
[2] 罗华建,刘星辉.水分胁迫条件下枇杷若干生理指标的变化[J].亚热带植物科学, 2004, 33(1):19-21, 25.
[3] Stellfeldt A, Hueso J J, Cuevas J. No need for further fruit thinning in water-deprived loquat trees at preharvest[J]. Scientia Horticulturae, 2013, 162(3):144-149.
[4] Ashraf M. Inducing drought tolerance in plants:recent advances[J]. Biotechnology Advances, 2009, 28(1):169-183.
[5] 杨再强,谢以萍,张旭东,等.水分胁迫对枇杷果实发育阶段的光合特性和果实品质的影响[J].灌溉排水学报, 2007, 6(26):89-92.
[6] Fernández M D, Hueso J J, Cuevas J. Water stress integral for successful modification of flowering dates in Algerie loquat[J]. Irrigation Science, 2010, 28(2):127-134.
[7] Hueso J J, Cuevas J. Loquat as a crop model for successful deficit irrigation[J]. Irrigation Science, 2008, 26(3):269-276.
[8] Yang Y, Xu M, Luo Q, et al. De novo transcriptome analysis of Liriodendron chinense petals and leaves by Illumina sequencing[J]. Gene, 2013, 534(2):155-162.
[9] 贾新平,孙晓波,邓衍明,等.鸟巢蕨转录组高通量测序及分析[J].园艺学报, 2014, 41(11):2329-2341.
[10] Seki M, Narusaka M, Ishida J, et al. Monitoring the expression profiles of 7000 Arabidopsis genes under drought, cold and high-salinity stresses using a full-length cDNA microarray[J]. The Plant Journal :for Cell and Molecular Biology, 2002, 31(3):279-292.
[11] Rabbani M, Maruyama K, Abe H, et al. Monitoring expression profiles of rice genes under cold, drought, and high-salinity stresses and abscisic acid application using cDNA microarray and RNA gel-blot analyses[J]. Plant Physiology, 2003, 133(4):1755-1767.
[12] Priest H D, Fox S E, Rowley E R, et al. Analysis of global gene expression in Brachypodium distachyon reveals extensive network plasticity in response to abiotic stress[J]. PLoS One, 2014, 9(1):e87499.
[13] 王 洁.干旱胁迫马铃薯叶片转录组分析[D].兰州:甘肃农业大学, 2014.
[14] Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7):644-652.
[15] Hayano-Kanashiro C, Calderón-Vázquez C, Ibarra-Laclette E, et al. Analysis of gene expression and physiological responses in three Mexican maize landraces under drought stress and recovery irrigation[J]. PLoS One, 2009, 4(10):69.
[16] 宋 杨,刘红弟,张红军,等.越橘果实转录组及R2R3-MYB转录因子分析[J].园艺学报, 2015, 42(12):2383-2394.
[17] 费 元,韩 雪,余 红,等.大岩桐花萼和幼叶转录组研究[J].园艺学报, 2015, 42(12):2519-2525.
[18] 麻文俊.楸树优良无性系2-8苗期生理变化与基因表达对干旱胁迫的响应[D].北京:中国林业科学研究院, 2013.
[19] Wu Y, Wei W, Pang X, et al. Comparative transcriptome profiling of a desert evergreen shrub, Ammopiptanthusmongolicus, in response to drought and cold stresses[J]. BMC Genomics, 2014,15(1):1-16.
[20] Lata C, Sahu P P, Prasad M. Comparative transcriptome analysis of differentially expressed genes in foxtail millet (Setaria italica L.) during dehydration stress. Biochemical and Biophysical Research Communications[J],2010,393(4):720- 727.
[21] 蔡国华.玉米促分裂原活化蛋白激酶激酶基因ZmMKK1的分离及功能分析[D].泰安:山东农业大学, 2014.
[22] Li J, Tax F. Receptor-like kinases:key regulators of plant development and defense[J]. Journal of Integrative Plant Biology, 2013, 55(12):1184-1187.
[23] Hassan N, El-Bastawisy Z, El-Sayed A K, et al. Roles of dehydrin genes in wheat tolerance to drought stress[J]. Journal of Advanced Research, 2013, 6(2):179-188.
[24] Zhang G, Chen M, Li L, et al. Overexpression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco[J]. Journal of Experimental Botany,2008,60(13):3781-3796.
[25] Li J S, Fu F L, An M, et al. Differential expression of MicroRNAs in response to drought stress in maize[J]. Journal of Integrative Agriculture, 2013, 12(8):1414-1422.
[26] Kumar V, Malik S K, Pal D, et al. Comparative transcriptome analysis of ovules reveals stress related genes associated with nucellar polyembryony in citrus[J]. Tree Genetics & Genomes, 2014, 10(3):449-464.
[27] Chen L, Song Y, Li S, et al. The role of WRKY transcription factors in plant abiotic stresses[J]. Biochimica et Biophysica acta, 2012, 1819(2):120-128.
[28] 巩 檑,张 丽,聂峰杰,等.干旱胁迫和复水处理后马铃薯转录因子的转录组分析[J].分子植物育种, 2015, 13(8):1745-1756.
[29] Nambara E, Marion-Poll A. Abscisic acid biosynthesis and catabolism[J]. Annual Review of Plant Biology, 2005,56(56):165-185.
[30] 黄彬城,季 静,王 罡,等.植物类胡萝卜素的研究进展[J].天津农业科学, 2006, 12(2):13-17.
[31] Umezawa T, Okamoto M, Kushiro T, et al. CYP707A3, a major ABA 8′-hydroxylase involved in dehydration and rehydration response in Arabidopsis thaliana[J].Plant Journal,2006, 46(2):171-182.
[32] Okamoto M, Kuwahara A, Seo M, et al. CYP707A1 and CYP707A2, which encode abscisic acid 8′-hydroxylases, are indispensable for proper control of seed dormancy and germination in Arabidopsis[J]. Plant Physiology, 2006, 141(1):97-107.
[33] 裴帅帅.干旱胁迫对谷子生理特性的影响及赤霉素代谢关键酶基因表达分析[D].晋中:山西农业大学, 2014.
[34] Divi U K, Krishna P. Overexpression of the brassinosterid biosynthetic gene AtDWF4 in Arabidopsis and increases cold tolerance in transgenic seedlings[J]. Plant Growth Reg,2010,29(4):385-393.
[35] 杨作仁.棉花T-DNA激活标签突变体pag1分子机制的研究与应用[D].北京:中国农业科学院,2014.