土壤的盐碱化是一个世界性的难题,全球盐渍土面积约10亿hm2,中国盐渍土面积约占世界盐渍土面积的10%[1-3]。随着人类活动的增加,环境的恶化,不合理的灌溉等因素使盐碱化土地的面积不断扩大。随着世界人口的不断增加,对粮食的需求也不断增加[4-5],人口、粮食、耕地的矛盾不断加剧,所以探索农作物的耐盐机理,培育耐盐品种是一项十分重要的任务。
植物在进化过程中形成了各种生理生化机制来适应盐胁迫,其中盐离子的转运和吸收是一个很重要的耐盐机制。液泡是成熟植物细胞中最大的细胞器,在细胞渗透压的调节、离子的吸收和转运、有毒离子的区域化以及信号转导系统的调控等方面具有重要作用[6-7],而液泡膜上的质子泵是实现这些生物功能的关键因素。定位于液泡膜上的H+-PPase的质子泵,能够利用焦磷酸(PPi)为底物,催化H+由细胞质进入液泡,形成电化学梯度,用于各种离子的运输和积累,维持细胞的离子平衡和渗透压,保证植物能够在盐胁迫下正常生长[8-9]。H+-PPase基因已经从多种植物中得到了克隆,如黄瓜、玉米、梭梭、陆地棉、费尔干猪毛菜、刚毛柽柳等[10-15]。大量的研究表明,通过对H+-PPase基因的遗传转化能够显著提高转基因作物的耐盐性。Gaxiola等[16]在2001年首次将AVP1 基因转入拟南芥中,结果发现,超表达AVP1 基因的转基因植株抗盐性得到增强。Duan等[17]通过在烟草中过量表达液泡膜H+-PPase基因,发现在盐胁迫下转基因烟草比野生型烟草叶肉细胞液泡中的Na+含量更高,耐盐性更强。
红麻(Hibiscus cannabinus)是一年生韧皮纤维作物。红麻纤维是重要的纺织原料,可以用于地毯,麻袋的生产,随着红麻多用途开发利用的研究,红麻还可以用于建筑板材、纸浆、饲料、麻骨炭等方面[18]。随着人们对粮食需求的不断增加,红麻的生产必须向盐碱地、滩涂、干旱坡地转移而避免与粮棉油争地。所以对红麻耐盐机理的探索,培育红麻耐盐品种对红麻产业的发展具有重要意义。目前,红麻的H+-PPase基因还未见报道,本研究从红麻耐盐转录组测序结果中发现了该基因,克隆了其全长cDNA序列,并对该基因进行了盐胁迫表达分析,这将为红麻耐盐分子设计育种奠定坚实的基础。
1 材料和方法
1.1 试验材料和试剂
中红麻16号的种子由中国农业科学院麻类研究所红麻育种课题组提供。选取籽粒饱满,大小均匀的种子,消毒,播种在发芽盒,置于光照培养箱,昼夜温度为28,26 ℃,光照13 h/d,培养7 d后,选取植株健壮,长势一致的红麻幼苗,移入1/2 Hoagland 营养液中继续培养,每天更换营养液。在营养液中培养5 d后进行NaCl胁迫处理,NaCl浓度梯度为0,75,150,250 mmol/L。每个处理设置3个重复,每个重复选用10株幼苗。NaCl胁迫处理5 d后取样,每个处理选取3株幼苗叶片,液氮速冻后保存在-80 ℃冰箱,用于总RNA的提取。
TRIzol购自Invitrogen 公司, Reverse Transcriptase、Rever Transcriptase inhibitor、Power2(SYBR Real-time PCR Premixture、pMD19-T载体购自TaKaRa 公司,DH5α感受态细胞购自天根生化科技有限公司;其余试剂为国产分析纯。
1.2 引物设计
依据红麻转录组序列利用Primer premier 5.0进行引物设计,NCBI Blast验证引物准确性,序列由上海生工合成。基因克隆的引物序列:Primer-F(5′-3′):ATGTCGGATCATGGATTTA;Primer-R(5′-3′):
TCATGGTTCGTGTTTAAGGA。
1.3 总RNA的提取
参照天根生化科技有限公司,植物总RNA提取试剂盒的说明书提取总RNA, 然后用1.0%的琼脂糖凝胶电泳检测提取总RNA的完整性和纯度。
1.4 Hcvp1基因的克隆
提取250 mmol/L NaCl胁迫下红麻幼苗叶片的总RNA,并进行反转录合成首链cDNA, 以1 μL反转录产物为模板进行PCR扩增。PCR反应体系:10×LA PCR Buffer(Mg2+ plus) 2.5 μL,LA Taq(TaKaRa) 0.25 μL, dNTP(2.5 mmol/mL)1 μL,F-primer(10 μmol/mL)0.25 μL,R-Primer(10 μmol/mL)0.25 μL,cDNA 1 μL,dH2O 补足25 μL。PCR反应程序:94 ℃ 2 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 60 s,30个循环;72 ℃ 3 min,反应终止于4 ℃。反应结束后,取10 μL PCR产物进行琼脂糖凝胶电泳。PCR产物连接到pMD19-T载体上,转化DH5α感受态细胞,送华大基因进行测序。
1.5 Hcvp1基因的生物信息学分析
利用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)寻找Hcvp1基因的最大开放读码框;在NCBI网站((http://www.ncbi.nlm.nih.gov/blast)对Hcvp1基因的核苷酸序列进行BlastN序列比对,同时利用推测的氨基酸序列进行BlastP比对;利用ClustalW软件进行Hcvp1基因氨基酸序列同源性比对;用MEGA 6.0构建进化树,分析该基因的进化关系。用TMHMM-2.0(http://www.cbs.dut.dk/services/TMHMM-2.0/)预测Hcvp1基因编码蛋白质的跨膜结构,用(http://www.expasy.ch/tools/pi_tool.html)推测Hcvp1基因编码蛋白质的等电点和分子量。
1.6 Hvcp1基因在不同NaCl浓度胁迫下的表达特征分析
采用实时荧光定量PCR(qRT-PCR)技术分析Hvcp1 在不同的NaCl浓度胁迫下的表达特征。荧光定量PCR的引物Primer-F(5′-3′):GGTGCGGAGGAATTAGTA,Primer-R(5′-3′):ATGAAAGCAAATCGTGGT;内参基因为Actin, 引物为:Primer-F (5′-3′) CAGGCAGTTCTTTCTTTGT;Primer-R (5′-3′) ATCCTCCAATCCAGACACT。qRT-PCR的反应体系为(10 μL):Power 2×SYBR Real-time PCR Premixture 5 μL,正向引物(Forward primer)0.3 μL,反向引物(Reverse primer)0.3 μL,cDNA 2 μL,H2O 2.4 μL。qRT-PCR的反应程序为:95 ℃ 2 min; 95 ℃ 15 s, 61 ℃ 15 s, 72 ℃ 20 s,40个循环。
2 结果与分析
2.1 红麻叶片总RNA的提取
根据天根生化科技有限公司的植物总RNA提取试剂盒说明书提取250 mmol/L NaCl浓度胁迫的红麻幼苗叶片的总RNA, 然后用1.0%的琼脂糖凝胶电泳检测提取总RNA的完整性和纯度。结果如图1所示28S、18S和5S 3条带清晰可见,并且28S条带的亮度约是18S条带亮度的2倍,表明RNA结构完整,无污染和降解可以作为反转录的模板。
2.2 Hcvp1基因的克隆
以红麻耐盐转录组获得的Hcvp1基因的片段序列为基础,设计特异性引物,以cDNA为模板进行PCR扩增,扩增产物进行琼脂糖凝胶电泳检测结果如图2所示。经PCR获得产物连接到pMD19-T载体上,转化DH5α感受态细胞,经培养后提取质粒,送华大基因公司进行测序。将测序获得的cDNA序列,通过Blast比对发现,其与H+-PPase基因高度相似,将其命名为Hcvp1,在GenBank中的登录号为:KX687919。
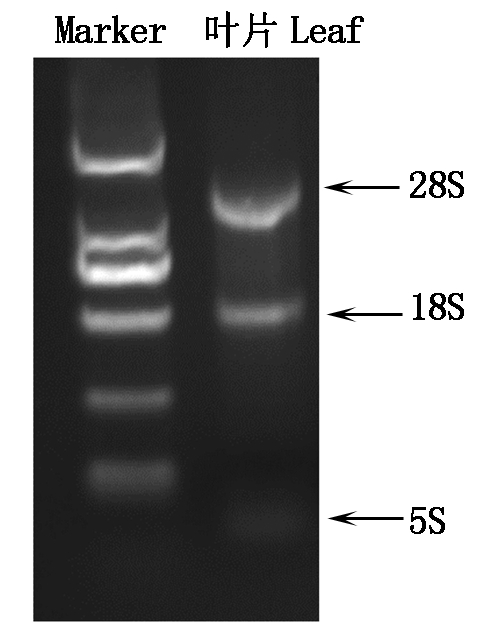
图1 红麻叶片总RNA凝胶电泳图
Fig.1 Agarose gel electrophoresis of total RNA of kenaf leaf
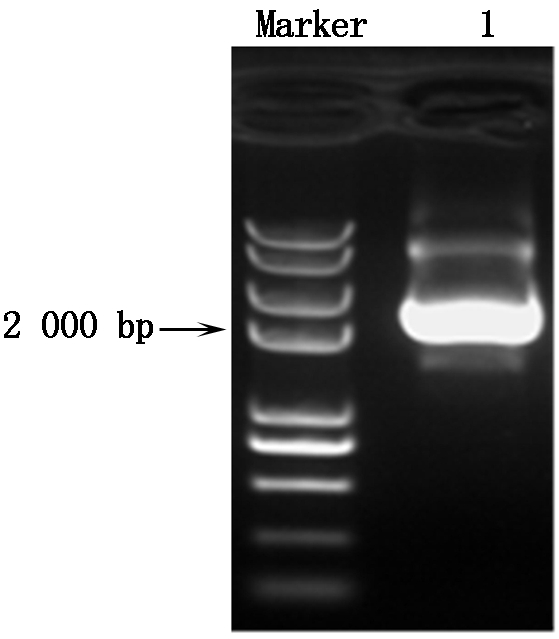
Marker.2k plus Ⅱ;1.PCR产物。
Marker.2k plus Ⅱ; 1.PCR production.
图2 Hcvp1基因的cDNA全长片段
Fig.2 cDNA full length fragment of Hcvp1

图3 Hcvp1基因的核苷酸序列和氨基酸序列
Fig.3 Nucleotides sequence and predicted amino acids sequence of Hcvp1 gene
2.3 Hcvp1基因的生物信息学分析
经过Sanger测序,该基因cDNA全长2 298 bp,开放阅读框为2 298 bp,推测其编码765个氨基酸的蛋白质(图3),推测其分子量和等电点分别为80.2 kDa和5.25。用TMHMM-2.0预测表明Hcvp1基因编码蛋白质具有13个跨膜结构(图4)。
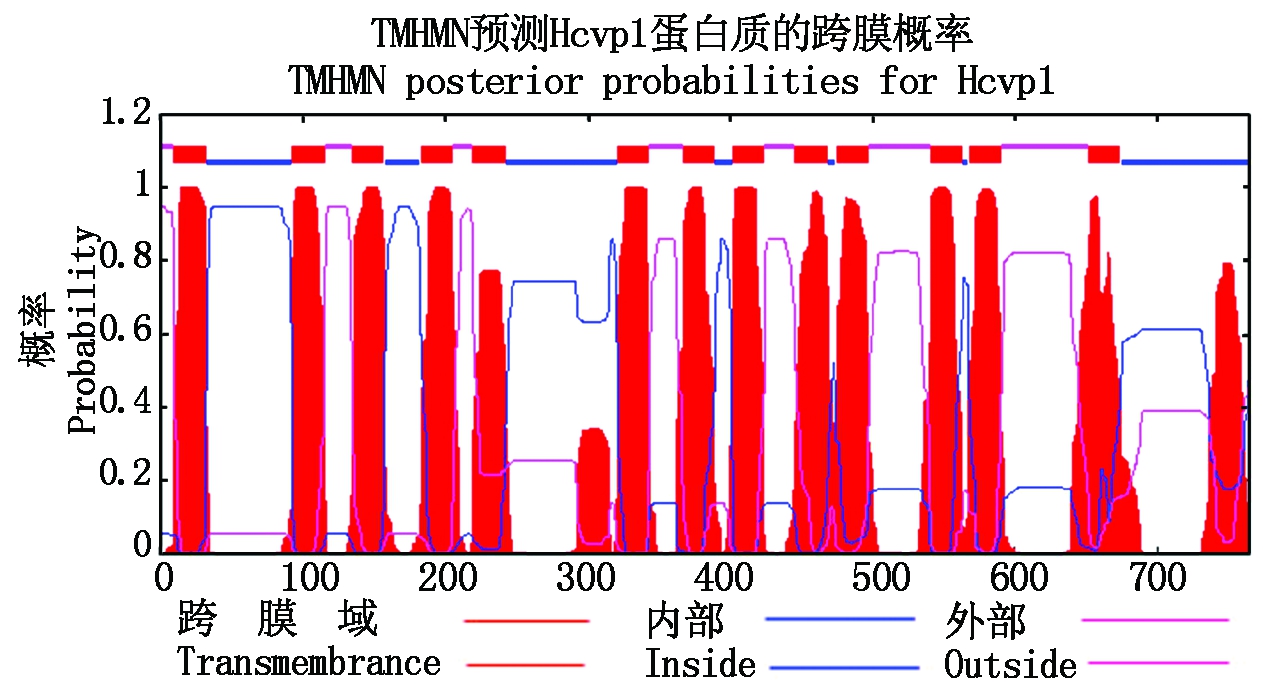
图4 Hcvp1 蛋白质的跨膜结构预测
Fig.4 Transmembrane region prediction of Hcvp1
将该基因核苷酸序列通过BlastN 比对分析,发现其与雷蒙德氏棉(XM 012587702)、甜橙(XM 006492028 )、毛果杨(XM 006389360 )、烟草(NM001325218)、和大豆(XM 003528254)的H+-PPase基因核苷酸序列的相似性分别为90%,85%,85%,83%,83%,通过BlastP比对发现蛋白质序列的相似性分别为96%,93%,91%,93%,94%。
利用ClustalW软件分析Hcvp1基因氨基酸序列与具有代表性的雷蒙德氏棉(XM 012587702)、甜橙(XM 006492028 )、毛果杨(XM 006389360)、烟草(NM001325218)、红麻(KX687919)和大豆(XM 003528254)的H+-PPase基因氨基酸序列进行比对分析(图5)。结果表明,Hcvp1基因氨基酸序列与其他植物氨基酸序列的一致性为94.6%,保守的氨基酸多达738个,而非保守的氨基酸仅有27个。


多重比较采用ClustalW软件进行;不同的标记表示氨基酸残基不同的保守性;* .完全保守; :.75%以上;..50%以上和不足50%的保守。
Multiple alignment was generated using ClustalW program; The different markers indicate that the different levels of the conservation amino acid residues;
* .Complete conservation; :.The conservation is no less than 75%; ..The conservation is no less than 50% and no more than 50%.
图5 Hcvp1氨基酸序列与其他植物H+-PPase氨基酸序列的多重比较
Fig.5 Amino acid sequence alignment of Hcvp1 with those from other plants
利用MEGA 6.0软件分析Hcvp1基因的氨基酸序列与具有代表性的雷蒙德氏棉(XM 012587702)、甜橙(SM 006492028 )、毛果杨(XM 006389360 )、烟草(NM001325218)、红麻(KX687919)和大豆(XM 003528254)的H+-PPase基因氨基酸序列的进化关系(图6)表明,红麻与雷蒙德氏棉的距离最近,这可能是因为两者都是锦葵科的缘故。红麻与毛果杨的距离最远。

图6 Hcvp1 和其他H+-PPase基因的进化树分析
Fig.6 Phylogenetic tree of Hcvp1 and other H+-PPase genes
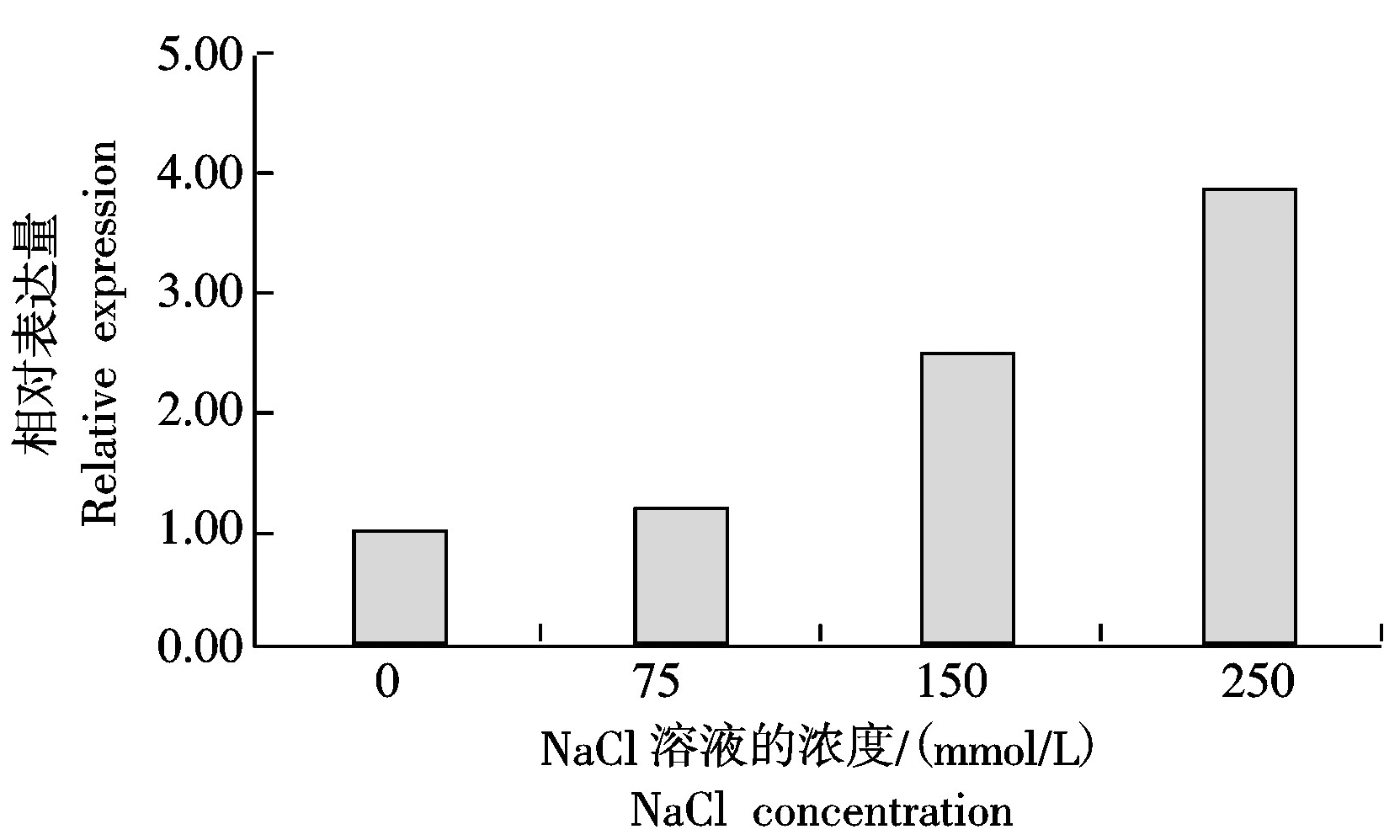
图7 qRT-PCR分析Hcvp1基因在不同NaCl浓度胁迫下的表达
Fig.7 Expression analysis of Hcvp1 under different concentration of NaCl by qRT-PCR
2.4 Hvcp1基因在不同NaCl浓度胁迫下的表达特征分析
分别提取0,75,150,250 mmol/L NaCl浓度胁迫下红麻幼苗叶片的总RNA,并反转录成cDNA作为qRT-PCR 反应的模板,以肌动蛋白做内参,进行qRT-PCR 分析,结果如图7所示。表明随着NaCl浓度的增加,Hvcp1基因的表达量不断增加。可见Hvcp1基因与NaCl胁迫直接相关。
3 讨论
根据红麻耐盐转录组测序获得的转录组序列设计引物[19],从红麻克隆得到Hcvp1基因,采用多种生物信息学的方法对Hcvp1基因进行分析,其cDNA全长为2 298 bp,开放阅读框为2 298 bp,编码765个氨基酸的多肽。同源序列比较发现,红麻的Hcvp1基因序列与雷蒙德氏棉、甜橙、毛果杨、烟草、和大豆的液泡膜H+-PPase 基因的序列高度同源,因此,推测获得的全长cDNA编码产物是红麻液泡膜H+-PPase。
液泡膜H+-PPase在植物耐盐抗旱、生长发育等方面具有重要的作用。液泡膜H+-PPase能够调节液泡的pH值,参与Na+、K+等溶质向液泡内运输;为Na+、Cl-液泡区域化提供运输动力,进而减轻盐害,降低细胞的渗透压[20-21],提高植物的耐盐能力。本研究通过qRT-PCR分析发现,随着NaCl浓度的提高,Hcvp1基因的表达量也随着升高。可见液泡膜H+-PPase基因的表达受NaCl胁迫的诱导,且表达量与NaCl浓度直接相关,所以液泡膜H+-PPase基因在红麻耐盐机制中具有重要的作用。
本研究克隆了红麻Hcvp1基因,并对其在不同NaCl浓度胁迫下的表达模式进行了qRT-PCR分析,这将为后续开展该基因的功能验证,揭示红麻耐盐机制奠定坚实的基础。
[1] Kronzucker H J, Coskun D, Schulze L M, et al. Sodium as nutrient and toxicant[J]. Plant and Soil, 2013, 369(1/2):1-23.
[2] Janz D, Polle A. Harnessing salt for woody biomass production[J]. Tree Physiology, 2012, 32(1):1-3.
[3] Zhang J L, Flowers T J, Wang S M. Mechanisms of sodium uptake by roots of higher plants[J]. Plant and Soil, 2009, 326(1/2):45-60.
[4] 鲁 燕,徐兆师,张瑞越,等.W6基因的过表达提高转基因烟草的耐盐性[J].作物学报, 2008,34(6):984-990.
[5] Munns R, Tester M. Mechanisms of salinity tolerance[J].Annual Review of Plant Biology, 2008, 59(59):651-681.
[6] Yang S, Maeshima M, Tanaka Y, et al. Modulation of vacuolar H+ -pumps and aquaporin by phytohormones in rice seedling leaf sheaths[J]. Biological & Pharmaceutical Bulletin, 2003, 26(1):88-92.
[7] 龚月桦,高俊凤.干旱胁迫下植物质膜功能蛋白研究现状[J].西北植物学报, 2002, 22(3):682-692.
[8] Qiu N W, Chen M, Guo J R, et al. Coordinate up-regulation of V-H+-ATPase and vacuolar Na+/H+ antiporter as a response to NaCl treatment in a C3 halophyte Suaeda salsa[J].Plant Science, 2007,172(6):1218-1225.
[9] Gao F G Q, Zhang J R.Cloning of an H+-PPase gene from Thellungiella halophila and its heterologous expression to improve tobacco salt tolerance[J]. Journal of Experimental Botany,2006,57(12):3259-3270.
[10] Kaba a K, K
a K, K obus G. Modification of vacuolar proton pumps in cucumber roots under salt stress[J]. Journal of Plant Physiology, 2008, 165(17):1830-1837.
obus G. Modification of vacuolar proton pumps in cucumber roots under salt stress[J]. Journal of Plant Physiology, 2008, 165(17):1830-1837.
[11] 宋丽艳,叶武威,赵云雷,等.陆地棉耐盐相关基因(GhVP)的克隆及分析[J].棉花学报, 2010, 22(3):285-288.
[12] 朱春利,张登峰,刘颖慧,等.玉米液泡膜焦磷酸酶基因ZmVPP1的克隆及逆境下的表达分析[J].植物遗传资源学报, 2011, 12(1):107-112.
[13] 甘晓燕,巩 檑,石 磊,等.梭梭液泡膜焦磷酸酶HaVVP基因克隆及序列分析[J].西北农业学报, 2014, 23(11):198-203.
[14] 贺 怡,钱雯婕,袁 磊,等.费尔干猪毛菜液泡膜氢离子焦磷酸酶基因SfVP的克隆、序列分析及表达[J].石河子大学学报:自然科学版, 2014, 32(3):319-324.
[15] 张春蕊,贾园园,王艳敏,等.刚毛柽柳液泡膜H+PPase基因的克隆与胁迫下的表达分析[J].西北植物学报, 2016, 36(5):881-887.
[16] Gaxiola R, Li J, Undurraga S, et al. Drought- and salt-tolerant plants result from overexpression of the AVP1 H+-pump[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(20):11444-11449.
[17] Duan X G Y A, Zhang J R. Heterologous expression of vacuolar H+-PPase enhances the flectrochemical gradient across the vacuolar membrane and improves tobacco cell salt tolerance[J].Protoplasma, 2007, 232(1):87-95.
[18] 李 辉,李德芳,陈安国,等.红麻雄性不育研究进展及展望[J].作物研究, 2015, 29(2):206-209.
[19] Hui L, Li D F, Chen A, et al. Characterization of the kenaf(Hibiscus cannabinus)global transcriptome using illumina Paired-End sequencing and development of EST-SSR markers[J].PloS One,2016,11(3):e0150548.
[20] Gaxiola R, Rao R, Sherman A, et al. The Arabidopsis thaliana proton transporters, AtNhx1 and Avp1, can function in cation detoxification in yeast[J].Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(4):1480-1485.
[21] Lin S, Tsai J, Hsiao C, et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase[J].Nature, 2012, 484(7394):399-403.