水稻是我国重要的粮食作物,种植面积和总产都位居第一。随着人们生活水平的提高,消费者对稻米品质需求越来越高。稻米品质主要包括加工品质、外观品质、蒸煮食味品质和营养品质,具体内容有:透明度、垩白、粒型、直链淀粉含量、胶稠度、糊化温度、蛋白质含量以及有无香味等。其中外观品质尤为重要,是水稻商品性的直接体现,决定了其在市场竞争中的地位[1-6]。垩白是衡量水稻外观品质优劣的重要指标之一,降低稻米的垩白是培育优质水稻品种的前提。
稻米垩白是指胚乳充实不足,胚乳中的淀粉和蛋白颗粒排列疏松,颗粒与颗粒之间存在空气,阳光透过其内部时光线发生折射而形成的一种光学特性[7-10]。根据其发生部位的不同可分为背白、腹白和心白[11-13]。水稻种子灌浆的顺序是从背部到腹部、从周边到中心[14],因此,水稻垩白中出现最多的是腹白,背白最少。相关研究表明,垩白与其他品质性状存在一定的相关性。在外观品质中,李欣等[15]认为籼稻稻米垩白与粒宽存在极显著的正相关性,与长宽比存在极显著的负相关性,而粳稻稻米的垩白面积与粒型、粒长没有明显的相关性。在加工品质上面,徐富贤等[16]认为垩白与整精米率呈显著负相关。在籼稻的蒸煮食味品质中,周少川等[17]认为在籼稻中稻米垩白与直链淀粉含量和糊化温度存在极显著正相关关系,与胶稠度存在极显著负相关关系,而在粳稻中,这些相关性不显著。这一结论与刘奇华等[18]的研究结果一致。
垩白是典型的数量性状,受多基因调控,以加性效应为主,同时也容易受到环境的影响,如温度、肥力水平等[19]。因此,对垩白性状的遗传研究比较困难。不同的研究者利用不同的遗传群体对垩白性状进行了大量的研究,鉴定了部分相关QTL。到目前为止共定位到85个与垩白相关的QTL( http://archive.gramene.org /qtl /),其中有13个与垩白大小有关,21个与垩白度相关的,51个控制垩白率,但多数研究尚停留在QTL初步和精细定位的层面上[20-24]。目前已克隆的与垩白相关的基因共8个,其中6个是通过T-DNA 插入、辐射诱变、化学诱变等方式创制人工突变体而分离克隆的,2个是利用自然突变的材料克隆的,前者的优点是能创造极端表型,等位基因效应大,较易分离克隆,但是限制了其在育种实践中的直接应用。GW2是第一个利用自然变异克隆的垩白相关基因,位于第2染色体上,编码一个E3泛素连接酶,它使籽粒变宽和粒重增加的同时籽粒的垩白也增加,可能是由于粒宽增大,籽粒灌浆速度加快,从而导致垩白的产生[25]。Chalk5是最近克隆的一个控制水稻垩白的主效QTL,该基因编码一种液泡上的有无机焦磷酸水解作用和H+转运作用的焦磷酸转移酶[26]。过量表达Chalk5能够使胚乳的垩白增加,可能是干扰了种子发育过程中膜运输系统的pH稳态,从而影响蛋白体的生物合成,使小囊泡结构大量增加,因此,在胚乳的贮藏部位形成垩白。与低垩白品种H94等位基因序列比较,高垩白品种珍汕97等位基因序列发生39处多态性变异,其中10处变异发生在翻译起始位点上游1.8 kb的启动子区域,包括替换、插入和缺失3种变异类型。5处变异发生在外显子区域,但仅在第1个和第4个外显子上发生2个氨基酸替换,内含子区域有24处SNP变异。对该基因进行功能性遗传变异分析发现,在珍汕97启动子-721和-485位的2个SNPs变异导致其与种子发育相关的2个顺式调控元件在H94启动子中发生了变异,位于-721位的是调控种子特异表达和ABA相应的RY/G-box[27-28],位于-485位的是调控种子发育过程中光合产物运输和代谢的CACT-box[29-31]。研究结果表明,这2个突变位点上与珍汕97具有相同等位基因型的Chalk5的表达量较高,与同一时期的珍汕97表达量相近,任意一个位点发生突变,Chalk5的表达量降低,与同一时期的H94表达量相近。垩白是典型的数量性状,容易受到环境的影响,且其表型是在成熟收获后才能进行选择,因此,获得Chalk5的功能标记能快速、准确鉴定目标基因的不同基因型,是开展Chalk5基因分子标记辅助选择的重要前提。
本研究根据这2个功能突变位点,每个位点分别开发了3对等位基因特异PCR (ARMS-PCR)引物,经过梯度PCR筛选,每个位点均得到1对能高效特异扩增的引物,Chalk12、NChalk12和Chalk22、NChalk22。通过PCR产物测序的方法对这2对标记的检测结果进行了验证,并利用该标记对不同地区的籼稻材料、江苏省历年来大面积推广的粳稻品种以及太湖流域粳稻地方资源进行了Chalk5基因型检测,快速准确地明确了该基因在这些品种和资源中的分布情况。
1 材料和方法
1.1 供试材料
供试水稻材料包括江苏省历年来大面积推广的粳稻品种65份;不同地区籼稻恢复系30份,常规籼稻品种10份,籼稻保持系3份;太湖流域粳稻地方资源179份。以含有Chalk5基因的9311和含有chalk5基因的Basmati作为对照。
1.2 Chalk5基因功能标记的设计与合成
在GenBank中(http://www.ncbi.nlm.nih.gov)搜索高垩白率显性等位基因Chalk5和低垩白率隐性等位基因chalk5的序列,来源于珍汕97的显性等位基因Chalk5的登录号为KJ363317.1,来源于H94的隐性等位基因chalk5的登录号为KJ363318.1。经过序列比对分析发现,与前人的研究结果一致[26],在起始密码子前第721位碱基由C突变为T,在起始密码子前第485位碱基由A突变为T,且这2个突变位点为功能突变,根据这2个功能突变位点以及基因序列,参照Ye等[32]的方法用Primer Premier 5.0 软件设计等位基因特异PCR (ARMS-PCR)引物,为了增强引物的特异性,第721位突变位点的后引在3′端引入一个人为错配,第485位突变位点的前引在3′端引入一个人为错配。由于尚不清楚哪一种碱基的错配能够在不影响扩增效率的前提下提高扩增特异性,因此,每个位点分别设计3种碱基错配方式,前引与后引配对,每个位点共6对引物以供筛选,引物具体序列及扩增产物大小见表1。
引物由Invitrogen公司合成,将合成的引物用TE缓冲液(pH值8.0)稀释为10 mol/L,-20 ℃保存备用。
1.3 Chalk5基因功能区段的测序和序列比对
为了进一步验证该功能标记的准确性以及明确常规粳稻和籼稻品种中目标基因的等位基因型,利用引物C1F和C2R分别为前引和后引组合配对,扩增目标基因功能区段,产物清晰,大小为846 bp,对其中9份品种的目标基因功能区段进行了测序。PCR 产物经琼脂糖凝胶电泳、回收、纯化、检测后送上海英骏生物技术有限公司进行测序,拼接后的序列与登录的chalk5进行比对。
1.4 DNA提取
在水稻分蘖盛期取新鲜幼嫩的叶片,采用SDS法提取水稻基因组DNA[33]。
1.5 PCR扩增和电泳
20 μL的反应体系包括:模板DNA(约15 ng/μL)2 μL,引物(4 pmol/μL)2 μL,10×缓冲液(25 mmol/L)2 μL,MgCl2(25 mmol/L)1.2 μL,dNTP(2.5 mmol/L)0.4 μL,Taq DNA聚合酶(5 U/μL)0.2 μL,灭菌双蒸水12.2 μL。在Eppendorf PCR仪上进行扩增,反应条件为:94 ℃预变性5 min;94 ℃,30 s,58 ℃,30 s,72 ℃,1 min,共32个循环;72 ℃再延伸10 min。其中在梯度反应中将退火温度设置为53~63 ℃。反应产物在2%的琼脂糖凝胶上进行电泳,DuRed染色,然后在紫外凝胶成像仪上观察并照相。
2 结果与分析
2.1 Chalk5基因序列的分析和功能标记的开发
对珍汕97中包含的高垩白率显性等位基因Chalk5和H94的低垩白率隐性等位基因chalk5的序列进行比对分析,在起始密码子前第721位碱基由C突变为T,在起始密码子前第485位碱基由A突变为T,且这2个突变位点为功能突变,根据这2个功能突变位点以及基因序列设计等位基因特异PCR (ARMS-PCR)引物,为了增强引物的特异性,第721位突变位点的后引在3′端引入一个人为错配,第485位突变位点的前引在3′端引入一个人为错配。由于尚不清楚哪一种碱基的错配能够在不影响扩增效率的前提下提高扩增特异性,因此,每个位点分别设计3种碱基错配方式,前引与后引配对,每个位点共6对引物,引物具体序列及扩增产物大小见表1。
将C1F与C1R1配对的引物命名为Chalk11,C1F与NC1R1配对的引物命名为NChalk11,C1F与C1R2配对的引物命名为Chalk12,C1F与NC1R2配对的引物命名为NChalk12,C1F与C1R3配对的引物命名为Chalk13,C1F与NC1R2配对的引物命名为NChalk13;将C2F1与C2R配对的引物命名为Chalk21,NC2F1与C2R配对的引物命名为NChalk21,C2F2与C2R配对的引物命名为Chalk22,NC2F2与C2R配对的引物命名为NChalk22,C2F3与C2R配对的引物命名为Chalk23,NC2F3与C2R配对的引物命名为NChalk23。
对与珍汕97具有相同等位基因型(高垩白率显性等位基因Chalk5)的水稻品种,引物Chalk11、Chalk12、Chalk13和Chalk21、Chalk22 、Chalk23分别能扩增出197,450 bp的片段,而引物NChalk11、NChalk12、NChalk13和NChalk21、NChalk22、NChalk23则无扩增产物;对与H94具有相同等位基因型(低垩白率隐性等位基因chalk5)的水稻品种,引物NChalk11、NChalk12、NChalk13和NChalk21、NChalk22、NChalk23分别能扩增出197,450 bp的片段,而引物Chalk11、Chalk12、Chalk13和Chalk21、Chalk22、Chalk23则无扩增产物。
表1 基于显性隐性等位基因序列单碱基功能突变位点设计的ARMS-PCR标记Tab.1 ARMS-PCR primers designed in this paper

引物名称Primer name引物序列(5'-3')Primer sequenceC1FCGGTTAGAACAGGATTGCC1R1ATAAAACAACTCTGGGTCAGGC1R2ATAAAACAACTCTGGGTCACGC1R3ATAAAACAACTCTGGGTCAAGNC1R1ATAAAACAACTCTGGGTCAGANC1R2ATAAAACAACTCTGGGTCACANC1R3ATAAAACAACTCTGGGTCAAAC2F1TCCAGACGGTGCCGTTTGCAC2F2TCCAGACGGTGCCGTTTGAAC2F3TCCAGACGGTGCCGTTTGGANC2F1TCCAGACGGTGCCGTTTGCTNC2F2TCCAGACGGTGCCGTTTGATNC2F3TCCAGACGGTGCCGTTTGGTC2RGACGAGCGAGTGATTGA
2.2 Chalk5基因功能标记的筛选
对这12对引物进行梯度筛选,梯度反应过程中退火温度为53~63 ℃,分别为53.0,53.2,53.8,54.7,55.8,57.1,58.4,59.7,60.9,61.9,62.6,62.9 ℃。根据前人的研究结果,DNA模板选用与珍汕97具有相同等位基因型的常规籼稻品种9311和与H94具有相同等位基因型的籼稻恢复系明恢63,扩增产物在1.5%琼脂糖电泳分析。
如图1所示,以9311为模板,引物Chalk11、Chalk12、Chalk13能扩增出197 bp的片段,对应引物NChalk11、NChalk12、NChalk13无扩增产物;以明恢63为模板,引物Chalk11、Chalk12、Chalk13无扩增产物,对应引物NChalk11、NChalk12、NChalk13能扩增出197 bp的片段,与预期结果完全一致。同时,从图1中可以看出第2个引物对Chalk12与NChalk12在退火温度为59.7 ℃条件下的扩增效率及扩增特异性最好,因此,选用等位基因特异PCR引物Chalk12与NChalk12作为第一个功能突变位点的筛选标记,可用于目标基因等位基因型的快速检测。

M. DL2000;A. 引物Chalk11、NChalk11扩增结果;B. 引物Chalk12、NChalk12扩增结果;C. 引物Chalk13、NChalk13扩增结果。
M.DL2000; A.Results of gradient PCR amplification by primers Chalk11 and NChalk11; B.Results of gradient PCR amplification by primers Chalk12 and NChalk12; C.Results of gradient PCR amplification by primers Chalk13 and NChalk13.
图1 等位基因特异PCR引物Chalk11、NChalk11、Chalk12、NChalk12、Chalk13、NChalk13对9311和明恢63梯度PCR的扩增结果
Fig.1 Results of gradient PCR amplification by ARMS-PCR primers Chalk11, NChalk11, Chalk12,NChalk12, Chalk13, NChalk13 from genomic DNA of 9311 and Minghui 63
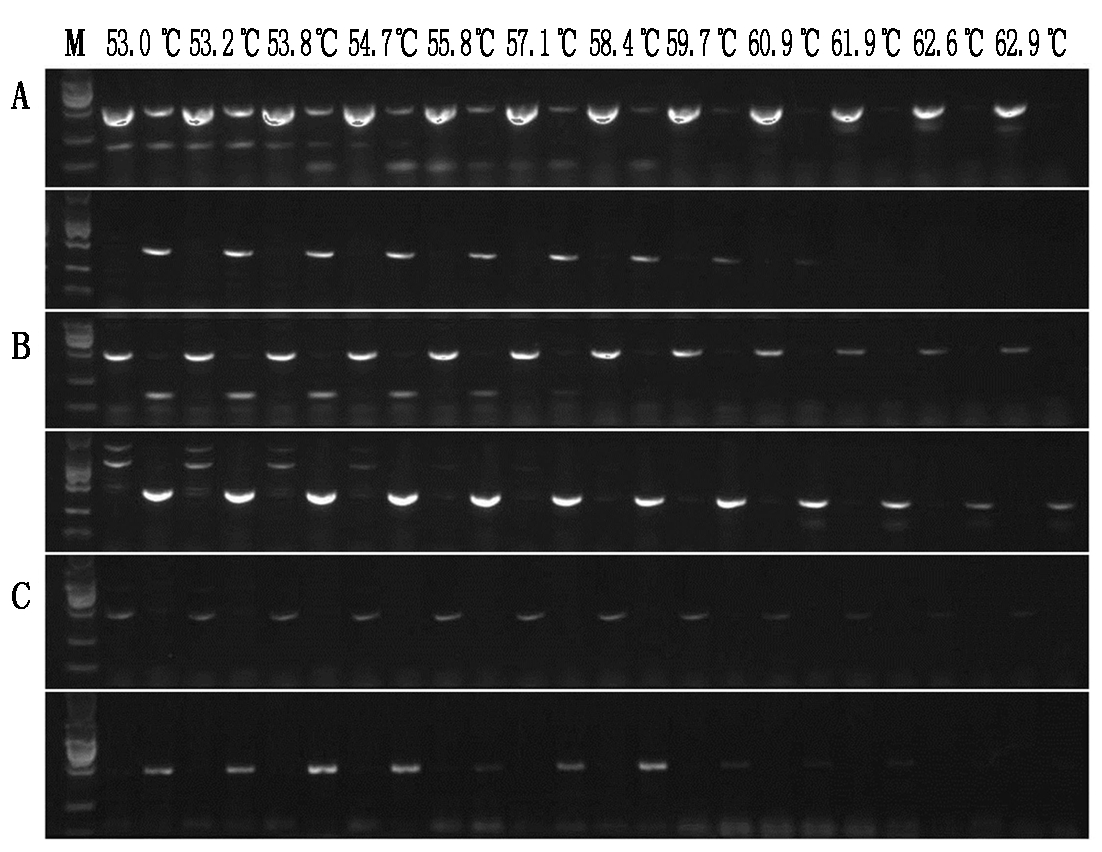
如图2所示,以9311为模板,引物Chalk21、Chalk22、Chalk23能扩增出450 bp的片段,对应引物NChalk21、NChalk22、NChalk23无扩增产物;以明恢63为模板,引物Chalk21、Chalk22、Chalk23无扩增产物,对应引物NChalk21、NChalk22、NChalk23能扩增出450 bp的片段,与预期结果完全一致。同时,从图2中可以看出第2个引物对Chalk22与NChalk22在退火温度为60.9 ℃条件下的扩增效率及扩增特异性最好,因此,选用等位基因特异PCR引物Chalk22与NChalk22作为第2个功能突变位点的筛选标记,可用于目标基因等位基因型的快速检测。
2.3 Chalk5基因功能标记的验证
为了进一步验证等位基因特异PCR引物Chalk12、NChalk12 和Chalk22、NChalk22对不同水稻品种包含的等位基因型检测的准确性,将引物C1F、C2R配对命名为ChalkCX,用于功能目标片段的测序分析,该引物能扩增出846 bp的目标片段,包含了上述2个功能突变位点。
利用引物ChalkCX对7个水稻品种9311、明恢63、日本晴、连粳4号、淮稻11号、华粳6号和盐粳11号进行扩增,PCR产物进行测序分析。如图3所示,与对照珍汕97和H94进行比对分析发现,9311、日本晴、连粳4号、淮稻11号、华粳6号和盐粳11号在2个功能突变位点的等位基因型与珍汕97一致,而明恢63在2个功能突变位点的等位基因型与H94一致,测序分析结果与等位基因特异PCR检测的结果完全一致,因此,利用Chalk12、NChalk12 和Chalk22、NChalk22中的任何一对引物均可以准确地鉴定出水稻品种中包含的Chalk5的不同等位基因型(图3)。
M. DL2000;A. 引物Chalk21、NChalk21扩增结果;B. 引物Chalk22、NChalk22扩增结果;C. 引物Chalk23、NChalk23扩增结果。
M.DL2000; A.Results of gradient PCR amplification by primers Chalk21 and NChalk21; B.Results of gradient PCR amplification by primers Chalk22 and NChalk22; C.Results of gradient PCR amplification by primers Chalk23 and NChalk23.
图2 等位基因特异PCR引物Chalk21、NChalk21、Chalk22、NChalk22、Chalk23、NChalk23对9311和明恢63梯度PCR的扩增结果
Fig. 2 Results of gradient PCR amplification by ARMS-PCR primers Chalk21, NChalk21, Chalk22, NChalk22, Chalk23, NChalk23 from genomic DNA of 9311 and Minghui 63
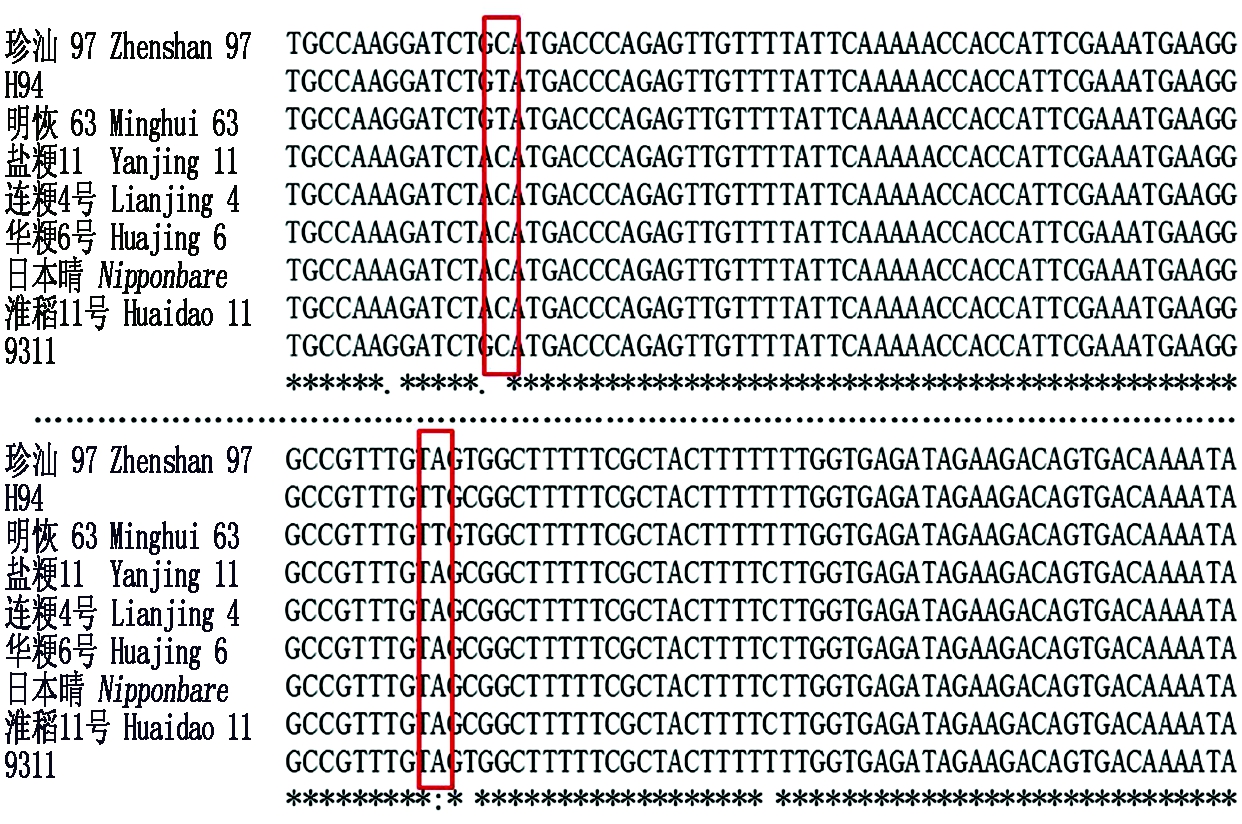
图3 部分粳稻和籼稻品种Chalk5位点功能区的测序比对结果
Fig.3 Sequence alignment of Chalk5 functional domain for partial rice varieties
2.4 ARMS-PCR标记对部分籼稻材料、江苏省粳稻品种和太湖流域粳稻地方资源的检测
利用2对ARMS-PCR标记Chalk12、NChalk12 和Chalk22、NChalk22分别对43份籼稻材料进行检测,其中有30份不同地区籼稻恢复系、10份常规籼稻品种以及3份籼稻保持系,2对标记的检测结果一致。其中,南京14号、圭630、苏农3037、明恢78等20份材料表现为chalk5低垩白率等位基因型,其余23份材料表现为Chalk5高垩白率等位基因型。这些含有chalk5低垩白率等位基因水稻材料在育种中可以作为优异亲本用于水稻稻米外观品质的改良(图4)。
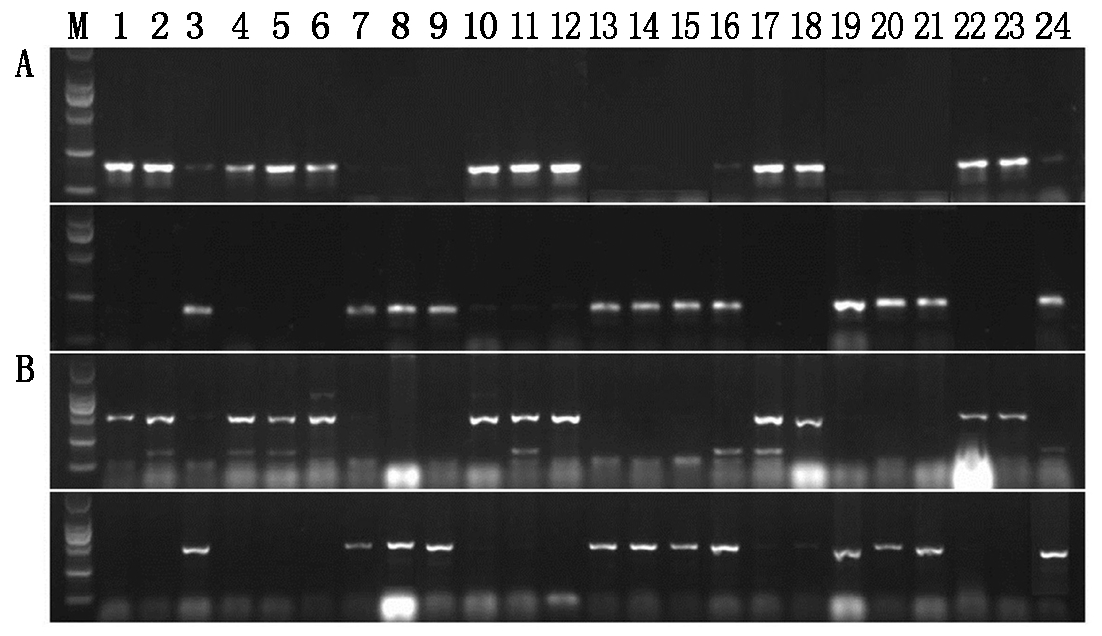
M. DL2000;A. 引物Chalk12、NChalk12 扩增的结果;B. 引物Chalk22、NChalk22扩增的结果; 1. 南京1号; 2. 南京11号;3. 南京14号;4. 南京15号; 5. 南京16号;6. BG90-2;7. 圭630;8. 苏农3037; 9. 明恢78; 10. 9311;11. IRBB21;12. 广陆矮4号;13. 粤晶丝苗;14. 浙恢7954;15. 乐恢188;16. 广恢128;17. IR64;18. 矮脚南特;19. 蜀恢527;20. 多恢1号;21. 绵恢725;22. Ⅱ-32B;23. 粤泰B;24. 华丰B。
M.DL2000; A. Results of PCR amplification by Chalk12 and NChalk12; B.Results of PCR amplification by Chalk22 and NChalk22; 1. Nanjing 1; 2. Nanjing 11; 3. Nanjing 14; 4. Nanjing 15; 5. Nanjing 16; 6. BG90-2; 7. Gui 630; 8. Sunong 3037; 9. Minghui 78; 10. 9311; 11. IRBB21; 12. Guangluai 4; 13. Yuejingsimiao; 14. Zhehui 7954; 15. Lehui 188;16. Guanghui 128; 17. IR64; 18. Aijiaonante; 19. Shuhui 527; 20. Duohui 1; 21. Mianhui 725; 22. Ⅱ-32B; 23. Yuetai B; 24. Huafeng B.
图4 引物Chalk12、NChalk12 和Chalk22、NChalk22对部分籼稻材料PCR的扩增结果
Fig. 4 Results of PCR amplification by Chalk12, NChalk12, Chalk22 and NChalk22 from genomic DNA of different indica materials
利用2对ARMS-PCR标记Chalk12、NChalk12 和Chalk22、NChalk22分别对江苏省历年来大面积推广的65份粳稻品种进行检测,2对标记的检测结果一致。65份粳稻品种均表现为Chalk5高垩白率等位基因型,说明江苏近几年大面积推广的粳稻品种中大部分包含的是Chalk5高垩白率等位基因,缺少chalk5低垩白率等位基因型的粳稻品种,部分品种的检测结果见图5。
利用2对ARMS-PCR标记Chalk12、NChalk12 和Chalk22、NChalk22分别对179份太湖流域粳稻地方资源进行检测,2对标记的检测结果一致。179份太湖流域粳稻地方资源中,仅有8份表现为chalk5低垩白率等位基因型,其余171份资源表现为Chalk5高垩白率等位基因型,说明这些粳稻地方资源中一小部分包含了chalk5低垩白率等位基因,可以作为水稻育种中稻米外观品质改良的重要资源,部分品种的检测结果见图6。
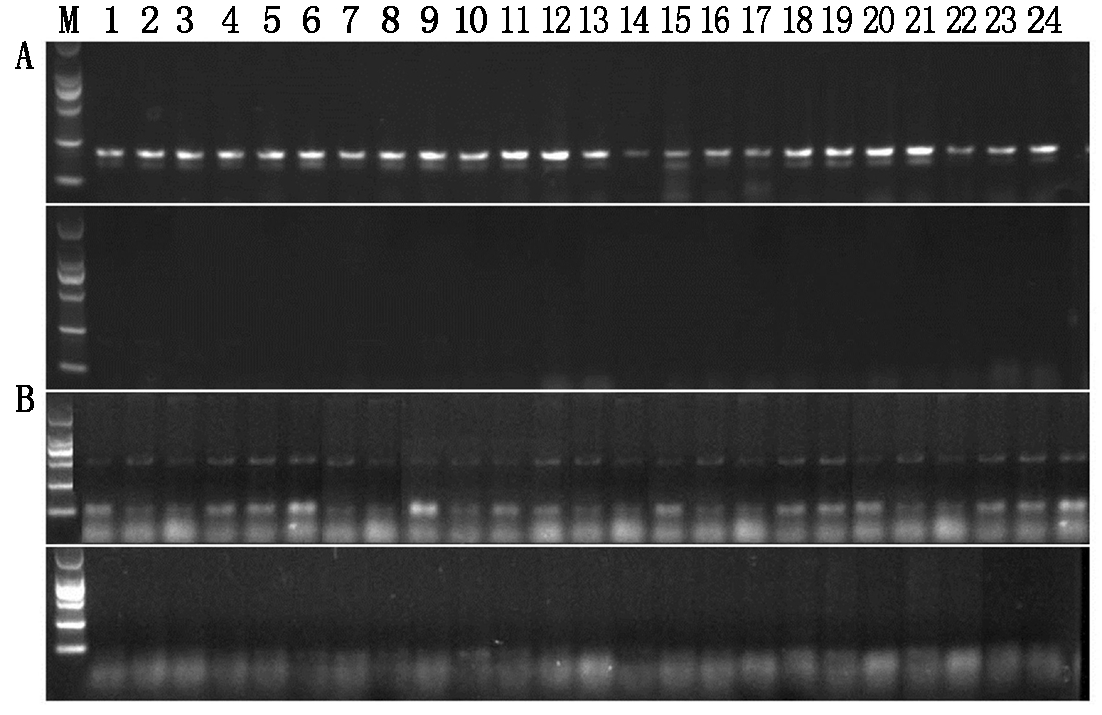
M. DL2000;A. 引物Chalk12、NChalk12 扩增的结果;B. 引物Chalk22、NChalk22扩增的结果;1. 连粳4号; 2. 武运粳21;3. 淮稻11号;4. 华粳6号; 5. 盐粳11号;6. 连粳6号;7. 连粳7号;8. 华瑞稻1号;9. 连粳11号; 10. 武运粳27;11. 淮稻14号;12. 泗稻785;13. 淮糯12号;14. 通糯3号;15. 扬农稻1号;16. 南粳45;17. 宁粳5号;18. 镇稻14号;19. 扬育粳2号;20. 苏稻5号;21. 南粳49;22. 宁9108;23. 扬粳805;24. 镇稻10号。
M.DL2000; A. Results of PCR amplification by Chalk12 and NChalk12; B.Results of PCR amplification by Chalk22 and NChalk22; 1.Lianjing 4; 2. Wuyunjing 21; 3.Huaidao 11; 4.Huajing 6; 5.Yanjing 11; 6.Lianjing 6; 7.Lianjing 7; 8.Huaruidao 1; 9.Lianjing 11; 10.Wuyunjing 27; 11.Huaidao 14; 12.Sidao 785; 13.Huainuo 12; 14. Tongnuo 3; 15.Yangnongdao 1; 16.Nanjing 45; 17.Ningjing 5; 18.Zhendao 14; 19.Yangyujing 2; 20.Sudao 5; 21.Nanjing 49; 22.Ning 9108; 23. Yangjing 805; 24.Zhendao 10.
图5 引物Chalk12、NChalk12 和Chalk22、NChalk22对部分粳稻品种PCR的扩增结果
Fig. 5 Results of PCR amplification by Chalk12, NChalk12, Chalk22 and NChalk22 from genomic DNA of different japonica varieties
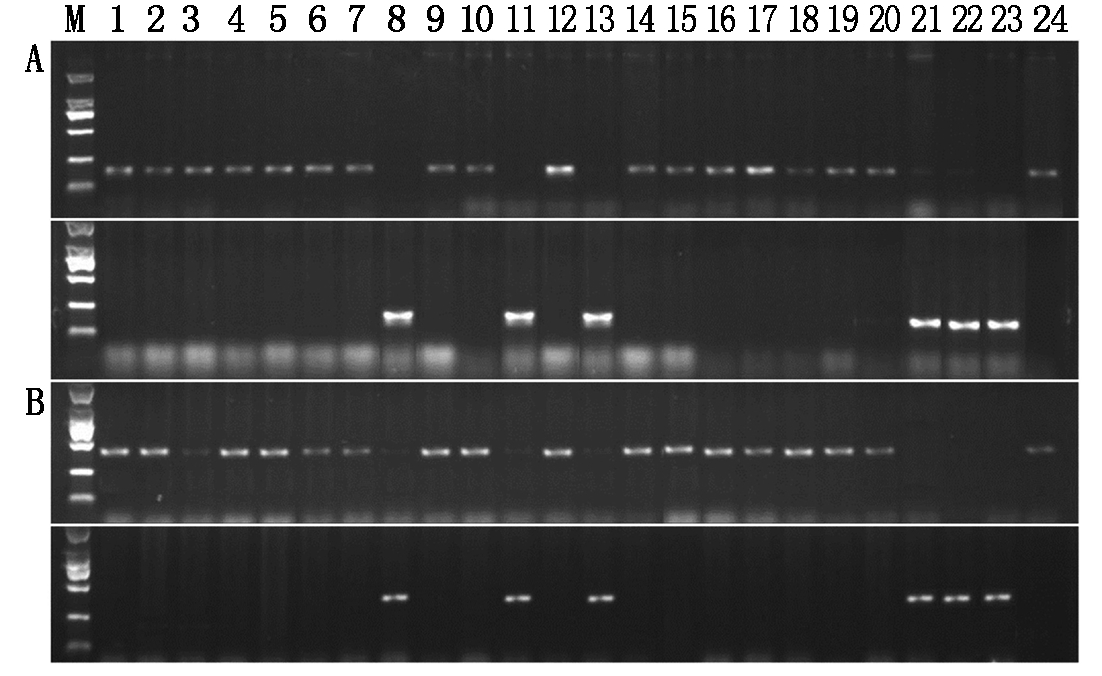
M. DL2000;A. 引物Chalk12、NChalk12 扩增的结果;B. 引物Chalk22、NChalk22扩增的结果;1. 上海青;2. 老来青;3. 立冬青;4. 笔杆青;5. 立更青;6. 盛郎青;7. 老来红;8. 长绿种;9. 红藩稻;10. 小罗汉;11. 短芒孟子粳;12. 牛芒黄;13. 糯稻;14. 白石稻;15. 大量稻;16. 白薄稻;17. 三光稻;18. 白稻头;19. 慢绿种;20. 葡萄种;21. 二粒病;22. 红壳糯;23. 黄金糯;24. 晚乱头。
M. DL2000; A. Results of PCR amplification by Chalk12 and NChalk12; B.Results of PCR amplification by Chalk22 and NChalk22; 1.Shanghaiqing; 2. Laolaiqing; 3.Lidongqing; 4.Biganqing; 5.Ligengqing; 6.Shenglangqing; 7.Laolaihong; 8.Changlüzhong; 9.Hongfandao; 10.Xiaoluohan; 11.Duanmangmengzijing; 12.Niumanghuang; 13.Nuodao; 14. Baishidao; 15.Daliangdao; 16.Baibodao; 17.Sanguangdao; 18.Baidaotou; 19.Manlüzhong; 20.Putaozhong; 21.Erlibing; 22.Hongkenuo; 23.Huangjinnuo; 24.Wanluantou.
图6 引物Chalk12、NChalk12 和Chalk22、NChalk22对部分太湖资源PCR的扩增结果
Fig.6 Results of PCR amplification by Chalk12, NChalk12, Chalk22 and NChalk22 from genomic DNA of different Taihu resources
3 讨论
作物常规育种是一门基于经验的艺术,为作物的改良做出了巨大的贡献。目前,生产上的品种基本上都是由常规育种培育的,但是常规育种的成功往往取决于经验和一定程度的机遇,“效率低、周期长、预见性差”是作物育种长期存在的老大难问题,当前水稻遗传改良中存在的问题很难通过传统育种得到解决。利用常规育种技术已经很难育成突破性新品种,而基于基因调控网络解析的分子育种已经成为突破传统育种瓶颈的唯一有效途径。
分子育种能够把表现型和基因型选择结合起来,可以实现单个或多个目标基因的直接选择和聚合,大幅度提高育种效率,缩短育种年限,在提高产量、改善品质、增强抗性等方面已表现出巨大潜力,成为现代作物育种的主要方向[34-35]。根据分子手段参与形式的不同,分子育种可分为分子标记育种、转基因育种和分子设计育种。分子标记育种即分子标记辅助选择,近年来被广泛运用到水稻育种中[36-38]。根据分子标记检测的结果与田间表型的相关性水平,分子标记辅助选择中采用的标记主要分为DNA标记、基因标记和功能标记3种类型。Andersen等[39]最早提出了功能标记的概念,通过鉴别基因序列的表型功能,挖掘该序列中的多态性信息,开发出能够区分和预测该基因的不同等位基因型及其表型的一种新型分子标记。由于是根据目标基因的功能序列差异设计出来的分子标记,检测目标基因等位基因型的结果准确,即使是不同的遗传背景也不需要进一步验证就可以确定目标基因的等位基因型。
水稻垩白性状是水稻的重要品质性状,是由多个微效基因共同控制的复杂数量性状,且极易受到环境等其他因素的影响,在水稻育种中难以直接通过表型来进行准确选择,而且稻米垩白性状要等成熟收获后才能进行鉴定,鉴定前需要出糙,费时费力。因此,通过设计目标基因的功能标记进行分子标记辅助选择能够快速准确的鉴定目标基因的不同等位基因型,能够快速准确筛选到包含目标基因的单株。本研究根据Chalk5基因的2个功能突变位点,每个位点分别开发了3对等位基因特异PCR引物,经过梯度PCR筛选,每个位点均得到一对能高效特异扩增的引物,Chalk12、NChalk12和Chalk22、NChalk22。通过PCR产物测序的方法对这2对标记的检查结果进行了验证,表明新开发的2对功能标记能够准确快速检测目标基因的等位基因型,能够很好地应用于分子标记辅助选择育种,提高选择效率。
利用2对标记对30份不同地区籼稻恢复系、10份常规籼稻品种、3份籼稻保持系、65份近年来江苏省历年来大面积推广的粳稻品种、179份太湖流域粳稻地方资源进行了Chalk5基因型检测,结果表明在43份籼稻材料中有20份材料表现为chalk5低垩白率等位基因型,其余23份材料表现为Chalk5高垩白率等位基因型;65份粳稻品种均表现为Chalk5高垩白率等位基因型;179份太湖流域粳稻地方资源中,仅有8份表现为chalk5低垩白率等位基因型,其余171份资源表现为Chalk5高垩白率等位基因型。以上结果说明该基因在籼稻材料中存在一定的分布,这些材料在籼稻育种中可以作为优异亲本用于水稻稻米外观品质的改良,而在常规粳稻品种中未检测到chalk5低垩白率等位基因型,仅在少数地方资源中检测到,缺乏可以直接应用的粳稻资源,这就存在目标基因在粳稻育种中难以直接应用的难题。如何将籼稻材料以及地方资源中的目标基因应用于粳稻稻米外观品质的改良是需要思考的一个问题,通过杂交后回交借助于分子标记辅助选择能否高效的实现目标基因的导入且能摆脱其他的不良基因,从而不影响其他性状,这还需要更多的实践工作来检验。
[1] Fitzgerald M, Mccouch S, Hall R. Not just a grain of rice:the quest for quality[J]. Trends in Plant Science, 2009, 14(3):133-139.
[2] Siebenmorgen T, Grigg B, Lanning S. Impacts of preharvest factors during kernel development on rice quality and functionality[J]. Annual Review of Food Science and Technology,2013,4(4):101-115.
[3] Bowles D. Towards increased crop productivity and quality[J]. Current Opinion in Biotechnology, 2012, 23(2):202-203.
[4] Zhang Q. Strategies for developing Green Super Rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(104):16402-16409.
[5] Xing Y, Zhang Q. Genetic and molecular bases of rice yield[J]. Annual Review of Plant Biology, 2010, 61(1):421-442.
[6] Fernie A, Tadmor Y, Zamir D. Natural genetic variation for improving crop quality[J]. Current Opinion in Plant Biology, 2006, 9(2):196-202.
[7] Kang H, Park S, Matsuoka M, et al. White-core endosperm floury endosperm-4 in rice is generated by knockout mutations in the C-type pyruvate orthophosphate dikinase gene (OsPPDKB) [J]. The Plant Journal :for Cell and Molecular Biology, 2005, 42(6):901-911.
[8] Singh N, Kaur L, Sandhu K S, et al. Relationships between physicochemical, morphological, thermal, rheological properties of rice starches[J]. Food Hydrocolloids, 2006, 20(4):532-542.
[9] Fujita N, Yoshida M, Kondo T, et al. Characterization of SSIIIa-deficient mutants of rice:the function of SSIIIa and pleiotropic effects by SSIIIa deficiency in the rice endosperm[J]. Plant Physiology, 2007, 144(4):2009-2023.
[10] Yamakawa H, Hirose T, Kuroda M, et al. Comprehensive expression profiling of rice grain filling-related genes under high temperature using DNA microarray[J]. Plant Physiology, 2007, 144(1):258-277.
[11] Satoh H, Omura T. New endosperm mutations induced by chemical mutagens in rice (Oryza sativa L.) [J].Japanese Tournal of Breeding,1981,31(3):316-326.
[12] Tan Y F, Xing Y Z, Li J X, et al. Genetic bases of appearance quality of rice grains in Shanyou 63, an elite rice hybrid[J]. Theoretical and Applied Genetics, 2000, 101(5/6):823-829.
[13] Li J M, Xiao J H, Grandillo S, et al. QTL detection for rice grain quality traits using an interspecific backcross population derived from cultivated Asian (O.sativa L.) and African (O.glaberrima S.) rice[J]. Genome, 2004, 47(4):697-704.
[14] 顾蕴洁,熊 飞,王 忠,等.水稻和小麦胚乳发育的比较[J].南京师大学报:自然科学版, 2001, 24(3):65-74.
[15] 李 欣,莫惠栋,王安民,等.粳型杂种稻米几个品质性状的遗传表达[J].中国水稻科学, 1999, 13(4):197-204.
[16] 徐富贤,熊 洪,朱永川,等.杂交中稻整精米率的影响因子及其间接鉴定方法研究[J].西南农业学报, 2013, 26(1):1-6.
[17] 周少川,李 宏,王家生,等.华南籼稻早造稻米蒸煮、外观和碾米品质与食味品质的相关性研究[J].作物学报, 2002, 28(3):397-400.
[18] 刘奇华,蔡 建,刘 敏,等.两个籼稻品种垩白对稻米蒸煮食味与营养品质的影响[J].中国水稻科学, 2007, 21(3):327-330.
[19] Lanning S B, Siebenmorgen T J, Counce P A, et al. Extreme nighttime air temperatures in 2010 impact rice chalkiness and milling quality[J]. Field Crops Research, 2011, 124(1):132-136.
[20] Fan C, Yu X, Xing Y, et al. The main effects, epistatic effects and environmental interactions of QTLs on the cooking and eating quality of rice in a doubled-haploid line population[J]. Theoretical and Applied Genetics,2005,110(8):1445-1452.
[21] Lin S, Tsai J, Hsiao C, et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase[J]. Nature, 2012, 484(7394):399-403.
[22] Yamakawa H, Ebitani T, Terao T. Comparison between locations of QTLs for grain chalkiness and genes responsive to high temperature during grain filling on the rice chromosome map[J]. Breeding Science, 2008, 58(3):337-343.
[23] Qin Y, Kim S M, Sohn J K. Genetic analysis and QTL mapping for grain chalkiness characteristics of brown rice (Oryza sativa L.) [J]. Genes & Genomics, 2009, 31(2):155-164.
[24] Zhou L J, Chen L M, Jiang L, et al. Fine mapping of the grain chalkiness QTL qPGWC-7 in rice (Oryza sativa L.) [J]. Theoretical and Applied Genetics, 2009, 118(3):581-590.
[25] Song X J, Huang W, Shi M, et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics, 2007, 39(5):623-630.
[26] Li Y B, Fan C C, Xing Y Z, et al. Chalk5 encodes a vacuolar H+-translocating pyrophosphatase influencing grain chalkiness in rice[J]. Nature Genetics, 2014, 46(4):398.
[27] Ezcurra I, Ellerström M, Wycliffe P, et al. Interaction between composite elements in the napA promoter:both the B-box ABA-responsive complex and the RY/G complex are necessary for seed-specific expression[J]. Plant Molecular Biology, 1999, 40(4):699-709.
[28] Ezcurra I, Wycliffe P, Nehlin L, et al. Transactivation of the Brassica napus napin promoter by ABI3 requires interaction of the conserved B2 and B3 domains of ABI3 with different cis-elements:B2 mediates activation through an ABRE, whereas B3 interacts with an RY/g-box[J].PubMed,2000, 24(1):57-66.
[29] Delgado-Alvarado A, Walker R, Leegood R. Phosphoenolpyruvate carboxykinase in developing pea seeds is associated with tissues involved in solute transport and is nitrogen-responsive[J]. Plant, Cell & Environment, 2007, 30(2):225-235.
[30] Gowik U, Burscheidt J, Akyildiz M, et al. cis-Regulatory elements for mesophyll-specific gene expression in the C4 plant Flaveria trinervia, the promoter of the C4 phosphoenolpyruvate carboxylase gene[J].Plant Cell, 2004, 16(5):1077.
[31] Walker R, Battistelli A, Moscatello S, et al. Metabolism of the seed and endocarp of cherry (Prunus avium L.) during development[J]. Plant Physiology and Biochemistry ,2011, 49(8):923-930.
[32] Ye S, Dhillon S, Ke X, et al. An efficient procedure for genotyping single nucleotide polymorphisms[J]. Nucleic Acids Research, 2001, 29(17):8.
[33] Dellaporta S L, Wood J, Hicks J B. A plant DNA minipreparation:Version Ⅱ[J]. Plant Molecular Biology Reporter, 1983, 1(4):19-21.
[34] 万建民.作物分子设计育种[J].作物学报, 2006, 32(3):455-462.
[35] 黎 裕,王建康,邱丽娟,等.中国作物分子育种现状与发展前景[J].作物学报, 2010, 36(9):1425-1430.
[36] Fjellstrom R, Conaway-Bormans C A, Mcclung A M, et al. Development of DNA markers suitable for marker assisted selection of three Pi genes conferring resistance to multiple Pyricularia grisea pathotypes[J]. Crop Science, 2004, 44(5):1790-1798.
[37] 李 军,李 白,高荣村.利用四引物扩增受阻突变体系 PCR 技术检测水稻光温敏核不育基因p/tms12-1[J].中国水稻科学, 2014, 28(4):442-446.
[38] 王 军,杨 杰,朱金燕,等.稻瘟病抗病基因Pi-kh功能标记的开发及江苏粳稻品种中Pi-kh的变异[J].中国水稻科学, 2014, 28(2):141-147.
[39] Andersen J R, Lübberstedt T. Functional markers in plants[J]. Trends in Plant Science, 2003, 8(11):554-560.