作者简介:李 辉(1980—),男,河北定州人,副教授,博士,主要从事作物遗传育种研究。
为了研究甲羟戊酸-5-磷酸激酶(PMK)在紫苏萜类物质生物合成代谢通路中的重要作用,对紫苏转录组数据进行分析,挖掘出紫苏PMK基因参考序列,采用基因克隆技术从紫苏中克隆了PMK基因,利用生物信息学和实时荧光定量PCR(qRT-PCR)的方法对紫苏PMK基因进行了分析。结果表明,紫苏PMK基因开放阅读框(ORF)全长为1 524 bp,编码507个氨基酸。生物信息学分析显示,PfPMK的分子质量为54.73 ku,等电点为5.20,为亲水蛋白;紫苏PfPMK氨基酸与SmPMK、SsPMK、SbPMK和PvPMK同源性较高,说明紫苏PfPMK蛋白在进化过程中保守性较强。亲缘关系分析显示,PfPMK与丹参和一串红的PMK亲缘关系很近,在线软件WoLF-PSORT预测PfPMK蛋白可能定位于质膜或内质网膜上。qRT-PCR结果表明, PfPMK基因在紫苏根、茎、叶中均有表达,在根中的表达量高于在叶和茎中的表达量;紫苏不同生长发育时期的qRT-PCR分析显示, PfPMK基因在9月中下旬表达量较高。首次从紫苏中克隆出 PfPMK基因,生物信息学分析该基因属于甲羟戊酸-5-磷酸激酶基因,参与了紫苏萜类物质的生物合成。
In order to investigate the function of 5-phosphomevalonate kinase PMK gene in biosynthesis pathway of terpenoids in Perilla frutescens L.,we analyzed the transcriptome data of Perilla frutescens and mined the reference sequence of PMK.The PMK gene was cloned from Perilla frutescens by gene cloning technique,and was analysed using bioinformatics and Real-time PCR(qRT-PCR).The results showed that the ORF of PfPMK gene was 1 524 bp,encoding 507 amino acids.Bioinformatics analysis showed that the molecular weight of PfPMK was 54.73 ku and the isoelectric point was 5.20,which was a hydrophilic protein.The amino acids of PfPMK were higher homologous with SmPMK,SsPMK,SbPMK and PvPMK,which indicated PfPMK protein was highly conserved in the evolutionary process.The phylogenetic analysis showed that PfPMK was closely related to PMK of Salvia miltiorrhiza and Salvia splendens.WoLF-PSORT predicted that PfPMK protein might be located in the plasma membrane or endoplasmic reticulum.Real-time fluorescence quantitative PCR results showed that PfPMK gene was expressed in roots,stems and leaves of Perilla frutescens,and the expression level in roots was higher than that in leaves and stems.The fluorescence quantitative PCR analysis showed that the expression level of PfPMK gene was higher in the middle and late September.The full length of PfPMK gene was first cloned from Perilla frutescens.Bioinformatics analysis showed that PfPMK gene belonged to 5-phosphomevalonate kinase gene and participates in the biosynthesis of terpenoids in Perilla frutescens.
紫苏(Perilla frutescens(L.)Britt.)是唇形科紫苏属一年生草本植物, 茎高0.3~2.0 m, 叶全绿、全紫或一面绿一面紫, 主要分布于中国、不丹、印度、中南半岛、印度尼西亚(爪哇)、日本、朝鲜[1]。紫苏适应性强, 在我国各地广泛栽培, 对土壤要求不严, 在排水较好的砂质壤土、壤土、黏土上均能良好生长, 适宜土壤pH值6.0~6.5, 生长适宜温度25 ℃。紫苏作为药食同源类药用植物, 其入药部分以茎、叶、种子为主, 叶具有镇痛、镇静、解毒等功效; 紫苏叶子又可食用, 可包生鱼片吃, 具有解毒和暖胃之功效, 和肉类煮熟可增加肉类的香气。茎有平气安胎之功效; 种子能镇咳、祛痰、平喘等, 紫苏种子榨出的油, 名苏子油, 可供食用, 又具有防腐作用[2]。
甲羟戊酸途径(Mevalonate pathway, MVA)是以乙酰辅酶A为原料合成异戊烯基焦磷酸和二甲基烯丙基焦磷酸的一条代谢途径, 存在于所有植物中, 是类固醇、类萜等生物分子的合成前体。甲羟戊酸-5-磷酸激酶(5-phosphomevalonate kinase, PMK)属于GHMP激酶超级家族成员, 是MVA途径上游的第5个关键酶, 也是MVA途径中依赖ATP的第2个限速酶, 其功能是将ATP上的γ 位磷酸基团转移到甲羟戊酸-5-磷酸上生成甲羟戊酸-5-二磷酸[3]。国内外有关植物PMK的研究报道较少, 国外的Sando等[4]从巴西橡胶树中克隆出PMK基因; 序列比对显示, 该基因与拟南芥、水稻同源基因相似性为61%~73%。Niu等[5]从檀香木中克隆出PMK基因, 基因表达分析显示, 该基因在根和成熟叶中高表达。国内的Xiao等[6]对拟南芥GHMP家族基因进行了全基因组鉴定、分类和表达分析, 共挖掘出12个GHMP家族基因, 其中包括甲羟戊酸激酶(MK)和甲羟戊酸-5-磷酸激酶(PMK)。Ma 等[7]对丹参基因组进行分析, 共挖掘出40个丹参萜类生物合成相关基因, 包括5个SmDXS、1个SmPMK等。此外, 已从阳春砂[8]、茉莉花[9]、独行菜[10]、香樟[11]等植物中分离克隆出PMK基因。PMK基因在紫苏萜类物质的生物合成过程中起着重要的调控作用, 但目前紫苏PMK基因还未见报道, 本研究首次从紫苏中克隆了甲羟戊酸-5-磷酸激酶基因, 对其开展了生物信息学分析和特异性表达模式分析, 旨在为进一步研究紫苏萜类物质的生物合成提供参考基因。
紫苏种子播种在营养钵中, 一个月后将紫苏根、茎、叶样品洗净液氮速冻, 用于紫苏RNA的提取。RNA提取试剂盒、cDNA反转录试剂盒、琼脂糖凝胶回收试剂盒、质粒提取试剂盒、荧光定量SYBR试剂盒等购自天根生化科技(北京)有限公司; pMD19-T载体、高保真DNA聚合酶PrimeSTAR DNA Polymerase购自宝生物工程(大连)有限公司; 大肠杆菌DH5α 购自北京蕾创生物科技有限公司。
1.2.1 紫苏RNA的提取及cDNA的合成 使用天根的RNAprep pure植物总RNA提取试剂盒(目录号:DP432)提取紫苏根、茎、叶的总RNA; 利用QuantScript RT Kit(天根, 北京)Quant cDNA第一链合成试剂盒(目录号:KR103)进行紫苏的第一链cDNA合成。
1.2.2 紫苏引物合成 在紫苏转录组数据基础上, 挖掘出紫苏萜类物质生物合成关键基因-甲羟戊酸-5-磷酸激酶的基因序列(c110706.graph_c0), 利用Primerer 5.0软件设计该基因的引物:PfPMK-F 5'-ATGGCAGTGGTTGCTTCTG-3', PfPMK-R 5'-TCACTCAATATGAATGGAAGAAAC-3', 由生工生物工程(上海)股份有限公司北京合成部进行引物的合成。
1.2.3 紫苏PfPMK基因克隆 以紫苏cDNA为模板, 利用TaKaRa公司高保真DNA聚合酶PrimeSTAR HS DNA Polymerase扩增PfPMK基因的开放阅读框(ORF)全长。50 μ L扩增体系如下:10.0 μ L 5× PrimeSTAR Buffer, 4.0 μ L dNTP Mixture, 1.0 μ L PfPMK-ORF-F, 1.0 μ L PfPMK-ORF-R, 1.0 μ L Template cDNA, 0.50 μ L PrimeSTAR DNA Polymerase, 32.5 μ L ddH2O。PCR扩增程序如下:94 ℃ 1 min; 98 ℃ 10 s, 55 ℃ 5 s, 72 ℃ 2 min, 共35个循环; 72 ℃ 10 min; 4 ℃ 保存。对PCR反应产物用1%的琼脂糖凝胶检测, 在含有适量核酸染料的1%(m/V)琼脂糖凝胶上, 以180 V/cm的电压在1× TAE电极缓冲液中电泳, 待电泳指示剂溴酚蓝至凝胶的2/3处, 取出凝胶置于紫外成像仪中检测。使用天根生化科技(北京)有限公司琼脂糖凝胶回收试剂盒(DP209)进行PCR产物的回收。PCR回收产物与pMD19-T载体连接转化大肠杆菌感受态DH5α , 并挑取阳性单克隆菌液送到生工进行序列测序。
1.2.4 紫苏PfPMK基因生物信息学分析 使用在线软件ProtParam(http://web.expasy.org/protparam)预测紫苏PfPMK基因的分子量、等电点; 利用NCBI数据库进行Blast同源性分析(http://www.ncbi.nlm.nih.gov); 利用Mega 4软件对PfPMK编码的氨基酸序列进行亲缘关系分析; 使用在线软件WoLF-PSORT(https://wolfpsort.hgc.jp)、NetNGlyc 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc)、NetPhos 3.1 Server(http://www.cbs.dtu.dk/services/NetPhos)、SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP-4.1)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)、SWISS-MODEL(https://swissmodel.expasy.org)对PfPMK蛋白进行亚细胞定位、糖基化位点、磷酸化位点和蛋白激酶结合位点、信号肽和氨基酸二、三结构等预测。
1.2.5 紫苏PfPMK基因表达分析 使用实时荧光定量PCR的方法对紫苏不同组织部位和5个不同生长时期(2020年8月21日、2020年8月31日、2020年9月10日、2020年9月20日、2020年9月30日)的紫苏样品进行PfPMK基因表达分析, 以紫苏肌动蛋白基因Actin(c86816.graph_c0)为内参基因, 利用在线软件Primer 3 Input(version 0.4.0)进行荧光定量PCR引物设计, 引物分别为PfActin-F 5'-TTAACACCCCCGCAATGTAT-3', PfActin-R 5'-TAGCCTCGCTCCGTCAGTAT-3'; PfPMK-RT-F 5'-GATCATCAACGCCATCACTG-3', PfPMK-RT-R 5'-TGCTCCGTCTCTCCACTTTT-3'。荧光定量PCR所采用的设备为ABI PRISM 7500(操作软件为7500 和7500 Fast Real-Time PCR Systems, v2.0.1, USA), 荧光染料为SYBR Green。参照RealMaster Mix(SYBR Green, 天根, 北京)试剂盒说明书草操作, 20 μ L反应体系如下:10 μ L SYBR Green PCR Master Mix, 0.5 μ L PfPMK-QRT-F, 0.5 μ L PfPMK-QRT-R, 2.0 μ L cDNA(1 ng), 7 μ L ddH2O。扩增条件:95 ℃ 30 s; 95 ℃ 10 s, 60 ℃ 10 s, 72 ℃ 10 s, 共35个循环; 72 ℃ 10 min, 4 ℃ 保存。每个反应3次重复, 使用2 -Δ Δ Ct 法分析紫苏PfPMK基因表达量。
根据紫苏转录组数据注释基因信息, 本研究发现一个紫苏PMK注释基因(c110706.graph_c0), 以此序列信息设计引物, 对紫苏PMK进行基因克隆。利用PCR法扩增获得一条1 500 bp左右的片段, 与参考序列c110706.graph_c0大小一致(图1-A)。将PCR产物胶回收连到测序载体pMD19-T上, 转化大肠杆菌DH5α 进行单克隆测序。对测序信息进行分析, 本研究获得紫苏PMK基因开放阅读框(ORF)全长为1 524 bp, 编码507个氨基酸(图1-B), 命名为PfPMK。PfPMK氨基酸组成中, 苏氨酸(Thr)含量为28.4%, 甘氨酸(Gly)含量为27.2%, 丙氨酸(Ala)含量为25.4%, 半胱氨酸(Cys)含量为19.0%, 使用ProtParam(http://web.expasy.org/protparam/)预测PfPMK基因的分子质量为54.73 ku, 等电点为5.20, 分子式为C2431H3874N646O746S20, 其半衰期为4.4 h, 不稳定指数为39.99, 归类为稳定蛋白, 脂肪族指数为27.56, 总平均亲水性为0.697。结合ExPasy-ProtSCale在线软件预测PfPMK蛋白亲/疏水性, 结果显示, PfPMK蛋白疏水性最强的位点是第474个氨基酸, 疏水最高值为2.344, 亲水性最强的位点是第326个氨基酸, 亲水性最低值为-2.833(图2-A), 但整个氨基酸预测值分析结果显示, 该蛋白整体趋向于亲水性, 为亲水蛋白。
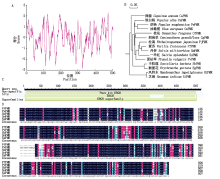
利用NCBI数据库对紫苏PfPMK基因进行Blast分析(http://www.ncbi.nlm.nih.gov/), 同源分析表明, PfPMK氨基酸与SmPMK(丹参, Salvia miltiorrhiza, AEZ55675.1)、SsPMK(一串红, Salvia splendens, XP_042024853.1)、SbPMK(半枝莲, Scutellaria barbata, QEY10159.1)和PvPMK(夏枯草, Prunella vulgaris, QEV81810.1)的同源性比较高, 同源性分别为90.77%, 88.41%, 85.27%, 85.77%(图2-B), 说明紫苏PfPMK蛋白在进化过程中保守性较强。通过NCBI蛋白质保守结构域数据库对PfPMK蛋白进行保守结构域预测, 结果显示, PfPMK蛋白存在一个典型的磷酸甲羟戊酸激酶结构域(图2-C)。利用Mega 4软件对PfPMK基因编码的氨基酸序列进行亲缘关系分析, 结果表明, PfPMK与丹参和一串红的PMK亲缘关系很近(图2-D)。
利用在线软件WoLF-PSORT进行PfPMK蛋白亚细胞定位预测, 结果显示, 该蛋白定位于质膜的系数为4.5, 内质网膜的系数为4.5, 内质网的系数为3.5, 叶绿体的系数为3。TMHMM Sever v2.0预测PfPMK蛋白跨膜结构域显示, 该蛋白整个序列都在膜外, 没有跨膜结构域, 不属于跨膜蛋白(图3-A)。NetNGlyc 1.0 Server在线软件预测PfPMK蛋白糖基化位点, 发现该蛋白不含有糖基化位点(图3-B)。利用NetPhos 3.1 Server预测PfPMK蛋白的磷酸化位点和蛋白激酶结合位点(图3-C), 预测值大于0.5的共发现81个磷酸化位点, 包括55个丝氨酸位点、15个苏氨酸位点和11个酪氨酸位点。SignalP 4.1 Server在线软件预测PfPMK蛋白的信号肽, 根据C值、S值和Y值都低于阈值0.5, 结果显示, 该蛋白不含有信号肽位点, 属于非分泌蛋白(图3-D)。
利用在线软件SOPMA对PfPMK基因编码氨基酸的二级结构进行预测, 结果显示, 该蛋白二级结构中α 螺旋(Alpha helix)有211个, 占比为41.62%; 无规则卷曲(Random coil)有202个, 占比为39.84%; 延伸链(Extended strand)有72个, 占比为14.20%; β 折叠(Beta turn)有22个, 占比为4.34%, 表明PfPMK蛋白主要以α -螺旋和无规则卷曲为主的蛋白二级结构(图4)。利用在线软件SWISS-MODEL对PfPMK基因编码氨基酸的三级结构进行模型建立, 以2r42.1.A蛋白为模板, 用于建立该模型的氨基酸残基为第2-501位, 覆盖范围为72%, 序列一致性为21.21%, 序列相似度为30%(图5)。
实时荧光定量PCR对紫苏不同组织部位的PfPMK基因表达进行分析, 结果表明, PfPMK基因在紫苏的根、茎、叶中有不同程度的表达, 但差异不显著, 其中PfPMK基因在紫苏根中的表达量高于在茎和叶中的表达量(图5-A)。为了研究紫苏PfPMK基因不同生长时期的表达情况, 本研究在紫苏收获前2个月进行采样, 共分5个时间点, 取样间隔时间为10 d, 每次3个生物学重复。实时荧光定量PCR结果显示, PfPMK基因在紫苏不同生长时期的表达量有差异, 以8月21日的样本为对照, 8月31日、9月20日和9月30日样本PfPMK基因表达量与对照有显著差异, 特别是9月20日的样本PfPMK基因表达量显著高于对照(图5-B)。
紫苏原名“ 苏” , 是我国卫生部首批颁发的既是药品又是食品的60种药食同源植物之一[12]。紫苏全草入药, 是常用中药之一。根据《名医别录》、《食疗本草》、《本草图经》和《本草纲目》记载 “ 紫苏, 除寒热, 治冷气, 治肺气喘急” [2]; 2020版《中国药典》记载的紫苏主要来源于唇形科植物紫苏, 具有降气消痰, 平喘, 润肠之功效[13]。目前, 国内外对紫苏的研究主要在紫苏不同采收期代谢物含量、挥发油成分、紫苏提取物对抗病机理的研究等方面, 如Luo等[14]对不同栽培时期紫苏叶中酚酸类物质含量的变化进行了研究; Ju 等[15]开展了不同紫苏种质资源精油活性成分的研究; Paradee等[16]对泰国紫苏进行研究表明, 其酒精提取物中含有大量的木犀草素、芹菜素、金圣草黄素以及葡萄糖苷衍生物, 能够抑制肿瘤坏死因子中微粒子的生成、降低细胞ICAM-1和IL-6因子的表达活性。
紫苏主要药效成分是紫苏醛(Perillaldehyde, PAE), 又称为二氢枯茗醛, 是从紫苏叶中提取出来的无色至浅黄色, 具有樱桃、辛香和桂醛似的特殊香气的单萜化合物的油状液体[17, 18]。紫苏醛属于单萜类化合物, 是植物次生代谢产物中最大的一个家族, 在自然界中广泛分布。甲羟戊酸途径是植物萜类物质生物合成的一条代谢途径, 其中PMK属于GHMP激酶超级家族成员, 其功能是将ATP上的γ 位磷酸基团转移到甲羟戊酸-5-磷酸上生成甲羟戊酸-5-二磷酸。甲羟戊酸途径中相关基因的研究主要集中在甲羟戊酸途径中下游基因的研究, 如檀香木中倍半萜烯合酶可以提高倍半萜类物质的生物合成甲羟戊酸途径[19, 20, 21, 22], 细胞色素P450加氧酶可以将倍半萜烯类碳水化合物转化为倍半萜烯醇类化合物[23, 24]。
最近几年随着高通量测序技术的发展, 萜类合成途径中的关键酶相继被挖掘。Song等[25] 利用转录组测序技术, 从银杏中挖掘出萜类化合物生物合成关键酶PMK, 从分子水平上研究了GbPMK基因在银杏叶中萜类物质生物合成中的功能。Woo等[26] 进一步研究表明, 过量表达PMK基因可以增加萜类物质的含量。
本研究根据紫苏转录组数据, 挖掘出紫苏萜类物质生物合成途径中的一个关键酶PMK, 利用基因克隆技术首次从紫苏中克隆了PfPMK基因, 该基因开放阅读框全长为1 524 bp, 编码507个氨基酸。生物信息学分析显示, PfPMK的分子质量为54.73 ku, 等电点为5.20, 分子式为C2431H3874N646O746S20, 为亲水蛋白。亲缘关系分析显示, PfPMK与丹参和一串红的PMK亲缘关系很近。在线软件WoLF-PSORT预测PfPMK蛋白定位于质膜和内质网膜的系数较高, 而Simkin等[27]研究长春花的PMK蛋白定位在过氧化物酶体上, Niu等[5]研究认为檀香木的PMK蛋白定位在胞质上。本研究将进一步构建PfPMK的亚细胞定位载体, 明确PfPMK在细胞中的定位情况。PfPMK基因在紫苏根中的表达量高于在叶和茎中的表达量, 这与人参[28]和檀香木[5]PMK基因表达情况相一致。紫苏不同生长发育时期的荧光定量PCR分析显示, PfPMK基因在9月中下旬高表达, 是否预示着紫苏萜类物质的含量会升高, 还有待于进一步验证。本研究结果将为紫苏萜类物质代谢通路相关基因的研究提供理论支撑。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|